環狀RNA hsa_circ_0003568通過miR-140-3p及miR-502-5p調控靶基因CDK6及NFYA在骨肉瘤中的作用
李康路,黃漢記,嚴國華,譚曼利,黃大計,朱博
(1. 廣西醫科大學,廣西 南寧 530021;2. 廣西百色市人民醫院,右江民族醫學院附屬西南醫院,廣西 百色 533000)
骨肉瘤(Osteosarcoma,OS)是最常見的原發性惡性骨腫瘤之一,多見于兒童、青少年,具有高侵襲性、高轉移性、進展迅速和化療耐藥等特點[1 ]。近年來,多藥化療加上手術切除可發現的病灶,已使局限性OS患者的長期生存率提高到65%~70%[2]。然而,目前由于早期診斷不足和缺乏有效的治療方法,OS患者的5年生存率和預后仍未明顯改善,尤其是對多藥耐藥、復發或肺轉移的患者[3]。因此,迫切需要識別新的臨床相關預后生物標志物,并探索腫瘤發生的潛在分子機制,以制定更有效的策略來治療OS。環狀RNA(circRNAs)是一種高度穩定、保守的特殊RNA,具有共價閉環特性,不易被內切酶降解,廣泛存在于各種組織和器官中[4]。越來越多的研究表明,circRNAs以多種方式廣泛參與疾病的發生發展,在癌癥中具有重要的作用[5],它們通過靶向許多不同基因或信號通路,并將相關基因緊密連接到調控網絡中從而影響腫瘤細胞的增殖、轉移、分化和其他生物學過程[6]。然而,在OS中,circRNAs的潛在分子機制尚不清楚。在本研究中,我們主要探討了環狀RNA hsa_circ_0003568在OS中的表達并進行生物信息學研究,以明確其表達和臨床意義,為進行OS分子標志物研究提供實驗依據,為臨床對OS的診斷和治療提供新的視角。
1 材料與方法
1.1 材料與設備 正常成骨細胞hFOB1.19、OS細胞系SJSA-1和HOS購買于深圳市豪地華拓生物科技有限公司(中國,深圳);胎牛血清購自杭州天杭生物公司;DMEM/F12培養基購自ThermoFisher Scientific公司(美國,馬薩諸塞州);TRIzol試劑盒購自Invitrogen 公司(美國,加利福尼亞州);cDNA反轉錄試劑盒、SYBR green PCR mix購自TAKARA生物技術公司(日本,東京);實時定量 PCR儀(LightCycler 9600)購自羅氏公司 (瑞士,巴塞爾)。
1.2 方法
1.2.1 細胞培養 hFOB1.19細胞用含10%胎牛血清的DMEM/F12 培養基,加入G418(0.3 mg/ml)以及青霉素(100 U/ml)和鏈霉素(100 U/ml),SJSA-1和HOS細胞系用含10%胎牛血清的DMEM/F12培養基,加入青霉素(100 U/ml)和鏈霉素(100 U/ml),于5%CO2、37℃恒溫培養箱中培養。每隔3 d換新鮮的完全培養基,細胞密度達90%左右時按1∶3比例傳代。
1.2.2 RNA 提取和RT-qPCR 采用TRIzol法提取細胞總RNA并利用反轉錄試劑盒合成cDNA,采用SYBR green PCR mix及hsa_circ_0003568的divergent引物進行RT-qPCR,反應條件如下:預變性95℃ 10 min,變性 95℃ 15 s,退火 60℃ 30 s,45個循環。GAPDH作為內參,采用2-△△Ct法計算hsa_circ_0003568相對表達量。同時,對PCR產物進行Sanger測序分析鑒定,測序由上海生工公司(Sangon Biotech (Shanghai) Co.,Ltd.)完成,引物見表1。

表1 RT-qPCR引物序列
1.2.3 生物信息學分析 通過circRNA的靶miRNA預測工具Circular RNA Interactome(https://circinteractome.nia.nih.gov/index.html)、CircBank Database(http://www.circbank.cn/index.html)和RegRNA2.0(http://regrna2.mbc.nctu.edu.tw/index.html)預測與hsa_circ_0003568相互作用的miRNA,并將其進行交集取3個預測工具同時靶向的miRNA,采用miRNA靶基因預測工具miRDB(http://mirdb.org/)、DIANA TOOLS(http://diana.imis.athena-innovation.gr/DianaTools/index.php?r=microT_CDS/index)、TargetScan(http://www.targetscan.org/vert_71/)和RegRNA2.0(http://regrna2.mbc.nctu.edu.tw/index.html)對交集到的miRNA進行靶基因預測,并將其進行交集取4個預測工具同時預測到的靶基因。通過GEPIA(http://gepia.cancer-pku.cn/index.html)對其交集的靶基因進行生存分析篩選生存預后顯著差異的靶基因(P<0.05)。通過篩選的circRNA-miRNA和miRNA-mRNA利用Cytoscape構建circRNA-miRNA-mRNA可視化調控網絡。
1.3 統計學方法 采用GraphPad Prism 8對實驗數據進行統計學分析,統計檢驗數據符合正態分布。兩組間比較采用t檢驗,以P<0.05為差異具有統計學意義。
2 結果
2.1 hsa_circ_0003568在OS細胞中的表達 我們通過RT-qPCR在人OS細胞系(SJSA-1及HOS)和人成骨細胞系(hFOB1.19)中檢測了hsa_circ_0003568的表達。結果顯示,與成骨細胞相比,在人OS細胞系中hsa_circ_0003568的表達顯著上調(P<0.05),見圖1A,并通過sanger測序驗證了hsa_circ_0003568成環的連接點為IST1前體RNA的第6和第7外顯子相接而成,見圖1B。
2.2 hsa_circ_0003568靶向miRNA的預測 為探索hsa_circ_0003568的高表達在OS中的作用機制,我們通過miRNA預測工具對hsa_circ_0003568靶向的miRNA進行預測。結果顯示CircInteractome數據庫預測到9個miRNA,CircBank預測到11個miRNA和RegRNA2.0預測到20個miRNA。對其預測的miRNA進行交集,發現3個預測工具同時預測到的2個miRNA為miR-140-3p和miR-502-5p,見圖2。
2.3 miRNA靶向基因的預測及分析 研究已經證明miRNA主要是通過調控其靶基因而發揮生物學功能[7]。因此,我們通過miRNA靶基因預測工具分別對miR-140-3p和miR-502-5p進行潛在的靶基因預測。對miR-140-3p靶基因的預測結果顯示,miRDB預測到697個靶基因,DIANA預測到881個靶基因,TargetScan預測到495個靶基因和RegRNA2.0預測到228個靶基因。進一步對其預測的靶基因進行交集,發現4個預測工具同時預測到7個潛在的靶基因(ACVR2B、NFYA、CDK6、VKORC1L1、NRIP1、DAZAP2、KIF5A),見圖3。對miR-502-5p靶基因的預測結果顯示,miRDB預測到457個靶基因,DIANA預測到782個靶基因,TargetScan預測到4120個靶基因和RegRNA2.0預測到74個靶基因。進一步對其預測的靶基因進行交集,發現4個預測工具同時預測到6個潛在的靶基因(ODF4、RAB1B、HOXC8、TNRC6C、NFYA、B3GALT5),見圖4。進一步分析發現NFYA基因同時是miR-140-3p和miR-502-5p的潛在靶基因。
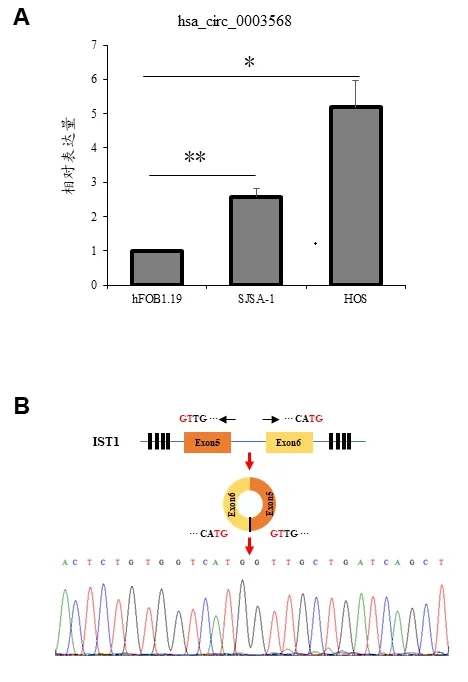
注:A:RT-qPCR的結果,*:P<0.05,**:P<0.01;B:has_circ_0003568形成示意圖及其PCR產物測序結果。
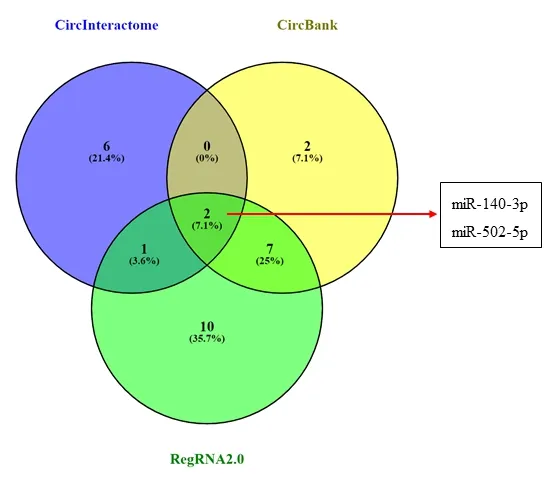
圖2 has_circ_0003568靶向的miRNA:CircInteractome、CircBank和RegRNA2.0數據庫預測的has_circ_0003568靶miRNA重疊的示意圖
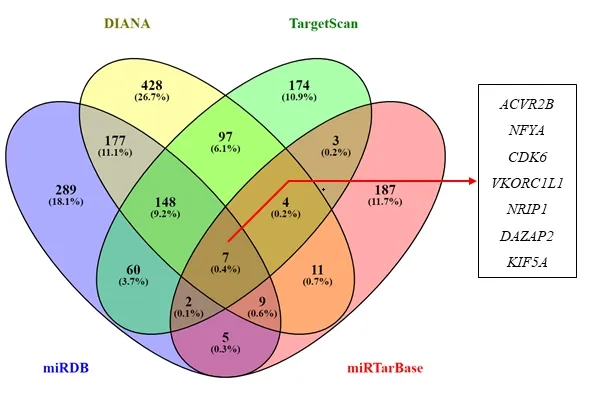
圖3 miR-140-3p靶向的mRNA:DIANA、TargetScan、miRDB和miRTarBase數據庫預測的miR-140-3p靶基因重疊的示意圖
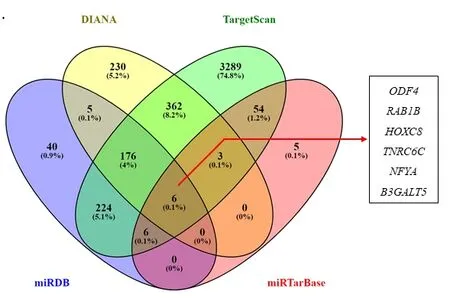
圖4 miR-502-5p靶向的mRNA:DIANA、TargetScan、miRDB和miRTarBase數據庫預測的miR-502-5p靶基因重疊的示意圖
2.4 生存分析 通過采用GEPIA數據庫對miR-140-3p和miR-502-5p分別交集到的7個和6個靶基因進行生存分析(見表2和表3),結果發現,除了CDF4無生存曲線記錄外,其余基因都有相關生存曲線記錄。更重要的是,我們發現CDK6和NFYA基因與OS患者的生存預后顯著相關,與低表達CDK6的OS病人相比,高表達CDK6的OS病人展現了更差的預后(P<0.001),見圖5A;同樣,與低表達NFYA的OS病人相比,高表達NFYA的病人生存率更低(P<0.05),見圖5B。
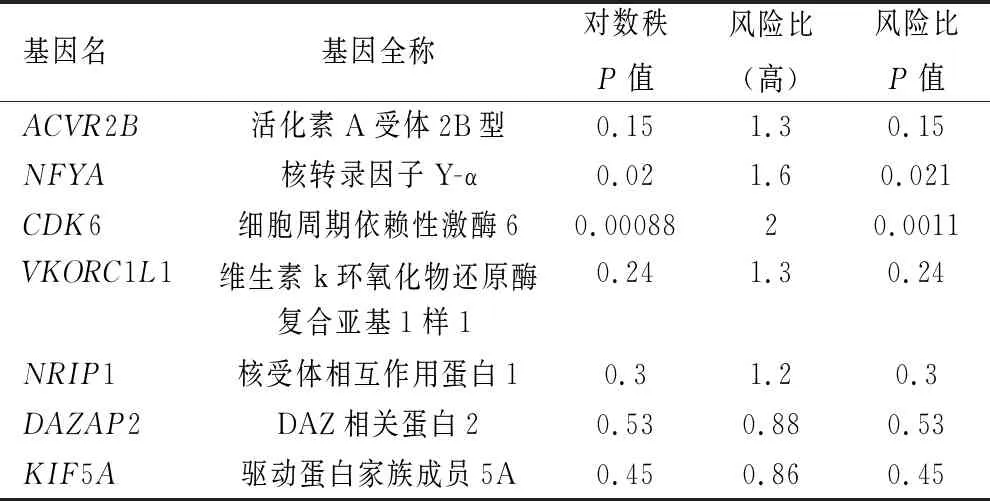
表2 miR-140-3p靶基因交集的7個mRNA的生存分析數據
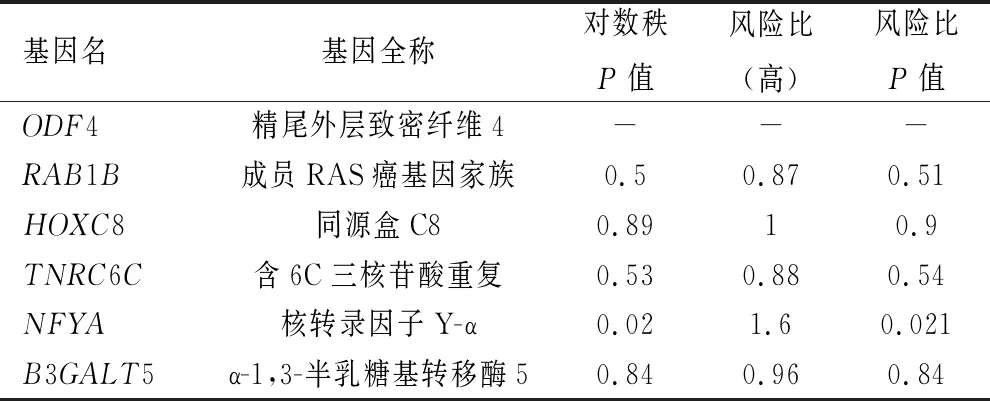
表3 miR-502-5p靶基因交集的6個mRNA的生存分析數據
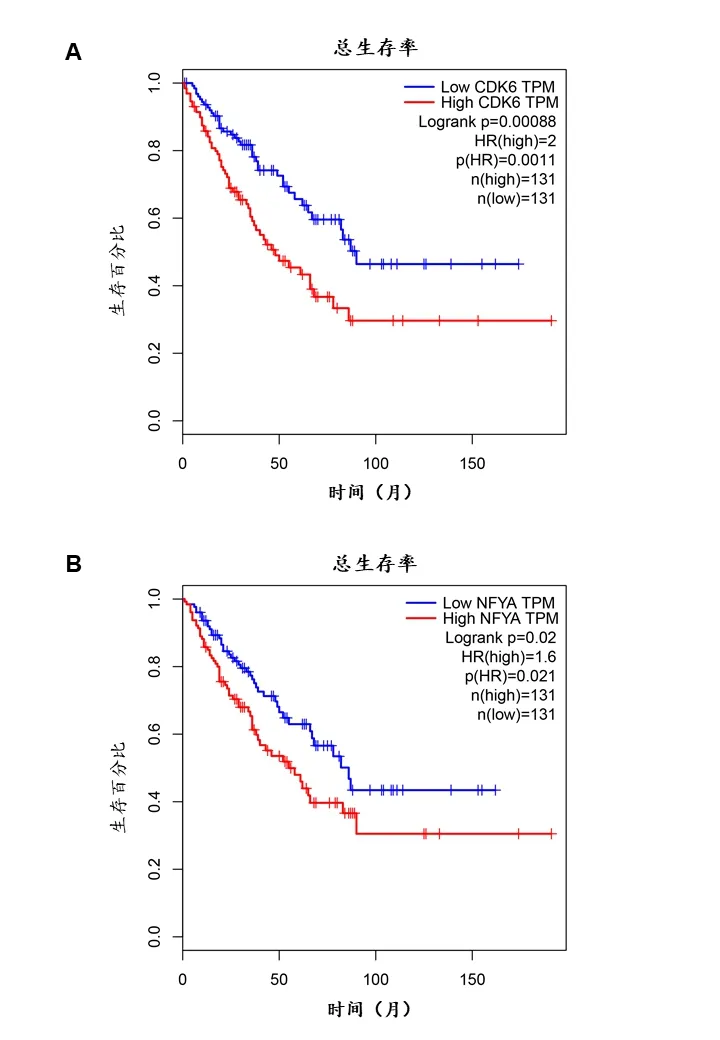
A:OS標本中CDK6基因高表達組較低表達組預后較差(P<0.001);B:OS標本中NFYA基因高表達組較低表達組預后較差(P<0.05)。
2.5 構建circRNA-miRNA-mRNA網絡 我們使用環狀RNA hsa_circ_0003568,及其靶向的2個miRNA和13個靶基因用Cytoscape構建了circRNA-miRNA-mRNA可視化網絡,見圖6A。同時,根據與生存預后顯著相關的mRNA,構建了hsa_circ_0003568-miR-140-3p/miR-502-5p-CDK6/NFYA可視化調控亞網絡,見圖6B。
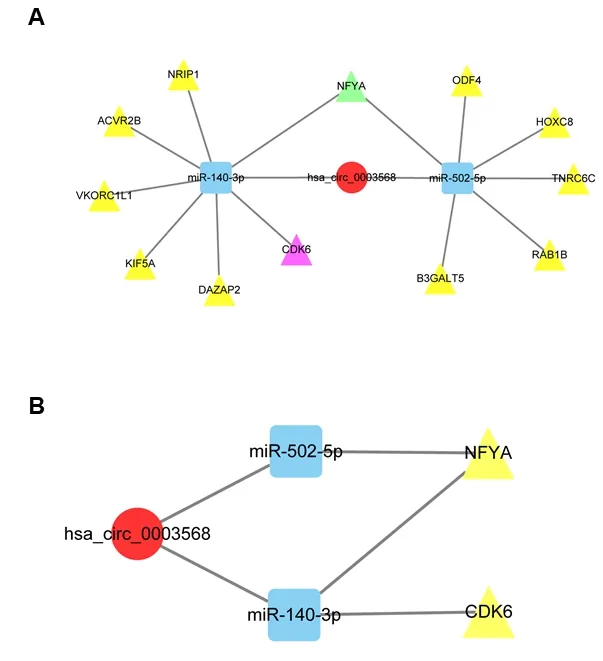
A:OS中hsa_circ_0003568-miRNA-mRNA相互作用的ceRNA網絡。紅色代表circRNA,藍色代表miRNA,黃色代表mRNA,粉色代表CDK6和綠色代表NFYA;B:基于有生存預后潛力的mRNA構建hsa_circ_0003568-miRNA-mRNA亞網絡。紅色代表circRNA,藍色代表miRNA,黃色代表mRNA。
3 討論
OS是一種常見的骨惡性腫瘤,如何治療OS仍然是困擾人類健康的一大難題。目前OS的治療主要是手術和化療,但患者預后仍不理想。盡管先前的研究[8]已經表明許多基因與OS的發生和發展有關,然而,OS的分子機制尚不清楚,闡明OS發展的分子機制仍是當務之急。近年來一些非編碼RNA被證實在OS的發病機制中發揮重要作用。circRNAs代表了一類新的保守內源性RNA,通過發揮miRNAs海綿作用調節下游靶基因的表達實現生物學功能[9]。與其他非編碼RNA如miRNAs和長非編碼RNA (long noncoding RNAs,lncRNAs)相比,circRNAs在哺乳動物細胞中具有高度保守性和高度穩定性[10-11]。這些特性使circRNAs有可能成為理想的生物標記物和潛在的治療靶點。ceRNA假說描述了轉錄組中不同類型的編碼成員和非編碼成員如何通過miRNAs相互通信,競爭與miRNAs結合,然后調控彼此間的表達,從而構建復雜的轉錄后調控網絡,有利于深入了解circRNAs在OS進展中的分子機制[12]。目前,雖然越來越多的研究人員已經開始研究circRNAs的潛在功能,但它們的臨床價值在很大程度上仍不為人所知。在本研究中,發現環狀RNA hsa_circ_0003568在OS細胞系中高度表達,并通過生物信息學分析預測miR-140-3p和miR-502-5p是hsa_circ_0003568的靶向miRNAs。研究發現miR-140-3p在結腸直腸癌中起到腫瘤抑制的作用,在體內過表達miR-140-3p可抑制結腸直腸癌腫瘤生長[13]。此外,miR-140-3p也被證明是肺癌的抑癌基因[14],在肝癌中可以增強肝癌細胞對索拉非尼的敏感性,抑制肝癌上皮-間質轉化、侵襲和轉移[15]。研究還發現miR-140-3p通過直接調控三聯體基序28的表達來抑制乳腺癌的增殖和遷移[16]。由此說明,miR-140-3p在許多癌癥中扮演著抑癌作用。同時,越來越多的研究表明miR-502-5p也是一種腫瘤抑制因子,可以增強細胞凋亡、抑制腫瘤細胞增殖,在許多癌癥中發揮重要的抑癌作用,包括乳腺癌、結腸癌、膀胱癌、胃癌、肝癌和非霍奇金淋巴瘤等[17-22]。因此,hsa_circ_0003568在OS中可能通過抑制miR-140-3p和miR-502-5p的表達加速了OS的生長。
本研究還進行了生物信息學分析預測了miR-140-3p和miR-502-5p的潛在靶點。結果表明CDK6及NFYA是miR-140-3p的潛在直接靶點,同時,我們還發現CDK6及NFYA在OS患者的生存分析中具有顯著的預后差異。CDK6在細胞周期進程中至關重要,抑制CDK6會導致腫瘤細胞不受控制的增殖,其途徑是導致不依賴正常細胞外信號的增殖,或超越確保基因組完整性和穩定性的檢查點[23]。CDK6在細胞進展中的作用已經在多種癌癥中被發現,包括膠質母細胞瘤、黏液纖維肉瘤和淋巴惡性腫瘤[24-25]。最近的多項研究表明,NFYA在多種癌癥中的表達增加與不良預后相關,包括肝癌、前列腺癌、結腸直腸癌等[26-28]。此外,研究發現增加NFYA的表達可促進OS細胞的惡性表型[29]。有趣的是,NFYA同時也是miR-502-5p的潛在靶點,由此暗示hsa_circ_0003568可能通過海綿抑制miR-140-3p和miR-502-5p調控CDK6和NFYA的表達從而參與OS的發生發展過程。
綜上所述,本研究驗證了hsa_circ_0003568在OS中高表達以及基于生物信息學預測構建了hsa_circ_0003568-miR-140-3p/miR-502-5p-CDK6/NFYA的可視化調控網絡,提示hsa_circ_0003568可能是OS潛在的治療目標和預后的生物標志物,為將來探索其在OS預后判斷中的作用提供了新的途徑。

