厄洛替尼耐藥的非小細胞肺癌細胞的差異表達mRNAs和lncRNAs分析
李樹斌 于 鴻 張耿月
肺癌是世界范圍內癌癥死亡的主要原因[1-3]。近年來,雖然臨床和實驗腫瘤學研究都取得了可喜的進展,但肺癌患者的預后仍不理想,5年總生存率僅在15%左右。非小細胞肺癌(non-small cell lung cancer, NSCLC)約占所有肺癌病例的85%[2-3]。2004年在NSCLC患者中首次發現表皮生長因子受體(epidermal growth factor receptor, EGFR)突變。研究表明,大多數攜帶EGFR突變的患者對EGFR酪氨酸激酶抑制劑(EGFR tyrosine kinase inhibitors, EGFR-TKIs)如厄洛替尼(erlotinib)和吉非替尼(gefitinib)治療敏感。然而這些對治療敏感的患者在治療10~16個月后會不可避免的獲得抗藥性,導致癌癥進展[4]。本文分析了1個NSCLC 厄洛替尼耐藥芯片數據集,篩選與厄洛替尼耐藥相關的差異表達mRNAs和lncRNAs,并通過生物信息學方法對獲得的結果進行初步驗證。
材料和方法
一、數據下載和mRNAs差異表達分析
在Gene Expression Omnibus(GEO)數據庫中挑選合適的基因表達譜數據集GSE80344[5]。該數據集的數據來自厄洛替尼耐藥的HCC827細胞和敏感細胞[6],技術平臺為GPL16699:Agilent-039494 SurePrint G3 Human GE v2 8×60K Microarray 039381(http://www.agilent.com)。首先下載全部注釋文件和原始數據,去掉不相關的樣本信息和數據,然后通過GEO2R分析差異表達基因,篩選標準為|Fold change|>1.5,Pvalue<0.01。對于多個探針對應一個基因的情況,取平均值作為這個基因的表達值。
二、厄洛替尼耐藥的HCC827細胞的差異表達mRNAs的gene ontology(GO)和Kyoto Encyclopedia of Genes and Genomes(KEGG)pathway分析
GO分析通過Panther(http://www.pantherdb.org)數據庫進行,篩選出的差異表達基因按照3個類別進行功能分類:molecular function(MF)、biological process(BP)、cellular component(CC)。然后通過KEGG(http://www.kegg.jp/)數據庫分析篩選得到的差異表達mRNAs通過哪些信號通路發揮作用。
三、構建蛋白相互作用(protein-protein interaction, PPI)網絡
通過STRING(http://string-db.org)數據庫將篩選得到的厄洛替尼耐藥的HCC827細胞的差異表達mRNAs轉化為基因編碼蛋白構建PPI網絡,將得到的結果用MCODE of Cytoscape軟件分析,得到蛋白相互作用關系圖并從中挑選出關鍵的候選基因[7]。
四、厄洛替尼耐藥的HCC827細胞的差異表達lncRNAs表達譜分析
通過https://www.gencodegenes.org/releases/current.html 下載lncRNAs注釋信息并與Agilent probe set ID匹配得到Agilent芯片的lncRNAs注釋信息,再將1.1中下載的原始表達數據與lncRNAs注釋信息匹配[8],得到厄洛替尼耐藥的HCC827細胞的差異表達lncRNAs。
五、厄洛替尼耐藥的HCC827細胞的差異表達lncRNA-mRNA相互作用的CNC(coding-non-coding)共表達網絡分析
六、厄洛替尼耐藥的HCC827細胞的差異表達lncRNAs的GO分析和KEGG分析 與1.2的分析方法相同。
七、驗證篩選得到的關鍵基因與肺癌患者臨床特征的相關性
使用蛋白表達數據庫驗證篩選得到的關鍵基因與肺癌患者預后(overall survival, OS)的相關性(https://cancergenome.nih.gov/),驗證數據來自TCGA(the Cancer Genome Atlas)。
結 果
一、篩選厄洛替尼耐藥的HCC827細胞的差異表達mRNAs結果
在數據集GSE80344中選擇厄洛替尼耐藥的HCC827細胞和敏感細胞作為分析對象,將樣本分為敏感細胞組(GSM2124850、GSM2124858)和耐藥細胞組(GSM2124846、GSM2124847、GSM2124849、GSM2124852、GSM2124853、GSM2124854、GSM2124855、GSM2124857、GSM2124860、GSM2124861)。通過GEO自帶的GEO2R分析工具,我們最終得到差異表達mRNAs共644個(耐藥細胞vs.敏感細胞),其中128 mRNAs表達上調,516 mRNAs表達下調,見表1。

表1 厄洛替尼耐藥的HCC827細胞的差異表達mRNAs(差異表達顯著的前100 mRNAs,耐藥細胞 vs.敏感細胞)
二、厄洛替尼耐藥的HCC827細胞的差異表達mRNAs的GO分析和KEGG結果
上述篩選得到的差異表達mRNAs的功能富集分析,見圖1。主要基于molecular function(MF)、biological process(BP)、cellular component(CC)三個類別。這些mRNAs通過參與與癌癥有關的信號通路發揮作用,這些信號通路主要包括癌癥的轉錄失調、Ras信號通路、PI3K-Akt信號通路、p53信號通路等。
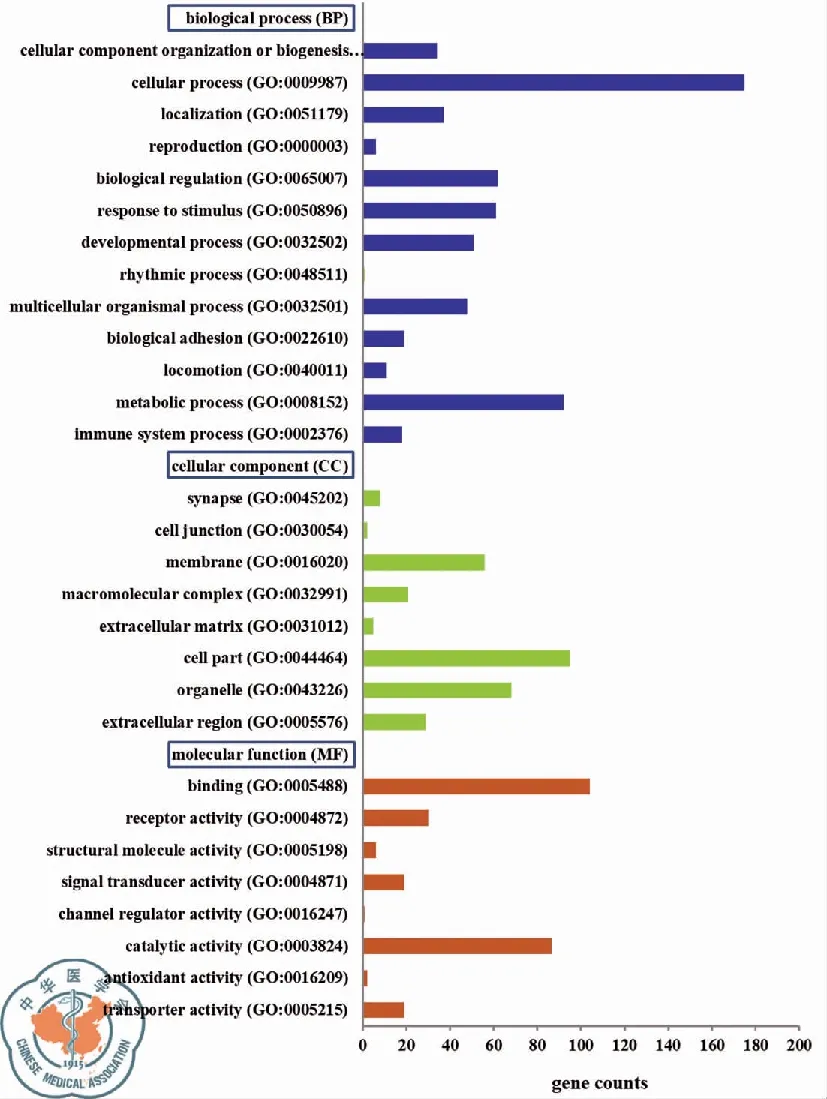
圖1 厄洛替尼耐藥的HCC827細胞的差異表達mRNAs的GO分析結果圖
三、構建PPI網絡
為了進一步挖掘厄洛替尼耐藥相關的關鍵基因,我們通過STRING(http://string-db.org)數據庫和MCODE of Cytoscape軟件構建了蛋白相互作用網路,計算結果來自414 nodes和427 edges。
四、厄洛替尼耐藥的HCC827細胞的差異表達lncRNAs結果
通過對探針的注釋匹配,我們共匹配到10353 Agilent probe set ID,對應6539 lncRNAs(包括TEC、sense_overlapping、sense_intronic、processed_transcript、lincRNA、bidirectional_promoter_lncRNA、antisense、3prime_overlapping_ncRNA)。再將獲得的結果與篩選的差異表達mRNAs的結果匹配,按照表達值│Fold change│> 1.0的標準,最終得到了差異表達的367 lncRNAs,其中137 lncRNAs表達上調,230 lncRNAs表達下調(耐藥細胞vs.敏感細胞)。我們注意到篩選出的lncRNAs數量大約是mRNAs的一半,這與文獻的報道一致,提示我們lncRNAs的表達在敏感和耐藥細胞之間可能更保守[9]。
五、厄洛替尼耐藥的HCC827細胞的差異表達lncRNA-mRNA的CNC共表達網絡構建
基于相關性分析的CNC網路可以用于分析lncRNAs的功能和作用機制,我們的結果表明有23 個關鍵的lncRNAs,包括表達上調的lncRNA RP11-620J15.3、RP11-227F19.2(LNC-LIMCH1-1)、LINC00472、CTD-3110H11.3、RP11-386G11.8(LNC-TUBA1C-2)、RP11-523L20.1(LINC02134)、AC005592.2(SPRY4-AS1)、CTD-3094K11.1(LNC-BLM-6)、ST7OT1(ST7-AS1)、RP1-232P20.1、RP4-781K5.5(LNC-COA6-6)、RP11-165M6.1(LINC00460)、RP11-442N24_B.1(LINC01715);表達下調的lncRNA RP11-115J16.1、RP4-798P15.3、AP001422.3(LINC01423)、AC107070.1(LINC01304)、RP11-783K16.14(LNC-TRPT1-1)、AC145676.2、RP11-498P14.4(LNC-CCDC180-4)、RP6-65G23.3(LNC-PCNX1-2)、AC097499.1(LINC01889)、AC084809.3(LNC-TMEM98-2)在CNC網絡中與mRNAs相互作用。這些lncRNAs中的大多數尚未見報道。一個lncRNA可以與多個mRNAs共表達,多個lncRNAs也可以與一個mRNA共表達,說明lncRNAs和mRNAs之間的相互作用的復雜性。
六、厄洛替尼耐藥的HCC827細胞的差異表達lncRNAs的GO和KEGG分析結果
篩選得到的lncRNAs的功能可能主要涉及免疫功能、代謝途徑以及癌癥相關信號通路。
七、篩選得到的mRNAs和lncRNAs與NSCLC患者臨床特征的相關性結果
為了驗證上述分析結果的可靠性,我們使用來自TCGA的肺腺癌患者(Lung Adenocarcinoma, LUAD)的臨床數據分析篩選得到的mRNAs和lncRNAs與患者臨床預后的相關性(https://cancergenome.nih.gov/, http://www.oncolnc.org/)。這些基因包括差異表達篩選中平均表達值倍數變化較大的和PPI網絡中的關鍵基因,見圖2。從圖2可見,肺腺癌患者GPR68和VNN1高表達,MUC1和PRODH低表達,這些患者的總生存期降低,預后差。篩選得到的差異表達的lncRNAs中LINC00087(SMIM10L2B)和H19低表達的肺腺癌患者的總生存期顯著降低。這些結果初步證明我們上述分析結果的可靠性。
討 論
本文的原始數據來自GEO數據庫。我們在GEO數據庫中挑選合適的表達譜數據集GSE80344[5]。該數據集的數據來自厄洛替尼耐藥的HCC827細胞和敏感細胞[6],技術平臺為Agilent-039494 SurePrint G3 Human GE v2 8x60K Microarray 039381。Agilent公司的SurePrint技術可以靈活地、大規模地原位合成60mer的寡核苷酸探針,每個探針的設計都經過反復試驗篩選優化,其CV值、檢測基因數目、與Taqman探針的重復性均處于國際同行業領先水平。

圖2 來自TCGA數據庫的肺腺癌患者的總生存期分析
通過篩選分析,我們最終得到差異表達mRNAs共644個(耐藥細胞vs.敏感細胞)。GO分析結果表明,這些mRNAs的功能主要涉及應激、代謝、免疫、黏附等生物學進程(biological process, BP),突觸、細胞連接、細胞外基質等細胞成分(cellular component, CC),受體活性、信號轉導、催化活性等重要的分子功能(molecular function, MF)。通過與癌癥有關的信號通路發揮作用,這些信號通路主要包括癌癥的轉錄失調、Ras信號通路、PI3K-Akt信號通路、p53信號通路等。其中涉及到的關鍵的mRNAs包括FGFR1、SATB2、PTX3、AXL、SOX7、H19、MUC1、ICAM2、TP53、IGF1等,這些基因中即有被廣泛研究報道的,也有未見報道的,這為我們深入研究EGFR-TKIs的耐藥機制提供了有價值的線索。
通過構建PPI網絡,我們對篩選得到的與厄洛替尼耐藥相關的差異表達mRNAs進行更深入的分析,得到處于相互作用網絡關鍵節點的基因,這些基因包括CXCR4、PMCH、HTR1A、OPRM1、GNG7、CXCL2、GNGT2、CXCL6、ADORA1、APLN、CXCL13、CD40、IL7、CD5、IL4R、CD19、IRF8、IL1A、KLRC1、MUC1、CSF2、TP53、IFNG等,這些基因主要在細胞因子及其受體的相互作用、PI3K-Akt信號通路、Jak-STAT信號通路、癌癥的轉錄因子失調、NF-κappa B信號通路等中發揮作用,進一步證明我們篩選得到的基因可能在NSCLC EGFR-TKIs耐藥中發揮關鍵作用。
與同類研究相比,我們篩選得到的結果中如CEACAM6、CALB1、ICAM2、CEACAM6、CKMT1A、ATP8A1、ADORA1等差異表達基因已有同類報道[9-10],其余大部分基因未見相關報道,這主要是由樣本選擇和分析方法的差異造成的。
近年來數以萬計的lncRNAs被發現,其表達模式的改變與不同類型癌癥的發生和惡性轉化有關,但lncRNAs表達譜的潛在用途仍然沒有被系統研究。在多種腫瘤類型中,異常表達的lncRNAs已經被研究作為潛在的生物標志物和治療靶點[11-12]。lncRNAs的長度大于200 nt(nucleotides),缺乏或沒有蛋白編碼能力,可參與多種生物學過程,包括X染色質印跡、細胞分化、選擇性剪接、細胞命運決定、免疫反應等[14]。lncRNAs也可以通過不同的機制,如表觀遺傳修飾、作為miRNA的競爭RNA等調節腫瘤細胞的生長和轉移[15]。
目前一些研究已經證明lncRNAs,如HOTAIR[16]、MEG3[17]、CCAT1[18]等在NSCLC化療耐藥中發揮作用。但是非小細胞肺癌EGFR-TKIs耐藥中lncRNAs的表達模式和潛在的功能仍然是未知的。Ma等[19]從GEO數據庫中下載了3個數據集,分別是吉非替尼耐藥的GSE34228,厄洛替尼耐藥的GSE38310,crizotinib或X-376耐藥的GSE49508。他們隨后分析了這3個數據集的lncRNAs表達譜,從表達失調的lncRNAs中選取了上調的CASC9和下調的EWAST1(LINC00227)作為研究對象,用Pearson相關系數計算與之共表達的蛋白編碼基因,推測CASC9和LINC00277可能通過與這些PCGs相互作用調節EGFR-TKIs耐藥。Cheng[14]等分析了EGFR-TKIs敏感的PC9和耐藥的PC9/R肺癌細胞的lncRNAs表達譜,發現耐藥細胞和敏感細胞相比共有22,587 lncRNAs和17,479 mRNAs差異表達。他們發現上調的lncRNAs-BC087858位于一個與癌癥發展相關的FOX轉錄因子家族成員FOXC1(forkhead box protein C1)附近。FOXC1可以通過抑制E-cadherin表達誘導上皮間質轉化(epithelial-mesenchymal transition, EMT),促進細胞遷移和侵襲。FOXC1的表達也可以被EGF/ERK(epidermal growth factor/extracellular signal-related kinase)信號通路激發[20]。因此推測BC087858可能通過EMT在EGFR-TKIs耐藥中發揮作用。Xue等[9]利用GEO的數據集GSE49644和GSE60189構建了肺癌耐藥的lncRNAs-mRNAs共表達功能性網絡。結果表明有32 lncRNAs和81 mRNAs相互調節,其中起關鍵作用的是COL1A1、COL27A1、BMPR2。作者接著分析了lncRNAs和mRNAs與肺癌患者預后的相關性,發現2對lncRNAs-mRNAs與耐藥高度相關。Wu等[10]分析了對吉非替尼耐藥的HCC827-8-1和敏感細胞HCC827的lncRNA表達譜,發現有1,476 lncRNAs和1,026 mRNAs表達失調。作者在構建的lncRNA-TF(transcription factors)網絡中,篩選出了4個關鍵基因:E2F4、E2F1、uSF1、TFAP2C。它們可能在EGFR-TKIs耐藥中發揮重要作用。
本文分析了厄洛替尼耐藥的HCC827細胞的差異表達lncRNAs表達譜并篩選出差異表達顯著的lncRNAs。參考文獻報道的方法[8],我們得到了與厄洛替尼耐藥相關的差異表達lncRNAs共367個。這些lncRNAs可能在耐藥發生中發揮潛在作用,其中一些lncRNAs被報道在癌癥或其它疾病中異常表達,如ST7OT1(ST7-AS1)和LINC00472,lncRNA-H19已被報道與吉非替尼耐藥相關[14]。我們的分析發現了很多新lncRNAs,到目前為止還沒有在肺癌或其它腫瘤中被研究過,它們的功能尚待確定。
分析mRNAs-lncRNAs的共表達可以預測lncRNAs的功能[9],通過構建差異表達lncRNA-mRNA的CNC共表達網絡,我們得到了23 個關鍵的差異表達lncRNAs,其中表達上調的lncRNAs共13個,表達下調的lncRNAs共10個。這些lncRNAs中的大多數尚未見報道。GO分析和KEGG分析結果表明,這些lncRNAs的功能可能涉及細胞因子及其受體的相互作用、P53信號通路、藥物代謝、Ecm受體的相互作用等與癌癥有關的重要信號轉導通路,進一步說明EGFR-TKIs耐藥以及lncRNAs和mRNAs之間的相互作用的復雜性。
篩選得到的lncRNAs既可以作為耐藥的標志,也可以作為診斷和治療的新靶點。迄今為止相關的研究尤其關于EGFR-TKIs耐藥相關的研究報道還比較少。Cheng等[21]研究了NSCLC中lncRNA UCA1在EGFR-TKIs獲得性耐藥中的作用。結果表明,UCA1可能通過活化AKT/mTOR和ERK通路和EMT 促進吉非替尼耐藥。已報道lncRNA BC087858在吉非替尼耐藥的NSCLC細胞中高表達[14]。Pan等[22]檢測了不同NSCLC細胞和EGFR-TKIs耐藥的腫瘤組織中lncRNA BC087858的表達,發現高表達lncRNA BC087858的NSCLC患者預后差。BC087858能夠通過上調Snail和ZEB1表達促進EMT,也能通過上調pEGFR、pAKT、pERK表達活化PI3K/AKT和MEK/ERK信號通路,從而促進EGFR-TKIs耐藥。Wang等[23]分析了對吉非替尼耐藥的PC9-R細胞和敏感細胞的lncRNA表達譜之間的差異。結果表明MIR31HG通過促進p-EGFR、p-PI3K、p-AKT、p-Mdm-2表達活化EGFR/PI3K/AKT信號通路在吉非替尼耐藥中發揮作用。一個已知的LncRNA,生長停滯特異性轉錄因子5(growth arrest-specific transcript 5, GAS5)被報道在肺癌組織中表達降低[24],并與較大的腫瘤體積、低分化、較高的TNM分期相關[25]。
為了驗證篩選得到的差異表達lncRNAs和mRNAs與肺癌患者臨床特征之間的相關性,我們通過來自TCGA的肺腺癌患者的臨床數據進行生存分析驗證,初步證明我們的數據分析的可靠性。
本文也存在不足之處。首先,對于許多已被鑒定出來的lncRNAs來說,有關它們的可能的作用途徑的信息仍然是有限的,雖然它們的差異表達模式提示在NSCLC EGFR-TKIs耐藥中有潛在作用,但缺乏直接的證據支持。第二,Agilent 芯片代表大部分但不是所有可能存在的lncRNAs。我們鑒定出的候選lncRNAs也不能代表全部可能在NSCLC EGFR-TKIs耐藥中發揮作用的lncRNAs。第三,我們使用的數據集是分類不均衡的,樣本量太少,這樣的數據不平衡會增加小樣本數據的一些重要屬性被忽略的可能性。第四,臨床樣本和細胞樣本的基因表達譜是有差異的。
對于后續的研究設想,綜合我們的分析結果和相關文獻報道,腫瘤耐藥與腫瘤細胞發生上皮間質轉化(EMT)密切相關。在NSCLC中,EMT也被報道與TKI類藥物的耐藥有關。在我們篩選得到的與厄洛替尼耐藥有關的差異表達lncRNAs和mRNAs中,GAS6/AXL通過調節EMT促進NSCLC EGFR-TKIs耐藥[26-27]。AXL是酪氨酸激酶受體,GAS6是AXL的配體。在多種類型的腫瘤中,AXL過表達與細胞生存、增殖、侵襲、遷移,細胞間黏附、抗凋亡等生物學過程相關[28],同時AXL可以誘導PI3K/AKT、STAT3、MAPK、nuclear factor κB下游活化。我們的結果表明與GAS6和AXL相互作用的lncRNAs有SPRY4-AS1(SPRY4 antisense RNA 1)、LINC00460、LINC01715、LINC00472、RP6-65G23.3、RP11-620J15.3、CTD-3110H11.3、RP11-165M6.1等。這些lncRNAs的功能尚未被深入研究,對于它們與GAS6/AXL之間的相互作用的研究可能有助于發現新的診斷和治療EGFR-TKIs耐藥的靶點。

