西藏不同產區曲拉細菌群落結構的比較分析
劉振東,畢娜,李哲,李梁,羅章,薛蓓*,汪雯翰
1(西藏農牧學院 食品科學學院,西藏 林芝, 860000)2(西藏自治區藏藥審評認證中心,西藏 拉薩,850000)3(上海市農業科學院食用菌研究所,上海, 201403)
曲拉(藏語,指奶干渣)是藏區傳統發酵乳制品之一,由牦牛奶脫脂后,在自然條件下發酵酪蛋白凝固并風干制成,屬于粗奶酪的一種[1-2]。作為傳統的藏族食品,曲拉具有蛋白質含量高、保存時間長、攜帶方便等優點,在整個藏區被廣泛食用[3-4]。
曲拉作為一種藏區典型的發酵型乳制品,其制作工藝多采用傳統的手工開放式自作,因此不同的制作環境直接關系到曲拉的微生物組成,進而影響曲拉的風味、安全及營養價值[5-7]。
基于微生物分離、純化、培養及鑒定的傳統研究方法難以獲得培養困難及痕量的微生物,而且難以展現環境中完整的微生物種群結構及生態關系[8-9]。目前,高通量測序技術已經逐漸成為研究微生物組成及結構的一種重要方法,近年該技術已廣泛用于原料乳和各類發酵乳等乳制品生態位的研究[10-11]。
本研究旨在利用高通量測序技術,全面了解西藏7個不同產區牦牛曲拉的細菌群落結構及相互差異,為西藏不同產區曲拉的安全性評價和功能微生物的挖掘奠定一定的理論基礎。
1 材料與方法
1.1 材料與試劑
21份牦牛曲拉樣品,分別來自西藏拉薩(LS)、日喀則(RKZ)、山南(SN)、昌都(CD)、林芝(LZ)、那曲(NQ)和阿里共7個地區;E.Z.N.A.Soil DNA試劑盒,美國OMEGA公司;Qubit2.0 DNA檢測試劑盒,美國Invitrogen公司;Q5?High-Fidelity DNA Polymerase,美國NEB公司;凝膠回收試劑盒,德國Qiagen公司。
1.2 儀器與設備
5424R高速冷凍離心機,德國Eppendorf公司;DYY-6C型電泳儀,北京六一儀器廠;T-100 PCR儀,美國Bio-rad公司;MiSeq高通量測序儀,美國Illumina公司;BG-gdsAUTO(130) 凝膠成像系統,北京百晶生物技術有限公司。
1.3 實驗方法
1.3.1 樣品的采集
2018年6月采集拉薩市林周縣、日喀則市薩迦縣、山南市隆子縣、昌都市八宿縣、林芝市米林縣、那曲市申扎縣、阿里地區改則縣7個地區的曲拉樣品,分別編號為LS、RKZ、SN、CD、LZ、NQ、AL。將所有樣品置于無菌離心管中,放入冰盒保存并立即運送到實驗室,置于-80 ℃冰箱中備用。
1.3.2 DNA提取和PCR擴增
對于不同來源的微生物組樣本,根據E.Z.N.A.?soil試劑盒說明書提取各牦牛曲拉樣品中的總DNA,然后通過0.8%瓊脂糖凝膠電泳的方法檢測總DNA的提取質量。本研究采用NEB公司的Q5高保真DNA聚合酶保證結果的可靠性,采用標準細菌V3-V4引物338F(5’-ACTCCTACGGGAGGCAGCA-3’)和806R(5’-GGACTACHVGGGTWTCTAAT-3’)進行PCR擴增。
PCR體系(25 μL):基因組DNA模板2 μL,dNTP 2 μL,ddH2O8.75 μL,緩沖液5 μL,Q5 DNA 0.25 μL,DNA模板2 μL,5×GC緩沖液5 μL。
PCR擴增條件:94 ℃預變性2 min;94 ℃變性30 s,55 ℃退火30 s,72 ℃延伸40 s,擴增40個循環;72 ℃延伸1 min。盡量保證同一批樣本的擴增條件一致。
1.3.3 文庫建立
采用三代測序技術制備測序文庫。把通過PCR擴增得到的產物進行末端修復,將黏性末端修整為平末端;在DNA序列的3′端添加A堿基以防止DNA片段自連;在DNA序列5′端添加含有文庫特異性標簽的測序接頭;對上述連上接頭的DNA片段進行PCR擴增,從而富集測序文庫模板,并采用BECKMAN AMPure XP Beads再次純化文庫富集產物;最后對文庫做最終的片段選擇與純化。
1.3.4 Illumina MiSeq測序
將Illumina MiSeq測序得到的雙端序列數據進行質量控制,以97%相似性為分類標準得到OTUs。然后在GenBank數據庫進行Blast比對,并提交到美吉生物的云分析平臺進行多樣性分析(http://www.i-sanger.com/)。
2 結果與分析
2.1 測序數據統計及Alpha多樣性分析
采用Illumina MiSeq平臺對7個地區曲拉樣品測序結果進行分析統計,結果如表1所示。由表1可知21個樣品總計測得有效序列條數為(241 148±23 200)條,以97%的序列相似度作為OTU劃分閾值,共產生(3 994±78)個OTUs,由序列數及OTUs可以看出,曲拉中細菌種類繁多、物種豐富,且不同來源的曲拉存在明顯差異。不同區域曲拉樣品間Alpha多樣性統計中的Chao1指數、Ace指數差異顯著(P<0.05)。

表1 不同樣本的測序數據統計
注:“-”表示無需統計;不同小寫字母表示差異顯著(下同)
2.2 稀釋曲線
由圖1可知,當樣品測序量較低時,每個樣品的Shannon指數隨著測序量增加呈顯著上升趨勢;但隨著測序量逐漸增加,各曲拉樣品的Shannon指數增加逐漸趨于緩慢;當樣品測序量到達5 000后各樣品的Shannon指數基本穩定,說明樣品測序量達到5 000時已足以反映21個曲拉樣品中細菌群落多樣性。

圖1 各樣品測序的稀釋曲線
2.3 在門水平上各樣品菌群分類及比較
由表2可知,7個產地的曲拉樣品共檢測出24個門,其中LS檢測出全部24個菌門,RKZ中檢測出6個菌門,SN中檢測出7個菌門,CD中檢測出6個菌門,LZ中檢測出12個菌門,NQ中檢測出20個菌門,而AL最少僅檢測出2個菌門。7個地區曲拉樣品的主要優勢菌門均為厚壁菌門和變形菌門,除LS和CD外,其他5個地區曲拉樣品的第一優勢菌均為Firmicutes,其豐度值在61.01%~96.54%,LS和CD的第一優勢菌Proteobacteria,其豐度值在40.60%~75.09%。
2.4 在屬水平上各樣品菌群分類及比較
如表3所示,對7個地區21份曲拉樣品從屬水平進行鑒定共鑒定出550個屬,其中LS中檢測出409個屬,RKZ中檢測出47個屬,SN中檢測出69個屬,CD中檢測出36個屬,LZ中檢測出99個屬,NQ中檢測出330個屬,AL中檢測出15個屬。各地區樣品在屬水平上檢出的種類差異較大,LS>NQ>LZ>SN>RKZ>CD>AL。

表2 各樣品門水平上菌群分布相對豐度 單位:%

表3 各樣品屬水平上菌群分布相對豐度 單位:%
如表4所示,從屬水平分析,多數產地曲拉樣品中優勢菌屬主要為乳球菌屬Lactococcus、乳桿菌屬Lactobacillus,其中RKZ中Lactococcus為(63.73±8.29)%,Lactobacillus為(4.15±1.38)%;SN中Lactococcus為(25.82±1.42)%;CD中Lactococcus為(46.62±4.60)%,Lactobacillus為(9.20±6.49)%;AL中Lactococcus為(85.13±2.99)%,Lactobacillus為(8.57±1.33)%,與曹磊等[12]研究的青海甘南地區牦牛曲拉群落結構存在共性的結果一致。乳桿菌屬和乳球菌屬均為乳酸菌,可在曲拉制作的初期產生大量的乳酸、乙酸和一定量的胞外多糖影響曲拉風味[13-14],從優勢菌角度分析,以上幾個地區的曲拉風味特點存在一定的相似性。而LS中優勢菌屬與其他地區差異較為明顯,其第一優勢菌為雷爾氏菌屬Ralstonia為(17.25±7.05)%,且致病菌假單胞菌屬Pseudomonas相對豐度達(16.05±3.22)%、蒼白桿菌Ochrobactrum達(9.52±3.19)%,均明顯高于其他地區,從優勢菌角度分析其風味及品質特點相對其他地區具有一定特殊性。
2.5 物種豐富度熱圖
根據OTU的豐度數據,熱圖可在屬水平上將不同的OTU分塊聚類,并根據顏色梯度反應不同樣品中細菌群落相似性、差異性及物種聚類關系。利用R語言繪制熱圖,可直觀地顯示不同產區曲拉樣品中細菌的差異。由圖2可知,上側為樣品聚類樹,左側為OTU聚類樹,西藏7個地市的21個曲拉樣品可分為2個大聚類:LS1和LS3可聚為一類,其余19個樣品聚為一類;物種豐富度上紅色越亮代表該菌屬在該樣品中的相對含量高于橫向的其他樣品,綠色越亮則相反,不同地區主要優勢菌屬存在一定差異。

表4 不同地區曲拉優勢菌分布表 單位:%
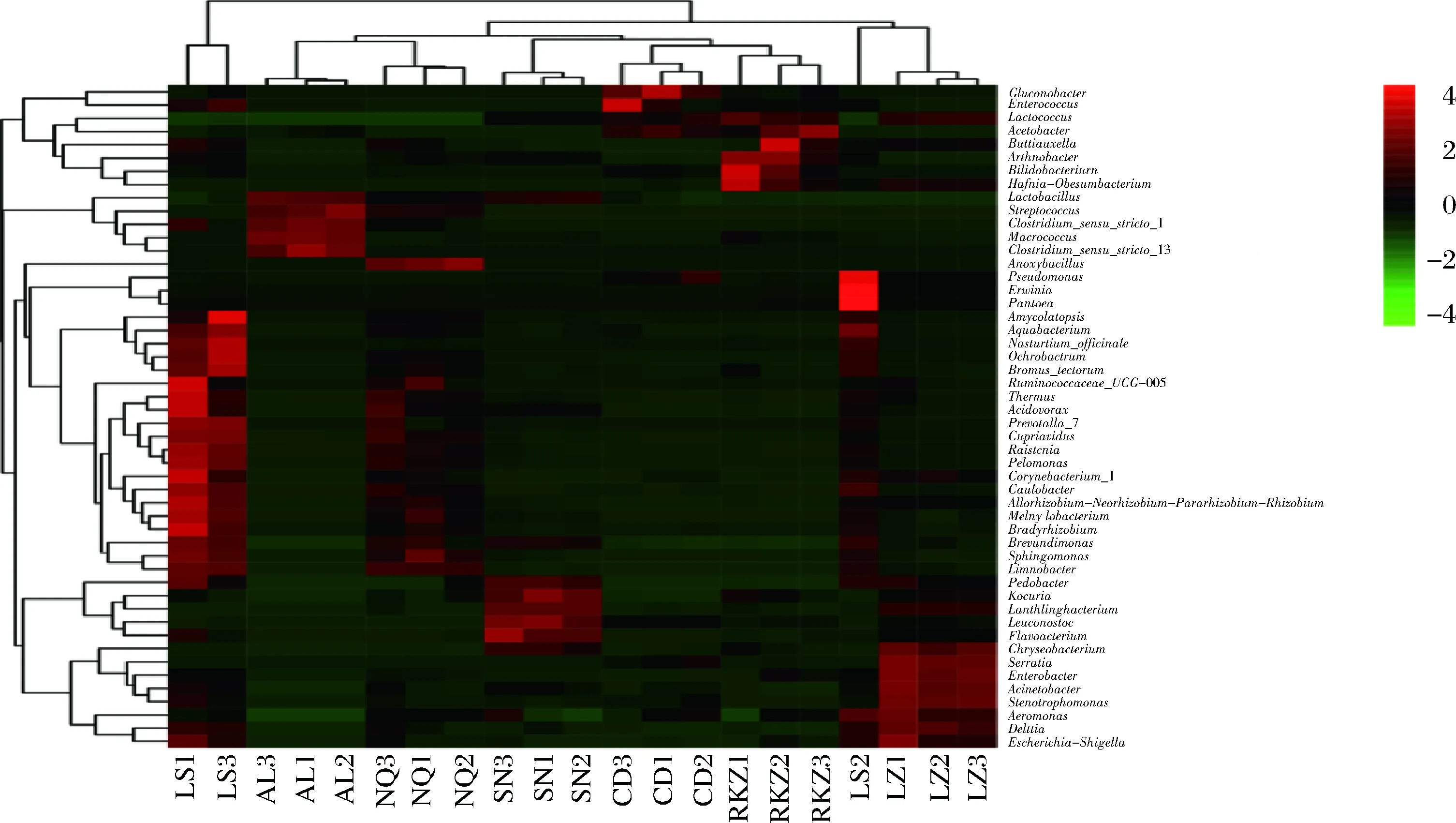
圖2 各樣品不同屬組成和相對豐度熱圖
2.6 基于UniFrac距離的樣本聚類分析
由圖3可知,細菌群落第1主成分(PC1)的貢獻率是64.87%,細菌群落第2主成分(PC2)的貢獻率是21.35%,兩者之和為86.22%,大于70%,表明兩個成分較好地代表了曲拉樣品中的細菌群落信息。不同曲拉樣本的菌群與地理位置存在一定關系,同一個采樣區的樣本具有一定的聚類趨勢,同時發現LZ、RKZ和CD可聚為一類,而其他4個地區的樣品較為離散,各自獨立。

圖3 PCA分析的樣本二維排序圖
2.7 UPGMA聚類分析圖
聚類分析主要指層次聚類(hierarchical clustering)的分析方法,以等級樹的形式展示樣品之間的相似度,通過聚類樹的分枝長度衡量聚類效果的好壞。樣本根據彼此之間的相似度聚類,樣本間的分枝長度越短,兩樣本越相似。由圖4可知,不同采樣地的樣品聚集到不同的類別,說明不同來源樣品的微生物多樣性存在一定的差異性,這與PCA分析的結果一致。

圖4 各曲拉樣品聚類分析圖
2.8 樣品功能類群分布統計
通過將現有的16S rRNA基因測序數據與代謝功能已知的微生物參考基因組數據庫相對比,從而實現對細菌和古菌代謝功能的預測。由圖5可知,7個樣品中細菌群落的代謝功能主要集中在氨基酸代謝(amino acid metabolism)、碳水化合物代謝(carbohydrate metabolism)和能量代謝(energy metabolism),其中AL和SN樣品在氨基酸代謝豐度上明顯低于其他5個地區樣品,而在碳水化合物代謝豐度上LS樣品顯著地區其它6個地區樣品,而在能量代謝豐度上7個地區樣品差異不顯著。

圖5 各曲拉樣品功能類群分布圖
3 討論
本研究采用高通量測序技術對西藏7個地市共計21份曲拉樣品的細菌群落結構進行分析,從屬水平上群落結構分布可知,不同地區曲拉細菌多樣性存在一定差異且存在一定的地域獨立性,如拉薩(LS)和那曲(NQ)樣品的細菌種類遠高于其他5個地區,細菌豐度上除拉薩地區革蘭氏陰性植物病原細菌雷爾氏菌屬(Ralstonia)和致病菌假單胞菌屬(Pseudomonas)占主要優勢菌群外,其他6個地市的優勢菌均主要集中于發酵乳制品常見菌乳球菌屬(Lactococcus)和乳桿菌屬(Lactobacillus),這6個地區曲拉的主要優勢細菌群落結構與SUN等[15]對青海牦牛酸奶進行純化培養和16S rRNA鑒定的結果一致,同時與其他傳統發酵乳制品如酸牛奶、酸馬奶、酸駝奶等細菌群落多樣性類似[16-17],在門水平上常見原料乳的細菌組成差異不明顯[18-19]。而本研究發現拉薩地區樣品存在一定的植物病原菌菌屬和條件致病菌菌屬。通過功能分析發現曲拉中細菌群落有助于人體消化過程中的氨基酸和碳水化合物代謝。
由于本研究采樣點較單一及樣品量相對較少,并不能直接反映出制備環境直接造成的影響,為進一步探討拉薩地區牦牛曲拉的細菌多樣性及安全性,應增加采樣點和采樣量,進一步開展細致研究。

