基于PCR-DGGE技術分析宣威火腿中真菌群落結構
鄒穎玲,劉姝韻,王桂瑛*,普岳紅,葛長榮,廖國周*
1(云南農業大學 食品科學技術學院,云南 昆明,650201)2(云南農業大學 云南省畜產品加工工程技術研究中心,云南 昆明,650201)
宣威火腿是云南省傳統名特優產品,其以“身穿綠袍,色香味美,咸淡相宜,風味獨特,食而不膩”等特點名揚海內外,距今已有270年的歷史[1],它是選用宣威當地傳統烏金豬的后腿,利用宣威獨特的氣候及地理條件在當地經過腌制、發酵、成熟制作而成[2]。云南省宣威市獨特的地理環境和氣候條件造就了宣威火腿特定的品質[3]。獨特的天然環境,促進火腿微生物的大量繁殖,從而使火腿持續發酵[4]。微生物對肉制品的風味形成及品質有著獨特的作用,發酵時期,由于微生物的作用會使原料中的蛋白質、脂肪等發生生化變化,進而影響發酵肉制品的品質,促進其產生芳香化合物,提升肉制品的風味[5]。
聚合酶鏈式反應-變性梯度凝膠電泳(polymerase chain reaction-denaturing gradient gel electrophorsis,PCR-DGGE)是一種分析微生物群落的有效工具,MUZYERS[6]等首次將PCR-DGGE技術應用于研究微生物多樣性,近年來該項技術被應用于研究食品微生態、鑒定微生物、評價食品質量如益生菌制品[7]、酸面團[8]、奶酪[9]、肉制品[10]、甜酒曲[11]等。目前,利用PCR-DGGE技術分析不同加工時間宣威火腿中真菌群落結構還未見報道。基于此,本研究通過PCR-DGGE技術分析不同加工年份(1、2和3年)宣威火腿中的真菌群落結構,為進一步探討宣威火腿品質奠定基礎。
1 材料與方法
1.1 材料與試劑
宣威火腿樣品由云南省宣威市宣泰火腿有限公司提供,并分別在加工1、2和3年火腿的表面和內部(距肉表面5 cm)取約10 g肉樣品,標記為:A1、A2(1年腿的表面和內部),B1、B2(2年腿的表面和內部),C1、C2(3年腿的表面和內部)。后將6個樣品真空包裝,置于-20℃保存以待分析。
乙醇、冰醋酸、硝酸銀、37%甲醛、NaOH,國藥集團化學試劑有限公司;環境微生物DNA提取試劑盒、50×TAE buffer、ddH2O,北京博友順有限公司;DNA純化試劑盒、DH5α感受態細胞,天根生化科技公司;dNTPs、rTaq、pMD18-T Cloning Kit,日本Takara公司;40% Acrylamide/Bis,美國Bio-Rad公司;Formamide (deionized)、Urea、APS,美國Amresco公司;TEMED,美國Sigma公司;Poly-Gel DNA Extraction Kit,美國OMEGA公司。
1.2 儀器與設備
Centrifuge 5415D 離心機,德國Eppendorf公司;T-gradient PCR儀,德國Biometra公司;SDC-6恒溫槽,寧波新芝有限公司;Gel-Doc2000凝膠成像儀,美國伯樂Bio-Rad公司;JY-SPFT CTAB,北京軍意東方儀器有限公司;Dcode變形梯度凝膠電泳儀,美國伯樂Bio-Rad公司;DHP-9052恒溫培養箱,上海一恒有限公司;ZHWY-100C搖床,上海智城有限公司。
1.3 實驗方法
1.3.1 樣品中總DNA的提取
根據環境微生物DNA提取試劑盒方法對6個樣品進行總DNA提取。具體操作步驟如下,取肉樣品0.5 g置于離心管中,加入緩沖液沒過離心管,漩渦振蕩5 min,后用多層無菌紗布過濾收集濾液,轉入2 mL離心管;加入100 mg玻璃珠,在高速振蕩器上振蕩約5 min;加入蛋白酶K,充分混勻,55 ℃水浴消化30 min;12 000 r/min離心2 min直至沒有沉淀;取上清液加入200 μL DNA提取試劑盒溶液B和200 μL無水乙醇,混勻后轉移到吸附柱中靜置2 min;12 000 r/min離心2 min后,棄廢液,然后漂洗吸附柱2次;12 000 r/min離心2 min,將吸附柱放在50 ℃恒溫箱一段時間后加洗脫液,室溫放置5 min后,12 000 r/min離心1 min;往吸附柱中加入離心所得洗脫液,室溫放置2 min,12 000 r/min離心2 min,即可得到基因組DNA,所得DNA置于-20 ℃保存以待分析。
1.3.2 真菌18S rDNA基因的PCR擴增
真菌通用引物[12]如表1所示。

表1 用于18S rDNA擴增的通用引物
以樣品基因組DNA為模板,以GC-Fung和NS1為引物擴增樣品,得到18S rDAN高變區序列。PCR擴增體系(25 μL)為10× Ex Taq buffer(緩沖) 2.5 μL;dNTP(2.5 mmol/L)2 μL;ExTaqPolymerase(5 U/μL)0.25 μL;GC-FungF(10 mmol/L)1 μL;NS1(10 mmol/L)1 μL;模板DNA 0.05 μg;補ddH2O至25 μL。
PCR擴增程序為:94 ℃預變性5 min;94℃變性30 min,60 ℃復性30 s,72 ℃延伸15 s,30個循環;最終72 ℃延伸10 min。PCR產物采用OMEGA公司DNA Gel Extraction Kit純化回收。
1.3.3 變性梯度凝膠電泳(DGGE)
參考郭繼平等[13]的方法,稍作修改。取10 μL PCR的產物進行變性梯度凝膠電泳分析。變性梯度凝膠梯度為20%~40%,膠濃度為6%,電泳溫度控制在60 ℃,用220 V電壓預電泳5 min,接著在150 V條件下電泳8 h。電泳結束后、采用銀染法染色,條帶顯現后用Gel-Doc2000凝膠成像儀拍照、記錄。
1.3.4 條帶切膠回收與克隆測序
參考鄭艷等[14]的方法,用經過滅菌的手術刀切下待回收DGGE條帶,采用OMEGA 公司Poly-Gel DNA Extraction Kit回收目的條帶。以回收產物(2 μL)為模板,以Fung/NS1為引物進行PCR擴增。PCR擴增體系(50 μL)為10× PCR buffer 5 μL;dNTP(2.5 mmol/L)3.2 μL;rTaq(5 U/μL)0.4 μL;Fung(10 mmol/L)1μL;NS1(10 mmol/L)1 μL;模板DNA 1 μL;補ddH2O至50 μL。PCR擴增程序為94 ℃預變性4 min;94 ℃變性30 s,55℃復性30 s,72 ℃延伸30 s,30個循環;最后,72 ℃延伸10 min。將重新擴增的DNA片段切膠回收、純化后,連接到Pmd18-T載體上,并轉化至DH5α感受態細胞中,篩選陽性克隆,進行序列測定。
1.4 真菌多樣性指數計算
真菌多樣性指數是研究群落物種數和個體數以及均勻度的綜合指標。根據電泳圖譜中樣品條帶數目及每個條帶的強度(灰度),對各樣品中真菌多樣性指數(H)、均勻度(E)和豐富度(S)等指標進行分析。利用Quantity one軟件分析DGGE圖譜,而香農指數(H)、豐度(S)和均衡指數(E)等指標被用來比較不同樣品間的多樣性情況[15]。其算法參考鄭艷等[14]的方法,計算如公式(1)、公式(2)所示:

(1)
(2)
式中:pi為樣品中單一條帶的強度在該樣品所有條帶總強度中所占的比率;N為DGGE圖譜單一泳道上所有條帶的豐度;Ni為第i條帶的豐度;S為某樣品中總條帶數目。
2 結果與分析
2.1 宣威火腿中真菌18S rDNA的PCR擴增
以GC-Fung和NS1為引物,以從不同加工年份宣威火腿的表面和內部6個樣品中提取的總DNA為模板進行18S rDNA片段擴增,得到220 bp左右的片段(圖1),符合DGGE分析。
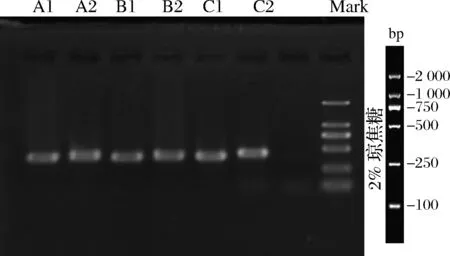
圖1 GC-PCR檢測
2.2 DGGE指紋圖譜及分析結果
對宣威火腿中的真菌總DNA進行DGGE分析,DGGE圖譜如圖2所示,泳道中每1條亮帶代表1種真菌,不同位置的條帶代表不同種屬真菌,條帶越亮代表相應的種屬真菌相對數量越多[16]。由圖2和圖3可知,共檢測到30條不同條帶,表明宣威火腿中豐富的真菌種類,不同加工年份宣威火腿樣品的電泳條帶分離效果較好,條帶的亮度也各有不同,其中1年腿表面有22個主要條帶,內部有19個;2年腿表面有17個,內部有22個;3年腿表面有23個,內部有17個。

圖2 宣威火腿樣品的DGGE圖譜

圖3 宣威火腿樣品的DGGE條帶強度示意圖
2.3 主要電泳條帶的測序比對結果分析
將宣威火腿樣品主要的30條電泳條帶進行克隆與測序,測序結果與GenBank中的序列進行比對得到表2。由表2可知,切膠測序的30個條帶中有27條可以在GenBank數據庫中找到,與其序列的同源性均在96%以上。
結合DGGE圖譜與表2可知,加工1年宣威火腿表面最亮的條帶為1、11和27,其中條帶1為Aspergilluspseudoglaucus(假灰綠曲霉),條帶11為Aspergillusoryzae(米曲霉),條帶27為Wallemiasebi。加工1年宣威火腿內部最亮的條帶為1、22和27,其中條帶22為Yamadazymatriangularis。加工2年宣威火腿表面最亮的條帶為1和5,其中條帶5為Aspergilluspenicillioides(帚狀曲霉)。2年宣威火腿內部最亮的條帶為1和14,其中條帶14為Aspergillusglaucus(灰綠曲霉)。加工3年宣威火腿表面最亮的條帶為1、3、16、22和27,其中條帶3為Phialosimplexcaninus,條帶16為Aspergillusniger(黑曲霉),條帶22為Yamadazymatriangularis。加工3年宣威火腿內部最亮的條帶為1、5和27,其條帶分別為Aspergilluspseudoglaucus(假灰綠曲霉)、Aspergilluspenicillioides(帚狀曲霉)和Wallemiasebi。由PCR-DGGE圖譜可知,不同加工年份宣威火腿的表面及內部均檢測出Aspergilluspseudoglaucus(假灰綠曲霉),由此可以推斷,Aspergilluspseudoglaucus(假灰綠曲霉)是宣威火腿的優勢菌種。

表2 宣威火腿樣品DGGE主要條帶的序列比對結果
2.4 樣品間真菌群落結構相似性指數與聚類分析
DGGE圖譜的相似性是通過Quantity One軟件進行分析的,通過分析得到不同加工年份宣威火腿真菌群落相似性指數。由表3可知,加工2年宣威火腿內部與加工3年表面的真菌群落結構相似性最高,為69.3%,加工1年內部與加工2年表面的真菌群落結構相似性最低,為42.7%。從宣威火腿表面看,加工1年的與加工3年表面真菌群落結構相似性為60.9%,分別比加工1年與加工2年表面真菌群落結構相似性、加工2年與加工3年表面真菌群落結構相似性高13.2%、15%。從宣威火腿內部看,加工1年的與加工2年內部真菌群落結構相似性為60.5%,分別比分別比加工1年與加工3年內部真菌群落結構相似性和加工2年與加工3年內部真菌群落結構相似高12.4%、4.9%。
圖4為不同加工年份宣威火腿表面及內部的條帶相似性聚類分析圖,由圖4可知,加工2年宣威火腿內部與加工3年火腿表面相似度為69%,說明兩者間差異較小。加工2年火腿表面與其他加工年份表面及內部5個樣品間的相似度為46%,說明2年火腿表面與其他5個樣品間差異較大。從整體看,不同加工年份火腿表面及內部真菌群落結構間存在一定的差異,這可能是隨著時間的變化,空氣對火腿的氧化,導致真菌群落結構發生了變化。

表3 戴斯系數比較宣威火腿樣品PCR-DGGE圖譜的相似性 單位:%

圖4 宣威火腿樣本間的聚類圖
2.5 宣威火腿樣品的真菌群落多樣性
微生物多樣性指數由于評估微生物群落的豐富度。香農指數(H)用于評估微生物群落的多樣性,H值越大,說明微生物的種類和數量越多,微生物群落多樣性越高;均勻度指數(E)用于評估微生物分布的均勻程度,E值越大,說明微生物群落分布越均勻;豐富度指數(S)是指一個群落或生境中物種數目的多寡[17]。由表4可知,不同加工年份宣威火腿表面及內部真菌群落多樣性有差異。加工3年宣威火腿表面及內部的香農指數分別是最高和最低的,說明其表面及內部真菌群落多樣性分別是最高和最低的,加工1年腿表面真菌群落分布最均勻,而加工3年腿表面真菌群落豐富度指數最大。

表4 宣威火腿各樣品間的真菌群落多樣性分析
3 討論
宣威火腿所具有的特殊風味與其漫長的發酵過程密切相關,微生物的代謝活動以及火腿表面大量霉菌的生長是宣威火腿獨特風味形成的基礎[18]。本研究發現,假灰綠曲霉、帚狀曲霉、假絲酵母屬存在于整個發酵期間的表面和內部。霉菌是宣威火腿發酵過程中的優勢真菌,這與黃艾祥等[19]對云南干腌火腿和甄宗圓等[20]對金華火腿的研究結果一致。霉菌的生長對火腿風味形成無直接作用,但對防止火腿氧化變質、抑制有害微生物的生長、提高火腿質量有間接作用[21]。酵母菌的存在不但是火腿整個發酵期的優勢有益菌群,而且對成熟火腿的VE、脯氨酸、色氨酸等香甜成分的增加及風味的形成起重要作用[22]。NUNEZ等[23]對伊比麗亞干腌火腿中酵母菌的研究發現,誕沫假絲酵母是原料腿的優勢菌,德巴利漢遜氏酵母是發酵腿、成熟腿的優勢菌。MUHAMED等[24]研究了從香腸中分離出來的微生物菌株抗輻射情況,經過抗輻射處理后,發現誕沫酵母菌(eandidazeylanoides)具有較強的抵抗力。本試驗經對云南宣威火腿分離鑒定出的酵母菌數量相對霉菌較少,最主要的有德巴利酵母屬和假絲酵母屬,這與黃艾祥[25]對云南干腌火腿的研究結果一致。
利用現代分子生物學技術,對微生物種群進行結構分析與鑒定,能克服傳統方法的缺點與不足。傳統的微生物種群結構分析方法是先進行菌種分離培養再進行單一菌種的鑒定,耗時長,過程繁瑣,且自然界中有85%~99%的微生物至今還不可純培養,而利用DGGE技術可以直接對微生物進行多種種群的鑒定[26]。但由于DGGE只能對微生物群落中數量>1%的優勢種群進行分析[27],并且由于嵌合體、共遷移、異源雙鏈體等會導致結果產生偏差,所以該技術也有一定的局限性[28]。因此充分了解該技術的基礎背景和局限性,結合其他分子生物學技術如酶學技術、熒光原位雜交技術等,才能從不同方面客觀的反映宣威火腿中真菌多樣性信息,為宣威火腿品質的研究提供更好的技術支持。
4 結論
從不同加工年份宣威火腿中共檢測到了27種真菌,宣威火腿中主要的真菌為Aspergilluspseudoglaucus(假灰綠曲霉)、Phialosimplexcaninus、Aspergilluspenicillioides(帚狀曲霉)、Yamadazymatriangularis、Wallemiasebi、Candidaglucosophila等,其中Aspergilluspseudoglaucus(假灰綠曲霉)是宣威火腿的優勢菌種。多樣性分析表明,加工3年的宣威火腿表面真菌群落多樣性最高,加工2年火腿表面真菌群落多樣性最低。加工2年火腿內部真菌群落多樣性最高,加工3年火腿內部真菌群落多樣性最低。相較于傳統的分離培養法,采用PCR-DGGE技術分析宣威火腿的真菌群落結構,結果能夠更全面、更直觀,也為進一步研究宣威火腿獨特風味的形成提供了技術支持。

