基于Cre/loxP系統的無篩選標記轉耐低磷轉錄因子GmPTF1大豆種質創制與分析
張小芳 董秋平 喬 瀟 喬亞科 王冰冰 張 鍇 李桂蘭
?
基于Cre/系統的無篩選標記轉耐低磷轉錄因子GmPTF1大豆種質創制與分析
張小芳1,**董秋平1,**喬 瀟2喬亞科1,*王冰冰1張 鍇1李桂蘭1,*
1河北科技師范學院農學與生物科技學院, 河北昌黎 066600;2河北科技師范學院物理系, 河北秦皇島 066004
篩選標記基因在轉基因植物應用中存在一定潛在安全風險, 在轉基因植物改良中如何合理消除該基因非常必要。轉錄因子PTF1具有改善植物在低磷脅迫下吸收磷效率的作用。本研究用根癌農桿菌介導法將Cre/-導入大豆品種豫豆22, 利用β-雌二醇誘導Cre/系統刪除篩選標記基因, 獲得了無篩選標記的轉基因大豆。用PCR法擴增刪除標記基因后的重組序列并測序顯示, 篩選標記基因在大豆基因組中已經被完全刪除, 重組序列中目的基因序列正確并保持了正確開放讀碼框;位點重組出現一種新的拼接類型, 重組后2個序列全部缺失, 新重組拼接位點長38 bp, 與NCBI數據庫的其他序列均無同源性, 并且重組涉及2個位點的外側翼序列, 造成Cre/盒上游和下游外側翼序列部分缺失。經過RT-PCR和Western雜交驗證顯示無篩選標記轉基因大豆植株中能夠正常轉錄和翻譯, 在根系、葉片及莖中的GmPTF1蛋白表達量均高于野生型對照, 而在種子中與對照無顯著差異。沙培試驗表明, 在低磷條件下無篩選標記轉基因大豆苗期根系指標、生物干重、葉綠素含量和磷含量均顯著高于野生型對照, 而丙二醛含量低于對照。利用Cre/重組系統可以有效刪除轉基因大豆中的篩選標記基因。
Cre/; GmPTF1; 無篩選標記; 轉基因大豆
自Hinchee等[1]在1988年用農桿菌浸染法獲得了轉基因大豆植株以來, 轉基因技術已作為一種高科技成功應用于大豆新種質的培育[2]。抗草甘膦轉基因大豆于1994年首次被美國批準商業化種植, 目前轉基因大豆已成為種植面積較大的轉基因作物之一[3]。隨著轉基因植物商業化推廣, 轉基因植物的生物安全問題一直是人們爭議的焦點[4-7], 其中篩選標記基因是轉基因作物潛在安全風險主要來源之一。篩選標記基因主要有抗生素抗性基因和除草劑抗性基因。常用的抗生素抗性基因有(潮霉素磷酸轉移酶)和基因(新霉素磷酸轉移酶), 除草劑抗性基因有抗草丁膦和雙丙氨膦的和、抗草甘膦的基因等。轉基因植物中篩選標記基因轉移至環境中會造成環境安全問題, 例如帶有一些抗除草劑基因的植物中其抗性基因可能漂流到其野生近緣種上, 出現超級雜草; 轉基因植物中的抗生素抗性基因是否會通過食物在腸道中水平轉移至微生物來影響人的健康等[7-8]。這些不可預測的風險引起人們對轉基因植物安全問題的廣泛關注[8], 也影響了轉基因作物的應用及發展。因此作物轉基因后刪除選擇標記基因, 對推動轉基因作物的商業化具有重要的價值和意義。
目前位點特異性重組是刪除選擇標記基因的一種有效途徑, 已經廣泛應用于不同植物的轉基因體系。其中來自噬菌體P1的Cre/系統是在所有位點特異性重組系統中應用最廣、可控性較高的轉化系統[8]。Cre/位點特異性重組系統中的Cre重組酶專一性識別由34對堿基對組成的特異序列(位點), 使2個位點片段發生重組, 2個位點之間的基因或DNA序列從而被剔除[9]。目前在煙草[10]、番茄[11]等獲得篩選標記刪除植株。Zuo等[12]構建了一種化學誘導與特異性位點重組相結合的高效DNA切除載體PX6-GFP, 應用β-雌二醇誘導Cre酶表達, 刪除了擬南芥中的標記基因。
大豆是一種需要磷較多的作物, 利用現代生物技術與常規育種相結合的手段, 獲得磷高效利用品種, 是解決大豆對磷需求的一個有效途徑。目前已發現并克隆出多個與磷代謝相關的基因[13]。研究較多的是轉錄因子類的基因,基因是bHLH轉錄因子家族的成員之一[14], 在植物逆境脅迫應答中起重要的作用[15]。在水稻中過表達基因其植株磷含量和干物質重高出野生型30%, 大豆中基因轉入擬南芥中超表達, 增強了擬南芥的耐低磷能力[16]。本研究旨在通過Cre/系統獲得無篩選標記基因的轉耐低磷基因大豆, 探討Cre/系統刪除轉基因大豆中篩選標記基因的效果, 同時為大豆耐低磷育種創制新種質資源。
1 材料與方法
1.1 材料
植物表達載體為px-(圖1-A), 其為c DNA; 轉化受體材料為大豆栽培品種“豫豆22”。
1.2 方法
1.2.1 轉基因大豆的獲得 用農桿菌介導大豆子葉節遺傳轉化法, 按照李桂蘭等[17-18]方法進行。
1.2.2 篩選標記基因刪除 轉Cre/-的大豆種子, 消毒后接種在含4 μmol L–1β-雌二醇的MS培養基中。培養25~30 d。將大豆苗移栽至營養土中, 提取基因組DNA, 進行檢測。
1.2.3 轉基因植株的PCR檢測 轉基因植株篩選標記基因刪除前及刪除后插入序列示意圖見圖1, 設計PCR引物檢測轉化及刪除效果, 根據PX6載體序列設計載體骨架引物, P1: 5¢-CCATCTCCACTGA CGTAAGGGAT-3¢, P2: 5¢-CTCGTCAATTCCAAGG GCATCGGT-3¢, P1在啟動子內, P2在下游的內, 預計擴增產物長度為653 bp。根據基因序列設計擴增引物, P3: 5¢-AGACAATCGGCT GCTCTGA-3¢, P4: 5¢-TCATTTCGAACCCCAGAGT C-3¢, 預計擴增產物長度為740 bp。根據目的基因序列設計特異性引物對, P5: 5¢-ATGGACC AAGCTCCGGGAGG-3¢, P6: 5¢-CCGTCCCTGATAT CTTGTCG-3¢, 預計擴增產物長度為727 bp。篩選標記基因刪除后重組序列使用P1/P6引物對擴增(圖1-B), 預計產物長度為901 bp。PCR擴增體系為20 μL, 包含10×buffer (緩沖液) 2 μL、dNTP (2.5 mmol L–1) 1.6 μL、上下游引物(10 μmol L–1)各0.8 μL、酶(5 U μL–1) 0.2 μL、模板DNA 2 μL、滅菌雙蒸水12.6 μL。擴增程序為95℃預變性10 min; 95℃變性30 s, 55℃退火1 min, 72℃延伸1 min, 共31個循環; 最后72℃延伸10 min。PCR產物經0.8%瓊脂糖凝膠電泳檢測并拍照。分別對引物P5/P6、P1/P6所擴增的產物進行切膠回收, 送北京華大基因有限公司測序。
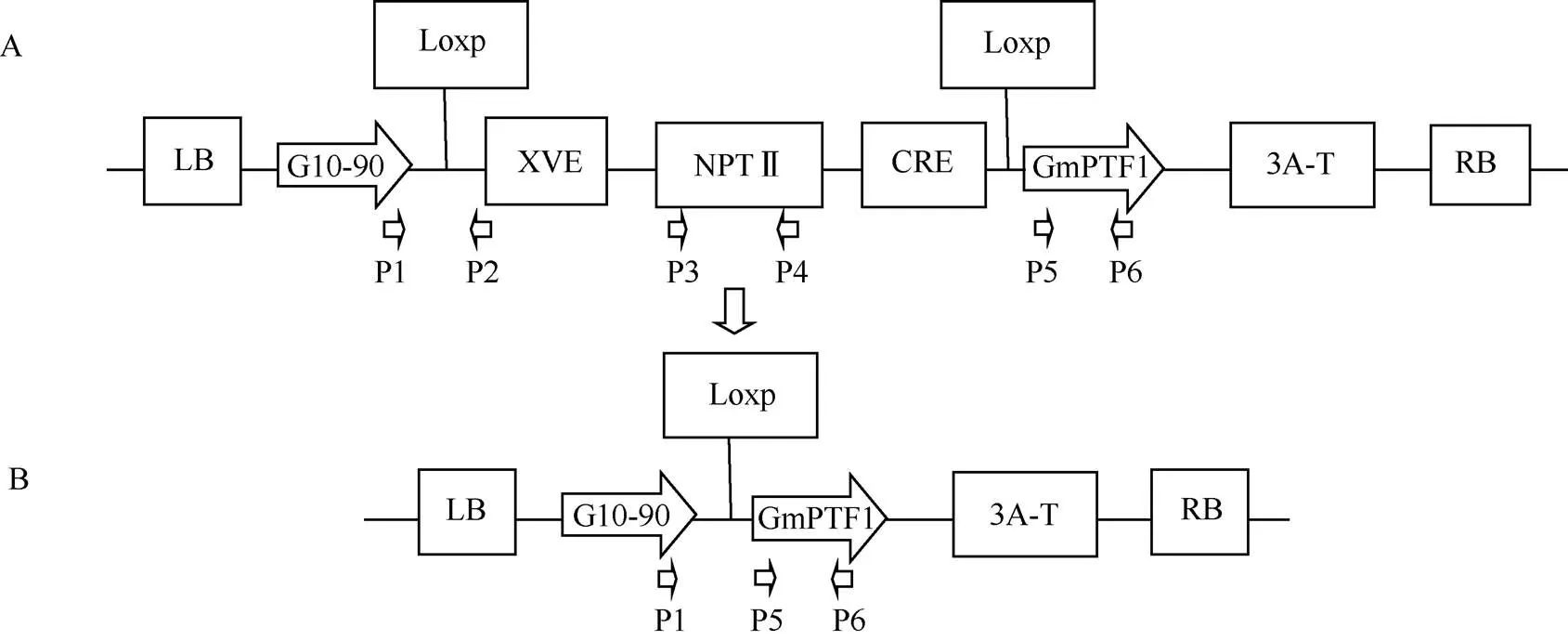
圖1 標記基因刪除前(A)和刪除后(B)轉基因植株插入序列及PCR鑒定示意圖
1.2.4 目的基因的Southern雜交檢測 用CTAB法提取大豆基因組DNA, 取100 μg左右DNA樣品, 分別選用I和I酶進行酶切, 野生型材料的總DNA作為陰性對照, 雜交程序按照羅氏Southern雜交試劑盒提供的說明書進行。以轉入的基因序列為模板, 應用特異引物P5/P6, PCR法合成Dig標記的探針。
1.2.5 熒光定量PCR 使用Bio-Rad iQ5熒光定量PCR儀, 內參基因為看家基因, 采用2–DDCt法對目的基因表達量進行相對定量分析。設計目的基因與內參基因的實時定量PCR引物(PTF-F: 5'-GTCCCAGGCTGCGACAAG-3', PTF-R: 5'-ACTGCTGCCAGTTTCATTGATAAG-3';n-F: 5'-GAGCTATGAATTGCCTGATGG-3',- R: 5'-CGTTTCATGAATTCCAGTAGC-3')。使用TaKaRa Mini BEST Plant RNA Extraction Kit提取刪除篩選標記基因PCR陽性植株葉片總RNA。取部分RNA根據Prime Script RT reagent Kit步驟進行反轉錄。將各樣品cDNA稀釋10倍, 取2 μL作模板, 分別用目的基因引物和內參基因引物進行擴增。擴增程序為95℃預變性10 min; 95℃變性15 s, 60℃延伸60 s, 循環40次。熒光定量PCR體系為20 μL, 包含2.5×Real master Mix 8 μL、20×SYBR Green solution 1 μL、上下游引物(10 μmol)各0.4 μL、cDNA模板2 μL、水8.2 μL。利用熒光定量PCR分析系統對樣品進行相對表達量分析, 計算出相對表達量, 同時在60~95℃進行溶解曲線分析。
1.2.6 轉基因植株的Western blot 提取無篩選標記轉基因大豆的根、莖、葉及成熟種子的蛋白, 測定蛋白質濃度, 上樣量為50 μg, 進行Western blot分析, 參考王關林等[19]的操作流程。以β-actin蛋白為內參, WB雜交圖像用Quantity one圖像分析系統進行各條帶平均灰度值的分析, 以目的條帶的灰度值與內參β-actin灰度值的比值表示GmPTF1蛋白的相對表達量。
轉基因材料中GmPTF1蛋白相對表達量= (轉基因材料目的條帶的灰度值/轉基因材料內參β-actin灰度值)/(對照目的條帶的灰度值/對照內參β-actin灰度值)
1.2.7 轉基因植株的測定項目與方法 對轉基因植株進行沙培, 定期澆灌霍格蘭氏營養液, 設營養液含低磷(0.05mmol L–1)和正常磷(1 mmol L–1) 2個處理, 3次重復, 每重復10株, 在處理30 d后, 測定各指標。用EPSON的PERFECTION V700型掃描儀測定根系各指標, 測量植株高度及上部第一完全展開三出復葉的中間小葉的長和寬。用丙酮法測定光合色素含量, 硫代巴比妥酸法測定丙二醛(MDA)含量。沖洗干凈植株根系上的沙子, 將莖、葉和根系分離, 分別裝入紙袋, 于105℃殺青20 min, 60℃烘干至恒重, 經硫酸-過氧化氫消煮, 釩鉬黃比色測定樣品含磷量。
1.3 數據分析
采用Microsoft Excel進行數據處理, DPSv7.05統計分析軟件進行分析。
2 結果與分析
2.1 無篩選標記轉GmPTF1基因大豆的獲得
應用農桿菌介導法轉化大豆品種“豫豆22”子葉節, 獲得目的基因PCR檢測為陽性的植株(圖2)。選擇純合株系后代進行雌二醇處理誘導篩選標記基因刪除, 篩選標記基因刪除后P1/P2和P3/P4則不能擴增出產物(圖1)。PCR擴增檢測刪除處理后的轉基因大豆植株顯示, P1/P2和P3/P4分別表現陰性, 而P5/P6和P1/P6表現陽性, 獲得符合預期大小的片段(圖3)。回收P1/P6擴增段, 以此為模板利用引物P5/P6進行PCR擴增并測序驗證, 測序結果顯示, 擴增長度為727 bp完全符合預期大小, 與目的基因序列比對完全相同, 進一步確定P1/P6擴增片段包含轉入的基因。說明標記基因已經被刪除并且保留了目的基因。
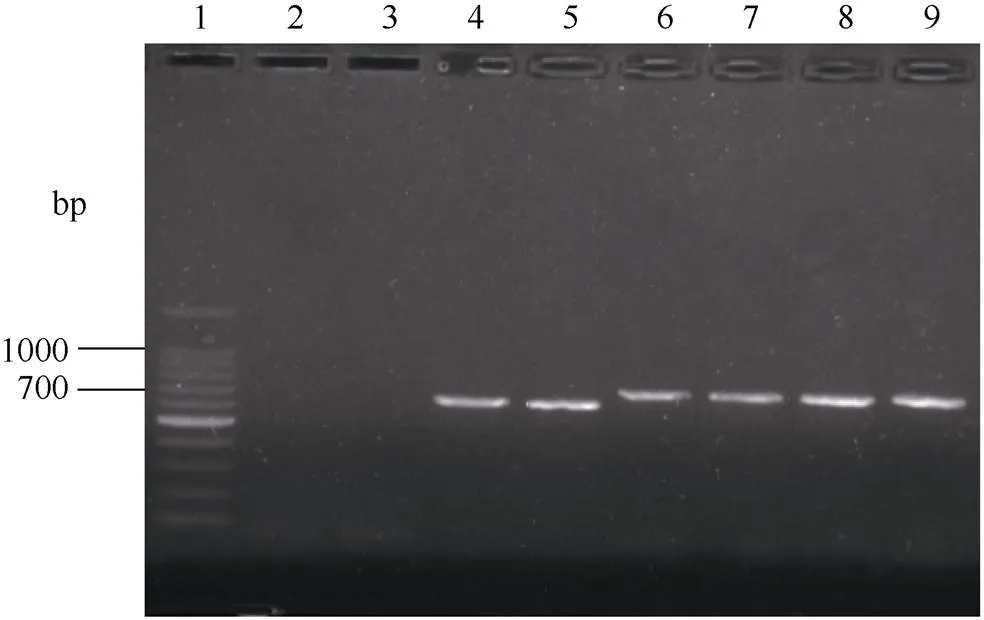
圖2 轉基因植株刪除標記之前的PCR檢測圖
1: 100 bp DNA ladder marker; 2: 水對照; 3: 野生型對照; 4, 5: P1/P2 PCR產物; 6, 7: P3/P4PCR產物; 8, 9: P5/P6基因PCR產物。
1: 100 bp DNA marker ladder; 2: blank control; 3: wild type control; 4, 5: PCR products of P1/P2; 6, 7:PCR products of P3/P4; 8, 9:gene PCR products of P5/P6.

圖3 轉基因植株刪除標記之后的PCR檢測圖
1, 12: 100 bp DNA ladder marker; 2: 水對照; 3: 野生型對照; 4, 5: P1/P2載體骨架片段PCR產物; 6, 7: P3/P4PCR產物; 8, 9: P5/P6基因PCR產物; 10, 11: P1/P6 PCR產物, 刪除標記后重組DNA片段。
1, 12: 100 bp DNA marker ladder; 2: blank control; 3: wild type control; 4, 5: PCR products of P1/P2; 6, 7:PCR products of P3/P4; 8, 9:gene PCR products of P5/P6; 10, 11: PCR product of P1/P6, recombinant DNA fragment after deletion of markers.
2.2 篩選標記基因刪除后的重組序列分析
對重組后的P1/P6擴增產物測序顯示(圖4), 擴增長度為859 bp, 陰影標出部分為P1和P6引物序列, 黑線框出的為篩選標記基因刪除后啟動子與基因之間的拼接位點。比對分析表明, P1/P6重組后的序列中包含目的基因序列(P5/P6之間序列)。重組序列中第1~94 bp與所用轉化載體中的G10-90啟動子序列一致, 而G10-90啟動子3¢末端的12 bp缺失, 其位于Cre/系統上游位點的外側; 第133~854 bp與轉入的基因的序列一致, 轉化載體下游位點與基因起始密碼ATG之間的30 bp的序列缺失。說明Cre/系統的重組可以涉及位點外的側翼序列。在第95~132 bp之間為拼接位點的重組序列, 長38 bp, 該序列經過比對發現與所用載體中的位點序列及NCBI數據庫的其他序列均無同源性, 并且拼接位點序列中出現了一個起始密碼子。分析開放閱讀框發現, 新出現的起始密碼子不改變原有基因的讀碼框, 從新出現的起始密碼子開始, 翻譯出的氨基酸序列比目的序列增加8個氨基酸(圖4)。
通過3個對比實驗,并綜合3個評價指標來看,IOCAP算法在整體上較K-Means算法、Spectral Clustering算法和AP算法有一定的優勢.該算法在一定程度保證聚類效率的同時,也較好地提高了準確度.因此,綜合考慮聚類的時間、精確度及通信開銷等因素,IOCAP算法的優勢較為明顯,也更適合大數據集的高效精準聚類.

圖4 標記基因刪除后重組序列P1/P6 PCR產物測序結果分析
陰影部分為P1和P6引物序列, 線框內為重組拼接位點。
The shaded parts were sequences of P1 and P6 primer, and therecombination splice sites were in the frame.
2.3 轉基因植株目標基因遺傳穩定性分析
對上述P1/P6 PCR陽性的植株按單株逐年種植, 對T5代純合株系進行Southern blot檢測, 提取全基因組DNA, 分別用限制性內切酶I和I進行酶切, 用地高辛標記的探針雜交。結果表明(圖5), 野生型對照材料沒有出現雜交信號, 兩種酶切的轉基因材料均出現單一的雜交信號條帶, 確認目的基因以單拷貝插入大豆基因組并且可以穩定遺傳。
2.4 無篩選標記轉基因大豆的GmPTF1表達分析
2.4.1基因的轉錄水平分析 對標記基因刪除后的轉基因大豆根據基因設計引物進行逆轉錄, 擴增出了符合預期大小的cDNA片段(圖6)。由于轉基因植株轉入的基因為大豆來源, 野生型對照內具有內源基因, 野生型出現一定的本底信號。
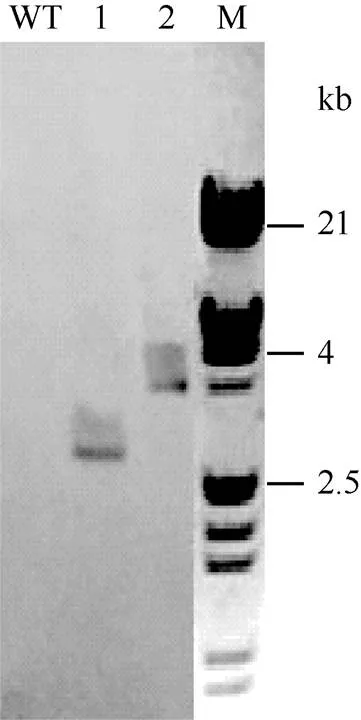
圖5 無篩選標記轉GmPTF1基因大豆T5代Southern blot檢測圖
1: 轉基因材料I酶切Southern blot; 2: 轉基因材料I酶切Southern blot; WT: 野生型對照I酶切Southern blot; M: λDNA/d III marker。
1: Southern blot of transgenic soybean digested byI; 2: Southern blot of transgenic soybean digested byI; WT: Southern blot of wild type controls digested byI; M: λDNA/d III marker.
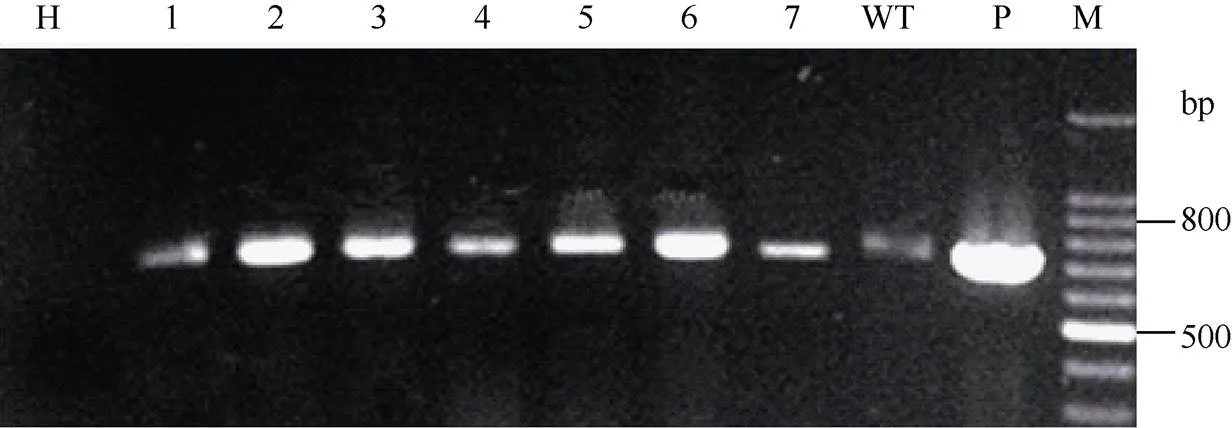
圖6 無篩選標記轉基因大豆GmPTF1基因RT-PCR
H: 空白對照; WT: 野生型對照; 1~7: 轉基因植株; P: 質粒陽性對照; M: 100 bp ladder DNA marker。
H: blank control; WT: wild type control; 1-7: transgenic soybean; P: plasmid positive control; M: 100 bp DNA marker ladder.
轉基因株系植株中的目的基因均能夠正常轉錄表達, 表達量顯著高于野生型, 是野生型對照的5.74倍(圖7)。
2.4.2 無篩選標記轉基因大豆不同器官中GmPTF1蛋白的Western blot分析 對轉基因大豆不同器官中目的基因表達GmPTF1蛋白進行Western blot分析(圖8), 在轉基因株系的根系、葉片和莖中GmPTF1蛋白水平的相對表達量均增加, 分別是野生型對照的3.17、2.16和1.19倍, 并且差異顯著; 而成熟種子中的GmPTF1蛋白相對表達量在轉基因株系和對照之間差異不顯著(圖9)。

圖7 無篩選標記轉基因大豆GmPTF1轉錄水平分析
1: 野生型; 2: 轉基因大豆。
1: wild type controls; 2: transgenic soybean plants.

圖8 無篩選標記轉基因大豆不同器官中GmPTF1蛋白的Western雜交結果
1: 野生型對照; 2: 轉基因材料; A: GmPTF基因表達蛋白; B: Actin基因表達蛋白。
1: wild type controls; 2: transgenic plants; A: GmPTF1 protein; B: Actin protein.
2.5 低磷脅迫下無篩選標記轉GmPTF1基因大豆苗期形態表現
結果表明, 正常磷條件下, 轉基因株系的根系指標與野生型對照的指標差異不顯著; 低磷條件下, 轉基因株系的根總長、根總表面積、根平均直徑和根體積均極顯著高于對照(表1)。說明轉基因植株在低磷條件下根系受低磷影響較小。
正常磷條件下, 轉基因大豆與野生型對照的單株干物質重差異不顯著(表3)。低磷脅迫下, 大豆幼苗干物質重均下降, 轉基因株系下降幅度顯著低于對照; 并且轉基因株的干物重顯著高于對照, 根和莖葉干物重分別比對照高60.5%和85.9%。表明基因過表達提高了低磷脅迫下大豆干物質合成與積累。
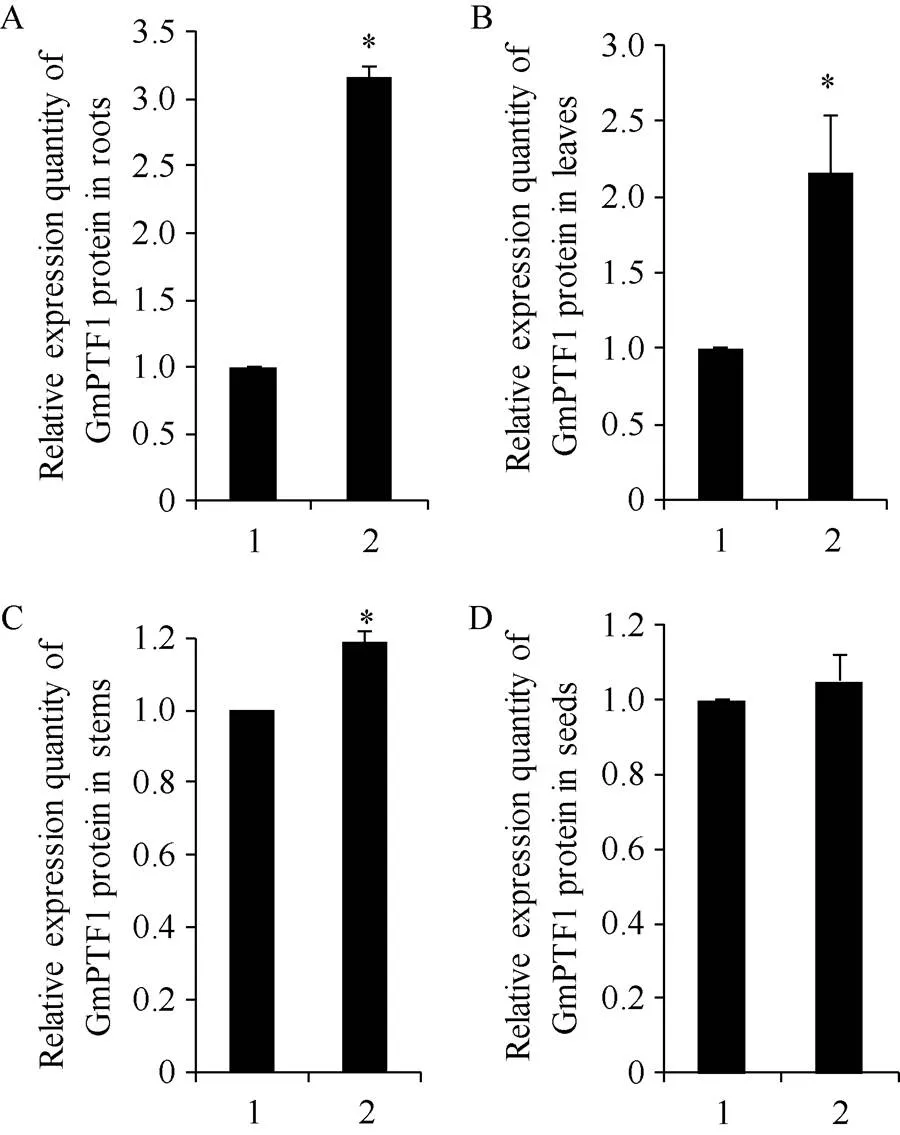
圖9 無篩選標記轉基因大豆不同器官中GmPTF1蛋白相對表達量
1: 野生型對照; 2: 轉基因大豆; A: 根; B: 葉; C: 莖; D: 種子。
1: wild type controls; 2: transgenic soybean plants; A: root; B: leaf, C: stem; D: seed.
2.6 無篩選標記轉GmPTF1大豆苗期對低磷脅迫的生理響應
丙二醛含量上升和光合色素含量下降是植物在低磷條件下的生理生化響應。正常磷處理條件下, 轉基因株系與野生型對照的丙二醛和光合色素無顯著差異。在低磷條件下, 轉基因株系的丙二醛含量及增幅顯著低于野生型對照, 而光合色素含量顯著高于對照, 降幅低于野生型對照(圖10和表4)。表明轉基因株系增加了對磷的利用能力, 進而減輕了低磷脅迫的不利影響。
2.7 無篩選標記轉GmPTF1大豆磷含量分析
正常磷處理下, 野生型與轉基因株系葉片磷含量無顯著差異。低磷處理下, 野生型磷含量顯著降低, 比正常磷處理下降了20.3%; 而轉基因株系葉片磷含量與正常磷處理無顯著差異, 與野生型對照相比提高了46.17% (圖11)。表明轉大豆增強了對磷的利用能力。
3 討論
Cre/重組系統可以定位定向地刪除轉基因作物中位于2個位點中的選擇標記基因, 在基因工程領域中有巨大的應用前景[20]。Cre重組系統是通過誘導重組酶啟動, 使兩側的位點重組, 刪除中間的標記基因, 重組后保留1個位點。在重組序列的研究中, Cheng等[21]構建利用Cre/重組系統在伴放線桿菌()中實現了標記基因的刪除,重組序列拼接位點只剩下1個loxp位點序列, 沒有出現其他序列變化[21]; Sreekala等[22]利用該系統成功獲得了無選擇標記基因的水稻, 重組序列拼接位點只剩下1個位點序列; Togawa等[23]利用Cre/系統對嗜熱菌()進行篩選標記基因敲出, 雖然篩選標記基因完全敲除, 但同時觀察到多個位點之間的染色體區域的不理想的缺失和反轉[23]。Zhang等[24]利用該系統刪除了轉基因番茄的標記基因, 重組序列拼接位點同樣只剩下1個位點序列; Li等[25]利用胚胎特異啟動子構建Cre/自刪除系統, 轉化后發育到胚狀體形成階段啟動Cre刪除轉基因大豆標記基因后, 重組序列中拼接位點長83 bp, 包括1個34 bp的位點以及其上游的22 bp下游27 bp的載體序列。在本研究中, 刪除標記基因后重組序列在啟動子與目的基因之間出現38 bp拼接位點序列, 該拼接位點序列與載體、和大豆基因組均無同源性, 為1個新位點重組的拼接類型; 并且重組涉及位點外的側翼序列, 位于Cre/系統中2個位點外側的部分載體序列均缺失, 這與上述報道不同, 說明Cre/系統重組后拼接位點序列的多樣性; 新的拼接序列中出現了一個起始密碼子, 但未改變原有目的基因的讀碼框; Real-time PCR和Western Blot結果顯示, 轉基因植株由于的拷貝數增加致使相應的表達量提高, 說明重組后G10-90啟動子3¢末端的12 bp缺失沒有影響啟動子的功能, 同時驗證了Cre/重組系統定位定向刪除的可靠性。鑒于重組可能涉及位點外的側翼序列, 構建載體時在目的基因及調控元件與連接處應增加相應保護序列。
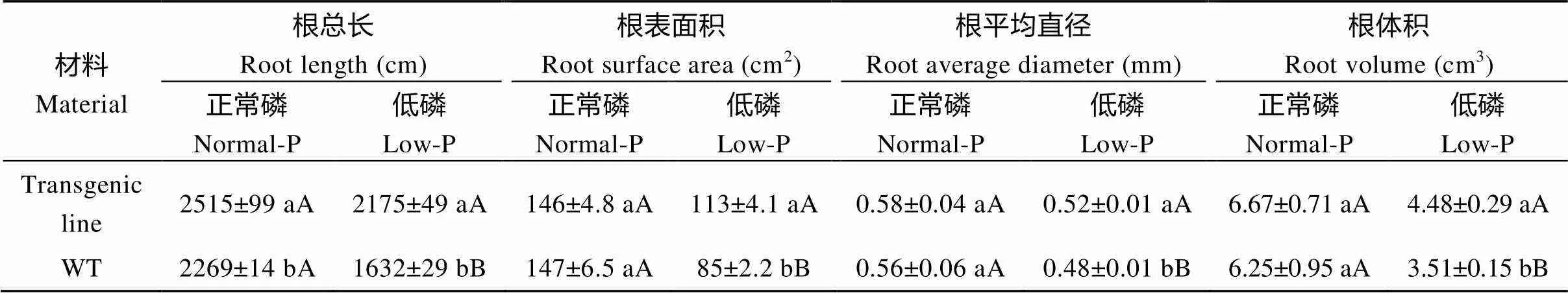
表1 不同磷濃度下不同大豆植株的根系性狀
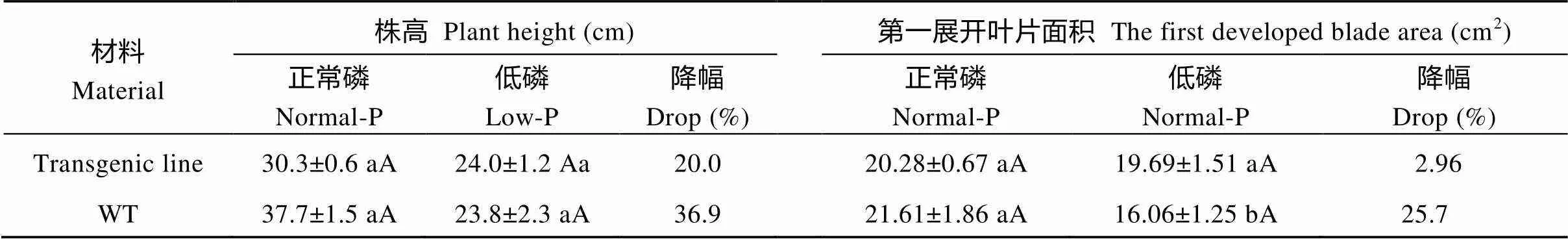
表2 低磷脅迫對不同大豆幼苗地上部的影響
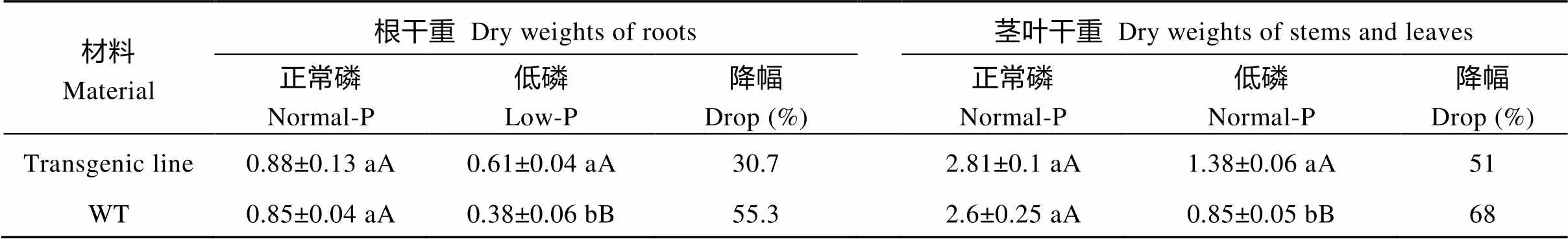
表3 低磷對不同轉基因大豆干物質積累的影響
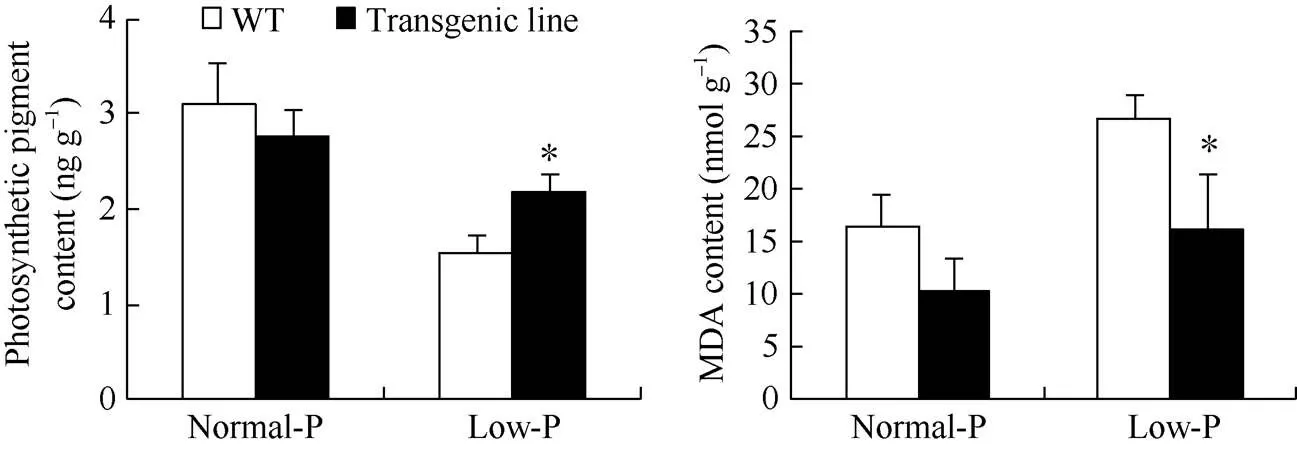
圖10 大豆葉片中光合色素與丙二醛含量

表4 大豆葉片丙二醛與光合色素變幅
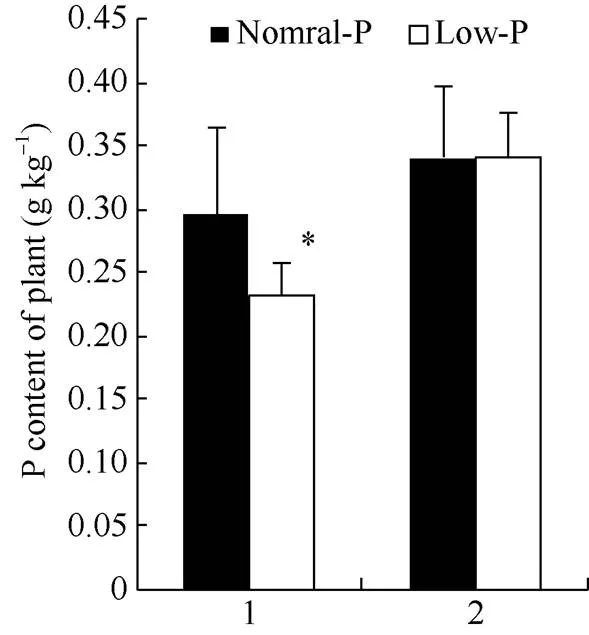
圖11 不同磷濃度處理下大豆葉片磷含量
1: 野生型對照; 2: 轉基因大豆。
1: wild-type control; 2: transgenic line.
有研究表明低磷脅迫條件下, 根總長、根總表面積、根寬、根干重、地上干重、地上磷含量可以作為綜合評價大豆苗期磷效率篩選的重要指標[26-27]。本研究低磷脅迫下無篩選標記轉基因大豆苗期的根總長、根總表面積、根寬、根總體積、根干重及地上干重均極顯著高于野生型對照豫豆22植株, 證明轉入的基因在植株體內成功表達, 提高了磷的利用效率, 從而促進了轉基因大豆植株的根系的生長發育, 使植株生長更加健壯。張彥麗等[26]研究認為大豆苗期相對株高受低磷脅迫的影響較小, 變異系數較小, 不適合作為大豆苗期磷效率的篩選指標。本研究結果亦得到相似結果, 表現為低磷脅迫下苗期不同材料株高無差異, 苗期株高對低磷不敏感。植物在低磷脅迫后發生一系列的生理生化反應, 從而調節植物的生長發育[28]。光合色素含量的下降是植物受到脅迫所表現的關鍵生理特征, 在逆境條件下, 植物體內葉綠素受到破壞, 光合作用受到抑制, 最終導致植物不能正常生長。丙二醛是膜脂過氧化分解的終產物, 在逆境脅迫下, 超氧自由基不斷累積并提高了植物組織脂膜氧化的程度, 使其最終產物丙二醛含量增加[29-30]。本研究無篩選標記轉基因大豆材料在低磷條件下葉片光合色素含量顯著高于野生型對照, 而MDA含量低于野生型對照。說明在低磷條件下基因的過量表達調控相關磷代謝基因表達, 減少了低磷環境給大豆植株帶來的傷害。本研究中基于Cre/系統刪除選擇標記的轉大豆在苗期低磷條件下的磷利用率提高, 根系、生物干重、植株含磷量顯著高于野生型對照, 與鐘磊等[31]獲得的帶篩選標記轉大豆表現一致, 證明Cre/系統刪除選擇標記的可靠性及有效性, 并再次證明轉錄因子在大豆磷代謝中的作用。
4 結論
利用Cre/系統有效刪除篩選標記基因, 獲得無篩選標記的轉大豆。位點重組出現一種新的拼接類型, 38 bp的拼接位點重組序列與其他序列均無同源性, 重組后2個序列全部缺失;位點重組涉及位點外的側翼序列, Cre/重組系統之外的側翼序列部分缺失, 構建載體時在目的基因及啟動子調控元件與之間應增加保護序列。無篩選標記的轉大豆植株提高了基因在根系及葉片中的表達, 提高大豆苗期低磷條件下的磷利用率, 根系、生物干重、植株含磷量顯著高于野生型對照。
[1] Hinchee M A W, Connorward D V, Newell C A, McDonnell R E, Sato S J, Gasser C S, Fischhoff D A, Re D B, Fraley R T, Horsch R B. Production of transgenic soybean plants usingmediated DNA transfer., 1988, 6: 915–922.
[2] 楊樂坤. 中國轉基因農產品的標識規制策略. 南京大學碩士學位論文, 江蘇南京, 2016. Yang L K. China’s Strategic Labeling of GM Food Based on Two-stage Dynamic Game Model. MS Thesis of Nanjing University, Nanjing, Jiangsu, China, 2016 (in Chinese with English abstract).
[3] 崔寧波, 宋秀娟. 國外轉基因大豆種植與育種研究進展國外轉基因大豆種植與育種研究進展. 東北農業大學學報, 2015, 46(8): 103–108. Cui N B, Song X J. Research progress on development and planting of foreign genetically., 2015, 46(8): 103–108 (in Chinese with English abstract).
[4] 陳曉亞, 楊長青, 賈鶴鵬, 凌飛. 中國轉基因作物面臨的問題. 華中農業大學學報, 2014, 33(6): 115–117. Chen X Y, Yang C Q, Jia H P, Ling F. Issues confronting GMO crops in China., 2014, 33(6): 115–117 (in Chinese with English abstract).
[5] 石紅璆, 查代明. 轉基因食品的道德風險及其解決對策. 安徽農業科學, 2018, 46(8): 7–9.Shi H Q, Zha D M. Moral risks of genetically modified foods and their countermeasures., 2018, 46(8): 7–9 (in Chinese with English abstract).
[6] 黃大昉. 我國轉基因作物育種發展回顧與思考. 生物工程學報, 2015, 31: 892–900. Huang D F. Review of transgenic crop breeding in China., 2015, 31: 892–900 (in Chinese with English abstract).
[7] 逄金輝, 馬彩云, 封勇麗, 胡瑞法. 轉基因作物生物安全:科學證據. 中國生物工程雜志, 2016, 36(1): 122–138. Peng J H, Ma C Y, Feng Y L, Hu R F. Biosafety of genetically modified crops: scientific evidence., 2016, 36(1): 122–138 (in Chinese with English abstract).
[8] 祁永斌, 劉慶龍, 陸艷婷, 金慶生. 轉基因植物中刪除選擇標記基因的研究進展. 浙江農業學報, 2014, 26: 1387–1393.Qi Y B, Liu Q L, Lu Y T, Jin Q S. Research progress of the selectable marker genes eliminated in the transgenic plants., 2014, 26: 1387–1393 (in Chinese with English abstract).
[9] Guo F, Gopaul D N, Van Duyne G D. Structure of Cer recombinase complexes with DNA in a site-specific recombination synapse., 1997, 389: 40–46.
[10] Dale E C, Ow D W. Intra- and intermolecular site-specific recombination in plant cells mediated by bacteriophage P1 recombinase., 1990, 91: 79–85.
[11] Ma B G, Duan X Y, Niu J X, Ma C, Hao Q N, Zhang L X, Zhang H P. Expression of stilbene synthase gene in transgenic tomato using salicylic acid-inducible Cre/recombination system with self-excision of selectable marker., 2009, 31: 163–169.
[12] Zuo J, Niu Q, Moller S, Chua N H. Chemical-regulated, site-specific DNA excision in transgenic plants., 2001, 19: 157–161
[13] 李喜煥, 常文鎖, 張彩英. 提高植物磷營養效率(候選)基因研究進展. 植物遺傳資源學報, 2012, 13: 83–97. Li X H, Chang W S, Zhang C Y. Research progress of candidate genes for improving plant phosphorus-effiency., 2012, 13: 83–97 (in Chinese with English abstract).
[14] 張全琪, 朱家紅, 倪燕妹, 張治禮. 植物bHLH轉錄因子的結構特點及其生物學功能. 熱帶亞熱帶植物學報, 2011, 19: 84–90. Zhang Q Q, Zhu J H, Ni Y M, Zhang Z L. The structure and function of plant bHLH transcription factors., 2011, 19: 84–90 (in Chinese with English abstract).
[15] 楊致榮, 毛雪, 李潤植. 植物次生代謝基因工程研究進展. 植物生理與分子生物學學報, 2005, 31: 11–18. Yang Z R, Mao X, Li R Z. Progress in studies on genetic engineering of secondary metabolites in plant., 2005, 31: 11–18 (in Chinese with English abstract).
[16] 吳冰. 大豆耐低磷轉錄因子與功能分析. 河北農業大學碩士學位論文, 河北保定, 2013. Wu B. Functional Analysis of Transcription FactorandResponsed to Low Phosphorus in Soybean. MS Thesis of Agricultural University of Hebei, Baoding, Hebei, China, 2013 (in Chinese with English abstract).
[17] 李桂蘭, 劉晨光, 喬瀟, 楊曉倩, 王迪, 喬亞科.共培養條件對農桿菌轉化大豆子葉節的影響.核農學報, 2014, 28: 1567–1575. Li G L, Liu C G, Qiao X, Yang X Q, Wang D, Qiao Y K. Conditions of co-culture affecting on the efficiency ofmediated transformation of cotyledonary node of soybean., 2014, 28: 1567–1575 (in Chinese with English abstract).
[18] 李桂蘭, 喬亞科, 楊少輝, 靳朝霞, 李明剛.農桿菌介導大豆子葉節遺傳轉化的研究. 作物學報, 2005, 31: 170–176. Li G L, Qiao Y K, Yang S H, Jin Z X, Li M G. Study of the-mediated transformation systems of soybean cotyledonary node., 2005, 31: 170–176 (in Chinese with English abstract).
[19] 王關林, 方宏筠. 植物基因工程原理與技術. 北京: 科學出版社, 1998. pp 634–636.Wang G L, Fang H J. Plant Gene Engineering Principle and Technique. Beijing: Science Press, 1998. pp 634–636 (in Chinese).
[20] 龍定沛, 譚兵, 趙愛春, 許龍霞, 向仲懷. Cre位點特異性重組系統在高等真核生物中的研究進展. 遺傳, 2012, 34: 177–189. Long D P, Tan B, Zhao A C, Xu L X, Xiang Z H. Progress in Cre/site-specific recombination system in higher eu-karyotes., 2012, 34: 177–189 (in Chinese with English abstract).
[21] Cheng Y A, Jee J, Hsu G, Huang Y Y, Chen C, Lin C P. A markerless protocol for genetic analysis of., 2014, 113: 114–123.
[22] Sreekala C, Wu L, Gu K, Wang D, Tian D, Yin Z. Excision of a selectable marker in transgenic rice (L.) using a chemically regulated Cre/system., 2005, 24: 86–94.
[23] Togawa Y, Nunoshiba T, Hiratsu K. Cre/-based multiple markerless gene disruption in the genome of the extreme thermophile., 2018, 293: 277–291.
[24] Zhang Y, Li H, Ouyang B, Ye Z. Chemical-induced autoexcision of selectable markers in elite tomato plants transformed with a gene conferring resistance to lepidopteran insects., 2006, 28: 1247–1253.
[25] Li Z, Xing A, Moon B P, Burgoyne S A, Guida A D, Liang H, Lee C, Caster C S, Barton J E, Klein T M, Falco S C. A Cre/-mediated self-activating gene excision system to produce marker gene free transgenic soybean plants., 2007, 65: 329–341.
[26] 張彥麗, 賈健輝, 趙月琪, 谷思玉, 許景鋼. 大豆苗期耐低磷篩選指標的研究. 安徽農業科學, 2010, 38: 5506–5507. Zhang Y L, Jia J H, Zhao Y Q, Gu S Y, Xu J G. Screening index of soybean for low phosphorus tolerance at seedling stage., 2010, 38: 5506–5507 (in Chinese with English abstract).
[27] 梁泉, 尹元萍, 嚴小龍, 廖紅. 不同磷水平下大豆根系性狀的遺傳特性分析. 分子植物育種, 2009, 7: 321–329. Liang Q, Yin Y P, Yan X L, Liao H. Genetic analysis of root characters in soybean using a recombinant inbred line population at two phosphorus levels., 2009, 7: 321–329 (in Chinese with English abstract).
[28] 敖雪, 謝甫綈, 劉婧琦, 張惠君. 不同磷效率大豆品種光合特性的比較. 作物學報, 2009, 35: 522–529. Ao X, Xie F T, Liu J Q, Zhang H J. Comparison of photosynthetic characteristics in soybean cultivars with different phosphorus efficiencies., 2009, 35: 522–529 (in Chinese with English abstract).
[29] 李俊, 張春雷, 秦嶺, 馬霓, 李峰. 不同磷效率基因型油菜對低磷脅迫的生理響應. 中國油料作物學報, 2010, 32: 222–228. Li J, Zhang C L, Qin L, Ma N, Li F. Physiological response to low phosphorus stress for different P-efficiency genotype of rapeseed., 2010, 32: 222–228 (in Chinese with English abstract).
[30] 王晶, 韓曉日, 站秀梅, 侯玉慧, 趙偉力. 低磷脅迫對番茄葉片脂膜過氧化及保護酶活性的影響. 植物營養與肥料學報, 2005, 11: 851–854. Wang J, Han X R, Zhan X M, Hou Y H, Zhao W L. Influence of low-phosphorus stress on membrane lipid peroxidation and protective enzyme activities in tomato leaves., 2005, 11: 851–854 (in Chinese with English abstract).
[31] 鐘磊, 喬亞科, 喬瀟, 李桂蘭, 王林紅, 劉晨光. 轉基因大豆在低磷脅迫下的表現. 核農學報, 2013, 27: 1041–1047. Zhong L, Qiao Y K, Qiao X, Li G L, Wang L H, Liu C G. Performance of transgenic soybean withgene under low phosphorus stress., 2013, 27: 1041–1047 (in Chinese with English abstract).
Creation and analysis of marker free transgenic soybean germplasm with low phosphate tolerance transcription factor GmPTF1based on Cre/system
ZHANG Xiao-Fang1,**, DONG Qiu-Ping1,**, QIAO Xiao2, QIAO Ya-Ke1,*,WANG Bing-Bing1, ZHANG Kai1, and LI Gui-Lan1,*
1College of Agronomy and Biotechnology, Hebei Normal University of Science and Technology, Changli 066600, Hebei, China;2Department of Physics, Hebei Normal University of Science and Technology, Qinhuangdao 066004, Hebei, China
The screening marker genes in transgenic crops have potential safety risks, which are necessarily eliminated in the improvement of transgenic plants. The transcription factorPTF1 has the effect improving phosphorus uptake by plants under low phosphorus stress. So Cre/-GmPTF1 was transferred into soybean cultivar Yudou 22 [(L.) Merr] via-mediated cotyledonary node transformation method. And then, the marker-free transgenic soybean withwas obtained by means of induced Cre/site specific recombination with β-estradiol. After the marker gene was deleted, the recombinant sequences were amplified by PCR and then sequenced. The sequencing results indicate that the marker genes were completely deleted out of the transgenic soybean genome. The recombinant maintained with correct ORF correct target gene sequence, and a new splicing type appeared inrecombination. Twosequences were missing in the recombination. The length of the new recombinant in splicing site was 38 bp which was no homology with other sequences in NCBI database. And the recombination was involved in the flank sequence outside the twosites, which lead partially deleted in the outer flank sequences of Cre/cassette. The results of RT-PCR and Western blot showed thatcould be transcribed and translated normally in marker-free transgenic soybean plants.The expression level ofGmPTF1 in roots, stem and leaves was higher than that in wild type, which was not significantly different in seeds. in the sand culture experiment under low phosphorus condition, the root indexes, dry biomass, chlorophyll content and phosphorus content in marker-free transgenic soybean were significantly higher than there in the wild type, while the MDA content was lower than that in the control. We conclude that screening marker genes in transgenic soybean could be effectively deleted by Cre/loxP recombinant system.
Cre/; GmPTF1; marker-free; transgenic soybean
2018-08-27;
2018-12-24;
2019-01-16.
10.3724/SP.J.1006.2019.84118
李桂蘭, E-mail: lgl63@126.com; 喬亞科, E-mail: qiaoyake@126.com
**同等貢獻(Contributed to equally this work)
張小芳, E-mail: zxf13214@163.com; 董秋平, E-mail: qiuping.dong@kejidalong.com
本研究由國家轉基因生物新品種培育科技重大專項(2014ZX0800404B)和現代農業產業技術體系河北省創新團隊建設項目(HBCT2018090203)資助。
This study was supported by the National Transgenic Major Project of China (2014ZX0800404B) and Modern Agricultural Industrial Technology System Hebei Province Innovation Team Construction Project (HBCT2018090203).
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20190115.1603.004.html

