利用CRISPR-Cas9系統構建新型異戊酰螺旋霉素Ⅰ產生菌
張曉婷,張妍,戴劍漉,王以光,赫衛清
1 中國醫學科學院 醫藥生物技術研究所 衛健委抗生素生物工程重點實驗室,北京 100050
2 沈陽同聯集團有限公司,遼寧 沈陽 110042
必特螺旋霉素 (Bitespiramycin,BT) 是將耐熱鏈霉菌Streptomyces thermotolerans的4″-異戊酰基轉移酶基因 (4″-Isovaleryltransferase gene,ist) 導入到螺旋霉素 (Spiramycin,SP) 產生菌螺旋鏈霉菌Streptomyces spiramyceticus中進行表達,所產生的發酵產物[1]。BT的主組分是異戊酰螺旋霉素(Isovalerylspiramycin,ISP) Ⅰ、Ⅱ和Ⅲ,其結構式如圖1所示。因為SP的3位酰基化酶的底物特異性不強,可以同時識別乙酰基和丙酰基底物,導致出現3種主組分。3位只是羥基的為Ⅰ組分,Ⅱ組分是3位羥基乙酰化產物,Ⅲ組分是丙酰化產物。BT是多組分藥物,在發酵產物提純和質控方面難度較大。經研究初步證實ISP Ⅰ的藥物活性與BT相當,因此可以作為一種單組分藥物進行后續開發。前期研究中通過阻斷SP的3位酰基化酶基因bsm4,獲得只產SPⅠ的基因工程菌株[2],再導入2個拷貝的ist基因[3],得到只產ISP Ⅰ的工程菌株[4]。但此菌株ISP Ⅰ的產量很低,發酵液的效價只有200 μg/mL。為了提高ISP Ⅰ的發酵產量,又將ist基因和其正調控基因acyB2整合到ISP Ⅰ菌株的染色體上,通過育種篩選,獲得效價在800 μg/mL左右的高產菌株S.spiramyceticusWSJ-IA[5–6]。此高產菌株中已經包含硫鏈絲菌素和阿普拉霉素兩種抗性基因,利用現有的鏈霉菌質粒很難再對其進行定向遺傳操作。因此采用等離子誘變和傳統誘變相結合的方法對WSJ-IA菌株進行誘變育種,獲得效價在2 000 μg/mL左右的高產菌株[7],基本滿足大型發酵的要求。此菌株共進行3次遺傳操作,特別是將bsm4基因進行部分刪除后,菌株的產量下降為原株的50%左右。在菌株染色體的2個不同位置中還包含3個ist基因,在傳代過程中有發生同源重組的可能性,不利于菌株的遺傳穩定。
為了獲得遺傳更加穩定的ISP Ⅰ菌株,利用近幾年新出現的CRISPR-Cas9基因編輯系統[8-9],可以方便地對靶基因進行阻斷或替換。本研究擬將組成型強啟動子ermEp*控制下的ist基因替換bsm4基因,從而獲得新的ISP Ⅰ菌株。
1 材料與方法
1.1 材料
1.1.1 菌種與質粒
螺旋霉素產生菌S.spiramyceticus1941由本實驗室保存。大腸桿菌Escherichia coliDH5α感受態細胞、基因克隆和重組質粒構建的受體菌,購自北京全式金生物技術有限公司。pKC1139[10]鏈霉菌和大腸桿菌穿梭質粒,帶有Apramycin抗性基因,本實驗室保存。CRISPR-Cas9基因組編輯質粒pKCcas9do[11],由中國科學院上海生命科學研究院姜衛紅研究員惠贈。pKCcas9do是以鏈霉菌溫敏質粒pKC1139為出發質粒,在培養溫度為37 ℃時停止復制。pKCcas9do質粒含有優化的cas9基因,由硫鏈絲菌素誘導的啟動子tipA進行調控;人工合成的j23119啟動子控制引導RNA(Single-guide RNA,sgRNA) 基因的轉錄;sgRNA由crRNA和tracrRNA組成,在crRNA中人工設計20 bp的靶結合序列,sgRNA 引導Cas9蛋白對靶位點進行切割。之后可以通過在37 ℃培養和傳代,從宿主菌中去除此質粒,獲得沒有抗性而靶基因被改造的目的菌株。實驗中用于構建質粒的引物序列見表1。
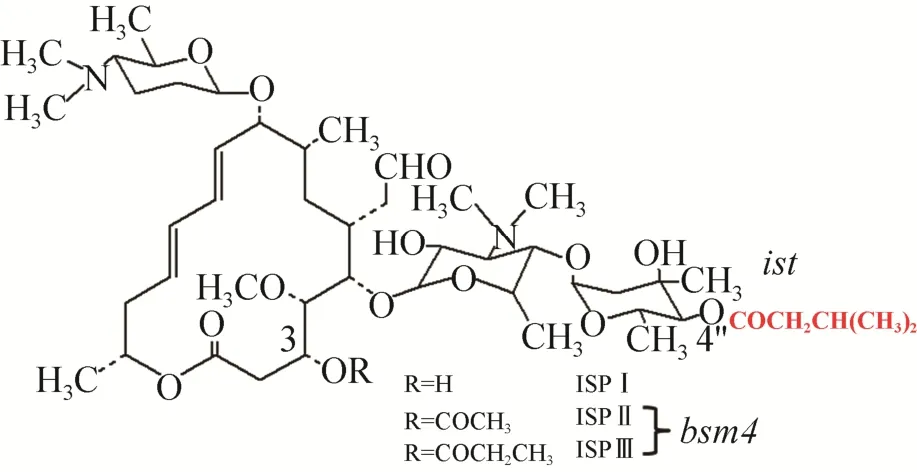
圖1 異戊酰螺旋霉素的結構Fig.1 Structure of isovalerylspiramycin.
1.1.2 試劑
BT和ISP Ⅰ的對照品,本實驗室自制。阿普拉霉素 (Apramycin,Am) 購自武漢遠城科技發展有限公司。硫鏈絲菌素 (Thiostrepton,Tsr),購自Sigma公司。限制性內切酶、T4 DNA連接酶購自TaKaRa公司。KOD FX Neo DNA聚合酶 (TOYOBO) 購自北京天佑恒遠生物科技有限公司。
1.1.3 培養基
S.spiramyceticus1941及其突變株的固體培養基 (g/100 mL):黃豆餅粉2,葡萄糖1,淀粉3,CaCO30.5,NaCl 0.4,瓊脂粉1.5,自然pH值。1 μg/mL的Tsr誘導Cas9基因表達進行定點切割。R2YE培養基用于S.spiramyceticus1941的原生質體的制備和轉化[12]。
S.spiramyceticus1941及其突變株的種子和發酵培養基 (g/100 mL) 詳見文獻[13]。
S.spiramyceticus1941及其突變株的生物檢定培養基 (g/L):牛肉膏3.0,酵母膏3.0,蛋白胨(F 403)10.0,葡萄糖1.0,氯化鈉5.0,瓊脂12.0,pH 8.0。
1.2 方法
1.2.1 分子克隆
鏈霉菌總DNA的提取按文獻[14]進行。E.coliDH5α與E.coliET12567感受態細胞的制備、分子克隆與鑒定按文獻[15]進行。PCR反應采用KOD FX Neo DNA聚合酶反應體系。DNA測序由中美泰和生物技術 (北京) 有限公司完成。

表1 本文中所用引物Table 1 Primers used in this study
1.2.2 構建bsm4基因替換質粒
利用3對引物bsm4-LF/LR、ei-F/R和bsm4-RF/RR分別以S.spiramyceticus1941基因組DNA為模板進行擴增,獲得左同源臂 (Left arm),ermEp*-ist和右同源臂 (Right arm) 3種PCR產物。將這些產物純化后,進行重疊延伸PCR,獲得 6.6 kb的目的片段,再進行XbaⅠ和HindⅢ酶切消化,與用同樣酶切的pKC1139載體進行連接,得到pKC-ei重組質粒,如圖2所示。
利用CRIPR-Cas9系統進行基因替換,先通過sgRNA-1和sgRNA-R引物擴增出引導Cas9進行特異性切割的引導序列sgRNA-1,用SpeⅠ和XbaⅠ進行雙酶切后回收;利用sgRNA-2和sgRNA-R引物擴增出另一個引導序列sgRNA-2,也用SpeⅠ和XbaⅠ進行雙酶切后回收;以pKC-ei為模板,利用Hist-F/R引物擴展出4.0 kb片段,用XbaⅠ和HindⅢ進行雙酶切后回收,將sgRNA-1和sgRNA-2分別與4.0 kb片段克隆到pKCcasdo載體 (SpeⅠ/HindⅢ) 上,獲得替換質粒pKcas-ei1和pKcas-ei2。
1.2.3 篩選目的菌株
將構建好的重組質粒通過原生質體轉化法導入到S.spiramyceticus1941中,利用1 μg/mL Tsr誘導Cas9 表達,由sgRNA引導Cas9對靶位點進行切割,在質粒上有設計好的ermEp*-ist替代bsm4基因的同源片段。將發生同源雙交換的目的菌株置于37 ℃培養,使質粒停止復制,之后通過Am和無抗性平板進行篩選,獲得ermEp*-ist替代bsm4基因的目的菌株,進行PCR鑒定和測序驗證。
1.2.4 發酵產物檢測
含S.spiramyceticus1941轉化子的斜面置于28 ℃培養 5–7 d,挖塊至種子培養基 (50 mL培養基/250 mL三角瓶),28 ℃振蕩 (200 r/min) 培養48 h,按1∶50的比例轉接至發酵培養基中 (50 mL培養基/500 mL三角瓶),28 ℃振蕩 (200 r/min)培養4 d,發酵液離心取上清液,用5.0 mol/L NaOH調至pH 8.5–9.0,加等體積乙酸乙酯萃取,8 mL萃取液離心濃縮至干燥,溶于200 μL乙腈,0.22 μm的濾膜過濾后,取3 μL進行HPLC檢測 (日本島津高效液相色譜儀,LC-10ATvp CLASS-VP V6.10):色譜柱為Kromasil C18,5 μm,4.6 mm×250 mm,流動相為乙腈/10 mmol/L pH 7.5–8.0的乙酸銨溶液 (65/35),流速為1 mL/min,檢測波長231 nm。LC-MS (美國Thermo公司LTQ型液相色譜-質譜聯用儀) 檢測在本所儀器室進行,質譜條件:ES I源;正離子檢測;鞘氣 (N2) 流速0.3 L/min;輔助氣 (He) 流速 0.1 L/min;源電壓4.5 kV;毛細管溫度350 ℃;毛細管電壓4.5 V。
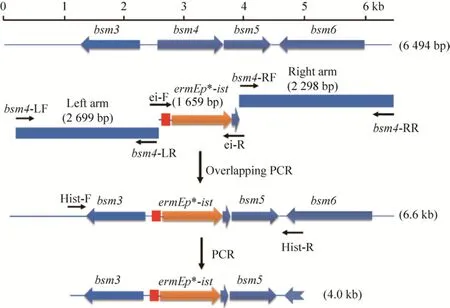
圖2 bsm4基因替換質粒的構建Fig.2 Construction of bsm4 replacement plasmids.
2 結果與分析
2.1 bsm4上下游基因分析
BT產生菌S.spiramyceticusWSJ已經完成全基因組測序,并從序列中鑒定出的螺旋霉素生物合成基因簇 (GenBank登錄號:MH 460451),3位的酰基轉移酶基因命名為bsm4(圖2),它的上游基因為bsm3,根據同源性推測其產物為23S rRNA甲基轉移酶,參與核糖體23S rRNA的甲基化,與抗生素的抗性相關。下游基因bsm5為O-甲基轉移酶基因,為螺旋霉素的后修飾基因,它與bsm4的開放閱讀框 (Open reading frame,ORF)之間只有5 bp的基因間序列,推測二者共轉錄。要確保bsm5的正常轉錄,在使用ermEp*-ist替代bsm4部分序列時,不能在ist后面設計轉錄終止信號,同時需要保留bsm5翻譯所需元件,如核糖體結合位點。因此在設計時保留了bsm4基因最后部分的111 bp序列。bsm6基因與bsm5的ORF方向相反,推測為crotonyl-CoA還原酶。ermEp*-ist替代bsm4后對其轉錄和翻譯沒有明顯影響。
2.2 構建重組質粒
利用 3對引物bsm4-LF/LR、eiF/eiR和bsm4-RF/RR以S.spiramyceticus1941基因組DNA為模板進行PCR,分別得到2 699 bp的左同源臂、1 659 bp的ermEp*-ist片段和2 298 bp的右同源臂3種目的片段 (圖3A),將這3種PCR產物通過重疊延伸PCR,獲得3個片段拼接的產物 (6.6 kb)。泳道4和5是利用bsm4-LF/RR引物鑒定構建好的pKC-ei重組質粒,目的片段大小與預期相符 (圖3B),并通過測序進行確證。將重組質粒pKC-ei通過原生質體轉化的方法導入到S.spiramyceticus1941,利用自然發生的同源重組來獲得ermEp*-ist替代bsm4的目的菌株。因為沒有抗性標記只能利用傳代和PCR進行篩選,經過幾百株次的篩選仍然無法獲得目的菌株。
之后利用CRISPR-Cas9系統的質粒pKCcas9do構建2種新的替換質粒pKcas-ei1和pKcas-ei2。圖3C中是pKcas-ei1 (泳道6和7) 與pKcas-ei2(泳道8和9) 進行XbaⅠ和HindⅢ酶切的電泳圖,均獲得了符合預期大小的4.0 kb片段;泳道10和11利用Hist-F/R引物驗證2種重組質粒中的4.0 kb的目的片段;利用sgRNA-1/R和sgRNA-2/R引物分別從pKcas-ei1和pKcas-ei2質粒中擴增出130 bp左右編碼sgRNA的小片段。最后經過測序證實2種質粒構建成功。通過原生質體轉化的方法導入到S.spiramyceticus1941中。
2.3 篩選出目的菌株
利用Am抗性篩選出含有pKcas-ei1和pKcas-ei2重組質粒的陽性轉化子。經過Tsr誘導Cas9表達后,通過傳代和在37 ℃培養使質粒停止復制,篩選出Am抗性消失的菌株 (圖4),再利用PCR篩選出既消除質粒又發生同源雙交換的目的菌株。只有pKcas-ei1的轉化子篩選出了陽性菌株ΔEI,而含pKcas-ei2的轉化子均未篩選出目的菌株。通過bsm4-LF/RR引物在原株S.spiramyceticus1941和ΔEI突變株中都擴增出了大約6 kb的特異性條帶 (圖3F),經過序列測定,證實在ΔEI突變株中目的基因bsm4確實被ermEp*-ist取代,而且其上下游基因也未發生突變。
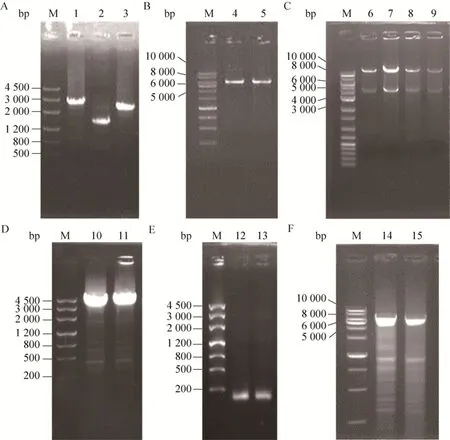
圖3 三種重組質粒的電泳檢測圖Fig.3 Identification of the three recombinant plasmids by electrophoretic detection.1: left arm; 2: ermEp*-ist; 3: right arm; 4–5: pKC-ei (bsm4-LF/RR); 6–7: pKcas-ei1 (Hind Ⅲ/XbaⅠ); 8–9: pKcas-ei2 (Hind Ⅲ/XbaⅠ); 10: pKcas-ei1(Hist-F/R); 11: pKcas-ei2 (Hist-F/R); 12: pKcas-ei1 (sgRNA-1/R); 13: pKcas-ei2 (sgRNA-2/R); 14: S.spiramyceticus 1941; 15: ΔEI; M: DNA marker.
2.4 檢測目的菌株ΔEI的發酵產物
通過HPLC檢測S.spiramyceticus1941原株和ΔEI突變株的發酵產物,與原株 (圖5B) 相比,在ΔEI突變株中沒有保留時間與SP Ⅱ和SP Ⅲ組分相似的產物出現,只有產物a保留時間為4.749 min,與SPⅠ的保留時間和紫外吸收光譜一致;與BT標準品 (圖5A) 對比,ΔEI中產物b的保留時間為16.596 min,與ISP Ⅰ的保留時間一致(圖5C)。ΔEI突變株的發酵產物經一級質譜全掃描,并未發現ISP Ⅱ和Ⅲ組分分子量相同的化合物峰。在ΔEI突變株中化合物a的離子峰[M+H]+為m/z843.5,與SP Ⅰ一致;產物b的離子峰[M+H]+為m/z927.62 (圖5D),與ISP Ⅰ的一致。而且產物b的二級碎片離子分別為768.36、699.39、540.37及402.28,也與ISP Ⅰ的裂解規律 (圖6) 和碎片峰數據一致 (圖5E和表2)。在ΔEI突變株的發酵產物中保留時間為26 min左右的峰為其他發酵產物,其[M+H]+為m/z279,遠小于螺旋霉素的分子量。因此ΔEI突變株發酵產物中只有SP Ⅰ和ISP Ⅰ組分,證實新的ISP Ⅰ菌株構建成功。

圖4 通過Am抗性和溫度篩選出的目的菌株Fig.4 The target strains screened by Am resistance and temperature.
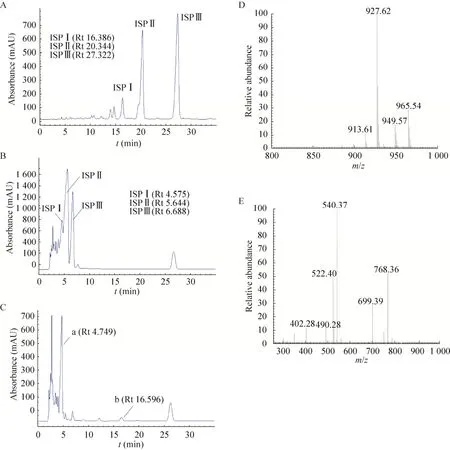
圖5 ΔEI突變株發酵產物的高效液相檢測圖和質譜檢測圖Fig.5 The profiles of HPLC and MS for the fermentation products of ΔEI strain.(A–C) HPLC detection.A: BT; B:S.spiramyceticus 1941; C: ΔEI.(D) The MS spectrum of compound b in ΔEI strain.(E) Product ion spectrum of [M+H]+of compound b at m/z 927.
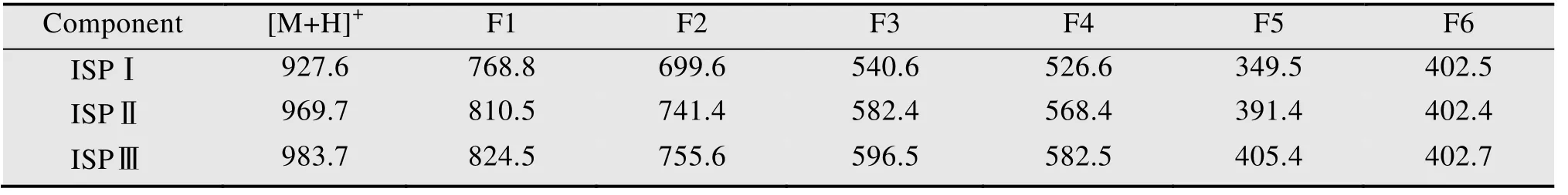
表2 異戊酰螺旋霉素的二級質譜碎片峰Table 2 The secondary MS data of isovalerylspiramycin
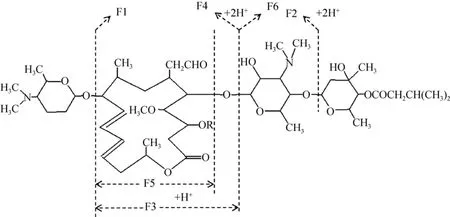
圖6 異戊酰螺旋霉素離子的裂解規律圖[16]Fig.6 The cleavage pattern of isovalerylspiramycin’s ion[16].
3 討論
ISP Ⅰ是BT中的一個主組分,其抗菌活性與BT相似,但其發酵產物質控更容易,還可以做成注射劑型。本研究獲得ISP Ⅰ新產生菌,只通過一次基因改造就實現目的基因的替換,而且不帶有抗性標記,可以持續進行遺傳操作來優化菌種。
實驗前期選擇經典同源重組的方法,通過替換質粒pKC-ei導入目的菌株進行同源雙交換,因為沒有抗性選擇壓力,自然發生同源雙交換的概率比較低,很難篩選到目的菌株。CRISPR-Cas9作為一種新的基因編輯技術可以通過sgRNA指導Cas9蛋白完成對靶DNA的雙鏈剪切而被廣泛使用,近幾年已經成功將該技術應用于腺病毒[17]、果蠅[18]、擬南芥[19]、水稻[20]、斑馬魚[21]等模式物種的基因修飾或改造中。在前期工作的基礎上利用CRISPR-Cas9基因編輯系統完成了bsm4基因的替換。開始時將6.6 kb的外源片段均克隆至pKCcas9do質粒上,重組質粒總長度近20 kb,因質粒過大導致原生質體轉化試驗失敗。縮短左右同源片段后,構建出的新質粒順利導入S.spiramyceticus1941中。經過Tsr的誘導、Cas9的切割及同源片段的互補后,最終篩選出發生同源雙交換的目的菌株。但只有pKcas-ei1篩選出了轉化子,pKcas-ei2一直未能成功篩選出目的菌株,原因可能是第2個切割位點不太適合CRISPR-Cas9系統,還有可能是發生脫靶效應導致重要基因失活。
ermEp*是鏈霉菌中組成型強啟動子,包含有2個-35區和-10區,屬于多重啟動子,可能還具有雙方向調控轉錄的功能[22],在3位酰基轉移酶基因的上游是23S rRNA的甲基轉移酶基因,是一種修飾23S rRNA而獲得抗生素抗性的基因,抗生素產生菌的產抗生素能力與其對抗生素的抗性之間具有一定的相關性,抗性基因的高表達通常能提高抗生素的產量[23–25]。用ermEp*-ist基因替換bsm4基因可以同時實現2個目標:1) 將3位酰基轉移酶基因刪除,菌株只產生SP Ⅰ;2) 高表達的ist基因整合到宿主染色體上穩定存在,而且位于螺旋霉素生物合成基因簇之中,有利于進行異戊酰基化修飾。另外,ermEp*還可能會提高上游的抗性基因的表達,從而增加螺旋霉素的產量。而且只經過1次遺傳操作和含有1個ist基因,對菌株的影響較小,菌株的遺傳穩定性更好。
從HPLC檢測結果來看,尚有大量的SP Ⅰ沒有轉化成ISP Ⅰ,說明ermEp*控制下的ist基因并沒有獲得理想的高表達,但其效價為420 μg/mL,比原始的ISP Ⅰ菌株提高了2倍多。通常來講,經過基因工程操作的菌株的產量開始時一般都偏低,還需要進行相應的育種分離操作,才能篩選出效價和ISP Ⅰ都較高的新菌株。將來還可利用CRISPR-Cas9系統把已經構建的ist高表達盒[26]導入到ΔEI菌株中,將會進一步提高SP Ⅰ轉化為ISP Ⅰ的產量,還可進行其他遺傳操作來獲得更加優質高產ISP Ⅰ的菌株。

