鹽地堿蓬PsaH基因的克隆及生物信息學分析
郝曉燕,李建平,常曉春,足木熱木,高升旗,陳果,黃全生
(新疆農業科學院核技術生物技術研究所,烏魯木齊 830091)
0 引 言
【研究意義】光系統I(Photosystem I,PS I)是由至少13個獨立亞基多肽組成的嵌于光合膜上的色素蛋白復合物,在植物中主要分布于葉綠體類囊體膜基質膜區[1-2]。在光合反應中主要起催化光電子傳遞的作用。在高等植物中所有已知的編碼PS I蛋白亞基的基因都已經被克隆,依據這些基因被發現的先后順序依次命名為PsaA-PsaN。真核生物中PS I蛋白亞基基因由葉綠體或細胞核基因組編碼,其中H亞基(PsaH)是由核基因組編碼的低分子量膜外周蛋白,主要存在于復合蛋白的邊緣[3-4]。鹽地堿蓬(suaedasalsaL.)隸屬藜科(Chenopodiaceae)堿蓬屬(suaeda),是一種葉片高度肉質化的一年生真鹽植物[5],主要分布于新疆、山東、遼寧等省區海濱、平原荒漠區湖邊潮濕鹽土上,是一種典型的鹽堿地指示植物,也是我國最重要的鹽土荒漠建群種之一。隨著土壤鹽漬化日趨嚴重與人口數量急劇增長矛盾的突出,開發和有效利用鹽漬土逐漸成為研究熱點,而增強植物的耐鹽性是提高鹽漬土利用效率有效的方法之一。因此,利用現代生物學技術,通過對鹽生植物耐鹽基因的發掘和功能研究能夠為改良作物耐鹽性提供理論基礎,還將對有效地改良和利用鹽漬土提供思路。對于農業生產、環境保護和生態治理都具有重要的意義。【前人研究進展】有關鹽地堿蓬的研究工作雖然早已起步,涉及了葉片結構、離子平衡、滲透調節、抗氧化系統和光合特性等方面[6-7]。在分子水平上,構建了堿蓬鹽脅迫cDNA文庫,篩選出很多可能與耐鹽相關的基因[8]。目前從堿蓬中克隆的有INPS、P5CS、BADH、CMO、NHX1、TypA1、H+/Ca2+等耐鹽相關基因[9-12],且轉化了擬南芥、水稻、煙草等植物中并顯著提高了轉基因后代的耐鹽能力。【本研究切入點】有關鹽地堿蓬中PsaH基因的研究尚未見報道。利用RT-PCR技術從鹽生植物堿蓬中獲得耐鹽相關基因SsPsaH全長,并對其進行生物信息學分析,以便進一步的了解其分子生物學功能。【擬解決的關鍵問題】 研究以鹽地堿蓬為材料,從鹽脅迫cDNA文庫中獲得PsaH基因cDNA全長序列,通過生物信息學軟件對其結構和功能進行預測,為開展該基因的功能研究提供基礎。
1 材料與方法
1.1 材 料
1.1.1 植物材料
鹽地堿蓬(suaedasalsa)種子收獲于阜康水庫鹽堿地,生長至4周大小,以400 mM NaCl脅迫處理48 h取樣,迅速置于液氮中冷凍,-70℃冰箱保存備用。
1.1.2 質粒、菌株、試劑
大腸桿菌(Escherichiacoli)Trans-T1感受態細胞、DNA Marker、TaqDNA聚合酶、pEASY-Blunt Zero Cloning Kit購自TransGen Biotech;;PowerScriptTMII 反轉錄試劑盒、限制性內切酶均購自NEB公司,DNase 酶Ⅰ、LATaq酶、ExTaq酶、dNTP均購自TaKaRa 公司,T4DNA 連接酶購自Promega 公司,DNA 回收試劑盒購自于OMEGA,引物由北京華大生物科技有限公司合成。
1.2 方法
1.2.1 RNA的提取與純化,cDNA的合成
材料的總RNA提取按照Life Technologies公司的PureLinkTMRNA Mini Kit 說明書進行操作。以DNaseⅠ純化處理后,通過瓊脂糖凝膠(1.0%)電泳檢驗獲得的各處理樣品總RNA的完整性,并用紫外分光光度計(Eppendorf)檢測各樣品總RNA的純度和濃度。
第一鏈cDNA的合成按照Fermentas公司的First Strand cDNA Synthesis Kit操作說明進行,采用的反轉錄引物為Oligo(dT)15。取1 μg 總RNA 作為模板進行RT-PCR,20 μL體系中總RNA 1 μg,DEPC水6 μL,Oligo(dT)151 μL,65℃ 5 min,冰上放置5 min,RNase inhibitor 1.0 μL,5×Reaction buffer 4 μL,dNTP(10 mmol/L)2 μL 和M-MLV(200 U/μL)1 μL,42℃溫育60 min,70℃變性5 min,冰上放置5 min,-70℃保存備用。
1.2.2 SsPsaH基因的克隆
使用Primer Primer 5.0軟件在開放閱讀框兩側設計一對能有效擴增SsPsaH的特異性引物(SsPsaH-ORF-F:5’-ATGGCTTCTCTAGCAACCTTTGCC-3’;SsPsaH-ORF-R:5’-TTAGATCTTGCCACGAGGTCCG-3’,擴增片段長度為438 bp)。以合成的第一鏈cDNA為模板,進行PCR擴增。反應體系為50 μL:cDNA 1 μL,引物各1 μL,ExTaq酶0.5 μL,10×PCR Buffer (Mg2+plus)5 μL,dNTP Mixture 4 μL,加ddH2O至50 μL。PCR程序:94℃預變性3 min;94℃變性30 s,60℃復性30 s,72℃延伸30 s,30個循環;72℃延伸10 min; 4℃保存。瓊脂糖凝膠電泳檢測后用DNA回收試劑盒回收目的片段連接到pEASY-Blunt Zero載體進行克隆,陽性克隆經酶切鑒定后送北京華大基因科技有限公司完成DNA測序。
1.2.3 SsPsaH基因的生物信息學分析
SsPsaH基因的核苷酸和氨基酸序列分別用NCBI的BLASTn和BLASTp(http://bladt.ncbi.nlm.nih.gov/Blast.cgi)進行相似性分析;利用在線軟件Protparam (htpp://www.expasy.org/protparam/)分析蛋白的理化性質;用TMPred軟件預測蛋白的跨膜區;用SignalP 4.1 Server 在線軟件預測蛋白的信號肽;用GOR4軟件分析蛋白的二級結構。在NCBI數據庫中查找與PsaH基因同源性較高的其他物種的同源基因進行多序列比對,用MEGA5.0通過鄰接算法(Neighbor-Joining)完成系統進化樹構建[13-15]。
1.2.4 SsPsaH基因的組織特異性表達分析
分別提取鹽地堿蓬根、莖、葉3種不同組織的總RNA(方法參見ThermoFisher PureLink RNA Mini Kit使用手冊)。以總RNA為模板,Oligo(dt)18為引物反轉錄合成第一鏈cDNA(方法見Fementers公司的 RevertAid First-strand cDNA Synthesis Kit for RT-PCR試劑盒使用手冊)。獲得的cDNA稀釋10倍后作為RT-PCR定量檢測的模板。SsPsaH基因的表達分析引物(SsPsaH-RT-F:5’-AGGATGTTCTACCCATCACAAA-3’;SsPsaH-RT-R:5’-ATGATACACCAAAAGGAAAGAAAT-3’),同時以鹽地堿蓬內參基因Actin為對照,引物序列(Actin-RT-F:5’-GGGCGAGTACGACGAATCTG-3’,Actin-RT-R:5’-CCTCTCCATCTCTCTCGACCAAAT-3’),反應條件為94℃預變性1 min,隨之94℃ 30 s,56℃ 30 s,72℃ 30 s,28個循環,最后72℃延伸7 min。
2 結果與分析
2.1 鹽地堿蓬SsPsaH基因的獲得
對400 mM NaCl脅迫處理48 h的堿蓬cDNA文庫進行耐鹽篩選后測序過程中,發現了1個與菠菜PsaH基因(XP_021B62899.1)同源性高達91%的序列(EST),對其相應的克隆進行完全測序,ORF Finder軟件分析發現該片段包含完整的CDS序列,將其命名為SsPsaH(GenBank Accession Number:KC4048847)。該基因全長770 bp,含有1個長為438 bp的開放閱讀框,編碼145個氨基酸。圖1

注:M:起始密碼子;*:終止密碼子
Note: M: start codon; *: stop codon
圖1 獲得的SsPsaH基因的cDNA全長序列及其氨基酸序列
Fig.1 Cdna sequence and amino acid sequence of SsPsaH gene obtained
2.2 SsPsaH蛋白的同源性及功能結構域
在NCBI上通過Blast對SsPsaH蛋白序列同源性進行分析。研究表明,SsPsaH蛋白與藜麥(chenopodiumquinoa) 相似性最高為94%,與菠菜(Spinaciaoleracea) 同源性為91%,與甜菜(Betavulgarissubsp)相似性為90%,與木薯(Manihotesculenta)、蓖麻(Ricinuscommunis) 相似性為85%,與毛果楊(Populustrichocarpa)的相似性為84%,與其它植物的氨基酸序列有著高度保守性。由此確定,研究所克隆的SsPsaH基因是屬于光系統I(PSI) H亞基成員。
利用DNAMAN軟件對包含SsPsaH基因在內的6種植物的氨基酸序列進行保守結構域分析發現,SsPsaH基因與其他植物中的PsaH基因一樣包含4個保守的結構功能域典型特征:1個N端的高酸性富集結構域NH-KYGDK,1個轉移肽(101-121)代表該基因編碼的氨基酸可能具有跨膜結構域,1個中心疏水結構域和1個C端的高度保守結構域PPKLGPRGKI。這些分析結果表明克隆的SsPsaH基因屬于PsaH超級家族。圖2

圖2 SsPsaH蛋白與其他物種的氨基酸序列
Fig.2 Comparison of amino acid sequences homology of SsPsaH with different plant PsaH
2.3 SsPsaH蛋白質的系統進化樹分析及理化性質預測
將鹽地堿蓬與藜麥、菠菜、甜菜、擬南芥、木薯、蓖麻等的PsaH氨基酸序列,用相鄰連接法通過MEGA5.0軟件構建系統進化樹,迭代值為1 000。可以看出,堿蓬與菠菜、甜菜、藜麥、擬南芥聚為一組,且與同屬藜科的菠菜、甜菜、藜麥聚在同一個分支,其中與菠菜親緣關系最近。而蓖麻、毛果楊、木薯、煙草樟子松、大豆、花生聚為一組,其中蓖麻和毛果楊親緣關系最近且在同一分支。這與NCBI中氨基酸同源性分析的結果相一致。用在線軟件ExPasy預測SsPsaH基因所編碼的蛋白質理化性質。該蛋白為不穩定的親水性蛋白。圖3,表1
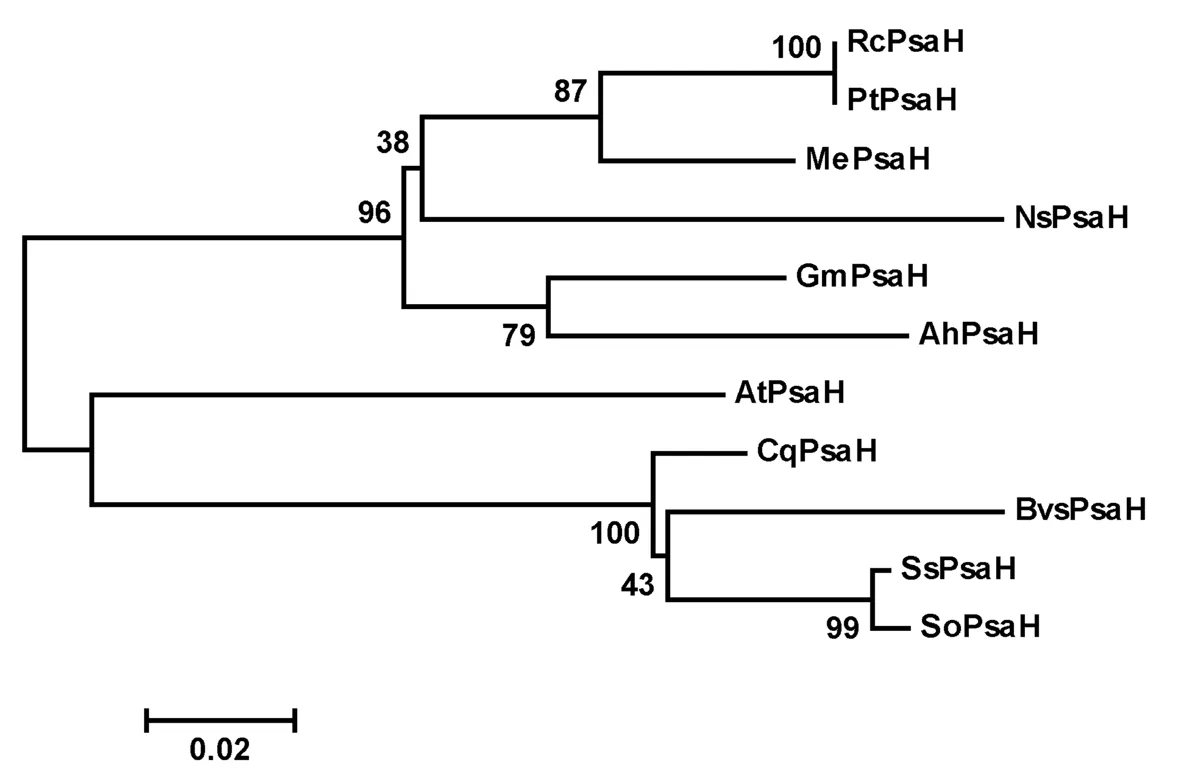
RcPsaH (蓖麻Ricinuscommunis, XP002516025);PtPsaH (毛果楊Populustrichocarpa, XP002304206);MePsah(木薯Manihotesculenta,XP021627101);NsPsaH (煙草樟子松Nicotianasylvestris, BAA04634); GmPsaH (大豆Glycinemax, ACU13848);AhPsaH (花生Arachishypogaea,AB184258); AtPsaH (擬南芥Arabidopsisthaliana,NP175633);CqPsaH (藜麥Chenopodiumquinoa,XP02136868); BvSPsaH (甜菜Betavulgarisstubsp. XP010691337); SoPsaH (菠菜Spinaciaoleracea, XP021862899)
圖3 SsPsaH系統進化樹構建
Fig.3 Phylogenetic tree analysis of SePsaH protein
表1 SsPsaH蛋白的理化性質預測
Table 1 Physicochemical property analysis of SsPsaH protein

蛋白理化特性characteristics預測結果Predicted result編碼的氨基酸個數 Number of coding amino acids145等電點 Theoretical PI9.84分子量 Molecular weight (Mr/103)15372.66負電荷殘基Total number of negatively charged residues (Asp+Glu)8正電荷殘基Total number of positively charged residues (Arg+Lys)16分子式 FormulaC699H1109N183O204S1不穩定系數 Instability index (II)47.81平均疏水性 Grand average of hydropathicity (GRAVY)-0.148脂肪系數 Aliphatic index (AI)83.45
2.4 SePsaH蛋白質二級結構預測
使用GOR 4在線分析軟件預測SePsaH蛋白二級結構發現,α-螺旋(Helix)占8.97%,延伸鏈(Extended strand)占25.52%,無規則卷曲(Random coil)占65.52%,無β-螺旋結構存在。圖4

注:A:大寫字母代表SePsaH蛋白質的氨基酸序列;h、c、e分別代表α-螺旋、無規則卷曲、延伸鏈. B:短豎線區(紫色線),無規則卷曲;中豎線區(紅色線),延伸鏈;長豎線區(藍色線),α-螺旋
Note: A. capital letters represent the amino acids sequence of SsPsaH protein; h, c and e represent the alpha helix, random coil and extended strand, respectively. B. short vertical bar area (purple wire): random coil; medium vertical bar area(red wire): extended strand; long vertical bar area(blue wire): alpha helix
圖4 SsPsaH蛋白質二級結構預測
Fig.4 Secondary structure prediction of SsPsaH
2.5 SsPsaH蛋白質的信號肽預測
使用SignalP 4.1 Server 在線軟件,對SsPsaH蛋白進行信號肽預測,研究表明,第21位蘇氨酸殘基原始剪切位點最高分值0.167,綜合剪切位點最高分值0.165,第1位蛋氨酸信號肽最高分值0.228,遠遠小于5,推測SsPsaH蛋白不存在信號肽。圖5,表2
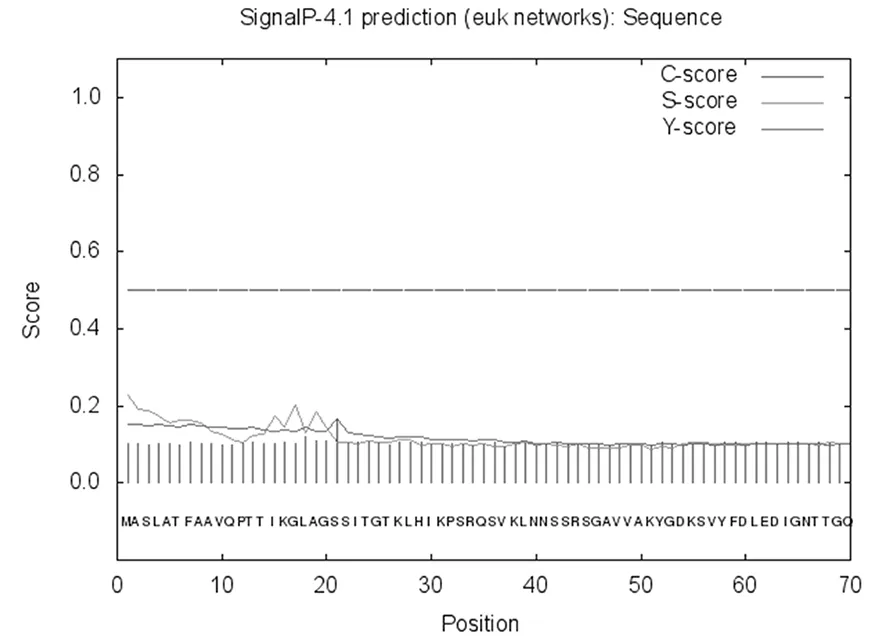
圖5 SsPsaH蛋白的信號肽預測
Fig.5 Prediction of the SsPsaH protein signal P-NN
表2 SsPsaH蛋白的信號肽預測數據
Table 2 Signal P-NN data prediction of the SsPsaH protein

指標Measure位點Sit得分Score有無信號肽Signal peptideMax. C210.167—Max. Y210.165—Max. S10.228—Mean S1-200.156—D1-200.160無No
Max. C:原始剪切位點的分值;Max. Y:綜合剪切位點的分值;Max. S: 信號肽的分值;Mean S:信號肽分值的平均值;D score:mean S和max. Y 的加權平均值
Max. C: scores of putative cleavage site; Max. S: scores of signal peptide; Max. Y: scores of synthesis cleavage site; Mean S: average S-score; D score: weighted average of the Mean S and the Max. Y scores
2.6 SsPsaH蛋白質跨膜結構預測
通過在線軟件TMPred分析SsPsaH蛋白的跨膜性預測,可知該蛋白具有1個跨膜結構域(位于第101~121個氨基酸殘基之間)。同時利用PredictProtein對 SsPsaH蛋白進行亞細胞定位預測分析,發現該蛋白可能定位于葉綠體膜系統上。這些結果與該基因氨基酸序列的結構功能域分析結果相符。圖6
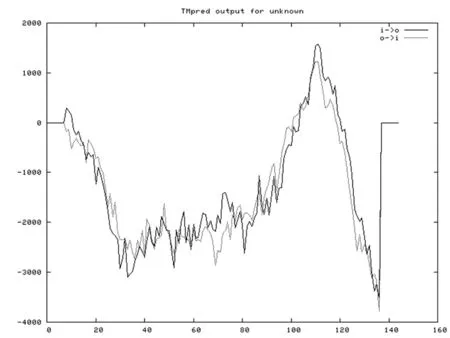
圖6 SsPsaH蛋白的跨膜結構域預測
Fig.6 The transmembrane domain prediction of the SsPsaH protein
2.7 SsPsaH基因的組織特異性表達
以鹽地堿蓬的根、莖、葉3種不同組織的RNA為模板進行RT-PCR分析。研究表明,SsPsaH基因在根、莖、葉中均有表達,但在葉和莖中表達量明顯高于根中的表達量。圖7
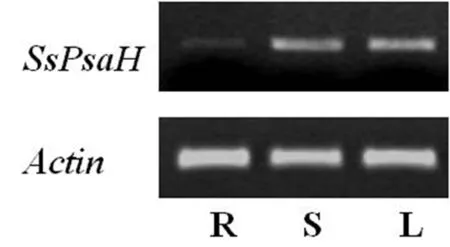
R: root; S:stem; L:leaf
圖7 SsPsaH基因的組織特異性表達
Fig.7 The expression analysis of the SsPsaH gene
3 討 論
光合作用是植物賴以生存的基礎,也是植物對外界環境刺激最敏感的生理過程之一。PS I作為光系統復合體蛋白在光合作用中起維持單子傳遞效率和狀態轉化等作用。研究表明,組成PS I 光系統各亞基的變化會影響它們之間的相互作用及蛋白復合體的結構形成。PsaH蛋白作為PS I復合體的一個組成亞基,主要通過與其它幾個亞基PsaD、PsaG、PsaI、PsaL、PsaP、PsaO相結合以及光合系統的環狀捕光天線LHCII形成相互作用區域,從而完成光合作用的同化過程[4、16、17]。Helle等[18]在PSI-H亞基缺乏的擬南芥突變體中發現,H亞基的缺失會引起PSI-L亞基組份的降低和NADP+酶活性降低至61%,此外還會影響到光電子傳遞的效率。鄭成超等克隆了裂葉牽牛(Pharbitis nil Choisy)PsaH基因[19],并發現其受內生晝夜節奏的調控,明顯受光誘導表達。江玉梅等[20]從(Lycoris radiata)石蒜中克隆了PsaH基因,張林華等[21]從天山雪蓮(Sasussured involucrate Kar.et Kir)中克隆了PsaH基因,Lunde等[22]研究發現,PsaH基因在維持PS I的穩定和光電子傳遞過程中起重要作用。對于該基因的研究大多都集中在光合作用本身。研究是在通過耐鹽脅迫文庫篩選后測序中獲得堿蓬SsPsaH基因全長cDNA序列,而PsaH基因在參與鹽地堿蓬應答鹽脅迫的研究還未見報道。
4 結 論
以鹽地堿蓬為實驗材料,獲得PsaH基因。通過生物信息學軟件分析結果顯示,該基因編碼蛋白定位于葉綠體膜系統上,是一種不穩定的親水性蛋白,不含信號肽;二級結構中無規則卷曲所占比例最高;有PsaH基因家族的保守結構域,屬于光系統I(PSI)復合蛋白H亞基。系統進化樹分析表明其與藜科的菠菜、甜菜聚為一個組,且同源性高達90%,該基因具有很強的保守性,且在親緣關系較近的物種中保守性更高。RT-PCR對堿蓬組織特異性表達分析結果表明,SsPsaH基因主要在堿蓬的莖和葉中表達。

