蘇丹草轉錄組SSR分子標記開發及遺傳多樣性評價
朱永群,彭丹丹,林超文,聶剛,許文志,黃琳凱,羅付香,彭建華,張新全*
(1.四川省農業科學院土壤肥料研究所,四川 成都 610066;2.四川農業大學動物科技學院,四川 成都 611130;3.四川省農業科學院,四川 成都 610066)
蘇丹草(Sorghumsudanense)系高梁屬一年生草本植物,染色體數為20(2n=20),其開花習性與一般禾本科植物不同,屬于異花授粉植物,因原產于非洲北部蘇丹地區而得名。蘇丹草具有耐旱、高產、營養價值豐富等特點,作為一種優質的禾本科牧草在世界各地廣泛栽培和使用[1-2]。自20世紀30年代由美國引入后,蘇丹草在我國的種植區域不斷擴大,主要分布在內蒙古、新疆、甘肅和寧夏等北部省區以及長江中下游地區,不僅可用于放牧、刈割青飼、調制青貯等為家畜所喜食,還具有“漁業青飼料之王”的美稱[3-4]。蘇丹草與高粱的雜交種高丹草(Sorghumbicolor×Sorghumsudanenes)因具有明顯的雜種優勢,亦是草業生產中一種重要的牧草,具有顯著的經濟效益[5]。我國開展蘇丹草品種選育和整理工作始于20世紀70年代,但由于缺乏品種在不同條件下生長表現的系統性評價和研究,其選育多憑個人經驗,缺乏一定的科學預見性,導致目前為止通過我國草品種審定的蘇、高丹草品種數還屈指可數,加之市面上銷售的草種子多存在魚龍混雜的現象,使得這一優良牧草的利用、推廣和品種的更新受到嚴重的阻礙,與目前全國畜牧業和養殖業的大力發展對蘇、高丹草生產規模的需求極不協調[6-7]。
簡單重復序列(simple sequence repeats,SSRs),是一類由1~6個核苷酸堿基為基本單元,組成的較長串聯重復的DNA序列[8]。SSR分子標記具有多態性高、重復性好、覆蓋面廣、共顯性和易于操作等特點,廣泛應用于牧草和作物的基因定位、遺傳圖譜構建、遺傳多樣性分析及分子標記輔助育種等方面[9-10]。然而,傳統SSR分子標記開發過程具有盲目性和隨機性,且開發成本高,開發效率低。雖然Zhan等[11]根據Gramene在線數據庫設計引物,利用SSR標記對多份蘇丹草及高粱材料間的多樣性及系統發育關系進行了研究,盧杰等[12]和李杰勤等[13]利用了高粱(Sorghumbicolor)表達序列標簽(expressed sequence tags, EST)序列信息建立EST-SSR標記應用于蘇丹草證明了標記的可行性,但迄今為止關于專門針對蘇丹草開發的SSR分子標記還鮮有報道,且缺乏與性狀緊密相關的特異性標記,嚴重制約了蘇丹草分子育種工作的進程。基于高通量測序技術(next generation sequencing, NGS)獲得的基因組轉錄信息用于分子標記的開發,具有簡單快捷、高效準確、引物通用性好、成本低等顯著優點,此外,還被證明標記可以有效與植物非生物逆境脅迫下抗性和重要的農藝性狀等緊密關聯[14-16]。因此,利用轉錄組信息開發SSR分子標記被認為是新時代提高作物遺傳多樣性和分子標記輔助育種研究準確性的有效途徑[17],目前已成功運用于苜蓿(Medicagosativa)[18]、鴨茅(Dactylisglomerata)[19]、芒草(Miscanthus)[20]等牧草種質資源的研究中。在蘇丹草測序工作研究方面,雖然Li等[21]先前根據S772蘇丹草轉錄組測序信息開發了SNP和Indel標記,但關于蘇丹草轉錄組SSR標記的開發及應用還有待進一步的研究,這為本工作的開展提供了契機。
本研究基于蘇丹草轉錄組測序結果擬分析SSR位點分布特征,設計開發EST-SSR分子標記,對蘇、高丹草共36份材料進行EST-SSR標記的遺傳多樣性分析,為豐富蘇丹草分子標記類型、開發與利用轉錄組EST資源提供基礎,也為進一步開展蘇丹草種質資源評價、遺傳圖譜構建和分子標記輔助育種提供參考。
1 材料與方法
1.1 供試材料與DNA提取
本試驗材料為來源于農業部全國畜牧總站國家牧草種質基因庫的蘇丹草及高丹草種質資源共36份,材料編號及來源地見表1。試驗從2017年1月起,對蘇丹草種子采用沙培法進行培育。種子經1% 次氯酸鈉消毒5 min并用蒸餾水沖洗幾遍后播種于盛有石英砂的培育盆中,放入光照培養箱中發芽,待發芽4 d后更換為Hoagland全營養液培養,溫度設置為白天25 ℃,夜晚22 ℃,時長各為12 h。待幼苗長至18 d左右(2片成熟葉片),每份種質資源隨機選取20個單株的幼嫩葉片等量混合并用植物DNA提取試劑盒(Qiagen)提取DNA(每份材料3個重復)。所提DNA經1.0%的瓊脂糖凝膠電泳和紫外分光光度計檢測純度和濃度,將檢測合格的DNA樣品置于-20 ℃冰箱保存。試驗開始前將各DNA樣品取出一部分用TE(Tris-EDTA)緩沖液稀釋至10 ng·μL-1,置于4 ℃冰箱備用。
1.2 SSR引物設計
在土壤肥料研究所土壤與耕作飼草研究室對烏拉特1號(Wulate No.1)蘇丹草轉錄組測序結果(SRR登錄號5183088)得到的13574條包含SSR位點的EST序列的基礎上,利用MISA 1.0軟件根據設定的單位大小和最小重復參數如1-10、2-6、3-5、4-5、5-5和6-5對位于非重復序列基因(unigenes)上的EST序列設計引物。用Primer 5.0引物設計軟件設定參數:鳥嘌呤和胞嘧啶(guanine and cytosine, GC)含量40%~60%,退火溫度50~60 ℃,且上下游引物熔解溫度差值不超過5 ℃,引物長度18~23 bp,預期擴增產物長度范圍為100~300 bp獲得300對EST-SSR引物。所有引物由金斯瑞生物科技有限公司合成。
1.3 SSR擴增及多態性檢測
從36份DNA樣品中隨機選取6份對300對SSR引物進行篩選,經PCR擴增和電泳分離鑒定后最終獲得54對條帶清晰、多態性良好的有效性引物用于進一步遺傳多樣性研究(表2)。25 μL PCR擴增體系包括2 μL模板DNA,0.5 μL Taq DNA 聚合酶 (2.5 U·μL-1),12 μL 2×Taq PCR Master Mix,上下游引物各1 μL,ddH2O 8.5 μL。擴增程序為94 ℃預變性4 min,接下來94 ℃變性 45 s,58 ℃退火45 s,72 ℃ 延伸1 min,共30個循環,最后72 ℃延伸10 min。PCR產物在200 V下經6%聚丙烯酰氨凝膠電泳預分離30 min后再轉入350 V下電泳1.5 h。并用0.1% AgNO3進行銀染和50 bp marker 鑒定PCR產物大小,顯影完后用凝膠成像系統拍照保存。
1.4 數據統計與分析
根據PCR產物在凝膠電泳上的情況,選取清晰可辨的條帶進行統計,對相同遷移位置或長度一致的條帶有帶記為“1”,無帶記為“0”,構成標準的0/1矩陣,統計PCR產物擴增總帶數(total number of bands,TNB)和多態性條帶數(number of polymorphic bands,NPB),計算多態位點百分率(percentage of polymorphic bands,PPB)和引物多態信息含量(polymorphic information content,PIC)。其中,PPB=K/N×100%,式中:K為多態位點數目;N為所測位點總數;PICi=1-∑Pij2,式中:PICi表示引物i的多態性信息含量,Pij表示引物i的第j個帶型出現的頻率,引物i的總帶型從 1 到n。利用NTSYS-pc2.10e軟件對原始矩陣進行分析,基于similarity程序計算遺傳相似系數(GS),用clustering程序中的SHAN進行聚類分析,再根據Graphics程序中的tree plot繪制親緣關系樹形圖。
2 結果與分析
2.1 蘇丹草轉錄組SSR分布頻率
從蘇丹草轉錄組的80686條Unigene序列中共鑒定出17548個SSR位點,分布在13574條Unigene序列中,發生頻率為16.82%(含有SSR的Unigene 數量占總Unigene 數量的百分比),其中2965條Unigene序列含一個以上SSR位點,SSR分布頻率為21.75%(SSR 的個數與總Unigene 的數量比),平均每43.14 kb出現一個SSR位點(SSR 的個數占序列總長的百分比)。


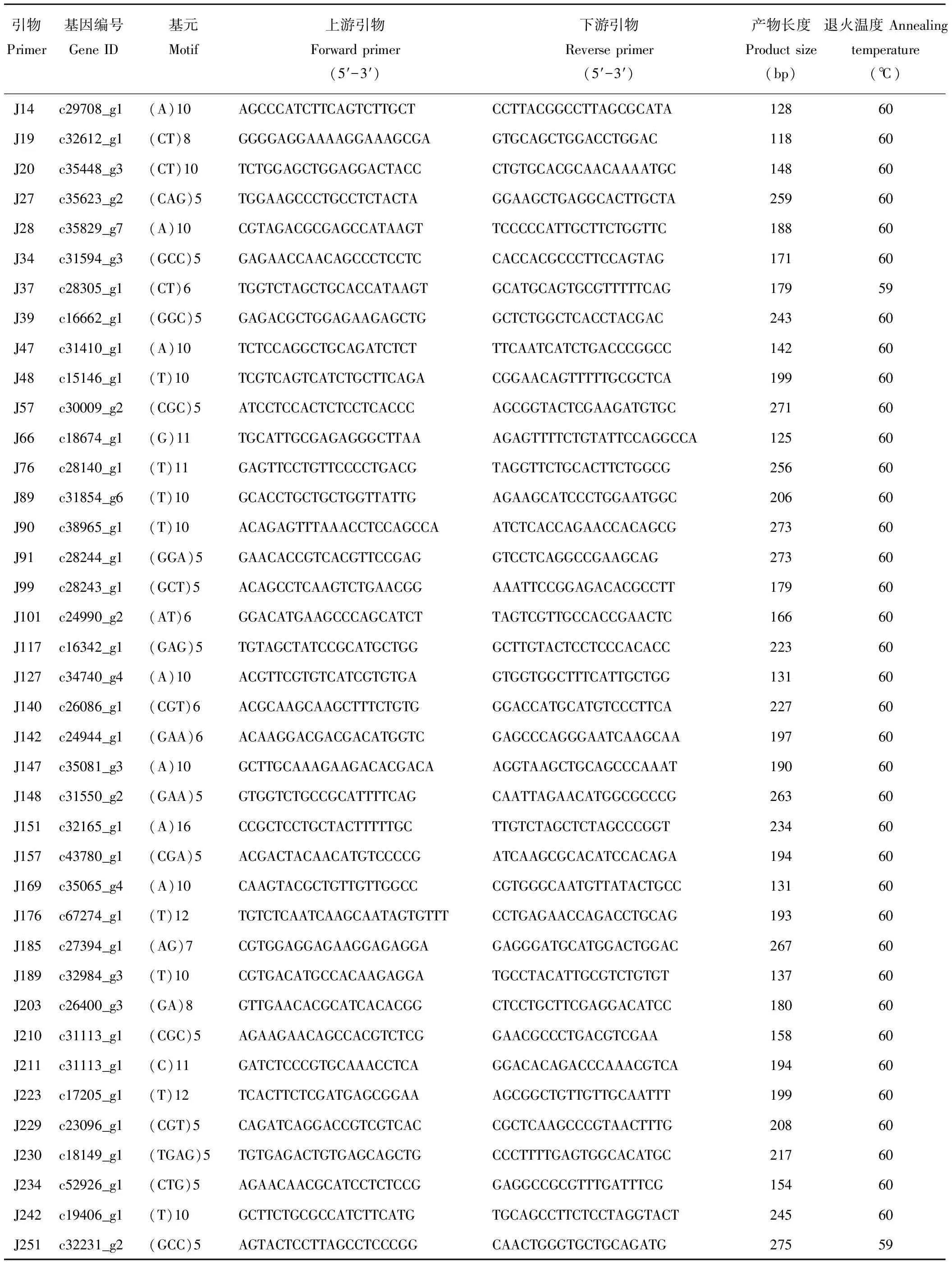
表2 引物信息及序列Table 2 Primer and sequences information applied in this study
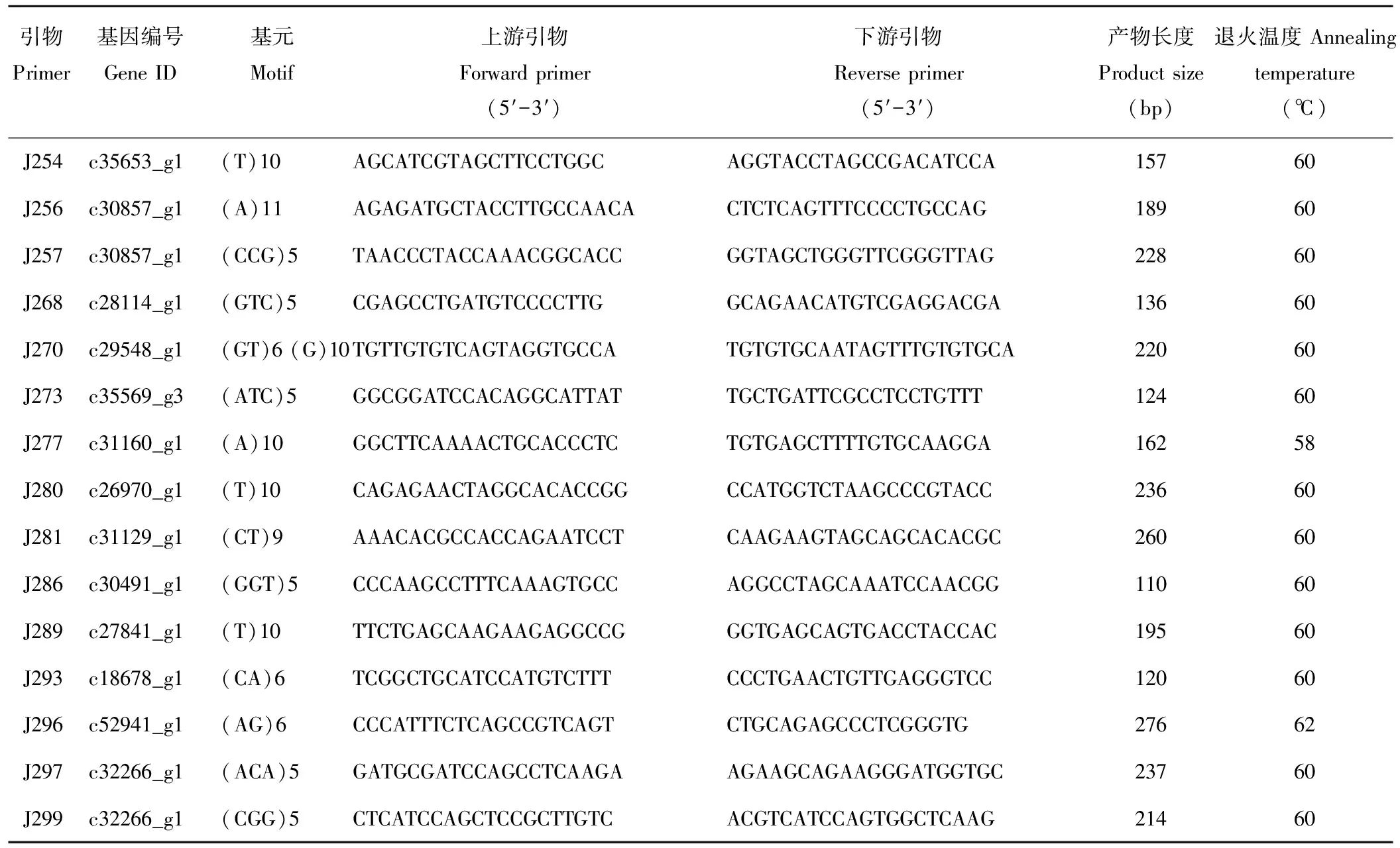
續表2 Continued Table 2
蘇丹草轉錄組SSR種類包括一至六核苷酸6種重復類型,以單/一核苷酸重復和三核苷酸重復類型最為豐富,分別占38.59%(6771)和39.09%(6860),其次為二核苷酸重復,有3366個,占19.18%,四、五和六核苷酸重復遠低于前3種重復類型,總計占3.14%(551)(表3)。
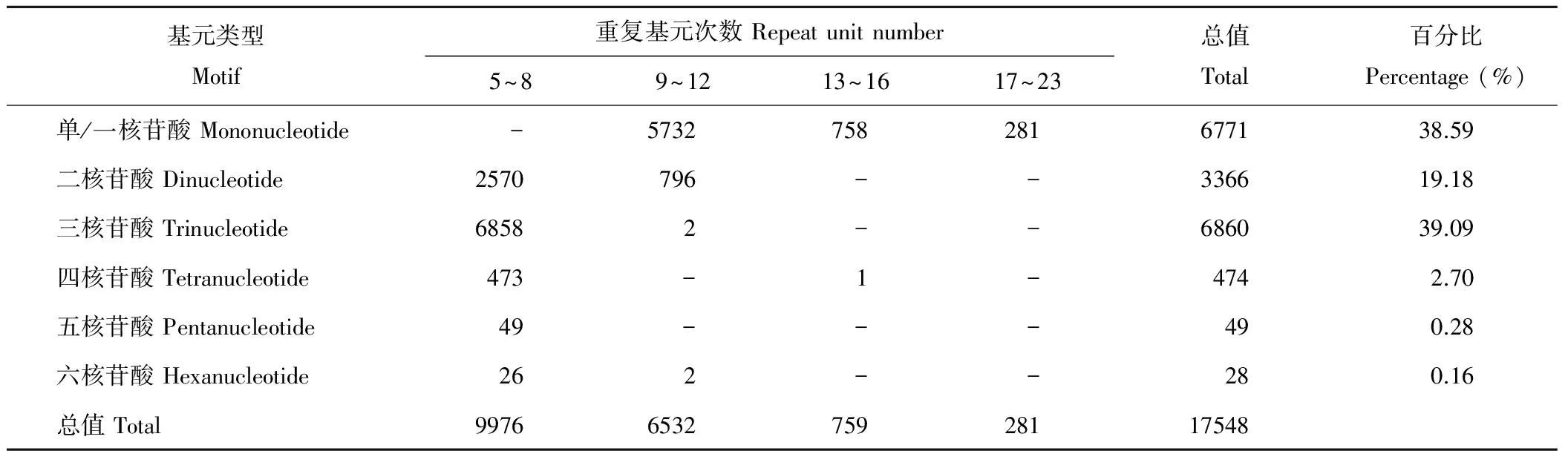
表3 基于重復基元的重復次數與重復數量分布Table 3 Frequencies of different SSR repeat motif types related to variation of repeat unit numbers
2.2 蘇丹草轉錄組SSR位點的分布特點
蘇丹草SSR重復單元的重復次數分布在5~23次,其中5~8次重復的有9976個SSR,其次為9~12次重復,有6532個SSR,往后逐漸減少。單核苷酸重復基元的重復次數跨度最大,在9~23次均有分布,其余核苷酸重復基元的重復次數主要集中于5~8次,在13~23次幾乎沒有分布(表3)。
17548個SSR位點中,占優勢重復的一、二和三核苷酸重復基元分別有2、4和10種,其中以A/T、AG/CT和CCG/CGG出現的頻率最高,分別占該重復基元類型的91.06%、46.67%和39.21%(表4)。
2.3 蘇丹草轉錄組SSR引物篩選與多態性檢測
隨機選擇的300對引物經初步篩選有77對引物未能擴增出清晰的條帶,擴增成功率為73.67%。對擴增成功的223對引物做進一步的篩選驗證,得到54對(24.22%)在供試材料間表現出擴增效果好、條帶清晰且在目標條帶范圍內的多態性引物,如圖1所示。54對有效引物在36份供試材料中共擴增出275條清晰帶,平均每對引物擴增5.09條帶,單個引物對最多擴增條帶數為12,最少擴增條帶數僅為2,擴增片段大小介于110~273 bp,其中多態性條帶數有205條,多態位點百分率(PPB)平均值為73.02%,不同引物所揭示的供試材料的多態信息含量(PIC)介于0.10~0.94,平均值為0.41(表5)。
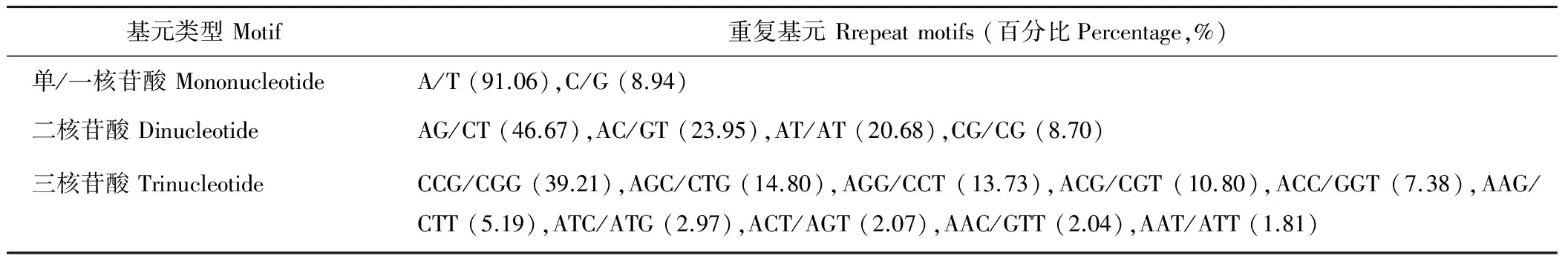
表4 重復基元類型的重復基元分布Table 4 Distribution of repeat motifs according to classified repeat types
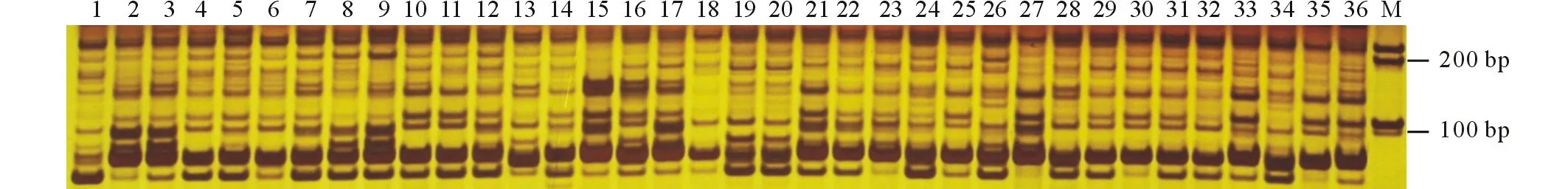
圖1 引物J268對36份材料的擴增結果Fig.1 The amplification results by primer J268 on 36 accessions
2.4 基于蘇丹草轉錄組SSR 的聚類分析
根據Nei[22]的方法用NTSYS-pc2.10e軟件統計分析36份供試蘇、高丹草材料間的遺傳相似系數(GS),并根據遺傳相似系數進行聚類分析。36份供試材料的GS介于0.3922~0.9020,平均為0.6471。其中,遺傳相似系數最大的是29和30與29和31,達到0.9020,其次為30和31,29和32,31和32及27和35,達到0.8922,表明29、30、31和32這4份材料間及27和35這兩份材料間的親緣關系很接近。
由聚類結果(圖2)可知,在相似系數為0.61,將36份供試材料分為3大類群:1、19和20號歸為一類,1號為來源于甘肅的引進品種,19和20號均為兩份栽培品種,分別來源于江蘇和遼寧;27和35號兩份高丹草材料以及來源于內蒙古的烏拉特2號共3份材料歸為第2類群;剩余30份材料歸為第3類,這一類群在相似系數為0.69處又可歸為兩個亞類,其來源地為川渝地區的24、26和34號以及內蒙古的28號均為引進品種的4份材料歸為第一亞類,其余26份材料歸為另一亞類,根據種子庫提供的已有信息,該類材料大部分皆來源于甘肅、寧夏、內蒙古和四川地區。由于先前部分種子入庫時的登記信息不夠完善,且個別材料如登記為烏拉特2號的13號材料登記信息有待進一步的核實。針對這一情況,可通過田間試驗結合表型調查、抗性鑒定等來進一步佐證。
3 討論
轉錄組測序技術具有不依賴于物種全基因組信息就能快速、準確的獲得全轉錄本序列信息的特點,近年來已成為各研究領域一種較為基礎的研究手段。本研究基于蘇丹草轉錄組測序結果分析可知,SSR位點廣泛分布于蘇丹草RNA序列中,占檢測序列的16.82%,與鴨茅(16.43%)[23]、黨參(Codonopsispilosula)(16.1%)[24]、芒草(14.44%)[20]等的測序結果基本一致。蘇丹草轉錄組SSR種類較為豐富,一至六核甘酸重復類型均有出現,單核苷酸重復僅次于三核甘酸,其次為二核苷酸重復,其他重復類型所占比例很小,這一分布規律在高粱EST-SSR中也有體現[13]。核苷酸的重復類型因植物種類的不同而存在一定的差異,主要體現在以二核苷酸和三核苷酸重復為主導所占比例的差別上。本研究蘇丹草中三核苷酸重復所占比例最高,與Yates等[25]、Huang等[26]和Cardoso-Silva等[27]分別在紅三葉(Trifoliumpratense)、牛鞭草(Hemarthria)和甘蔗(Saccharumofficinarum)中的研究結果相一致。 由于密碼子是以三核苷酸為一個功能單位, 在一個DNA的閱讀框內三核苷酸發生遷移突變對氨基酸聚合物形成的蛋白質不會造成太大的影響[28],這可能合理的解釋了為何大多數植物中三核苷酸為最為豐富的重復類型的現象。在大多數的單子葉植物中,AG/CT和CCG/CGG分別為二核苷酸和三核苷酸的優勢重復基元[29],本研究結果也剛好印證了這一點。蘇丹草轉錄組SSR標記中單核苷酸和三核苷酸SSR重復基元的重復次數多,尤其以單核苷酸重復基元的跨度最大,在9~23次均有分布。研究表明,核苷酸的重復次數與SSR標記的多態性呈正相關[30],所以理論上這兩種核苷酸SSR具備更為豐富的多態性,可作為潛在的SSR重復基元進行引物設計和分子標記開發。
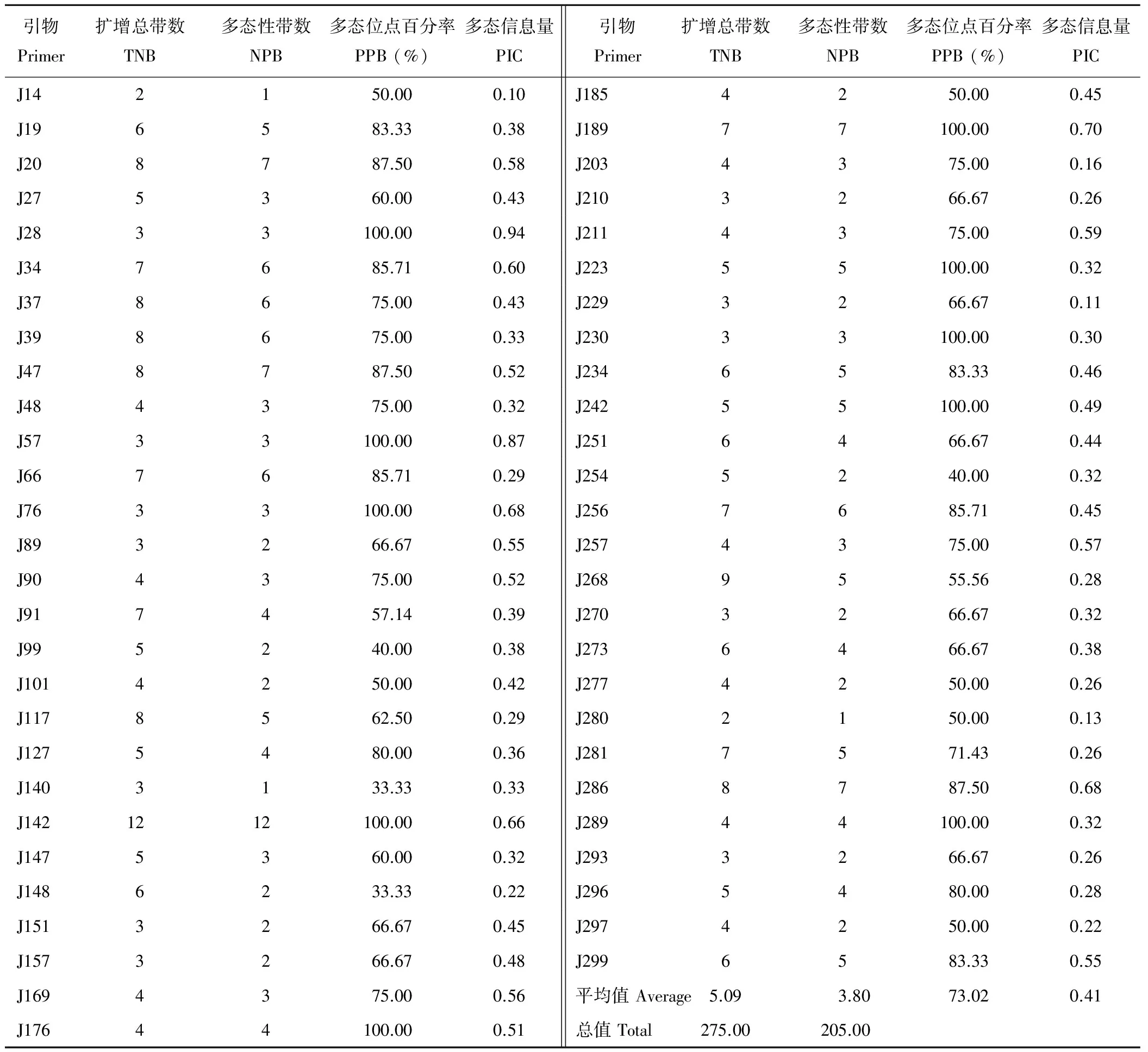
表5 54對引物擴增結果Table 5 The amplification results of 54 SSR primer pairs
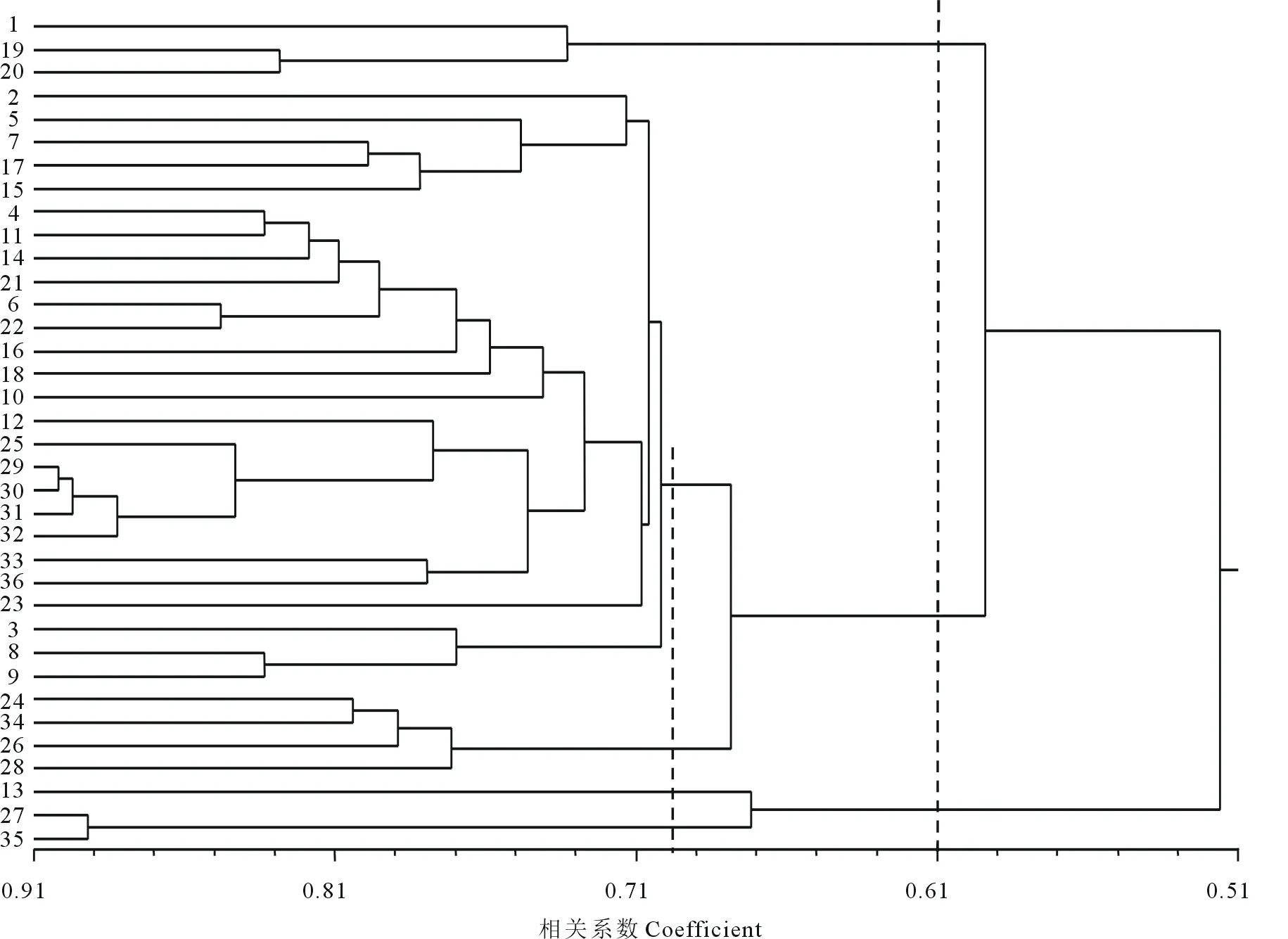
圖2 EST-SSR對36份材料的UPGMA聚類分析Fig.2 UPGMA cluster of 36 accessions based on EST-SSR markers
根據EST-SSR是對基因內部變異進行直接評價的這一特點,本研究利用包含不同重復基元的54對多態性引物應用于34份蘇丹草和2份高丹草材料中進行分子標記擴增分析,得到較多的變異位點,其中73.05%的擴增片段能夠揭示材料間的遺傳差異,均值為0.41的多態信息含量(PIC)反映了供試材料間的遺傳多樣性水平。一般來說,PIC含量越高,表明群體變異程度越大,物種的遺傳多樣性越豐富[31]。這說明基于轉錄組開發的EST-SSR對蘇丹草遺傳多樣性的檢測具有一定的有效性,同時也證明了供試材料具有較為豐富的遺傳變異類型。
基于遺傳相似系數(GS)的分析也能印證以上結論。36份供試材料的平均GS為0.6471,表明供試材料間具有較遠的親緣關系,而27和35號兩份高丹草材料其GS為0.8922,聚類結果也歸為一類,表明蘇丹草轉錄組開發的SSR引物擴增出的特異位點能夠有效地將蘇丹草與高丹草材料進行區別。另外,采自于川渝地區的29、30、31和32號4份材料間的GS值較高,且其聚類結果與地理來源基本一致,說明來自于同一地區的材料遺傳變異較小。以上結果均表明根據蘇丹草轉錄組開發的SSR標記對蘇、高丹草遺傳變異和親緣關系的鑒別能力具有一定的可行性,可有效應用于蘇、高丹草種質資源的遺傳多樣性評價中。
根據EST-SSR標記具有很好的通用性這一特點,EST-SSR引物不僅可運用于近源物種的遺傳多樣性研究,如將高粱EST-SSR標記應用于蘇丹草及高丹草[13],將高丹草EST-SSR標記應用于蘇丹草和高粱[31]等。此外,陳永霞等[32]還將玉米(Zeamays)、水稻(Oryzasativa)和高粱3種禾谷類作物的EST-SSR引物成功用于扁穗牛鞭草(Hemarthriacompressa)的跨種擴增研究其遺傳多樣性水平。本研究根據蘇丹草轉錄組數據開發大量本草種EST-SSR分子標記,通過遺傳多樣性分析揭示了所開發的引物具有良好的特異性,在這一基礎上,以后可將這54對特異性SSR引物運用到高粱、高丹草以及其他禾本科牧草、草坪草甚至是禾谷類作物等的遺傳多樣性研究工作中。由于EST-SSR來源于序列相對保守的基因組編碼區,相同引物因與不同材料的基因組互補DNA片段數目和位點而不同,擴增產物大小和數目也存在一定差異,故擴增產物的多態性反映了材料的遺傳多樣性,為基因功能多樣性的研究提供了可能[33]。因此,利用EST-SSR標記技術對草種質資源進行遺傳多樣性分析,可以更好地開發和利用優良基因資源,保護和鑒定基因的多樣性,并可通過對材料的親緣關系進行梳理[34],為雜交育種選育新品系奠定基礎,提高品種選育工作的準確性。
4 結論
本研究基于蘇丹草轉錄組測序分析明確了蘇丹草SSR位點分布的總體特征,利用SSR位點數目和種類均較為豐富的EST序列開發了54個特異的蘇丹草SSR分子標記,建立了高效開發蘇丹草特異標記的新途徑,為今后蘇丹草及近源物種遺傳組成的鑒定、遺傳多樣性研究、“三性”測試(DUS)和分子標記輔助育種奠定了重要基礎。
參考文獻References:
[1] Zhang X D, Liu J R, Ye T Y. The growth performance and cultivation techniques ofSorghumsudanense. Bulletin of Agricultural Science and Technology, 2004, (2): 38-39.
張希弟, 劉建榮, 葉挺云. 蘇丹草的生長表現及栽培技術. 農業科技通訊, 2004, (2): 38-39.
[2] Ke M, Zhu H, Liang W W,etal. Grey correlation analysis of main agronomic characters and its yield and quality traits inSorghumsudanense. Pratacultural Science, 2016, 33(5): 949-955.
柯梅, 朱昊, 梁維維, 等. 蘇丹草農藝性狀與產量、品質間的灰色關聯度分析. 草業科學, 2016, 33(5): 949-955.
[3] Xu Y P, Wu Z X, Zhao Z X. The adaptability and the developing foreground of Sudan grass in the produce of agriculture and animal husbandry in China. Pratacultural Science, 2003, (7): 23-25.
徐玉鵬, 武之新, 趙忠祥. 蘇丹草的適應性及在我國農牧業生產中的發展前景. 草業科學, 2003, (7): 23-25.
[4] Li C J, Fu Y B, Wan J C,etal. Genetic diversity of agronomic characteristics of 30Sorghumsudanensegermplasm. Pratacultural Science, 2015, 32(1): 85-93.
李陳建, 付彥博, 萬江春, 等. 30份蘇丹草種質資源農藝性狀的遺傳多樣性分析. 草業科學, 2015, 32(1): 85-93.
[5] Zhan Q W. Prospect analysis of Sorghum-sudangrass hybrids in bio-energy utilization. Seed, 2009, 28(8): 77-79.
詹秋文. 高粱-蘇丹草雜交種生物能源利用的前景分析. 種子, 2009, 28(8): 77-79.
[6] Wang J, Gao Q, Yang G F,etal. Fingerprint constructing and genetic diversity analyzing ofSorghumsudanenseandSorghumbicolor×Sorghumsudanensewith SSR markers. Acta Agrestia Sinica, 2016, 24(1): 156-164.
王杰, 高秋, 楊國鋒, 等. 國審蘇丹草和高丹草品種SSR指紋圖譜構建及遺傳多樣性分析. 草地學報, 2016, 24(1): 156-164.
[7] Wang Y W, Cao Z Z, Han J G,etal. Evaluation and cluster analysis on forage and seed performances of nineSorghumsudanensevarieties. Acta Pratacultural Sinica, 2005, (5): 117-123.
王赟文, 曹致中, 韓建國, 等. 9個蘇丹草品種生產性能的評價與聚類分析. 草業學報, 2005, (5): 117-123.
[8] Kalia R K, Rai M K, Kalia S,etal. Microsatellite markers: an overview of the recent progress in plants. Euphytica, 2011, 177(3): 309-334.
[9] Agarwal M, Shrivastava N, Padh H. Advances in molecular marker techniques and their applications in plant sciences. Plant Cell Reports, 2008, 27(4): 617-631.
[10] Luo R, Wu W L, Zhang Y,etal. SSR marker and its application to crop genetics and breeding. Genomics and Applied Biology, 2010, 29(1): 137-143.
羅冉, 吳委林, 張旸, 等. SSR分子標記在作物遺傳育種中的應用. 基因組學與應用生物學, 2010, 29(1): 137-143.
[11] Zhan Q W, Zhang T Z, Wang B H,etal. Diversity comparison and phylogenetic relationships ofS.bicolorandS.sudanenseas revealed by SSR markers. Plant Science, 2008, 174(1): 9-16.
[12] Lu J, Zhan Q W, Lu J B,etal. Development of EST-SSR maker in Sorghum and Sudangrass. Acta Laser Biology Sinica, 2009, 18(3): 389-394.
盧杰, 詹秋文, 陸景標, 等. 高粱與蘇丹草EST-SSR標記的開發. 激光生物學報, 2009, 18(3): 389-394.
[13] Li J Q, Wang L H, Zhan Q W,etal. Establishment of sorghum EST-SSR marker and its preliminary application to sudangrass. Pratacultural Science, 2010, 27(3): 112-117.
李杰勤, 王麗華, 詹秋文, 等. 高粱EST-SSR標記的建立及其在蘇丹草中的應用初探. 草業科學, 2010, 27(3): 112-117.
[14] Kane N C, Rieseberg L H. Selective sweeps reveal candidate genes for adaptation to drought and salt tolerance in common sunflower,Helianthusannuus. Genetics, 2007, 175(4): 1823-1834.
[15] Yu X Q, Bai G H, Luo N,etal. Association of simple sequence repeat (SSR) markers with submergence tolerance in diverse populations of perennial ryegrass. Plant Science, 2011, 180(2): 391-398.
[16] Nie G, Huang L K, Zhang X Q,etal. Marker-trait association for biomass yield of potential bio-fuel feedstockMiscanthussinensisfrom Southwest China. Frontiers in Plant Science, 2016, 7: 802.
[17] Huang H Y, Du H Y, Wu Y T N,etal. Development of SSR molecular markers based on transcriptome sequencing ofEucommiaulmoides. Scientia Silvae Sinicae, 2013, 49(5): 176-181.
黃海燕, 杜紅巖, 烏云塔娜, 等. 基于杜仲轉錄組序列的SSR分子標記的開發. 林業科學, 2013, 49(5): 176-181.
[18] Liu Z P, Chen T L, Ma L C,etal. Global transcriptome sequencing using the Illumina platform and the development of EST-SSR markers in autotetraploid alfalfa. PLoS One, 2013, 8(12): e83549.
[19] Huang L K, Yan H D, Zhao X X,etal. Identifying differentially expressed genes under heat stress and developing molecular markers in orchardgrass (DactylisglomerataL.) through transcriptome analysis. Molecular Ecology Resources, 2015, 15(6): 1497-1509.
[20] Nie G, Tang L, Zhang Y J,etal. Development of SSR markers based on transcriptome sequencing and association analysis with drought tolerance in perennial grassMiscanthusfrom China. Frontiers in Plant Science, 2017, 8: 801.
[21] Li J Q, Wang L H, Zhan Q W,etal. Transcriptome characterization and functional marker development inSorghumsudanense. PloS One, 2016, 11(5): 1-12.
[22] Nei M. Analysis of gene diversity in subdivided populations. Proceedings of the National Academy of Sciences, 1973, 70(12): 3321-3323.
[23] Li J, Huang L K, Jin M Y,etal. Development and verification of orchardgrass Genomic-SSR. Molecular Plant Breeding, 2017, (10): 4071-4079.
李季, 黃琳凱, 金夢雅, 等. 鴨茅基因組Genomic-SSR標記開發. 分子植物育種, 2017, (10): 4071-4079.
[24] Wang D, Cao L Y, Gao J P. Data mining of simple sequence repeats inCodonopsispilosulatranscriptome. Chinese Traditional and Herbal Drugs, 2014, 45(16): 2390-2394.
王東, 曹玲亞, 高建平. 黨參轉錄組中SSR位點信息分析. 中草藥, 2014, 45(16): 2390-2394.
[25] Yates S A, Swain M T, Hegarty M J,etal.Denovoassembly of red clover transcriptome based on RNA-Seq data provides insight into drought response, gene discovery and marker identification. BMC Genomics, 2014, 15(1): 453.
[26] Huang X, Yan H D, Zhang X Q,etal.Denovotranscriptome analysis and molecular marker development of twoHemarthriaspecies. Frontiers in Plant Science, 2016, 7: 496.
[27] Cardoso-Silva C B, Costa E A, Mancini M C,etal. De novo assembly and transcriptome analysis of contrasting sugarcane varieties. PloS One, 2014, 9(2): e88462.
[28] Kaur S, Pembleton L W, Cogan N O I,etal. Transcriptome sequencing of field pea and faba bean for discovery and validation of SSR genetic markers. BMC Genomics, 2012, 13(1): 104.
[29] Kantety R V, La Rota M, Matthews D E,etal. Data mining for simple sequence repeats in expressed sequence tags from barley, maize, rice, sorghum and wheat. Plant Molecular Biology, 2002, 48(5): 501-510.
[30] Gao L F, Tang J F, Li H W,etal. Analysis of microsatellites in major crops assessed by computational and experimental approaches. Molecular Breeding, 2003, 12(3): 245-261.
[31] Wen Y, Lu X P, Ren R,etal. Development of EST-SSR marker and genetic diversity analysis inSorghumbicolor×Sorghumsudanenes. Hereditas, 2013, 35(2): 225-232.
溫瑩, 逯曉萍, 任銳, 等. 高丹草EST-SSR標記的開發及其遺傳多樣性. 遺傳, 2013, 35(2): 225-232.
[32] Chen Y X, Zhang X Q, Xie W G,etal. Genetic diversity ofHemarthriacompressagermplasms from southwestern China based on EST-SSR markers. Acta Prataculturae Sinica, 2011, 20(6): 245-253.
陳永霞, 張新全, 謝文剛, 等. 利用EST-SSR標記分析西南扁穗牛鞭草種質的遺傳多樣性. 草業學報, 2011, 20(6): 245-253.
[33] Wang R J, Li P Y, Zhang Y H. Development ofElytrigiarepensEST-SSR marker and its application. Pratacultural Science, 2016, 33(8): 1526-1535.
王瑞晶, 李培英, 張延輝. 偃麥草EST-SSR標記開發及應用. 草業科學, 2016, 33(8): 1526-1535.
[34] Wang F, Gao Q, Wang J,etal. Analysis of genetic diversity inSorghumgermplasm collections using SSR markers. Acta Prataculturae Sinica, 2016, 25(5): 125-133.
王芳, 高秋, 王杰, 等. 利用SSR標記分析高粱屬種質資源的遺傳多樣性. 草業學報, 2016, 25(5): 125-133.
DOI:10.11686/cyxb2017262http://cyxb.lzu.edu.cn
成啟明, 格根圖, 尹強, 等. 苜蓿干草捆安全貯藏條件的研究. 草業學報, 2018, 27(5): 190-200.
Cheng Q M, Ge G T, Yin Q,etal. Effect of moisture, bale density and CaO on storage life and quality of alfalfa hay. Acta Prataculturae Sinica, 2018, 27(5): 190-200.

