高通量測序法對劍南春大曲真菌群落結構的分析
徐占成,徐姿靜,劉孟華,唐清蘭
(四川劍南春集團有限責任公司,四川綿竹618200)
曲為“酒之骨”,大曲作為我國傳統大曲酒釀造的糖化、發酵、生香劑,是我國傳統釀造的特征與精華之所在[1]。劍南春作為中國濃香型白酒的典型代表,其生產用曲采用自然接種、開放式發酵生產,導致大曲中微生物種類繁多,群系復雜,采用傳統的分離培養方法研究,將會遺漏很多微生物種類,其中甚至可能有關鍵性功能微生物[2-3]。
宏基因組學作為一種研究環境樣品中的微生物總基因組信息的新的微生物研究方法,能通過微生物總基因組信息來反映群落的生物多樣性、種群結構、功能活性、相互協作關系及與環境之間的關系[4]。該技術已在許多領域應用,在“腸道微生物”[5]“土壤微生物”[6]和“海洋微生物”[7]方向上取得了顯著成就。為認識環境微生物群落結構及篩選新基因提供了一條新的方法和思路。
前期本課題組利用宏基因組學的16S rDNA V4區高通量測序對劍南春大曲細菌多樣性進行了系統研究。為了更加深入的探索大曲糖化生香機理,本研究以劍南春大曲曲藥為研究對象,通過大曲總DNA的提取、PCR擴增及構建ITS基因文庫,對劍南春大曲真菌群落組成進行了全面研究,為進一步解析劍南春大曲的功能微生物、篩選優良釀造微生物提供堅實的理論依據。
1 材料與方法
1.1 實驗材料
劍南春集團有限公司的中溫大曲曲藥(由劍南春集團公司提供),MO BIO強力土壤DNA提取試劑盒(MO BIO Laboratories,Carlsbad,CA,USA),其他分子實驗耗材(由上海生工提供)。
1.2 實驗方法
1.2.1 基因組DNA提取
采用MO BIO強力土壤DNA提取試劑盒(MO BIO Laboratories,Carlsbad,CA,USA)對樣本的基因組DNA進行提取,之后利用瓊脂糖凝膠電泳和Nanodrop檢測DNA的純度和濃度,取適量的樣品于離心管中,使用無菌水稀釋樣品至10 ng/μL,保存在-40℃冰箱中以備后續實驗使用。
1.2.2 PCR擴增
以稀釋后的基因組DNA為模板,根據測序區域的選擇,使用帶Barcode的特異引物和高效高保真的酶(TaKaRa,Dalian)進行PCR,確保擴增效率和準確性。
引物對應區域[8]:
ITS4(5'-TCCTCCGCTTATTGATATGC-3');
gITS7F(5'-GTGARTCATCGARTCTTTG-3')。
PCR反應體系(25 μL)的組成:
10 × PCR buffer 2.5 μL,MgCl2(25 mM)6 μL,10 mM dNTP mix 1 μL,雙向引物(10 μM)2 μL,Taq DNA聚合酶(TaKaRa,Dalian)0.5 U,無菌去離子水補足至25 μL。PCR擴增程序:94℃預變性5 min;94 ℃變性30 s,56 ℃退火30 s,68 ℃延伸45 s,34個循環;72℃延伸10 min。每個樣品做2個PCR擴增平行樣,最后根據PCR產物濃度進行等量混樣。
1.2.3 PCR產物純化和定量
PCR產物使用1%濃度的瓊脂糖凝膠進行電泳檢測;根據PCR產物濃度進行等量混樣,充分混勻后使用1%的瓊脂糖凝膠電泳檢測PCR產物,對目的條帶使用生工公司提供的膠回收試劑盒(Sangon Biotech,China,Cat#SK8132)回收產物,并用Nanodrop進行濃度和質量的測定。
1.2.4 文庫構建和上機測序
使用TruSeq?DNA PCR-Free Sample Preparation Kit建庫試劑盒進行文庫構建,構建好的文庫經過Qubit和qPCR定量,文庫合格后,使用v2測序試劑盒(2×250 bp)和Miseq測序儀進行上機測序。
1.3 生物信息學分析
1.3.1 Alpha多樣性分析
Alpha Diversity用于分析樣品內(Within-community)的微生物群落多樣性,通過單樣品的多樣性分析(Alpha多樣性)可以反映樣品內的微生物群落的豐富度和多樣性,包括用稀釋曲線和一系列統計學分析指數來評估各樣品中微生物群落的物種豐富度和多樣性。
1.3.2 Beta多樣性分析
Beta多樣性指沿環境梯度不同生境群落之間物種組成的相異性或物種沿環境梯度的更替速率,也被稱為生境間的多樣性。
2 結果和分析
2.1 Alpha多樣性指數表(表1)

表1 Alpha多樣性指數(測序深度5786條序列/樣品)
從表1可以看出:3個大曲樣品的Chao1豐富度指數在37~43范圍內變化;Shannon指數(Index)在3.039~1.889范圍內變化;Simpson指數,其值的變化范圍介于0.567~0.795間。其中,最大值均出現在2號大曲,最小值由1號大曲檢測取得。三大指數檢測表明在大曲真菌多樣性方面:2號大曲>3號大曲>1號大曲。
2.2 群落組成分析
2.2.1 門水平大曲樣品群落組成圖
基于微生物ITS基因,對檢測樣品的有效序列進行聚類,以97%的序列相似性將序列在門水平上聚類成為OTU,其結果見圖1。圖1表明,在3類大曲中,Ascomycota、Zygomycota、Basidiomycota 3類微生物占絕對優勢,相對豐度總和均超過90%。其中,1號大曲中,3類微生物相對豐度總和最高,達到95.65%;而在2號大曲中,3類微生物相對豐度總和最低,為90.88%。由此可見,基于ITS基因的門水平分析結果,上述3類微生物是劍南春大曲中的優勢微生物。

圖1 大曲樣品群落組成圖(門水平)
在所研究樣品中,Ascomycota的相對豐度于30.07%~60.18%的范圍內變化。其中,最大值在1號大曲中獲得;最小值在2號大曲中獲得。利用方差分析(ANOVA)對3個不同大曲Ascomycota相對豐度的差異顯著性進行檢驗,結果表明,不同大曲中該類微生物的相對豐度差異并不顯著(p>0.05)。
從Zygomycota相對豐度值分析,其變化范圍介于28.22%~60.44%之間。其中,在2號大曲中,該類微生物的相對豐度最高,而在1號大曲中最低。而Basidiomycota相對豐度值分析發現,3個大曲相對豐度值差距不大(<0.2%)。
對3類優勢微生物的總相對豐度進行方差分析,結果發現,不同大曲間并不存在顯著差異(p>0.05)。由此可見,基于ITS基因的群落分析表明,劍南春大曲的優勢微生物,其相對豐度并無顯著差別。
2.2.2 科水平大曲樣品群落組成圖
利用數據庫聚類,在科水平上對不同大曲中微生物的種類進行分析,其結果見圖2。

圖2 大曲樣品群落組成圖(科水平)
從科的水平分析結果表明,在3類大曲中,Mucoraceae、Saccharomycopsidaceae、Trichocomaceae及Thermoascaceae 4類微生物占均對優勢,相對豐度總和均超過85%。其中,1號大曲4類微生物相對豐度總和最高,達到93.69%;而在2號大曲中,3類微生物相對豐度總和最低,為86.65%。由此可見,基于ITS基因的科水平分析結果,上述4類微生物是劍南春大曲中的優勢微生物。
從各科分析來看:2號大曲,在Mucoraceae的相對豐度值最高達到60.44%,1號和3號分別為28.22%、32.92%;1號大曲在Saccharomycopsidaceae有最大相對豐度值,為61.15%,2號大曲有最小值16.44%;而在Trichocomaceae及Thermoascaceae分布上3號大曲占據優勢。
對4類優勢微生物的總相對豐度進行方差分析,結果發現,不同大曲間并不存在顯著差異(p>0.05)。由此可見,基于ITS基因的群落科水平分析表明,劍南春大曲的優勢微生物,其相對豐度并無顯著差別。
2.2.3 屬水平大曲樣品群落組成圖
利用數據庫聚類,在屬水平對不同大曲中微生物的種類進行分析,其結果見圖3。
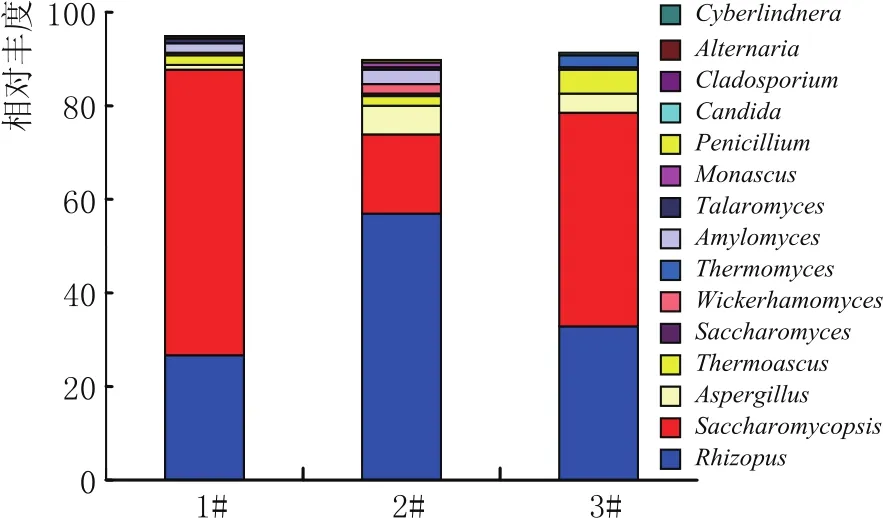
圖3 大曲樣品群落組成圖(屬水平)
根據各屬的相對豐度值,選擇數值最高的前10類微生物進行展示,分別為:Rhizopus、Saccharomycopsis、Aspergillus、Thermoascus、Saccharomyces、Wickerhamomyces、Thermomyces、Amylomyces、Talaromyces、Monascus。
在所有樣品中,Rhizopus的相對豐度值介于26.51%~57.17%之間。其中,最小值在1號大曲獲得;最大值在2號大曲獲得。方差分析(ANOVA)表明,不同大曲間的Rhizopus相對豐度并無顯著差異(p>0.05)。
Saccharomycopsis的相對豐度在16.44%~61.15%范圍內變化。其中,最小值在2號大曲獲得;最大值則在1號大曲獲得。根據方差分析(ANOVA),結果表明不同大曲間Saccharomycopsis的相對豐度并無顯著差異(p>0.05)。
Aspergillus的相對豐度在1.19%~6.22%范圍內變化。其中,最小值在1號大曲獲得;最大值則在2號大曲獲得。根據方差分析(ANOVA),結果表明不同大曲間Aspergillu的相對豐度并無顯著差異(p>0.05)。
Thermoascus在檢測大曲樣品中的相對豐度值介于1.711%~5.081%之間。其中,最小值由1號大曲獲得;最大值由3號大曲獲得。經方差分析(ANOVA),結果表明,大曲間的Thermoascus相對豐度并無顯著差異(p>0.05)。
由此可見,基于ITS基因的群落屬水平分析表明,劍南春大曲的優勢真菌群落多樣性分布在Rhizopus、Saccharomycopsis、Aspergillus、Thermoascus、Saccharomyces、Wickerhamomyces、Thermomyces、Amylomyces、Talaromyces、Monascus。這與前期采用PCR-DGGE技術研究結果相符[3]。
3 結論
劍南春大曲中真菌群落結構多樣性的研究結果表明,劍南春大曲的優勢真菌多樣性分布,從門水平說集中在Ascomycota、Zygomycota、Basidiomycota,相對豐度總和超過90%;從科水平說集中在Mucoraceae、Saccharomycopsidaceae、Trichocomaceae及Thermoascaceae,4類微生物相對豐度總和超過80%;從屬水平說主要分布在Rhizopus、Saccharomycopsis、Aspergillus、Thermoascus、Saccharomyces、Wickerhamomyces、Thermomyces、Amylomyces、Talaromyces、Monascus。
霉菌、酵母是白酒生產不可或缺的優良菌種,霉菌能產生豐富的葡萄糖淀粉酶,因而可以將原料中的淀粉直接降解為酵母菌可利用的還原糖,進而刺激酵母菌的生長,同時促進原料中的碳源向發酵終端產物酒精的代謝。胡承等[9]對濃香型大曲制曲過程中微生物的變化規律進行了研究,其結果表明,出房大曲中霉菌主要是根霉和曲霉。高亦豹[10]通過對不同工藝大曲酵母26S rRNA區基因DGGE圖譜分析發現,扣囊復膜孢酵母、異常威克漢姆酵母普遍存在于大曲中。本研究利用高通量測序法分析優勢菌種與他們的結果相符,而又更為全面,是對劍南春濃香型大曲真菌群落較為客觀真實的解析。
參考文獻:
[1]沈怡方.白酒生產技術全書[M].北京:中國輕工業出版社,2009:46-48.
[2]高亦豹,王海燕,徐巖.利用PCR-DGGE未培養技術對中國白酒高溫和中溫曲藥細菌群落結構的分析[J].微生物學通報,2010(7):999-1004.
[3]徐占成,唐清蘭,劉孟華,等.劍南春大曲曲藥真菌群落結構的分析[J].釀酒,2015,42(4):13-16.
[4]唐婧,蘇迪,徐小蓉,等.基于宏基因組學的茅臺酒酒曲細菌的多樣性分析[J].貴州農業科學,2014,42(11):180-183.
[5]YOUSSEF N,SHEIK C S,KRUMHOLZ L R,et al.Comparison of species richness estimates obtained using nearly complete fragments and simulated pyrosequencing-generated fragments in 16S rRNA genebased environmental surveys[J].Applied and environmental microbiology,2009,75(16):5227-5236.
[6]LANG K S,ANDESON J M,SCHWARZ S,et al.Novel florfenicol and chloramphenicol resistance gene discovered in Alaskan soil by using functional metagenomics[J].Applied and environmental microbiology,2010,76(15):5321-5326.
[7]KENNEDY J,FLEMER B,JACKSON S A,et al.Marine metagenomics:new tools for the study and exploitation of marine microbial metabolism[J].Marine drugs,2010,8(3):608-328.
[8]BOKULICH N A,MILLS D A.Improved selection of internal transcribed spacer-specific primers enables quantitative,ultra-high-throughput profiling of fungal communities[J].Applied and environmental microbiology,2013,79(8):2519-2526.
[9]胡承,鄔捷鋒,沈才洪,等.濃香型(瀘型)大曲的研究及其應用[J].釀酒科技,2004(1):33-36.
[10]高亦豹.聚合酶鏈式反應-變性梯度電泳技術(PCRDGGE)研究中國白酒大曲中微生物群落結構[D].無錫:江南大學,2010.

