未分化甲狀腺癌相關基因的生物信息學分析
趙靜,張家明,杜雅瑩,李興睿
(華中科技大學同濟醫學院附屬同濟醫院,武漢430000)
甲狀腺癌是最常見的內分泌惡性腫瘤,約占所有內分泌相關惡性腫瘤的95%[1],按其惡性程度可分為分化良好的甲狀腺癌(WDTC)、低分化甲狀腺癌(PDTC)和未分化甲狀腺癌(ATC)。多數甲狀腺癌發展緩慢,患者整體預后較好。ATC是惡性程度最高的甲狀腺癌,發病率約占甲狀腺癌總發病率的2%[2],其特點為發病突然、進展迅速和預后極差,確診時多已出現周圍組織器官侵犯,15%~50%的患者伴肺、骨、腦、肝等遠處轉移[3],患者中位生存期僅3~6個月,5年生存率低于10%[4]。由于ATC失分化而無攝碘能力,其生長不受促甲狀腺激素的影響,因而放射性131I治療和促甲狀腺激素抑制治療均無明顯效果[5],此外,大部分ATC難以完全切除,且對放化療敏感性較低。因此,分子靶向治療有望成為ATC治療的發展方向之一。生物信息學是一門結合分子生物學與計算機信息技術的新型交叉學科,基因表達譜芯片是一種高效率、大規模獲取基因表達數據的技術。應用生物信息學的方法分析基因表達譜芯片,可實現在全基因組水平上進行大規模、高通量、高靈敏度、高精確度的基因序列測定及功能研究。本研究通過對美國國家生物信息中心(NCBI)GEO數據庫中的ATC基因表達譜芯片進行挖掘,篩選得到ATC與正常甲狀腺組織的差異表達基因(DEGs),對差異基因進行功能富集、蛋白-蛋白相互作用網絡構建等生物信息學分析,初步預測靶向作用于差異基因的關鍵轉錄因子和microRNA,進一步在ATC細胞系與正常甲狀腺組織細胞系表達譜芯片中驗證關鍵DEGs和轉錄因子的表達,初步從分子水平探究ATC發病機制、尋找潛在的ATC治療靶點。
1 材料與方法
1.1芯片來源 基因表達譜芯片來源于美國國家生物信息中心(NCBI)的GEO數據庫(https://www.ncbi.nlm.nih.gov/geo/),3組芯片均為全基因組RNA表達譜芯片,序列號分別為GSE65144、GSE53072和GSE9115。GSE65144由von Roemeling CA等提交,研究包括12例人類ATC組織樣本及與之配對或非配對的13例正常甲狀腺組織樣本。GSE53072由Pita等提交,研究包括5例人類ATC組織樣本(3例樣本來自術中冰凍切片,3例樣本來自細針穿刺組織),3例配對的正常甲狀腺冰凍切片樣本和1例人類正常甲狀腺組織RNA雜交池樣本。GSE9115由Salvatore G等提交,研究包括5例人類ATC組織樣本和4例正常甲狀腺組織樣本。
1.2數據處理及差異基因篩選從GEO數據庫中下載原始芯片表達譜數據,使用R軟件(version 3.3.3)及R Bioconductor軟件的affty、limma、ggplot2、VennDiagram等軟件包,對表達譜數據進行質量控制、背景校正、標準化和數據匯總,使用經驗貝葉斯分析方法篩選出ATC和正常甲狀腺組織的差異表達基因。選取差異倍數(FC)和校正后P作為篩選指標,以|log FC|>1且adjustP<0.05作為篩選標準,分別選取3組芯片數據中表達差異顯著的基因合并取交集后,得到267個差異基因作為研究對象。
1.3差異基因富集分析及蛋白相互作用網絡分析將267個差異基因導入在線富集分析數據庫DAVID(https://david.ncifcrf.gov),分別從生物學過程、分子功能、細胞組分三個方面進行在線功能富集GO分析,應用在線數據庫KEGG(http://www.genome.jp/kegg/pathway.html )對差異基因進行生物學信號通路富集分析。通過STRING在線分析平臺(https://string-db.org )構建蛋白-蛋白相互作用網絡(PPI),分析差異基因編碼的蛋白質之間的相互作用關系,應用Cytoscape軟件(version 3.5.0)及其插件CytoHubba和MCODE對PPI網絡進行可視化和分析,計算單個蛋白質節點與其他節點間的相互作用關系強度,評估蛋白質間相互作用關系的密切程度,并創建PPI子網絡。
1.4轉錄因子-差異基因調節網絡分析和microRNA-mRNA調節網絡分析使用數據庫WebGstalt(http://www.webgestalt.org/option.php )預測microRNA與差異基因轉錄后mRNA的靶向作用關系,構建microRNA-mRNA靶向作用網絡,預測ATC中發揮關鍵作用的microRNA。
1.5ATC細胞系中潛在靶點基因表達的驗證從GEO數據庫中下載原始芯片表達譜數據集GSE85457,該芯片由Weinberger 等提交,樣本包括四個ATC細胞系和三個正常甲狀腺細胞系。通過R軟件(version 3.3.3)及R Bioconductor軟件的affty、limma軟件包和Affymetrix Expression Console Software(verversion1.4),使用經驗貝葉斯分析方法計算ATC細胞系和正常甲狀腺細胞系的基因表達水平,得到基因表達差異熱圖,并與ATC組織中預測得到的重要DEGs和關鍵轉錄因子的基因表達水平進行對比驗證,進一步證實潛在靶點基因在ATC中的表達水平,提高預測的可信度。
2 結果
2.1篩選的差異基因3組芯片差異基因分別篩選取交并集后共有273個表達差異基因,其中共同表達上調基因108個,共同表達下調基因159個,共267個表達差異基因。
2.2富集分析和蛋白相互作用網絡分析 DAVID數據庫GO富集分析結果顯示,差異基因主要參與細胞分裂、細胞增殖、調節細胞周期檢查點、有絲分裂中的核分裂和胞質分裂、DNA復制、細胞缺氧反應、細胞間黏附作用、上皮-間質轉化等生物學過程,差異基因主要與細胞質、外泌體、細胞膜、細胞中心體、著絲粒等細胞組分有關,其主要分子功能為ATP結合、蛋白質同源二聚化、蛋白絲氨酸/蘇氨酸激酶活性等。KEGG信號通路分析顯示,差異基因富集得到的信號通路主要有細胞周期、吞噬、精氨酸和脯氨酸代謝和HIF-1信號通路。通過對差異基因編碼的蛋白質進行STRING在線分析、Cytoscape軟件可視化處理,得到包含179個蛋白質節點和561條互作線的蛋白-蛋白互作(PPI)網絡。在PPI網絡中,有一部分蛋白質節點與其他節點有較多連接,稱之為中心節點(HUB)。使用Cytoscape插件CytoHubba計算蛋白節點間相互作用關系,得到相互作用關系度最高的10個中心節點, BUB1、CCNB2、AURKA、CCNA2、BUB1B、CDC6、CDH1、KIF23、CENPA和KIF2C。
2.3轉錄因子調節網絡分析和microRNA-mRNA調節網絡分析通過在線數據庫WebGstalt預測靶向作用于差異基因的轉錄因子和作用于差異基因mRNA的microRNA,Cytoscape可視化處理后得到轉錄因子-基因調節網絡(圖1)和microRNA-mRNA調節網絡(圖1),其中轉錄因子E2F、NFY、HFH4和E2F1DP2與差異基因相互作用關系密切,可能是ATC中調節差異基因表達的關鍵轉錄因子。microRNA15a、microRNA15b、microRNA16、microRNA195、microRNA424和microRNA497等與18個差異基因mRNA存在相互作用關系,可能通過調節基因轉錄后的mRNA水平,在ATC發生發展中發揮重要作用。
2.4ATC細胞系中潛在靶點基因的表達驗證 ATC組織中預測得到的潛在靶點基因,包括重要的DEGs(BUB1、CCNB2、AURKA、CCNA2、BUB1B、CDC6、CDH1、KIF23、CENPA、KIF2C)和關鍵轉錄因子E2F、NFY。在ATC細胞系中,重要的DEGs表達水平與其在ATC組織中的表達水平一致,均呈表達上調,轉錄因子E2F1、E2F2、E2F7、E2F6在ATC細胞系中表達均上調,轉錄因子NFY在ATC細胞系中的表達無統計學差異。進一步驗證了ATC與正常甲狀腺組織中預測所得潛在靶點基因的表達。
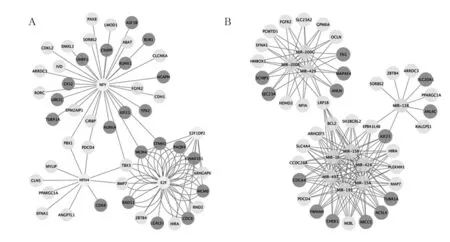
注:A:轉錄因子調節網絡;B:microRNA調節網絡。
圖1轉錄因子-基因調節網絡
3 討論
本研究通過生物信息學的方法篩選得到267個ATC與正常甲狀腺組織的差異基因,其中表達上調基因108個,表達下調基因159個。基因富集分析發現,差異基因主要參與細胞分裂增殖、細胞間黏附作用和上皮-間質轉化等生物學過程。細胞異常分裂和增殖失控是腫瘤發生發展過程中的關鍵環節,而細胞間黏附降低和上皮-間質轉化則在腫瘤細胞的遷移和侵襲過程中起到重要作用。上皮-間質轉化是腫瘤細胞以鵝卵石樣上皮表型向狹長狀成纖維細胞樣轉換并獲得間質表型過程,不僅參與腫瘤的遷移和侵襲,且與細胞抗衰老、抗凋亡和腫瘤細胞耐藥、耐放療等密切相關[6]。KEGG富集分析表明,差異基因主要涉及2條腫瘤相關通路:細胞周期相關通路和HIF-1信號通路。由于腫瘤細胞惡性增殖速率過快,局部血管網無法為瘤體提供充足氧氣從而形成了局部缺氧的微環境[7]。HIF-1是轉錄調節因子,通過調控一系列與缺氧有關的基因轉錄,使癌細胞克服缺氧環境。另外,HIF-1促進選擇性的線粒體自噬,誘導多能性腫瘤干細胞生成,促進上皮-間質轉化,降低E-cadherin表達等,從而促進腫瘤的生長和進展[8]。
通過對差異基因編碼的蛋白質之間的相互作用進行分析,構建出以BUB1、CCNB2、AURKA、CCNA2、BUB1B、CDC6、CDH1、KIF23、CENPA、KIF2C等為中心節點構成的蛋白-蛋白互作網絡。BUB1是一種絲氨酸/蘇氨酸激酶,調控細胞周期和染色體內聚,在有絲分裂中起到核心作用。BUB1還具有調控TGF-β通路的重要作用,正常情況下TGF-β是腫瘤抑制因子,BUB1與TGF-β受體結合導致TGF-β轉變為腫瘤促進因子,刺激正常細胞異常增殖進而惡變,促進腫瘤細胞的增殖和轉移[9]。CCNB2是細胞周期蛋白家族的一員,在細胞分裂的G2/M期進展中起關鍵作用,研究發現CCNB2在膀胱癌[10]、非小細胞肺癌[11]等惡性腫瘤中均為高表達。PPI第1個子網絡中,表達上調的蛋白PBK、KIFC1、TTK、NEK2、KIF2C、CHEK1、AURKA、CDC7、BUB1均屬于細胞分裂蛋白激酶家族,在細胞增殖和細胞周期調控中有重要作用,蛋白激酶過度激活可引起細胞異常增殖分化。第2個子網絡中,表達上調的蛋白NUSAP1、ZWILCH、CENPI、CENPF、CENPA、TPX2、ZWINT、MCM6均與細胞有絲分裂過程中染色體、紡錘體等細胞器相關,表明與正常細胞相比,ATC存在異常旺盛的細胞分裂和增殖。
轉錄因子可與基因特定序列靶向結合,調節目的基因的時間空間表達及表達強度。轉錄因子調節網絡中,轉錄因子E2F在差異基因的轉錄調控中起到重要作用。轉錄因子E2F在調控細胞周期進程方面具有重要作用,通過調控G1/S期DNA合成所需的時相基因控制細胞周期過程。WebGstalt在線數據庫是基于網絡的整合數據挖掘系統,可預測靶向結合并調節差異基因轉錄的轉錄因子,構建轉錄因子-mRNA靶向作用網絡,預測ATC中發揮關鍵作用的轉錄因子。研究表明,E2F與腫瘤發生發展、血管新生、轉移等有關,與乳腺癌預后密切相關[12],因此ATC發病可能與E2F轉錄調節活性失調有關。microRNA為一類轉錄后水平的調控因子,通過作用于基因轉錄出的mRNA來調節目的基因表達。microRNA-mRNA調節網絡中,microRNA-15家族參與調節凋亡、細胞周期調控、代謝等,在腫瘤發生發展中起重要作用[13]。
綜上所述,通過對ATC芯片數據的生物信息學分析,初步預測基因BUB1、CCNB2、AURKA、CCNA2、BUB1B、CDC6、CDH1、KIF23、CENPA、KIF2C及轉錄因子E2F和microRNA-15家族有望成為ATC分子靶向治療潛在靶點。
參考文獻:
[1] 孫嘉偉,許曉君,蔡秋茂,等.中國甲狀腺癌發病趨勢分析[J].中國腫瘤,2013,22(9):690-693.
[2] Yun H, Tao Y, Li X, et al. Bioinformatics analysis of key genes and latent pathway interactions based on the anaplastic thyroid carcinoma gene expression profile[J]. Oncology Letters, 2016,13(1):167-176.
[3] Are C, Shaha AR. Anaplastic thyroid carcinoma: biology, pathogenesis, prognostic factors, and treatment approaches[J]. Ann Surg Oncol, 2006,13(4):453-464.
[4] Xu B, Ghossein R. Genomic Landscape of poorly Differentiated and Anaplastic Thyroid Carcinoma[J]. Endocrine Pathology, 2016,27(3):205-212.
[5] 陳靜,何霞云.甲狀腺未分化癌的治療進展[J].中國癌癥雜志,2014,24(4):310-315.
[6] Nieto MA. The Ins and Outs of the Epithelial to Mesenchymal Transition in Health and Disease[J]. Annu Rev Cell Dev Biol, 2010,27(1):347-376.
[7] Soni S, Padwad YS. HIF-1 in cancer therapy: two decade long story of a transcription factor[J]. Acta Oncologica, 2017,56(4):503.
[8] Ajdukovic J. HIF-1 - a big chapter in the cancer tale[J]. Experimental Oncology, 2016,38(1):9.
[9] Nyati S, Schinskesebolt K, Pitchiaya S, et al. The kinase activity of the Ser/Thr kinase BUB1 promotes TGF-β signaling[J]. Sci Signal, 2015,8(358):ra1.
[10] Lei CY, Wang W, Zhu Y T, et al. The decrease of cyclin B2 expression inhibits invasion and metastasis of bladder cancer[J]. Urologic Oncology, 2015,34(5):237.e1.
[11] Qian X, Song X, He Y, et al. CCNB2 overexpression is a poor prognostic biomarker in Chinese NSCLC patients[J]. Biomed Pharmacother, 2015,74:222-227.
[12] Johnson J, Thijssen B, Mcdermott U, et al. Targeting the RB-E2F pathway in breast cancer[J]. Oncogene, 2011,35(37):4829-4835.
[13] 劉麗鳳,王禹.microRNA-15家族功能研究進展[J].生理學報,2012,64(1):101-106.

