云南主要貿易類干巴菌遺傳多樣性分析*
周 汐,馮云利,陳正啟,余金鳳,吳素蕊,羅 瑞,桂明英
(中華全國供銷合作總社昆明食用菌研究所,云南 昆明 650223)
干巴菌是屬于擔子菌門(Basidiomycota) 蘑菇綱(Agaricomycetes) 革菌目 (Thelephorales) 革菌科(Thelephoraceae) 革菌屬(Thelephora) 的一類可食用真菌[1]。作為云南珍貴的野生食用菌,干巴菌憑借其獨特的風味和豐富的營養成為云南貿易類野生菌市場中不可或缺的一員[2]。
在云南地區,傳統廣義上的干巴菌包括革菌屬的6個種,蓮座革菌(Thelephora vialisSchwein)、干巴菌(Thelephora ganbajunM.Zang)、橙黃革菌(Thelephora aurantiotinctaCorner)、淡褐革菌 [Thelephora fuscella(Ces.)Lloyd]、日本革菌(Thelephora japonicaYasuda) 和掌狀革菌 [Thelephora palmate(Scop.)Fr.]。常見的主要有4種,蓮座革菌(T.vialis)、干巴菌 (T.ganbajun)、橙黃革菌 (T.aurantiotincta) 和掌狀革菌(T.palmate),主要產地為昆明、玉溪、曲靖、楚雄、普洱、麗江、保山和大理,分布區域大約位于北緯 22°~27°,東經 99°~106°,海拔800m~2200m的范圍內[3-4]。干巴菌僅在滇中及鄰近地區有食用習慣,且作為一種共生真菌,其生長條件復雜,導致干巴菌的人工栽培難以實現,因此,國內外對此類真菌的研究成果不如其他菌類豐富,主要涉及分類鑒定、分離純化、營養成分測定、生態學研究幾個方面,在分子生物學方面有部分研究但相對較少[5]。
本文通過對2015年至2016年,采集于野生環境中的34份貿易類干巴菌樣品進行遺傳多樣性分析,以期了解貿易類干巴菌的種類、分布以及數量,從分子水平為云南貿易類干巴菌資源的開發與保護提供一定的理論依據。
1 材料和方法
1.1 試驗材料
自2015年至2016年,從云南貿易類干巴菌8個主產地中的普洱市、曲靖市馬龍縣、保山市、玉溪市元江縣、玉溪市峨山縣、玉溪市易門縣、玉溪市新平縣、大理州洱源縣、大理州祥云縣、楚雄市、楚雄州武定縣、昆明市富民縣、昆明市嵩明縣、昆明市宜良縣和昆明市尋甸縣采集貿易類干巴菌樣品40份。采集的樣本用硅膠干燥后備用。
1.2 試劑與儀器
1.2.1主要試劑
CTAB,TaKaRa Taq,ITS1/ITS4 引物,RV-M/M13-47引物,TaKaRa膠回收試劑盒,pMD18-T載體,大腸桿菌DH5α感受態細胞,Amp,IPTG,X-gal。
1.2.2主要儀器
凝膠成像儀,Gensnap公司;常溫高速離心機,SCILOGEX公司;PCR儀,ABI公司;電泳儀,六一生物科技有限公司;紫外透射儀,Benchtop公司;微型分光光度計,Nano Drop公司。測序服務由北京擎科新業生物技術有限公司提供。
1.3 試驗方法
1.3.1形態學鑒定
通過觀察形態特征和顯微結構,按照傳統分類方法,參考黃年來、卯曉崗[6-7]對于革菌屬各個種的形態描述,初步確定每份樣品的屬種名。
1.3.2DNA提取
去除樣品表面的泥土等雜質后用無菌去離子水和75%的酒精對樣品進行清洗并粗略切割,吸干樣品表面液體后將樣品置于經過高溫消毒的研缽中,加入液氮將樣品研磨至粉末,采用CTAB法提取樣品的DNA保存備用。
1.3.3PCR
利用真菌ITS通用引物對ITS1/ITS4對樣品進行PCR擴增,設置反應體系為Buffer 5μL,dNTP 4 μL,引物各1μL,Taq酶0.5μL,DNA模板1μL,無菌去離子水補足至50μL。設置PCR儀擴增程序為95℃預變性5 min;94℃變性30 s,55℃退火40 s,72℃延伸1min,35個循環;72℃延伸7min。
1.3.4克隆
使用1%的瓊脂糖凝膠電泳對樣品的PCR擴增產物進行檢測,割膠后使用膠回收試劑盒對含有目的DNA片段的回收膠條進行純化。將純化產物連接到pMD18-T載體上,轉化入大腸桿菌DH5α感受態細胞,使用含有Amp/IPTG/X-gal的LB平板篩選陽性單克隆,挑取單克隆于0.8mL含Amp的LB液體培養基中,37℃、200 r·min-1振蕩培養 14 h~16 h。
1.3.5測序
使用pMD18-T載體上的引物對RV-M/M13-47對菌液進行菌落PCR驗證,使用1%的瓊脂糖凝膠電泳對PCR產物進行檢測,選擇含有目的條帶的菌液保存。將PCR驗證有目的條帶的陽性克隆送公司進行測序。
1.3.6序列分析
使用DNASTAR軟件包中的SeqMan程序,對照測序峰圖對序列進行手動校對,去除序列兩端多余的堿基,以確保序列的準確性。通過Blast在線工具(http://www.ncbi.nlm.nih.gov/Blast.cgi)進行比對,將測序獲得的序列同GenBank中已錄入的干巴菌(T.ganbajun) 以及革菌科(Thelephoraceae) 其他種序列進行比對。
1.3.7系統進化樹的構建
以銀耳(Tremella fucifomis) 作為外群,使用MAGA5.05軟件中的系統分析法(phylogeny)對序列進行系統關系分析,采用鄰接法(neighbor-joining method,NJ) 構建系統發育樹。系統樹的每個分枝的統計學顯著性分析以Boot strap方法進行檢驗,重復次數為1000次,顯著性低于50%時,數值不在進化樹中顯示。
1.3.8遺傳距離的計算
將測得的序列和從GenBank中通過BLAST檢索獲得的參考序列進行多重對位排列,用MAGA5.05軟件中的Kimura-2-parameter(K2P) 模型計算遺傳距離,構建距離矩陣。
1.3.9遺傳多樣性分析
使用軟件DnaSP 5.10對來自5個地理群的25個同種樣品進行變異位點、單倍型數量、單倍型多樣性指數和核苷酸多樣性指數的統計。
2 結果和分析
2.1 ITS序列鑒定結果
對經形態學鑒定為貿易類干巴菌的34個樣品的ITS序列進行BLAST比對,結果列于表1。
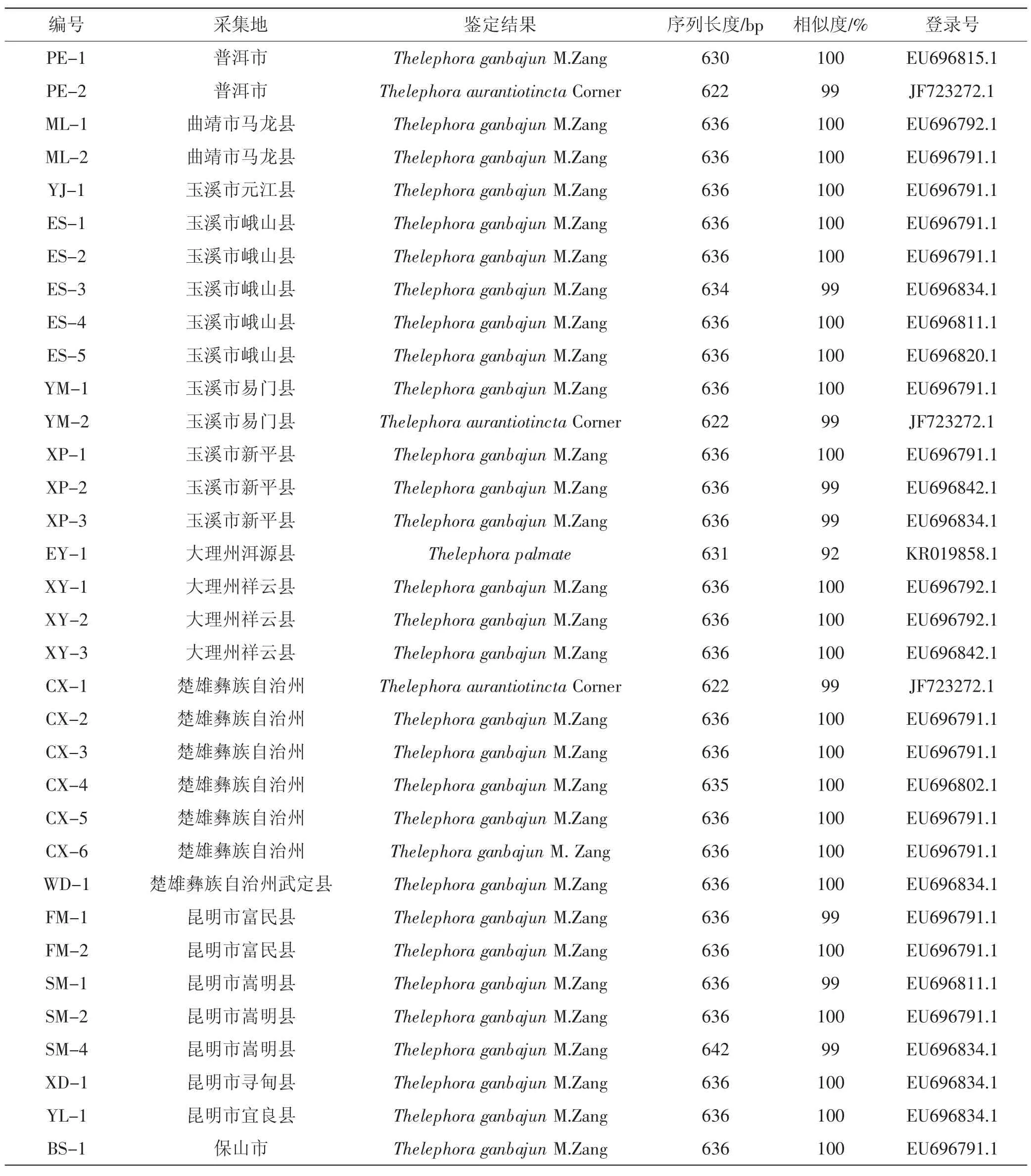
表1 ITS鑒定結果Tab.1 ITS identification result
在對測定序列進行BLAST比對的過程中,發現3個樣品在GenBank數據庫中只能比對到屬名(Thelephorasp.),為了確定其種名,根據文獻溯源,發現該種最初由Yagame等[8]在對小舌唇蘭與其伴生真菌的混合營養模式的研究中被測序并登錄,登錄號為JF723272.1。同時,由于食用菌數量廣且形態多樣,利用單一的手段對某個菌種進行鑒定會造成假陽性[9-11],影響后續研究,因此在本文的研究中,通過對采集的樣品進行宏觀形態鑒定和顯微結構觀察(圖1),將形態鑒定與ITS序列鑒定相結合,確定PE-2為橙黃革菌(T.aurantiotincta)。
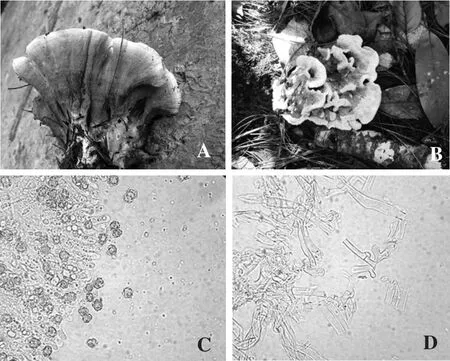
圖1 PE-2形態鑒定Fig.1 Morphological identification of PE-2
在上述結論的基礎上,由表1可知,34份樣品的ITS序列長度在640 bp左右,其中PE-2、YM-2和CX-1為橙黃革菌(T.aurantiotincta),EY-1以92%的相似率比對到掌狀革菌(T.palmate),其余30份樣品均為干巴菌(T.ganbajun)。
從地域分布看,昆明市周圍縣級地區8份樣品均為干巴菌(T.ganbajun);玉溪市11份樣品以干巴菌(T.ganbajun) 為主,且于易門縣采集到1份橙黃革菌(T.aurantiotincta);大理市的4份樣品除采集于洱源縣的1份掌狀革菌(T.palmate) 外,其他均為干巴菌(T.ganbajun);楚雄州7份樣品中發現一份橙黃革菌(T.aurantiotincta) 其余6份為干巴菌(T.ganbajun)。而普洱市的兩份樣品中1份為干巴菌(T.ganbajun),另一份為橙黃革菌(T.aurantiotincta)。曲靖市的2份樣品均為干巴菌(T.ganbajun)。
2.2 系統進化樹分析
樣品的系統進化樹分析見圖2。
由圖2可知,EY-1與登錄號為KR019858.1的掌狀革菌聚為一支且與其他樣品親緣關系較遠;其次CX-1和YM-2與登錄號為JF723272.1的1個革菌屬序列親緣關系較近且與其他樣品親緣關系相對較遠;其余樣品基本聚為一支,但仍呈現出一定的個體差異。
2.3 干巴菌遺傳多樣性分析
干巴菌(T.ganbajun)樣品的遺傳多樣性分析如表2所示。

圖2 基于ITS的云南主要貿易類干巴菌樣品間的NJ系統樹Fig.2 NJphylogenetic tree ofmain trading Thelephora samples in Yunnan based on ITS

表2 干巴菌遺傳多樣性分析Tab.2 Genetic diversity analysis of Thelephora ganbajun M.Zang
表2數據顯示,5個地區的干巴菌均顯示出一定的遺傳多樣性,尤其玉溪的樣品最為明顯,單倍型數、多態位點數最多,以9個樣品達到了0.722的單倍型多樣性。
2.4 貿易類干巴菌遺傳距離分析
主要貿易類干巴菌樣品間的遺傳距離見表3。

從表3可以看出,通過遺傳距離的計算得知,云南地區貿易類干巴菌的總體遺傳距離范圍為0.000~0.165;樣品EY-1和YM-2之間的遺傳距離與EY-1和CX-1之間的遺傳距離均達到了0.165,表明橙黃革菌(T.aurantiotincta) 與掌狀革菌(T.palmate)之間存在明顯的種間差異;EY-1與其他干巴菌(T.ganbajun) 樣品的遺傳距離分別能達到0.126、0.114、0.116、0.110、0.136 和 0.130,說明掌狀革菌(T.palmate) 與干巴菌(T.ganbajun) 之間存在較大的種間差異;YM-2/CX-1和其他干巴菌(Thelephora ganbajun) 樣品之間的遺傳距離范圍在0.091~0.111,說明掌狀革菌(T.palmate) 與干巴菌(T.ganbajun) 之間存在種間差異;YM-2與CX-2的遺傳距離有0.005,說明橙黃革菌(T.aurantiotincta) 種內存在一定差異;其他干巴菌(T.ganbajun) 樣品相互之間的遺傳距離范圍在 0.000~0.047,0.047的遺傳距離存在于SM-1和CX-4之間,說明云南各地干巴菌(T.ganbajun)種內也有顯著差異。
3 結論與討論
本文從云南8個貿易類干巴菌主產地采集了34份樣品,覆蓋了15個區縣。通過ITS序列分析,確認34份樣品中有30份樣品為干巴菌(T.ganbajun),說明云南地區貿易類干巴菌以干巴菌(T.ganbajung)為主要種。除干巴菌外,掌狀革菌(T.palmate) 和橙黃革菌(T.aurantiotincta)亦有分布但資源較為稀缺。說明云南地區貿易類干巴菌具有一定的多樣性,但本次研究中體現出多樣性不豐富。
系統發育樹及遺傳距離分析顯示,干巴菌(T.ganbajun)、掌狀革菌(T.palmate) 和橙黃革菌(T.aurantiotincta)分散為3支,且干巴菌的一個大支分為了3個小支,但與地域差異關聯不明顯,種間有明顯差距,種內有一定差距。
在本次研究中,掌狀革菌(T.palmate) 樣品EY-1采集于云南省大理州洱源縣的針闊混交林,片段長度為631 bp,以92%的相似性比對到NCBI數據庫中登錄號為KR019858.1的序列。另外3份橙黃革菌(T.aurantiotincta) 樣品中,PE-2采集自云南省普洱市倚象鎮石膏箐的混交林,片段長度為622,以99%的相似率比對到JF723272.1;YM-2采集自云南省玉溪市易門縣的松純林,片段長度為622,以99%的相似率比對到JF723272.1;CX-1采集自楚雄彝族自治州的針闊混交林,片段長度為622,同樣以99%的相似率比對到JF723272.1。參考前人的研究[12-14],本文認為從生物多樣性角度出發,這兩種菌種產量較小且分布零星,應加大保護力度并嘗試進行原生境菌種栽培方面的探索。蓮座革菌(T.vialis)、淡褐革菌(T.fuscella) 和日本革菌(T.japonica) 3個種在本次研究中并未發現,期望在今后的試驗中通過擴大采樣區域、采樣密度,增加樣品數量得以發現。
參考文獻:
[1]馮云利,郭相,楊珍福,等.革菌屬干巴菌研究進展[J].食用菌,2016(4):1-3.
[2]周彤燊,任瑋.云南可食的革菌屬真菌的分類研究[J].云南植物研究,1986,8(3):295-297.
[3]桂明英,劉蓓,朱萍,等.干巴菌生態學初步研究[J].西南農業學報,2005,8(3):325-327.
[4]郭安,胡慧蓉,趙鳳喜,等.云南優特野生食用菌-干巴菌的開發利用[J].西南林學院學報,1998,18(2):106-108.
[5]Corner EJH.A monograph ofThelephora(Basidiomycetes)[M].Cambridge:Verlag Von J.Cramer,1968.
[6]黃年來.中國大型真菌原色圖鑒[M].北京:中國農業出版社,1998.
[7]卯曉嵐.中國大型真菌[M].鄭州:河南科學技術出版社,2000.
[8]Takahiro Y,TakamichiO,Marc-Andre S,et al.Mixotrophy ofPlatantheraminor,an orchid associated with ectomycorrhizalforming Ceratobasidiaceae fungi[J].New Phytologist,2012,193(1):178-187.
[9]朱洪慶,王盼盼,張萌,等.rDNA-ITS序列分析法與傳統分類法相結合在真菌鑒定中的應用[J].2016,西華師范大學學報:自然科學版,37(3):264-268.
[10]燕勇,李衛平,高雯潔,等.rDNA-ITS序列分析在真菌鑒定中的應用[J].中國衛生檢驗雜志,2008,18(10):1958-1961.
[11]陳劍山,鄭服叢.ITS序列分析在真菌分類鑒定中的應用[J].安徽農業科學,2007,35(13):3785-3786,3792.
[12]Sha T,Xu JP,Malliya.Genetic diversity of the endemic gourmetmushroomThelephora ganbajunfrom south-western China[J].Microbiology,2008,154(11):3460-3468.
[13]Wang PF,Zhang Y,Sha TS,et al.The completemitochondrial genome of the edible Basidiomycete mushroomThelephora ganbajun[J].Mitochondrial DNA Part B,2017,2(1):103-105.
[14]Wang PF,Sha T,Zhang YR,et al.Frequent heteroplasmy and recombination in themitochondrial genomes of the basidiomycetemushroomThelephora ganbajun[J].Scientific R-eports,2017,7(1):1626.

