CD163雙等位基因編輯豬的制備及傳代
魏迎輝,劉志國,徐奎,Evanna Huyhn ,Paul Dyce ,李繼良,周偉良,董樹仁,馮保亮,牟玉蓮,Julang Li 2,,李奎
(1中國農業科學院北京畜牧獸醫研究所,中國北京 100193;2佛山科學技術學院,中國廣東佛山528000;3Department of Animal BioSciences, University of Guelph, Canada Ontario N1G 2W1;4天津市寧河原種豬場,中國天津 301504)
0 引言
【研究意義】豬繁殖與呼吸綜合征(porcine reproductive and respiratory syndrome,PRRS)是由豬繁殖與呼吸綜合征病毒(porcine reproductive and respiratory syndrome virus , PRRSV)引起的以妊娠母豬早產、晚期流產、死胎、弱胎和木乃伊胎等繁殖障礙及仔豬和生長豬的呼吸系統疾病為主要特征的一種高致死性的傳染病[1-2]。因該病在臨床上表現為耳部皮膚紫紺,所以又被稱為“藍耳病”。根據抗原性,基因組及致病性的差異,PRRSV可分為2個型,即歐洲型和美洲型,又被稱為1型和2型[3-5]。PRRS于1987年首次在美國被發現,緊接著于1989 年在歐洲爆發,并逐漸向世界其他地區擴散,其在世界范圍內的頻繁爆發對養豬業造成了極大的經濟損失[6-7]。自20世紀90年代初期,科學家們通過表型選擇、免疫遺傳標記輔助選擇等途徑開展了很多PRRSV的抗病育種工作,也取得了很大的進展,但并未從根本上杜絕疾病的發生。PRRSV在與機體互作時,能夠抑制天然免疫、延遲中和抗體的產生,再加上抗病性狀屬于多基因性狀,PRRSV基因組易發生變異,導致國內外市面上的疫苗效果并不理想。隨著分子育種手段的不斷發展和PRRSV致病機理的逐漸清楚,利用遺傳操作手段制備對 PRRSV引起的傳染性疾病具有抗性的動物育種新材料對畜牧業的發展具有重要意義。【前人研究進展】PRRSV在體內主要通過侵染豬肺泡巨噬細胞(porcine alveolar macrophages,PAM)表面的受體而發揮作用[8]。研究發現,硫酸乙酰肝素(heparin sulphate, HS)、唾液酸粘附素(sialoadhesin, Sn)和CD163(cluster of differentiation 163)分子是PAM上存在的能與PRRSV結合的3個重要受體分子[9]。其中,CD163是一種富含半胱氨酸的清道夫受體,屬于典型的Ⅰ型糖基化蛋白,也是一種巨噬細胞分化的抗原,因其分子大小為130kDa,故又被稱為M130蛋白[10]。有研究表明,在PRRSV非易感細胞系(BHK-21和PK-15)中轉染表達CD163分子可以使這些細胞系感染PRRSV并在細胞內產生子代病毒粒子,而抗人CD163的抗體可以阻斷 PRRSV 的感染,表明 CD163 是該病毒的必需受體[11]。VAN GORP等研究發現 CD163蛋白結構域scavenger receptor cysteine-rich domain 5(SRCR5)由第7外顯子所編碼,是病毒感染細胞所必需的,而氨基端的4個scavenger receptor cysteine-rich(SRCR)區域和胞質尾部是非必需的[12]。近幾年,科學家們成功通過敲除或者替換宿主內源的受體基因來達到抗PRRSV的目的。WHITWORTH等利用CRISPR/Cas9技術敲除豬內源CD163基因,活體攻毒試驗證明敲除豬具有抵抗1型和2型PRRSV的能力[5,13-14]。隨后,WELLS等獲得用人的同源基因CD163-like的SRCR8區域替換豬CD163的SRCR5區域的基因修飾豬,攻毒試驗發現該基因替換豬具有抵抗1型PRRSV和降低感染2型PRRSV的能力[5]。最近,BURKARD等獲得了刪除豬CD163的SRCR5結構域的基因編輯豬,細胞水平試驗證明,其巨噬細胞具有抵抗1型和2型PRRSV的表型,同時該編輯豬在標準飼養條件下生長發育正常[15]。另外,LI等發現 siRNA能夠引起特異性的抗病毒反應后,其通過攻毒實驗證明重組腺病毒介導的RNAi可以使豬抵抗PRRSV 的感染[16]。LU等制備的過表達histone deacetylase6(HDAC6)轉基因豬能夠阻礙病毒復制,延緩發病時間,減弱發病癥狀,并表現出對PRRSV的抗性[17]。【本研究切入點】目前,盡管關于CD163基因編輯豬已有研究報道,然而利用體細胞核移植技術獲得無篩選標記的 CD163雙等位基因編輯純合豬的研究國內未見報道。【擬解決的關鍵問題】利用CRISPR/Cas9基因編輯技術結合體細胞核移植制備CD163雙等位基因編輯豬,既無外源篩選標記引入同時又可以快速擴繁后代,可獲得具有抵抗PRRSV能力的育種新材料。
1 材料與方法
本研究于2015年9月到2017年12月在中國農業科學院北京畜牧獸醫研究所豬基因工程與種質創新團隊實驗室和天津市寧河原種豬場完成。
1.1 材料
試驗用到的細胞為中國農業科學院北京畜牧獸醫研究所豬基因工程與種質創新團隊 2013年分離的純種大白豬胎兒成纖維細胞,CRISPR/Cas9載體為pX330-U6-hSpCas9載體。
1.2 方法
1.2.1 豬CD163基因gRNA設計和載體構建 首先鎖定編碼豬CD163基因SRCR5結構域的第7外顯子的打靶區域,利用麻省理工學院張峰實驗室開發的網站(http://crispr.mit.edu)對其打靶區域進行評分,從候選的打靶位點中選擇一個合適的 gRNA,其序列為GGAAACCCAGGCTGGTTGGA。根據gRNA序列合成互補配對的寡聚核苷酸,合成的寡聚核苷酸序列如下:CD163-gRNA-F:caccGGAAACCCAGGCTGGTT GGA,CD163-gRNA-R:aaacTCCAACCAGCCTGGGT TTCC。載體構建步驟為:(1)將兩條寡聚核苷酸稀釋至 10μmol·L-1后各取 10μL, 98℃反應 10min,在自然冷卻至室溫的條件下對其進行退火。(2)37℃條件下,用限制性內切酶 Bbs I 對含有 Cas9序列的pX330骨架載體酶切2h,切膠回收線性化片段。(3)回收的線性化載體骨架與退火的寡聚核苷酸 16℃連接 1h,隨后轉化 Top10或 DH5α感受態細胞,涂布于含氨芐的LB平板進行生長,挑取單菌落擴大培養并測序,測序引物為U6-FWD。序列正確后進行擴大培養,用去內毒素大提質粒試劑盒提取pX330-CD163-gRNA質粒,所提的質粒純化后用于細胞的轉染。
1.2.2 轉染效率檢測和gRNA活性檢測 轉染前一天將原代大白豬胎兒成纖維細胞復蘇至6cm平皿中,當細胞達到70%—80%匯合度時即可進行細胞轉染。細胞分兩組進行轉染,一組轉染表達EGFP的質粒,另一組轉染pX330-CD163-gRNA質粒,轉染步驟嚴格按照 Basic Primary Fibroblasts Nucleofector Kit(Lonza)試劑盒說明書進行操作。轉染后 48h,熒光顯微鏡下觀察轉EGFP組的細胞發光情況用以檢測細胞的轉染效率。轉pX330-CD163-gRNA質粒48h后的細胞部分用于鋪板,另一部分用以提取基因組檢測gRNA活性。以提取的細胞基因組為模板,用Pre mixTaq DNA聚合酶進行PCR,擴增出300bp片段,引物為CD163-F:5′- AAGCCCACTGTAGGCAGAA-3′和 CD163-R:5′-GTGGTTTCCCTCCTGGGG-3′。擴增條件為 95℃,5min;95℃,30s;61℃,30s,36個循環;72℃,30s;72℃,10min;PCR產物送北京天一輝遠公司測序,根據靶位點附近序列的套峰情況即可對gRNA的活性進行檢測。
1.2.3 單克隆細胞培養 將電轉 48h后細胞鋪板于10cm培養皿中,每3d更換一次培養液。鋪板后10d左右可以觀察到合適大小的克隆點,將單克隆細胞進行擴大培養,同時取部分細胞提取基因組用以基因型鑒定。
1.2.4 單克隆細胞基因型鑒定 以提取的細胞基因組為模板進行PCR擴增,PCR按照1.2.2的條件進行操作。PCR產物送北京天一輝遠公司測序,根據測序結果,篩選具有移碼突變的純合編輯細胞用作核移植的供體細胞。
1.2.5 體細胞核移植及克隆豬基因型鑒定 以體外成熟40h的青年豬卵母細胞為核移植受體細胞,將核移植供體細胞移入去核的卵母細胞,經電融合與激活,構建成重組克隆胚胎,挑選發育狀態良好的克隆重組胚胎用手術法移入自然發情的經產大白母豬子宮內進行妊娠。胚胎移植后,技術人員注意觀察其返情情況,定期用B型超聲波檢查受體母豬妊娠情況。小豬出生后剪取部分耳樣,提取基因組后進行PCR和測序鑒定。
1.2.6 克隆豬的擴繁及 F1代仔豬基因型鑒定CD163雙等位基因編輯公豬與野生型大白母豬交配,擴繁后代并定期檢查交配母豬的妊娠情況。F1代仔豬出生后剪取部分耳樣,提取基因組后進行PCR和T-A克隆測序,鑒定其基因型。
2 結果
2.1 豬CD163基因gRNA設計及活性檢測
針對大白豬的第7外顯子序列設計了一條gRNA(圖 1-A),構建好的載體轉染胎兒成纖維細胞,靶位點PCR測序檢測gRNA活性。與未轉染質粒組對比,轉染 pX330-CD163-gRNA質粒組靶位點下游出現套峰(圖 1-B),可說明部分細胞靶位點附近序列處造成了堿基的隨機插入或缺失,說明 pX330-CD163-gRNA能夠對靶位點進行切割。
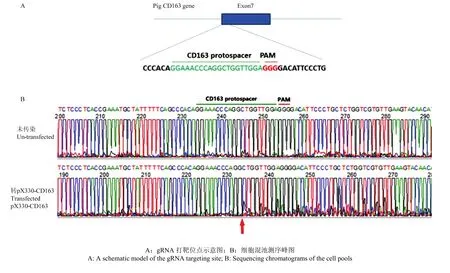
圖1 gRNA靶位點設計和打靶效率檢測Fig. 1 Design of gRNA and detection of the target efficiency
2.2 細胞轉染效率檢測
將表達 EGFP的質粒轉染豬胎兒成纖維細胞,48h后熒光顯微鏡下觀察細胞發光情況,結果顯示大部分細胞能夠表達 EGFP蛋白(圖 2),表明轉染效率較高。
2.3 單克隆細胞培養及基因型鑒定
通過PCR和測序鑒定獲得的127個單克隆細胞基因型,統計結果顯示共有21個細胞克隆的CD163基因發生移碼突變,突變效率為16.5%,其中有14個克隆(11.0%)出現單等位基因突變或雙等位基因雜合突變,7個克隆(5.5%)為雙等位基因純合突變(圖3-A)。7個雙等位基因純合突變克隆的基因型如圖3-B所示。
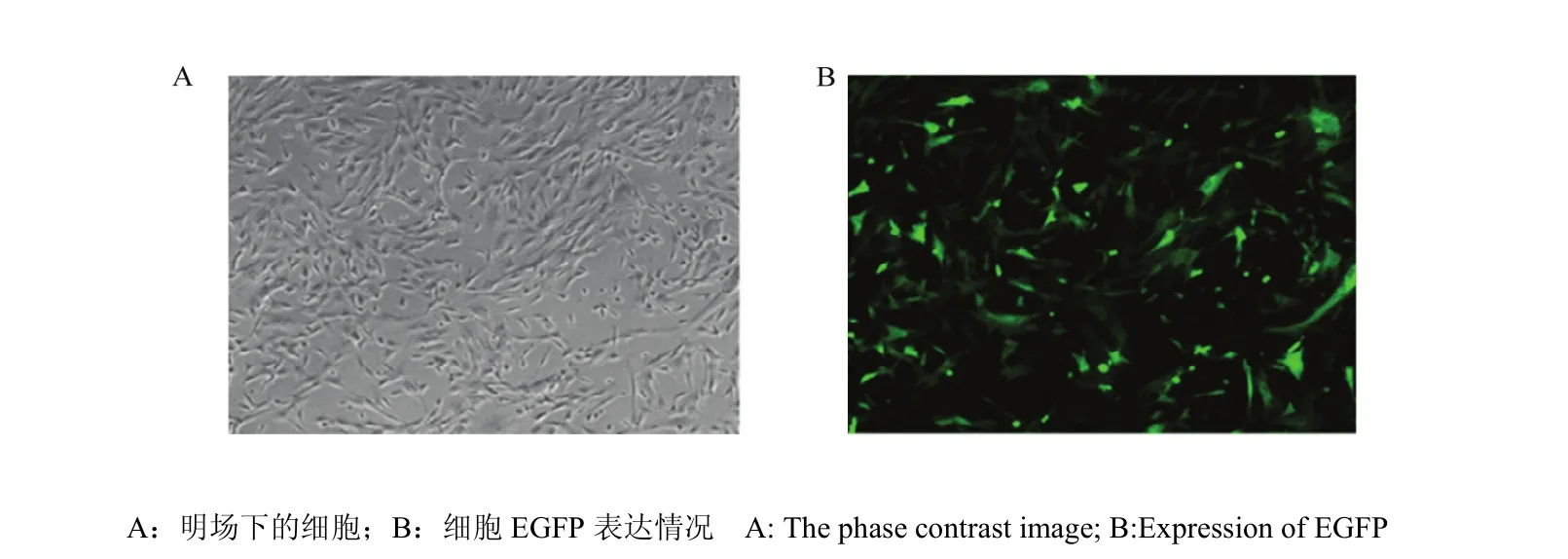
圖2 轉染效率檢測Fig. 2 Detection of cell transfection efficiency
2.4 克隆豬的基因型檢測及蛋白序列預測
選擇 CD163-34號細胞克隆(-50bp/-50bp)作為核供體進行體細胞核移植,移植受體母豬3頭,成功獲得CD163雙等位基因純合編輯的大白豬。采取豬耳組織進行基因型鑒定,PCR和測序結果顯示該豬CD163基因型與供體細胞克隆基因型一致,均為雙鏈缺失50bp(圖4-A、B)。CD163基因編輯豬缺失50bp后,第7外顯子缺失16個氨基酸,同時發生移碼突變并提前終止(圖4-C)。

圖3 克隆基因型鑒定Fig. 3 Detection ofclone genotyping
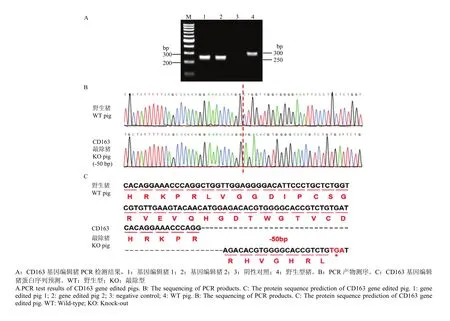
圖4 克隆豬基因型鑒定及蛋白序列預測Fig. 4 Detection of cloned pigs genotyping and prediction of protein sequence
2.5 基因編輯豬的種系傳代
CD163雙等位基因編輯公豬與野生型大白母豬繁殖獲得15頭F1代仔豬,其中5頭公豬,10頭母豬,仔豬平均出生重為 1.21kg(圖 5-A)。提取 F1代仔豬耳緣組織 DNA,進行 PCR擴增,對PCR產物進行T-A克隆并測序鑒定其基因型,結果表明為 CD163基因雜合(WT/-50bp)型(圖5-B、C)。
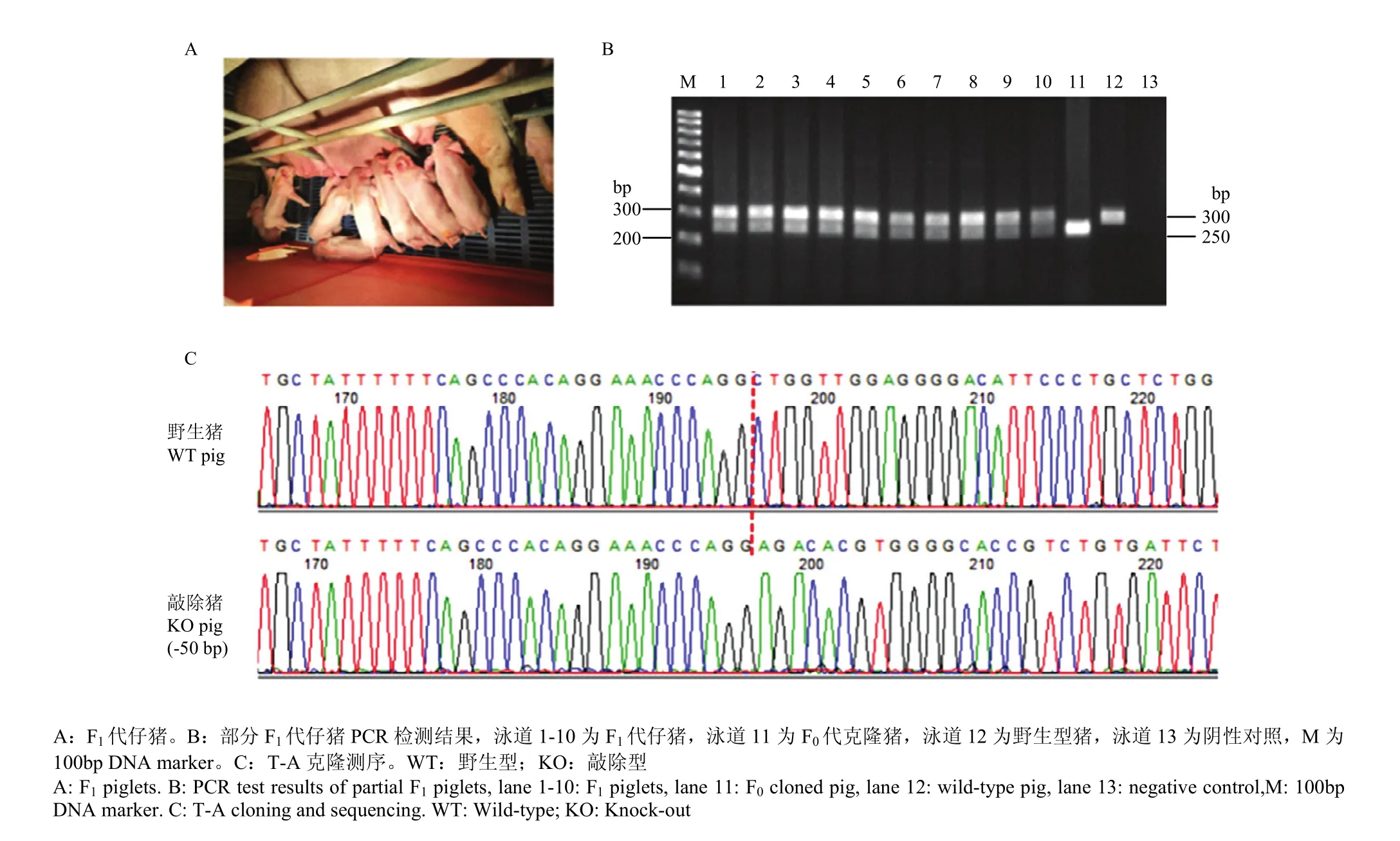
圖5 F1代仔豬基因型鑒定Fig. 5 Detection of F1 piglets genotyping
3 討論
CRISPR/Cas9 技術介導的基因突變是指 Cas9蛋白通過sgRNA靶向識別并定點切割基因組DNA,導致DNA雙鏈斷裂(double strand break, DSB),細胞通過非同源末端連接修復機制修復 DSB時所引發的切割位點處 DNA堿基的插入或缺失[18]。II型CRISPR/Cas系統已經在各物種中得到了較為廣泛的應用,如:小鼠[19-21]、大鼠[22]、豬[23-25]、牛[26-27]、羊[28]、猴子[29]、斑馬魚[30]、果蠅[31]、線蟲[32]等。與基因打靶、ZFN、TALEN等基因修飾技術相比,CRISPR/Cas9具有操作簡單、效率高等諸多優勢,利用CRISPR/Cas9對重要經濟性狀基因進行修飾已經成為家畜新品種培育的重要手段[33]。
最近幾年,科學家們通過CRISPR/Cas9技術已成功獲得抗 PRRSV的基因編輯豬[5,13-15]。密蘇里大學Prather團隊通過細胞水平和個體水平攻毒試驗發現制備的 CD163基因敲除豬具有同時抵抗 1型和 2型PRRSV的能力,而用人的同源基因 CD163-like的SRCR8區域替換豬CD163的SRCR5區域制備的基因替換豬具有抵抗1型PRRSV的能力,其對2型PRRSV易感性降低,但與對照相比差異不明顯[5,13]。愛丁堡大學ARCHIBALD團隊通過刪除CD163基因SRCR5區域獲得的基因敲除豬,細胞水平攻毒試驗發現其PAM細胞不受1型和2型PRRSV的感染,這一研究成果同 PRATHER團隊的結論一致[15]。本研究利用CRISPR/Cas9技術對PRRSV受體基因CD163進行修飾,用構建好的pX330-CD163-gRNA載體轉染豬胎兒成纖維細胞并檢測gRNA活性,結果表明在無任何篩選標記富集的情況下,pX330-CD163-gRNA能夠有效的對靶位點進行識別和切割。考慮到CRISPR/Cas9的高效編輯特性和篩選標記殘留引起的安全問題,本研究中細胞單克隆的篩選沒有使用藥物、流式等細胞富集方法,但是仍然得到了 21個移碼突變的細胞單克隆,整體突變效率達 16.5%(21/127),其中 7個細胞克隆為雙等位基因純合突變,效率達到5.5%。本研究利用CRISPR/Cas9基因編輯技術結合體細胞核移植技術成功制備了 CD163雙等位基因編輯的純合大白豬,并通過擴繁獲得了后代。綜合分析首批F1代基因編輯仔豬的出生數據,未發現CD163基因編輯對豬繁殖性能有不利的影響。該研究獲得的基因編輯豬不攜帶篩選標記,能夠安全地為培育抗 PRRSV的新品種豬提供育種材料,同時可為進一步闡明藍耳病發病機制的研究奠定堅實的基礎。
4 結論
本研究利用CRISPR/Cas9基因編輯技術結合體細胞核移植技術,成功制備出CD163雙等位基因純合編輯大白豬,并已獲得CD163F1代基因編輯豬,安全并快速地為培育抗PRRSV新品種豬提供了育種新材料。
[1] NIEDERWERDER M C, BAWA B, SERAO N V, TRIBLE B R,KERRIGAN M A, LUNNEY J K, DEKKERS J C, ROWLAND R R.Vaccination with a porcine reproductive and respiratory syndrome(PRRS) modified live virus vaccine followed by challenge with PRRS virus and porcine circovirus type 2 (PCV2) protects against PRRS but enhances PCV2 replication and pathogenesis compared to results for nonvaccinated cochallenged controls. Clinical and Vaccine Immunology:CVI, 2015, 22(12): 1244-1254.
[2] WENSVOORT G, TERPSTRA C, POL J M, TER LAAK E A,BLOEMRAAD M, DE KLUYVER E P, KRAGTEN C, VAN BUITEN L, DEN BESTEN A, WAGENAAR F, ET AL. Mystery swine disease in The Netherlands: The isolation of Lelystad virus.Veterinary Quarterly, 1991, 13(3): 121-130.
[3] NELSEN C J, MURTAUGH M P, FAABERG K S. Porcine reproductive and respiratory syndrome virus comparison: divergent evolution on two continents. Journal of Virology, 1999, 73(1): 270-280.
[4] ALLENDE R, LEWIS T L, LU Z, ROCK D L, KUTISH G F, ALI A,DOSTER A R, OSORIO F A. North American and European porcine reproductive and respiratory syndrome viruses differ in non-structural protein coding regions. Journal of General Virology, 1999, 80(2):307-315.
[5] WELLS K D, BARDOT R, WHITWORTH K M, TRIBLE B R,FANG Y, MILEHAM A, KERRIGAN M A, SAMUEL M S,PRATHER R S, ROWLAND R R R. Replacement of porcine CD163 scavenger receptor cysteine-rich domain 5 with a CD163-Like homolog confers resistance of pigs to genotype 1 but not genotype 2 porcine reproductive and respiratory syndrome virus. Journal of Virology, 2017, 91(2): e01521-01516.
[6] NEUMANN E J, KLIEBENSTEIN J B, JOHNSON C D, MABRY J W, BUSH E J, SEITZINGER A H, GREEN A L, ZIMMERMAN J J.Assessment of the economic impact of porcine reproductive and respiratory syndrome on swine production in the United States.Journal of the American Veterinary Medical Association, 2005, 227(3):385-392.
[7] ZHOU L, YANG H. Porcine reproductive and respiratory syndrome in China. Virus Research, 2010, 154(1-2): 31-37.
[8] JUSA E R, INABA Y, KOUNO M, HIROSE O. Effect of heparin on infection of cells by porcine reproductive and respiratory syndrome virus. American Journal of Veterinary Research, 1997, 58(5): 488-491.
[9] CALVERT J G, SLADE D E, SHIELDS S L, JOLIE R, MANNAN R M, ANKENBAUER R G, WELCH S K. CD163 expression confers susceptibility to porcine reproductive and respiratory syndrome viruses. Journal of Virology, 2007, 81(14): 7371-7379.
[10] VAN GORP H, DELPUTTE P L, NAUWYNCK H J. Scavenger receptor CD163, a Jack-of-all-trades and potential target for celldirected therapy. Molecular Immunology, 2010, 47(7-8): 1650-1660.
[11] PATTON J B, ROWLAND R R, YOO D, CHANG K O. Modulation of CD163 receptor expression and replication of porcine reproductive and respiratory syndrome virus in porcine macrophages. Virus Research, 2009, 140(1-2): 161-171.
[12] VAN GORP H, VAN BREEDAM W, VAN DOORSSELAERE J,DELPUTTE P L, NAUWYNCK H J. Identification of the CD163 protein domains involved in infection of the porcine reproductive and respiratory syndrome virus. Journal of Virology, 2010, 84(6):3101-3105.
[13] WHITWORTH K M, ROWLAND R R, EWEN C L, TRIBLE B R,KERRIGAN M A, CINO-OZUNA A G, SAMUEL M S, LIGHTNER J E, MCLAREN D G, MILEHAM A J, WELLS K D, PRATHER R S.Gene-edited pigs are protected from porcine reproductive and respiratory syndrome virus. Nature Biotechnology, 2016, 34(1): 20-22.
[14] WHITWORTH K M, LEE K, BENNE J A, BEATON B P, SPATE L D,MURPHY S L, SAMUEL M S, MAO J, O'GORMAN C, WALTERS E M, MURPHY C N, DRIVER J, MILEHAM A, MCLAREN D,WELLS K D, PRATHER R S. Use of the CRISPR/Cas9 system to produce genetically engineered pigs from in vitro-derived oocytes and embryos. Biology of Reproduction, 2014, 91(3): 78.
[15] BURKARD C, LILLICO S G, REID E, JACKSON B, MILEHAM A J, AIT-ALI T, WHITELAW C B, ARCHIBALD A L. Precision engineering for PRRSV resistance in pigs: Macrophages from genome edited pigs lacking CD163 SRCR5 domain are fully resistant to both PRRSV genotypes while maintaining biological function. PLoS Pathogens, 2017, 13(2): e1006206.
[16] LI G, JIANG P, LI Y, WANG X, HUANG J, BAI J, CAO J, WU B,CHEN N, ZESHAN B.Inhibition of porcine reproductive and respiratory syndrome virus replication by adenovirus-mediated RNA interference both in porcine alveolar macrophages and swine.Antiviral Research, 2009, 82(3): 157-165.
[17] LU T, SONG Z, LI Q, LI Z, WANG M, LIU L, TIAN K, LI N.Overexpression of Histone deacetylase 6 enhances resistance to porcine reproductive and respiratory syndrome virus in pigs. PLoS One, 2017, 12(1): e0169317.
[18] CONG L, RAN F A, COX D, LIN S, BARRETTO R, HABIB N,HSU P D, WU X, JIANG W, MARRAFFINI L A, ZHANG F.Multiplex genome engineering using CRISPR/Cas systems. Science,2013, 339(6121): 819-823.
[19] MIZUNO S, DINH T T, KATO K, MIZUNO-IIJIMA S, TANIMOTO Y, DAITOKU Y, HOSHINO Y, IKAWA M, TAKAHASHI S,SUGIYAMA F, YAGAMI K. Simple generation of albino C57BL/6J mice with G291T mutation in the tyrosinase gene by the CRISPR/Cas9 system. Mammalian Genome, 2014, 25(7-8): 327-334.
[20] WANG H, YANG H, SHIVALILA C S, DAWLATY M M, CHENG A W, ZHANG F, JAENISCH R. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell, 2013, 153(4): 910-918.
[21] FUJIHARA Y, IKAWA M. CRISPR/Cas9-based genome editing in mice by single plasmid injection. Methods in Enzymology, 2014, 546:319-336.
[22] GUAN Y, SHAO Y, LI D, LIU M. Generation of site-specific mutations in the rat genome via CRISPR/Cas9. Methods in Enzymology,2014, 546: 297-317.
[23] HAI T, TENG F, GUO R, LI W, ZHOU Q. One-step generation of knockout pigs by zygote injection of CRISPR/Cas system. Cell Rresearch, 2014, 24(3): 372-375.
[24] RUAN J, LI H, XU K, WU T, WEI J, ZHOU R, LIU Z, MU Y, YANG S, OUYANG H, CHEN-TSAI R Y, LI K. Highly efficient CRISPR/Cas9-mediated transgene knockin at the H11 locus in pigs. Scientific Reports, 2015, 5(14253).
[25] HUANG L, HUA Z, XIAO H, CHENG Y, XU K, GAO Q, XIA Y,LIU Y, ZHANG X, ZHENG X, MU Y, LI K. CRISPR-Cas9-mediated ApoE-/-and LDLR-/-double gene knockout in pigs elevates serum LDL-C and TC levels. Oncotarget, 2017, 8(23): 37751-37760.
[26] WEHNES C A, REHBERGER T G, BARRANGOU R, SMITH A H.Short communication: determination of salmonella clustered regularly interspaced short palindromic repeats (CRISPR) diversity on dairy farms in Wisconsin and Minnesota. Journal of Dairy Science, 2014,97(10): 6370-6377.
[27] GAO Y, WU H, WANG Y, LIU X, CHEN L, LI Q, CUI C, LIU X,ZHANG J, ZHANG Y. Single Cas9 nickase induced generation of NRAMP1 knockin cattle with reduced off-target effects. Genome Biology, 2017, 18(1): 13.
[28] CRISPO M, MULET A P, TESSON L, BARRERA N, CUADRO F,DOS SANTOS-NETO P C, NGUYEN T H, CRENEGUY A,BRUSSELLE L, ANEGON I, MENCHACA A. Efficient generation of myostatin knock-out sheep using CRISPR/Cas9 technology and microinjection into zygotes. PLoS One, 2015, 10(8):e0136695.
[29] KANG Y, ZHENG B, SHEN B, CHEN Y, WANG L, WANG J, NIU Y,CUI Y, ZHOU J, WANG H, GUO X, HU B, ZHOU Q, SHA J, JI W,HUANG X. CRISPR/Cas9-mediated Dax1 knockout in the monkey recapitulates human AHC-HH. Human Molecular Genetics, 2015,24(25): 7255-7264.
[30] VARSHNEY G K, PEI W, LAFAVE M C, IDOL J, XU L,GALLARDO V, CARRINGTON B, BISHOP K, JONES M, LI M,HARPER U, HUANG S C, PRAKASH A, CHEN W, SOOD R,LEDIN J, BURGESS S M. High-throughput gene targeting and phenotyping in zebrafish using CRISPR/Cas9. Genome Research,2015, 25(7): 1030-1042.
[31] GRATZ S J, UKKEN F P, RUBINSTEIN C D, THIEDE G,DONOHUE L K, CUMMINGS A M, O'CONNOR-GILES K M.Highly specific and efficient CRISPR/Cas9-catalyzed homologydirected repair in Drosophila. Genetics, 2014, 196(4): 961-971.
[32] ZHENG J, JIA H, ZHENG Y. Knockout of leucine aminopeptidase in Toxoplasma gondii using CRISPR/Cas9. International Journal for Parasitology, 2015, 45(2-3): 141-148.
[33] BARRANGOU R, DOUDNA J A. Applications of CRISPR technologies in research and beyond. Nature Biotechnology, 2016,34(9): 933-941.

