桑樹谷氨酸脫氫酶基因MaGDHs的克隆及表達分析
植爽,任艷紅,唐星,徐鳳翔,王傳宏,2,趙愛春,王茜齡
(1西南大學生物技術學院,重慶 400715;2安徽農業大學生命科學學院,合肥 230036)
0 引言
【研究意義】桑樹是最早的栽培林木之一,從海南島到黑龍江,桑樹有廣泛的分布和利用。長期以來,桑樹都是以養蠶為目的而栽培,其根、皮、枝、葉和富含次生代謝產物的桑果也已被用于保護肝臟,提高視力,促進排尿,改善便秘,降低血壓等[1-3]。此外,桑樹根系發達、耐貧瘠,抗旱、耐鹽堿等抗逆性強[4]。氮代謝在桑樹生長發育、化合物的合成和抗逆過程中起著重要作用,谷氨酸脫氫酶(GDH)普遍存在于植物中,在碳、氮代謝中既能催化銨與α-酮戊二酸合成谷氨酸,又能催化谷氨酸的氧化同時釋放銨。目前對其生理作用的認識還不清楚,因此研究桑樹 GDH的特點對闡述其生理作用具有重要意義。【前人研究進展】谷氨酸脫氫酶(GDH)曾被認為是在高等植物中銨同化的關鍵酶[5],自 1974年谷氨酰胺合成酶(glutamine synthetase,GS)和谷氨酸合酶(glutamate synthase,GOGAT)被發現以后,GS/GOGAT循環被公認為高等植物銨同化的關鍵酶[6],GDH催化α-酮戊二酸與NH4+合成谷氨酸被認為是除GS/GOGAT途徑外的另外一種NH4+同化途徑。此外,GDH存在于線粒體中,GS存在胞質中,因此谷氨酸脫氫酶被認為對光呼吸過程產生的 NH3再同化起著關鍵作用,但是用GS抑制劑磷絲菌素預處理或者缺乏GS2或Fd-GOGAT的突變體進行試驗表明,在光呼吸釋放銨的再同化過程中,GS2的作用是主要的[7-8]。MASCLAUXDAUBRESSE等[9]通過15N的銨鹽和谷氨酸示蹤研究證實,無論任何葉齡,銨鹽的同化在黑暗和光照的條件下都是經過GS/GOGAT途徑,不依靠GDH途徑,而谷氨酸分解為 α-酮戊二酸和銨鹽的過程中,GDH在起著重要作用。WANG等[10]研究表明,小麥在低濃度的鹽脅迫下,GS的活性增加,在高濃度的鹽脅迫下,GDH的酶活性增加,GS的酶活性降低。MIYASHITA等報道[11]擬南芥的兩個 GDH表達具有組織特異性,受到細胞內碳源調控,并且認為GDH在C缺乏條件下在氨基酸酸降解中起著重要作用。水稻OsGDHs基因家族成員在氮和磷脅迫下表達量不同[12]。蔗糖饑餓處理會誘導羽扇豆的GDH酶活性增加,Cd2+和Pb2+導致銨鹽積累,同時伴隨著 GDH酶活性增加,這些結果表明GDH主要在逆境條件下在C、N代謝中具有重要功能[13]。近幾年來,國內外涉及到的研究 GDH的物種越來越多,也越來越深入,主要集中在模式植物[14-16]和農作物[17-19]中進行 GDH 克隆、生物信息學及表達分析,中國科學院微生物研究所發現將真菌的GDH轉入到棉花中,可提高氮素利用率30%以上,轉GDH植物是屬于抗營養貧瘠類的新一代轉基因植物,它不僅可以節約氮肥30%—40%,降低生產成本,還有利于環境保護[20]。【本研究切入點】桑樹為多年生木本植物,生物產量高,耐肥性強,而GDH在桑樹中的特點及生物學功能未見報道。【擬解決的關鍵問題】本研究擬應用RT-PCR技術克隆MaGDHs,分析其結構和表達特點,構建原核重組表達載體在大腸桿菌BL21(DE3)中高效表達,并對誘導時間和IPTG濃度等參數進行研究和優化,建立高效原核表達體系,為下一步桑樹的GDH功能研究及其酶促動力學研究奠定基礎。
1 材料與方法
試驗于2016年3月至2017年5月在西南大學生物技術學院桑樹遺傳育種研究室及家蠶基因組生物學國家重點實驗室進行。
1.1 試驗材料
取桂桑優62號桑樹(廣西區審定的桑樹雜優組合,購于廣西蠶業技術推廣總站),屬于廣東桑,將種子消毒后播種于1/2MS培養基上,待其生長至四葉一心時,在對照培養基(MS +瓊脂7 g·L-1+蔗糖30 g·L-1,CK)培養10 d后轉移到不同的培養基中(表1),每個濃度處理4瓶,每瓶3—4株。在不含NH4+的培養基中,硝酸銨用硝酸鉀代替,增加的銨鹽以(NH4)2SO4提供。處理后0、12、24和48 h分別取材。取材時,每一組試驗隨機采取多株試管苗混合取材,重復3次。另外,從成熟桑樹(同一株,雌雄同株不同花穗)中收集根、莖、老葉、嫩葉、幼葉、雌花、雄花、果實、葉柄、表皮,以檢測不同組織的轉錄水平。收集的所有材料立即在液氮中冷凍帶回實驗室并儲存在-80℃冷凍箱中。
1.2 數據搜索和分析
為鑒定桑樹中GDH基因家族的成員,以“Glutamate dehydrogenase”為關鍵詞在川桑(Morus notabilis)數據庫(http://morus.swu.edu.cn/morusdb/)中檢索,并使用 BLASTN(www.ncbi.nlm.nih.gov)和 SMART(http://smart.embl-heidelberg.de/)鑒定候選基因,利用 GeneScript公司在線工具(http://www. genscript.com.cn/index.html)設計用于克隆和qRT-PCR的引物(表 2),通過在線軟件(http://web.expasy.org/compute_pi/)預測MaGDHs分子量和等電點。下載可可(Theobroma cacao)、獼猴桃(Actinidia chinensis)、擬南芥(Arabidopsis thaliana)、大豆(Glycine max)、花生(Arachis hypogaea)、水稻(Oryza sativa)、玉米(Zea mays)、小麥(Triticum aestivum)8個物種的GDH氨基酸序列,使用Genetyx-v7、Bioxm 26,Sequencher 4.2、BioEdit、GeneDoc等軟件對GDH基因序列和氨基酸序列進行分析和比對,系統發育樹由MEGA 5.0軟件構建。
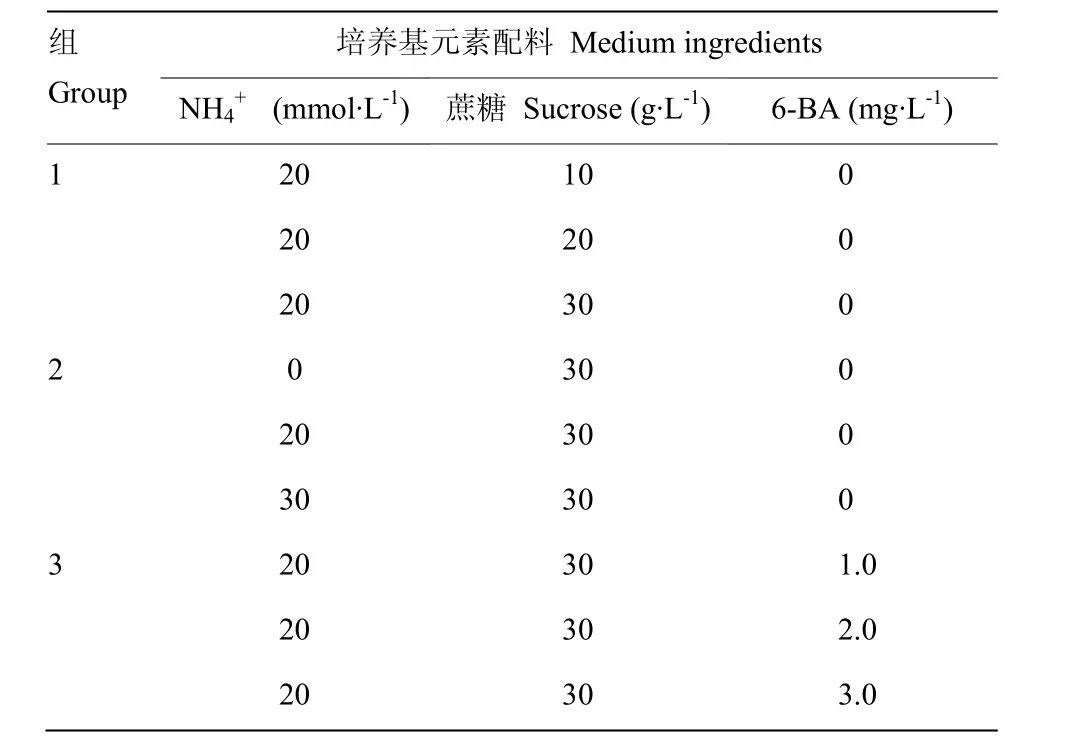
表1 培養基的成分Table 1 Medium ingredients
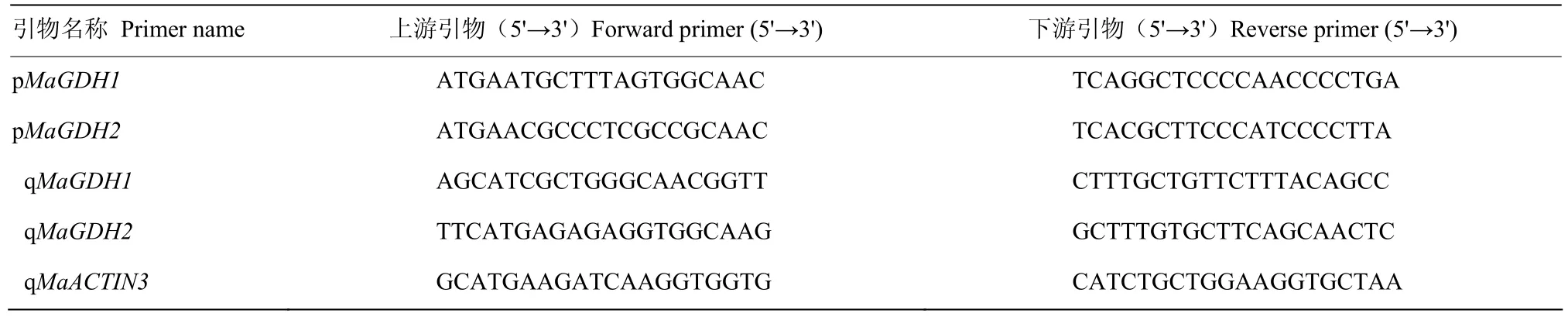
表2 MaGDH1和MaGDH2的克隆及熒光定量引物序列Table 2 Primers of MaGDH1and MaGDH2 for cloning and qRT-PCR
1.3 RT-PCR克隆MaGDHs
按照TaKaRa公司RNA抽提試劑盒說明書提取桂桑優62號桑葉總RNA。分別取適量體積RNA用1%的瓊脂糖凝膠電泳檢測,并用紫外分光光度計檢測總RNA的濃度。以總 RNA為模板,參照反轉錄酶M-MLV(Promega)說明書,并以Oligo(dT)18作為引物,合成 cDNA第 1鏈,存于-20℃備用。以cDNA為PCR模板,進行PCR擴增,擴增條件為94℃ 4 min;94℃ 40 s,51℃ 1 min,72℃ 2 min,循環 32 次;72℃ 10 min。DNA凝膠回收試劑盒回收PCR產物,目的片段與TaKaRa公司的PMD19-T載體16℃過夜連接,轉化大腸桿菌 DH5α,挑選陽性克隆,送至生工生物工程(上海)股份有限公司測序。
1.4 MaGDHs的原核表達及酶活性測定
本研究使用 pET-28a(+)、pET-32a(+)、pcold-TF載體。設計每個載體含有限制酶位點的引物用于PCR(表3)。將PCR產物克隆到載體中并轉化到大腸桿菌BL21細胞中以在大腸桿菌細胞中產生組氨酸標記的重組蛋白。分別用 0.1、0.5 和 1.0 mmol·L-1IPTG(異丙基-bD-硫代吡喃半乳糖苷)在大腸桿菌中誘導重組蛋白,pET-28a(+)和 pET-32a(+)載體 28℃培養 6 h,pcold-TF載體在16℃培養24 h。培養物4℃下5 000 r/min離心15 min,收集菌體并進行超聲破碎,使用不同濃度的咪唑,利用 Ni-NTA親和層析(Genscript,NJ,USA)純化在大腸桿菌中表達的重組蛋白質,用12%(w/v)SDS-PAGE進行電泳檢測。將純化的蛋白質用尿素梯度透析液進行梯度透析,收集復性蛋白,用Bradford法測定蛋白濃度,用索萊寶公司的谷氨酸脫氫酶試劑盒測定酶活性,具體方法如下:
酶標儀預熱30 min以上,調節波長至340 nm,蒸餾水調零。在試劑二中加入19 ml試劑一充分溶解混勻,25℃水浴5 min,在96孔板中加入10 μL樣本和190 μL試劑二,混勻,立即記錄340 nm處20 s的吸光值A1和5 min后的吸光值A2,計算△A=A1-A2。定義每毫升復性的蛋白溶液每分鐘消耗 1 nmol NADH定義為一個酶活力單位。計算公式如下:
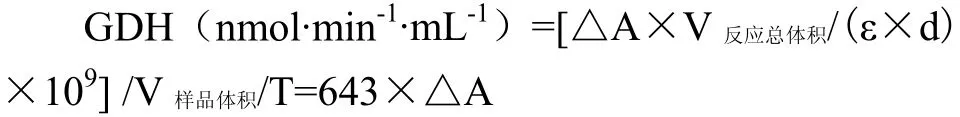
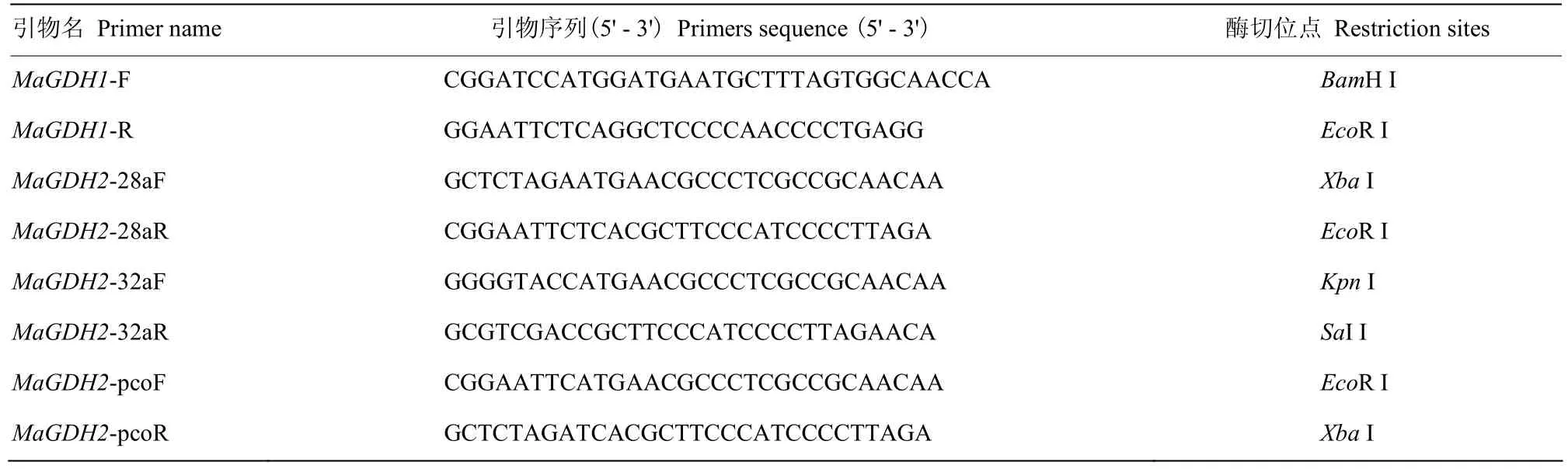
表3 MaGDH1和MaGDH2原核表達載體構建引物序列Table 3 The primer sequences of prokaryotic expression vectors for MaGDH1 and MaGDH2
1.5 定量PCR分析
分別從收集到的植物材料中提取總RNA。DNase I(TaKaRa,Japan)消化RNA樣品以去除DNA。參照反轉錄酶M-MLV(TaKaRa)說明書,以Oligo(dT)18為引物,合成cDNA第1鏈。將cDNA稀釋3倍,使用實時熒光定量PCR試劑SYBR? Premix Ex TaqTM(Tli RNaseH Plus)擴增,擴增體系為SYBR?Premix Ex TaqTM(Tli RNaseH Plus)(2×) 10 μL,ROX Reference Dye(50×) 0.4 μL,cDNA 2 μL,depc H2O 6 μL,上、下游引物各0.8 μL。程序為95℃預變性30 s;94℃ 5 s, 60℃ 30 s, 72℃ 10 min,循環40次,并以MaACTIN3(HQ163775.1)為內參。用 StepOne Software V2.1和GraphPad Prism 5軟件分析試驗數據,采用2-△△Ct法進行基因相對表達量分析。所有試驗均重復3次,采用SPSS16進行統計學分析。
2 結果
2.1 桑樹谷氨酸脫氫酶基因家族的鑒定和克隆
在川桑數據庫中搜索到川桑谷氨酸脫氫酶基因GDH(登錄號為Morus014749和Morus017087),以該序列設計引物,以栽培桑品種桂桑優 62號的葉片cDNA為模板,通過 RT-PCR,瓊脂糖電泳鑒定出兩條750—1 500 bp的目的條帶(圖1-A)。經回收,測序后發現兩條序列全長均為1 236 bp,編碼411個氨基酸,含一個開放閱讀框(ORF),命名為MaGDH1和MaGDH2。同源比較分析得知,川桑和桂桑優62號的GDH相似性為99%,MaGDH1和MaGDH2氨基酸水平相似度為81%,均含有9個外顯子,8個內含子(圖1-B)。在線軟件(http://web.expasy.org/compute_pi/)預測 MaGDH1蛋白質的分子量為 44.1 kD,理論等電點為5.84;MaGDH2蛋白質的分子量為44.2 kD,理論等電點為6.68。NCBI上分析氨基酸序列的保守結構域,發現MaGDH1和MaGDH2蛋白屬于ELFV-dehydrog superfamily超家族。

圖1 MaGDHs瓊脂糖電泳圖(A)及其外顯子內含子結構模式圖(B)Fig. 1 Agarose gel electrophoresis of MaGDHs and their scaled diagram of exon-intron structure
2.2 MaGDHs氨基酸序列比較及進化樹分析
在NCBI數據庫中下載擬南芥、水稻、大豆、可可、獼猴桃等8個物種的GDH氨基酸序列進行同源性分析結果顯示,MaGDH編碼的氨基酸序列與雙子葉植物中 GDH編碼的氨基酸序列同源性為 86%—93%,與單子葉植物GDH編碼的氨基酸序列同源性為84%—87%。多重序列比對結果表明這些GDH所編碼的氨基酸序列含有在N末端的線粒體轉移肽序列,包含 Glu/a-KG結合域的 PF02812結構和包含NAD(P)結合域的 PF00208結構。PF02812結構比PF00208結構的序列更保守,PF00208結構的 N-和C-端比中間區域序列更保守(圖2)。同時利用它們的氨基酸序列構建進化樹如圖 3所示,桑樹的兩個MaGDHs聚為兩支,驗證了克隆的MaGDHs分別為GDH1和 GDH2。MaGDH1與可可先聚為一支,然后與獼猴桃再聚為一支,再與其他雙子葉、單子葉植物聚類;MaGDH2并沒有與可可聚為一類,而是可可與獼猴桃先聚為一支,然后與桑樹再聚為一支,表明 MaGDH1和MaGDH2在進化順序上并不是一致的。
2.3 MaGDHs在大腸桿菌BL21(DE3)中原核表達
以 pET-28a(+)、pET-32a(+)、pCold-TF為載體構建融合蛋白重組質粒 pET-28a(+)-MaGDH1、pET-32a(+)-MaGDH1、pcold-TF-MaGDH1,pET-28a(+)-MaGDH2、pET-32a(+)-MaGDH2、pcold TF-MaGDH2,酶切電泳如圖4所示,除含有3種質粒大小的目的條帶,也含有MaGDH1和MaGDH2片段。以IPTG誘導后,pET-28a(+)-MaGDH1、pET-32a(+)-MaGDH1、pcold-TF-MaGDH1在大腸桿菌 BL21(DE3)中均能表達,且使用pET-28a(+)載體時以IPTG濃度為0.1 mmol·L-1表達效果最佳,pET-32a(+)載體時 IPTG 濃度為 1.0 mmol·L-1效果最佳,pCold-TF載體時 IPTG濃度對蛋白表達量影響不大(圖5-A、B、C)。MaGDH2只能以重組質粒pET-32a(+)-MaGDH2才能在大腸桿菌 BL21(DE3)中成功表達,IPTG 濃度為 1.0 mmol·L-1時效果最佳(圖 5-D、E、F)。融合蛋白 pET-28a(+)-MaGDH1,pCold-TF-MaGDH1大小接近44.3 kD,與預測結果基本一致,pET-32a(+)自帶標簽大于pET-28a(+)和pCold-TF,因而融合蛋白pET-32a(+)-MaGDH1、pET-32a(+)-MaGDH2大小接近66.4 kD。將菌液進行超聲破碎,將上清和沉淀分別進行 SDSPAGE電泳檢測,結果表明 pET-28a(+)-MaGDH1、pET-32a(+)-MaGDH1、pCold-TF-MaGDH1和pET-32a(+)-MaGDH2均以包涵體的形式存在(圖5-G、H)。
2.4 MaGDHs蛋白的純化及酶活性測定
用 Ni-NTA樹脂層析柱,利用不同濃度的咪唑洗滌蛋白沉淀,對目的蛋白進行純化SDS-PAGE電泳表明,pET-28a(+)-MaGDH1、pET-32a(+)-MaGDH1和pCold-TF-MaGDH1均在咪唑濃度為100 mmol·L-1時目的蛋白被大量洗脫下來(圖6-A、B、C),而pET-32a(+)-MaGDH2目的蛋白則在咪唑濃度為250 mmol·L-1時被大量洗脫下來(圖 6-D),獲得純化的MaGDH1蛋白和 MaGDH2蛋白。經過測定,不同載體重組質粒在最適 IPTG濃度條件下,表達的蛋白質含量不同(表4),活性也有差異,其中pCold-TFMaGDH1 表達的蛋白最高,為 633.6 μg·mL-1,但表達的蛋白沒有酶活性,而 pET-28a(+)-MaGDH1表達的蛋白活性最高,為 10.07 nmol-1·min-1·mL-1。

圖2 桑樹谷氨酸脫氫酶GDH的氨基酸與其他物種氨基酸序列同源性序列比對Fig. 2 Amino acid sequence alignment of glutamate dehydrogenase (GDH) mulberry and other species

圖3 GDH家族的系統樹Fig. 3 Phylogenetic tree of the glutamate dehydrogenase family

圖4 重組質粒的酶切電泳Fig. 4 Electrophoresis of recombinant plasmid after restriction digestion
2.5 MaGDH1和MaGDH2的組織表達
MaGDH1和MaGDH2在桑樹各個組織中都有表達(圖7),總體上MaGDH1在各個組織上表達量高于MaGDH2。MaGDH1在雄花中的表達量最高,其次為嫩葉、莖、葉柄、雌花,在果實中表達量低。MaGDH2也是在雄花中的表達量最高,其次為雌花、幼葉、嫩葉、莖、葉柄,在果實中幾乎不表達。桑樹的開花期在花和幼葉代謝生長發育旺盛,莖和葉柄起運輸作用,由此推測,MaGDH1和MaGDH2在各個組織中的表達量與桑樹的生長發育密切相關。
2.6 MaGDH1和MaGDH2的表達的調控
在培養基中添加 10、20和 30 g·L-1的蔗糖,MaGDH1和MaGDH2的表達量如圖8所示。隨著蔗糖濃度的增加,MaGDH1和MaGDH2表達量增加,MaGDH1在蔗糖濃度為30 g·L-1處理12 h時表達量最高,MaGDH2則在蔗糖濃度為30 g·L-1處理24 h時表達量最高,MaGDH2的相對表達量比MaGDH1表達量高。MaGDHs受NH4+調控表達如圖9所示,培養基中即使沒有外源NH4+,MaGDHs也有表達,添加20 mmol·L-1(常規量)NH4+時,MaGDHs表達量立即增加;當NH4+增加到30 mmol·L-1(過量)時,MaGDHs并沒有立即增加反而受到抑制,MaGDH1在24 h表達量超過常規NH4+濃度的表達量,MaGDH2在48 h表達量超過常規NH4+濃度的表達量。在培養基中添加細胞分裂素6-BA,MaGDH1和MaGDH2的表達量如圖10所示,添加6-BA 12 h后,MaGDH1和MaGDH2的表達量都受到抑制,濃度越高,抑制更加明顯;24 h后,MaGDH1的表達量受到濃度為1.0 mg·L-1和2 mg·L-1的6-BA促進,當濃度為3 mg·L-1時抑制其表達;48 h時,3個濃度的6-BA都促進MaGDH2的表達。
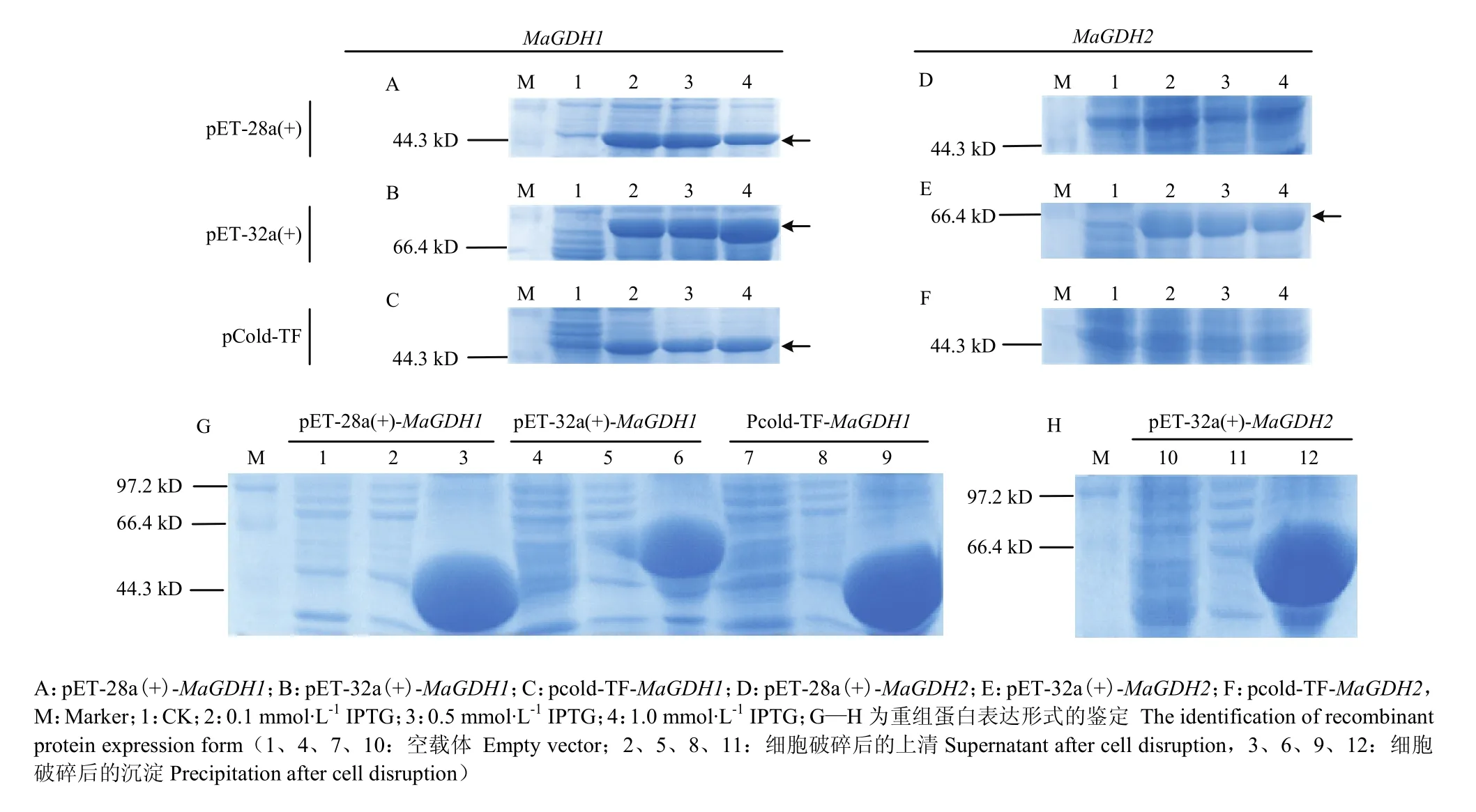
圖5 原核表達MaGDHs蛋白表達產物SDS-PAGE電泳(箭頭所指為目的蛋白)Fig. 5 SDS-PAGE electrophoresis of MaGDHs fusion protein product by prokaryotic expression (The arrow refers to the target protein strip)

圖6 重組MaGDHs蛋白的純化Fig. 6 Purification of recombinant MaGDHs fusion protein
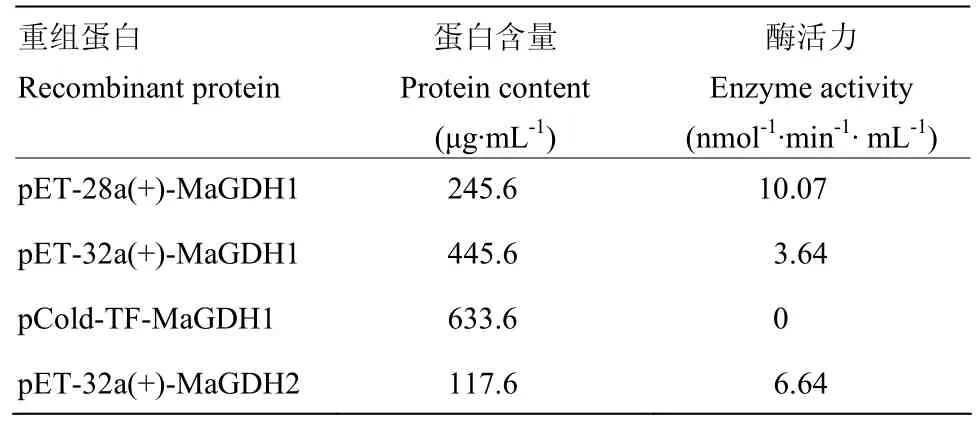
表4 MaGDHs重組蛋白含量及酶活性Table 4 The content and activity of MaGDHs protein
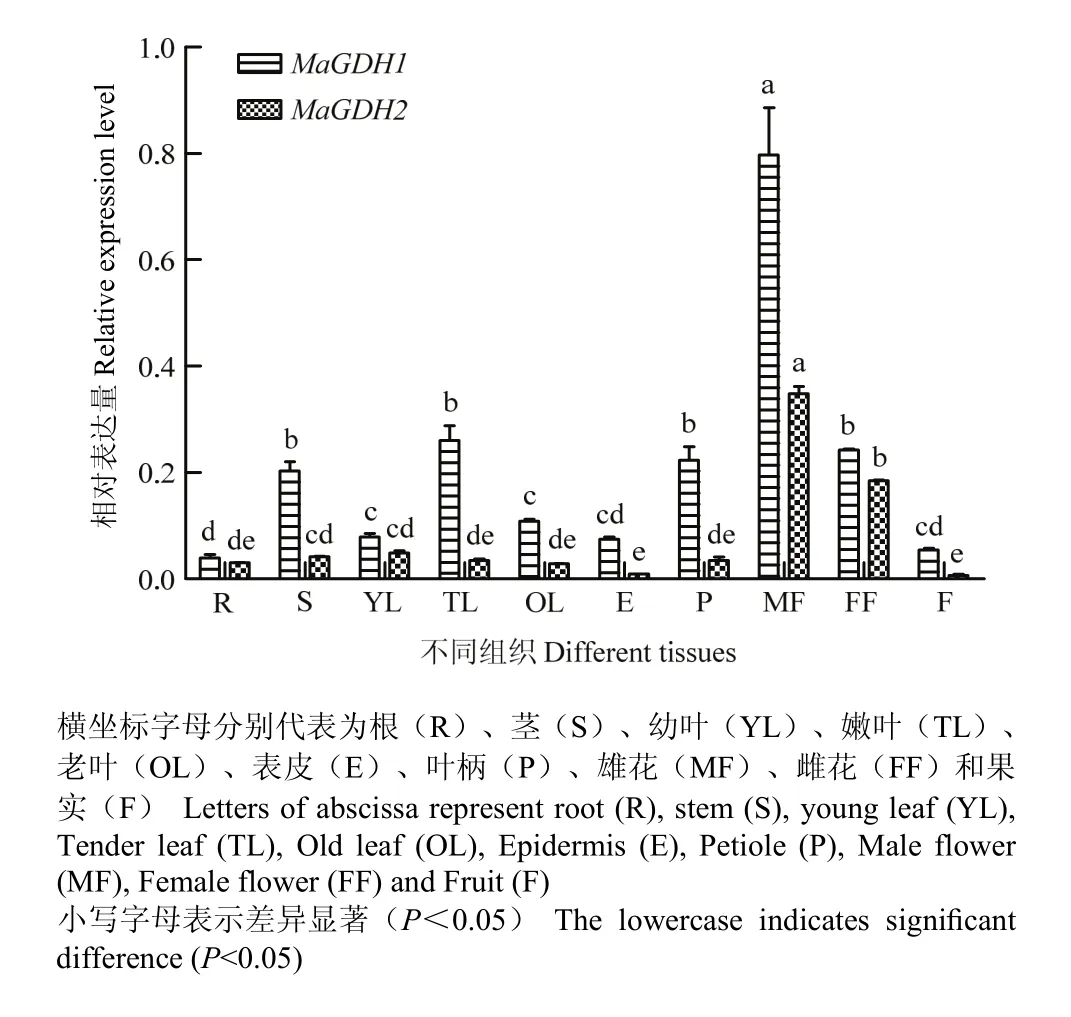
圖7 MaGDHs在各個組織中定量表達分析Fig. 7 Quantitative real-time PCR (qPCR) results for MaGDHs genes
3 討論
一般認為,高等植物GDH中的α和β亞基分別由不同的兩個基因編碼,GDH1(或 GDHB)基因編碼β亞基,GDH2(或GDHA)編碼α亞基[21],在擬南芥中發現了GDH3編碼γ亞基[22]。在有些植物中如黑小麥、黃羽扇豆、煙草、番茄等已克隆出了至少2個GDH,分別編碼α和β亞基[23-26],而一些植物中目前卻只克隆獲得了其中一個基因[27-29]。本研究結果表明,桑樹中有兩個GDH,進化樹分析表明MaGDHs蛋白屬于兩種類型,分別為MaGDH1和MaGDH2,分別編碼β亞基和α亞基。

圖8 MaGDHs在不同濃度蔗糖條件下定量表達Fig. 8 Quantitative real-time PCR (qPCR) results for MaGDHs genes under different concentration of sucrose
LEHMANN等[24]報道黃羽扇豆GDH1和GDH2的表達具有組織特異性,雖然兩個基因在黃羽扇豆的所有組織中都有表達,但GDH1的轉錄明顯高于GDH2,本研究的結果與其結果基本一致。此外,在檢測的桑樹組織中,MaGDH1和 MaGDH2均在花中表達量最高,與前人對水稻[12]和擬南芥[30]GDH的研究結果相似,他們認為花器官是能量需求較多的器官,因此AtGDH2和OsGDH2在花中表達量最高。谷氨酸脫氫酶的生理功能有較多的爭論,由于植物體內的 GDH對于銨具有很高的 Km值(5.2—70 mmol·L-1),使人們對于GDH在植物體內的銨同化功能提出質疑。也有人認為GDH的底物是NH3而不是NH4+,本研究在未加外源NH4+的情況下,檢測到GDH的表達,因此推測,GDH底物至少不會完全來源于外源NH4+也可能由植物自身代謝產生。本研究表明添加過量的NH4+能促進MaGDHs表達,且MaGDH1與響應比MaGDH2早,結果暗示MaGDHs可能對過量的 NH4+有一定的解毒作用。FOX等[31]以胡蘿卜懸浮細胞為材料發現培養基中碳源缺乏時GDH活性最大,當給培養基添加蔗糖時,GDH活性急劇降低;MELO-OLIVEIRA[32]以擬南芥為材料,發現在黑暗或碳源有限供給條件下,GDH1的mRNA大量積累,而光照條件或蔗糖供給都會抑制GDH1 mRNA積累,本研究發現隨著蔗糖濃度增加,MaGDH1和 MaGDH2的表達量增加,因此,碳源對 MaGDHs的調控還有待進一步研究。同時,MaGDHs還受到外源激素的調控,培養基中添加細胞分裂素6-BA 12 h后,抑制了MaGDH1和MaGDH2的表達,并且濃度越高,抑制作用越強。隨著時間延長,這種抑制作用減弱,推測 6-BA對MaGDHs的正調控可能是間接調控。
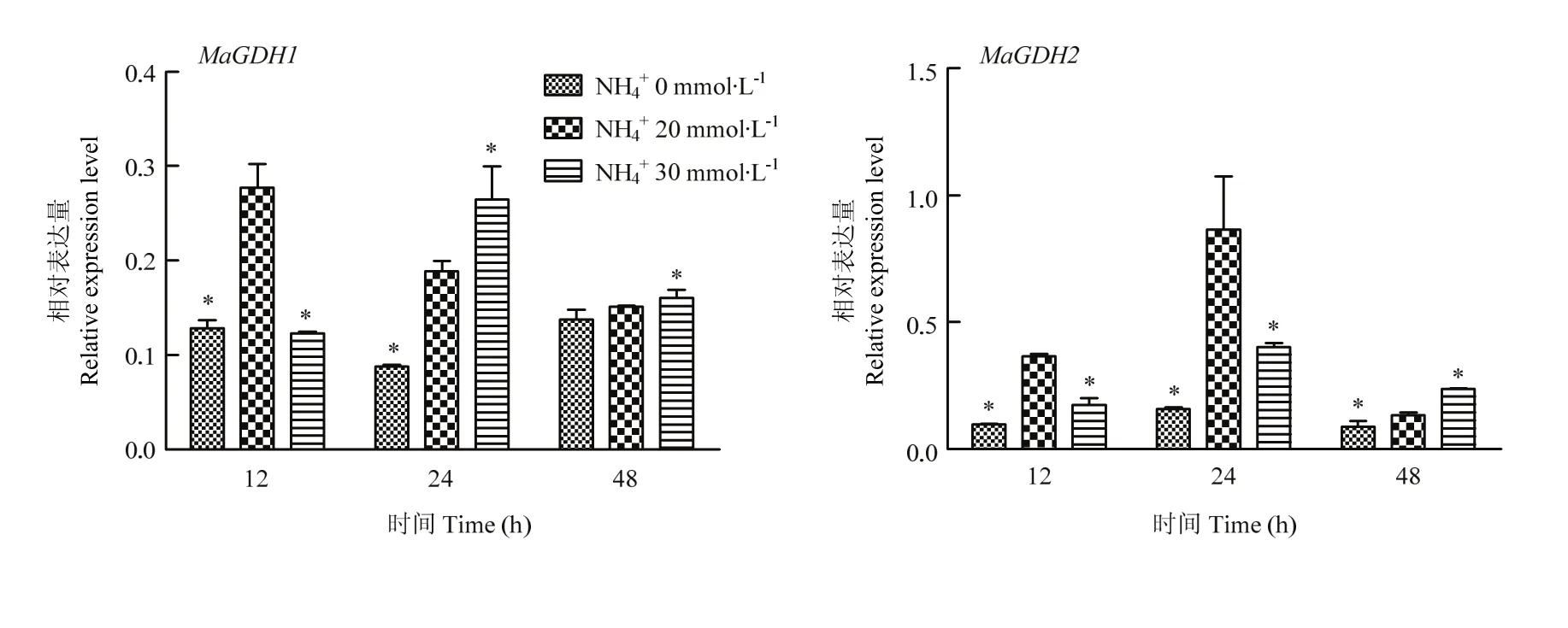
圖9 MaGDHs在不同濃度NH4條件下定量表達Fig. 9 Quantitative real-time PCR (qPCR) results for MaGDHs genes under different concentration of NH4+

圖10 MaGDHs在不同濃度6-BA條件下定量表達Fig. 10 Quantitative real-time PCR (qPCR) results for MaGDHs genes under different concentration of 6-BA
4 結論
獲得兩個MaGDHs,全長均為1 236 bp,編碼411個氨基酸,分別編碼β和α亞基,基因序列高度保守。MaGDHs表達具有組織特異性,在旺盛生長的組織中表達量較高,受到過量NH4+、細胞分裂素的調控。IPTG濃度和載體都會影響重組蛋白的高效表達,大腸桿菌表達的重組MaGDH蛋白以包涵體形式存在,不同載體的重組蛋白酶活性存在差異。
[1] DARIAS-MARTíN J, LOBO-RODRIGO G, HERNáNDEZ-CORDERO J, DíAZ-DíAZ E, DíAZ-ROMERO C. Alcoholic beverages obtained from black mulberry. Food Technology & Biotechnology, 2003, 41(2):173-176.
[2] VENKATESH KR, CHAUHAN S. Mulberry. Life enhancer. Journal of Medicinal Plants Research, 2008, 2(10): 271-278.
[3] WANG C, ZHI S, LIU C, XU F, ZHAO A C, WANG X, REN Y, LI Z,YU M. Characterization of stilbene synthase genes in mulberry(Morus atropurpurea) and metabolic engineering for the production of resveratrol in Escherichia coli. Journal of Agricultural & Food Chemistry, 2017. doi:10.1021/acs.jafc.6b05212.
[4] CHECKER V G, KHURANA P. Molecular and functional characterization of mulberry EST encoding remorin (MiREM) involved in abiotic stress. Plant Cell Reports, 2013, 32(11): 1729-1741.
[5] BULEN W A. The isolation and characterization of glutamic dehydrogenase from corn leaves. Archives of Biochemistry &Biophysics ,1956, 62(1): 173-183.
[6] LEA P J, MIFLIN B J. Alternative route for nitrogen assimilation in higher plants. Nature ,1974, 251(5476): 614-616.
[7] WALLSGROVE R M, TURNER J C, HALL N P, KENDALL A C,BRIGHT S W J. Barley mutants lacking chloroplast glutamine synthetase-biochemical and genetic analysis. Plant Physiology ,1987,83(1): 155-158.
[8] ROBINSON S A, SLADE A P, FOX G G, PHILLIPS R, RATCLIFFE R G, STEWART G R. The role of glutamate dehydrogenase in plant nitrogen metabolism. Plant Physiology, 1991, 95(2): 509-516.
[9] MASCLAUX-DAUBRESSE C, REISDORF-CREN M, PAGEAU K,LELANDAIS M, GRANDJEAN O, KRONENBERGER J,VALADIER M H, FERAUD M, JOUGLET T, SUZUKI A. Glutamine synthetase-glutamate synthase pathway and glutamate dehydrogenase play distinct roles in the sink-source nitrogen cycle in tobacco. Plant Physiology, 2006, 140(2): 444.
[10] WANG Z Q, YUAN Y Z, OU J Q, LIN Q H, ZHANG C F.Glutamine synthetase and glutamate dehydrogenase contribute differentially to proline accumulation in leaves of wheat (Triticum aestivum) seedlings exposed to different salinity. Journal of Plant Physiology, 2007,164(6): 695.
[11] MIYASHITA Y, GOOD A G. NAD(H)-dependent glutamate dehydrogenase is essential for the survival of Arabidopsis thaliana during dark-induced carbon starvation. Journal of Experimental Botany, 2008, 59(3): 667-680.
[12] QIU X, XIE W, LIAN X, ZHANG Q. Molecular analyses of the rice glutamate dehydrogenase gene family and their response to nitrogen and phosphorous deprivation. Plant Cell Reports, 2009, 28(7):1115-1126.
[13] LEHMANN T, SKROK A, DABERT M. Stress-induced changes in glutamate dehydrogenase activity imply its role in adaptation to C and N metabolism in lupine embryos. Physiologia Plantarum, 2010,138(1): 35-47.
[14] LAURA M, FRANCESCA D, EUGENIA P, THéRèSE TL,FRéDéRIC D, BERTRAND H, FRANCESCO MARIA R. Glutamate dehydrogenase isoenzyme 3 (GDH3) of Arabidopsis thaliana is regulated by a combined effect of nitrogen and cytokinin. Plant Physiology & Biochemistry, 2013, 73(6): 368-374.
[15] MARCHI L, POLVERINI E, DEGOLA F, BARUFFINI E, RESTIVO FM. Glutamate dehydrogenase isoenzyme 3 (GDH3) of Arabidopsis thaliana is less thermostable than GDH1 and GDH2 isoenzymes.Plant Physiology & Biochemistry, 2014, 83(83): 225-231.
[16] PURNELL M P, SKOPELITIS D S, ROUBELAKIS-ANGELAKIS K A, BOTELLA J R. Modulation of higher-plant NAD(H)-dependent glutamate dehydrogenase activity in transgenic tobacco via alteration of beta subunit levels. Planta, 2005, 222(2): 167-180.
[17] TSILIKOCHRISOS G, TSANIKLIDIS G, DELIS C, NIKOLOUDAKIS N, AIVALAKIS G. Glutamate dehydrogenase is differentially regulated in seeded and parthenocarpic tomato fruits during crop development and postharvest storage. Scientia Horticulturae, 2015,181: 34-42.
[18] LEHMANN T, RATAJCZAK L. The pivotal role of glutamate dehydrogenase (GDH) in the mobilization of N and C from storage material to asparagine in germinating seeds of yellow lupine. Journal of Plant Physiology, 2008, 165(2): 149-158.
[19] GRABOWSKA A, NOWICKI M, KWINTA J. Glutamate dehydrogenase of the germinating triticale seeds: Gene expression,activity distribution and kinetic characteristics. Acta Physiologiae Plantarum, 2011, 33(5): 1981-1990.
[20] 許宏濤, 計懷春, 李傳軍, 黃西林. 轉GDH基因棉花研究簡報. 中國棉花 2005, 32(2): 22-23.XU H T, JI H C, LI C J, HUANG X L. Study of cotton with GDH gene. China Cotton, 2005, 32(2): 22-23. (in Chinese )
[21] 黃國存, 田波. 高等植物中的谷氨酸脫氫酶及其生理作用. 植物學報, 2001, 18(4): 369-401.HUANG G C, TIAN B. The Physiological Role of Glutamate dehydrogenase in higher plants. Bulletin of Botany, 2001, 18(4):369-401. (in Chinese )
[22] MARCHI L, DEGOLA F, POLVERINI E, DUBOIS F, HIREL B,RESTIVO F M. Glutamate dehydrogenase isoenzyme 3 (GDH3) of Arabidopsis thaliana is regulated by a combined effect of nitrogen and cytokinin. Plant Physiology & Biochemistry, 2013, 73(6): 368-374.
[23] GRABOWSKA A, ZDUNEK-ZASTOCKA E, KUTRYN E, KWINTA J. Molecular cloning and functional analysis of the second gene encoding glutamate dehydrogenase in Triticale. Acta Physiologiae Plantarum, 2017, 39(1): 24.
[24] LEHMANN T, DABERT M, NOWAK W. Organ-specific expression of glutamate dehydrogenase (GDH) subunits in yellow lupine. Journal of Plant Physiology, 2011, 168(10): 1060-1066.
[25] RESTIVO F M. Molecular cloning of glutamate dehydrogenase genes of Nicotiana plumbaginifolia: Structure analysis and regulation of their expression by physiological and stress conditions. Plant Science,2004, 166(4): 971-982.
[26] FERRARO G, BORTOLOTTI S, MORTERA P, SCHLERETH A,STITT M, CARRARI F, KAMENETZKY L, VALLE EM.Novel glutamate dehydrogenase genes show increased transcript and protein abundances in mature tomato fruits. Journal of Plant Physiology,2012, 169(9): 899-907.
[27] 陳湘瑜, 徐日榮, 林栩松, 唐兆秀.花生谷氨酸脫氫酶基因AhGDH1的克隆與生物信息學分析. 福建農業學報 2016, 31(3):217-224.CHEN X Y, XU R R, LIN X S,TANG Z X. Cloning and bioinformatic analysis of AhGDH1 gene from Arachis hypogaea L.Fujian Journal of Agricultural Sciences, 2016, 31(3): 217-224. (in Chinese )
[28] 馮仁軍, 盧利方, 程萍, 袁克華, 張銀東. 巴西橡膠樹谷氨酸脫氫酶基因 cDNA片段克隆及表達分析. 西南大學學報(自然科學版),2008, 30(8): 114-118.FENG R J, LU L F, CHENG P, YUAN K H, ZHANG Y D. Cloning and expression analysis of glutamate dehydrogenase gene cDNA from Hevea brasiliensis. Journal of Southwest University (Natural Science Edition), 2008, 30(8): 114-118. (in Chinese )
[29] 陳琪, 孟祥宇, 江雪梅, 于淑偉, 宛曉春. 茶樹茶氨酸代謝相關基因表達組織特異性分析. 核農學報, 2015, 29(7): 1285-1291.CHEN Q, MENG X Y, JIANG X M, YU S W, WAN X C. Tissue specificity analysis of alliophytona-related gene expression in Camellia sinensis. Journal of Nuclear Agricultural Sciences, 2015,29(7): 1285-1291. (in Chinese )
[30] TURANO F J, THAKKAR S S, FANG T, WEISEMANN J M.Characterization and expression of NAD(H)-dependent glutamate dehydrogenase genes in Arabidopsis. Plant Physiology, 1997, 113(4):1329-1341.
[31] FOX G G, RATCLIFFE R G, ROBINSON S A, STEWART G R.Evidence for deamination by glutamate dehydrogenase in higher plants: Commentary. Canadian Journal of Botany, 1995, 73(7):1112-1115.
[32] MELO-OLIVEIRA R, OLIVEIRA I C, CORUZZI G M. Arabidopsis mutant analysis and gene regulation define a nonredundant role for glutamate dehydrogenase in nitrogen assimilation. Proceedings of the National Academy of Sciences of the United States of America, 1996,93(10): 4718-4723.

