相對熒光定量法檢測人體2′,5′-寡聚腺苷酸合成酶相對表達水平
張晶,王芳,陸春燕,孫詩琪,孫家鵬,邢美杰,章良弼,付潔,宋海峰
1.軍科正源(北京)藥物研究有限責任公司,北京 102206;2.軍事科學院 軍事醫學研究院生命組學研究所,蛋白質藥物國家工程研究中心,北京 102206;3.安徽安科生物工程(集團)股份有限公司,安徽 合肥 230088
生物標志物是指可用于檢測并評價生理病理的某種特定的指標物[1],包括生物大分子如酶、生長因子,或小分子如DNA、RNA、microRNA等。臨床上,可以通過檢測生物標志物篩查出相關疾病的早期患者。在藥物研發及臨床治療中檢測相關生物標志物并通過生物標志物的上升或下調來評價藥效或療效,判斷患者的預后以及預估生存期的長短。更重要的是對生物標志物的深入研究為新藥的研發和臨床應用提供了可能,例如肺癌相關的生物標志物p53基因[2-3]、乳腺癌相關標志物糖蛋白CA125[4]、正常細胞DNA甲基化與沉默基因突變[5]、干擾素療效相關的 2′,5′-寡聚腺苷酸合成酶等[6-8]。
干擾素類藥物是Kerr等發現的由干擾素誘導產生的抗病毒蛋白(antiviral protein,AVP),參與機體非特異抗病毒反應,是機體重要的抗病毒蛋白之一。2′,5′-寡聚腺苷酸合成酶(2′,5′-oli?goadenylate synthetase,2′,5′-OAS)的活性變化已經成為臨床上評價干擾素類藥物如派羅欣對機體作用的經典生物標志物分子之一[9-10]。
目前針對生物大分子如生長因子、酶類的檢測方法主要通過依賴于特異性抗體的酶聯免疫反應檢測其蛋白水平的表達,如ELISA、MSD、Gy?rolab等[11-13],雖然最為靈敏的MSD(Meso Scale Discovery)已經能將檢測靈敏度提升至皮克級,但其成本高,并且針對個性化大分子獲得理想的特異性抗體從而建立建立理想的實驗條件仍是巨大挑戰。相比之下,嘗試從核酸水平去檢測生物大分子是分析方法發展趨勢之一。聚合酶鏈式反應(polymerase chain reaction,PCR)是一種通過引物、聚合酶特異性擴增DNA片段的技術,逆轉錄-實時熒光定量PCR(reverse transcription quan?titative real-time PCR,RT-QPCR)是在這種擴增過程中加入熒光物質對每次PCR反應后的產物進行檢測的方法。根據內參或外參的選擇,常用的 QPCR 法又可分為相對定量法(2-ΔΔCt)、雙標準曲線相對定量法及絕對定量法。目前,RT-QPCR或QPCR法已廣泛應用于基因的RNA表達水平變化及核酸類藥物的藥代動力學檢測中[14-15]。
本研究基于受試品聚乙二醇化重組人干擾素α2b注射液臨床Ⅰa期的研究,以派羅欣為陽性參比藥物,建立了 RT-QPCR 2-ΔΔCt方法檢測受試者給予干擾素類藥物后不同時間點2′,5′-OAS相對于藥前點的表達水平變化倍數。與傳統蛋白水平檢測的ELISA方法相比,2種方法對于藥效學指標的整體趨勢與劑量關系類似。此外,基于核酸水平的研究結果表明可能給出更多生物標志物變化信息。證明RT-QPCR可以作為一種檢測外周全血中生物標志物的替代分析方法。
1 材料與方法
1.1 材料
QPCR 儀器為 ABI 7500 Fast(ThermoFisher Scientific公司);受試藥聚乙二醇化重組人干擾素α2b注射液由安徽安科生物工程(集團)股份有限公司生產并提供;對照藥物聚乙二醇干擾素α2a注射液(商品名:派羅欣)由安徽安科生物工程(集團)股份有限公司提供);TRI試劑購自Sigma公司;逆轉錄試劑購自Thermo公司;QPCR SYBG染料購自諾唯贊公司;ELISA檢測試劑盒購自美旋生物科技有限公司。
1.2 試驗方案
藥效學分析中受試品聚乙二醇重組人干擾素α2b注射液的給藥方法為160 μg/人,對照品聚乙二醇干擾素α2a注射液(派羅欣)為180 μg/人。分別在給藥前 30 min,給藥后 1、3、6、9、12、24、48、72、96、120、144、168、192、240、288、336、480 h采取樣品。
1.3 采樣方法及樣品前處理方法
1.3.1 血清樣品采取方法 取靜脈血放入真空試管中,室溫放置30 min后3000 r/min離心8 min,分離出血清,-80℃保存;
1.3.2 全血樣品 取200 μL新鮮全血加入含750 μL TRI試 劑 、20 μL 5 mol/L 醋 酸 的 1.5 mL EP管中,立即混勻,凍存于-80℃,按TRI全血RNA樣品提取方法提取RNA后逆轉錄成cDNA。
1.4 QPCR檢測方法
1.4.1 cDNA模板制備 將0.2 mL新鮮抗凝全血加入裝有 750 μL TRI試劑、20 μL 5 mol/L 醋酸緩沖液的EP管,立即混勻,凍存于-60℃~-80℃。用TRI試劑提取RNA,用Thermo試劑盒體外逆轉錄獲得cDNA,凍存于-80℃備用。
1.4.2 引物設計 根據NCBI Reference Sequence(NM_001032409.1、NM_016816.2、NM_002534.2),用DNAMAN分析同源序列(795 bp),在博邁德生物技術有限公司將序列合成到pBM19-B simple載體,并利用Primer 5.0軟件設計OAS、GAPDH上、下游引物,擴增片段長度分別為150、127 bp。2′,5′-OAS 上游引物為 5′-CAAGCTCAAGAG CCTCATCC-3′,下 游 引 物 為 5′-TGGGCTGTGTTG AAATGTGT-3′;GAPDH 上 游 引 物 為 5′-GAAGGT GAAGGTCGGAGTC-3′,下 游 引 物 為 5′-GAAGAT GGTGATGGGATTTC-3′。
1.4.3 精密度檢測 為了驗證QPCR方法的精密度,用質粒提取試劑盒提取OAS質粒,將質粒逆轉錄成cDNA后,用紫外分光光度計檢測其濃度、純度并計算其拷貝數。用1/10 TE將其稀釋到108拷貝/μL,保存在-20℃備用。將OAS質粒按1/10 梯度稀釋至 107、106、105、104、103、102拷貝/μL;另外將 OAS 質粒從 108拷貝/μL稀釋至 107、5×105、2.5×104、2.5×102、102作為質控樣品。20 μL QPCR反應體系包括模板 1 μL,SYBG 混合液 10 μL,上、下游 引 物(10 μmol/L 混 合 液)0.5 μL,用DEPC水補足。按說明書推薦程序進行檢測并分析該方法的準確度及精密度。
1.4.4 分析方法 用逆轉錄cDNA作為熒光定量PCR擴增的模板,用DEPC水將模板稀釋至30 ng/μL,上樣量分別為1、2 μL。反應體系包括SYBG混合液10 μL,上、下游引物(10 μmol/L混合液)0.5 μL,用 DEPC 水補至 20 μL。反應程序按說明書推薦進行(95℃ 5 min;95℃ 10 s,60℃ 30 s,40 個循環),熔解曲線分析。采用 2-ΔΔCt計算 2′,5′-OAS 相對表達倍數。
1.5 試劑盒(ELISA)檢測方法
根據試劑盒說明書,用標準品稀釋液將標準品梯度稀釋至200.0、100.0、50.0、25.0、12.5、6.25、3.125、1.563 U/mL;其中 200.0 U/mL同時為錨定點及定量上限,1.563 U/mL為錨定點,3.125 U/mL為定量下限。質控樣品濃度為150.0、20.0、9.4 U/mL。將每個個體相鄰的2個時間點的血清樣品混合后參照說明書進行檢測。
2 結果
2.1 引物特異性
用 2′,5′-OAS 及 GAPDH 基因的引物,以各樣品的cDNA為模板,用QPCR檢測樣品中這2個基因的表達情況,隨機挑選3個樣品的結果進行瓊脂糖凝膠電泳,檢測PCR擴增片段大小(圖1)。同時對每個QPCR反應孔的熔解曲線進行分析,結果顯示除了檢測結果Ct>33的某些樣品可能在72℃左右出現非特異峰,其他樣品的熔解曲線均在83±1℃左右單一峰(圖2),說明引物有良好的特異性,在整個反應體系中只有單一特異產物。
2.2 最佳模板量的確定
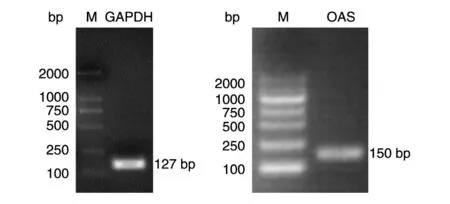
圖1 瓊脂糖凝膠電泳
用逆轉錄得到的cDNA樣品為模板進行PCR擴增,模板稀釋至30 ng/μL。為避免樣品處理過程中帶入的抑制性因素的影響,考察了不同上樣量對測樣的影響。結果顯示上樣量為1 μL時Ct值最小(圖3),因此確定樣品的上樣量為1 μL。
2.3 標準曲線線性、質控樣品靈敏度及精密度檢測
經過5次檢測,標準曲線的線性(R2)均在0.99以上,OAS質粒標準曲線的批間CV為5.1%~17.6%;質控樣品的批內精密度CV為0.2%~20%,批間CV為5.4%~18.5%,批內準確度RE為-31%~17%,批間準確度RE為-13.5%~9.8%。測樣過程中標準曲線的批間精密度為18%~25%,批間準確度為-11%~10%。

圖2 QPCR融解曲線
2.4 樣品檢測結果
在ELISA方法中,以給藥時間點為橫坐標、血清中2′,5′-OAS活性值為縱坐標做圖(圖4A),可以看到 160 μg劑量組(n=4)的 2′,5′-OAS在給藥后20 h左右微弱上升,180 μg劑量組(n=3)未見上升。在 QPCR結果中,用 2′,5′-OAS相對藥前值計算的相對表達變化倍數對時間做圖(圖4B),160 μg劑量組(n=4)2′,5′-OAS的相對表達倍數在給藥后明顯上升,12 h左右達到峰值,之后開始下降,192 h左右降低到基準值左右;180 μg劑量組(n=3)給藥后也有微弱上升,并在9 h左右上升的峰值,15 h左右降低到基準水平。因個體差異很大,2組數據間無統計學意義,但2種分析方法得到的結果趨勢是一致的。
3 討論

圖3 上樣量確定實驗

圖4 臨床Ⅰa期試驗中受試組(160 μg/人)和派羅欣組(180 μg/人)2′,5′-OAS生物標志物-時間圖
利用分子手段檢測生物標志物已經成為一種趨勢,并且隨著PCR技術的發展,更加精密、靈敏、高通量的PCR檢測方法被開發及用于臨床檢測,如數字PCR[17-18]、seminest-PCR、多重熒光定量PCR等。對于含量極低、容易降解、在蛋白水平檢測時受基質影響較大,以及其他不能在蛋白水平檢測的生物標志物,均可以嘗試在核酸水平對其進行檢測。在本實驗中,我們分別用QPCR和ELISA法對相同個體、相同給藥方法的血清樣品、全血樣品提取的RNA進行檢測并比較藥時關系。
核酸翻譯蛋白質的效率在不同個體間存在差異,本研究對標志物核酸水平和蛋白水平比較的結果,2種檢測方法得到的單個個體的藥時變化趨勢及劑量依賴性趨勢是一致的,甚至在QP?CR分析方法基礎上,獲得了給藥后48 h更多的變化水平信息(盡管因個體差異大無統計學差異),提示QPCR法相比較于ELISA法更加適用于檢測變化相對微弱但很關鍵的一些指標。
對于相對定量PCR來說,需要用對照樣品、內參基因進行歸一,通過2-ΔΔCt計算實驗樣品待測基因相對于對照樣品的相對表達倍數變化,會顯著提高樣品組與對照組之間的差異,并且與樣品內參基因、靶基因檢測出的Ct值有關。例如,若靶基因的Ct值在34甚至以上,理論上靶基因的表達量很低,但若內參基因的Ct也相對較高如為32,則按 2-ΔΔCt計算出的相對表達倍數很高,但理論上該樣品是不能滿足QPCR檢測標準的。因此,在本研究中,我們在保證大部分樣品可用的前提下,將GAPDH的可接受Ct值限定為≤30,若某個樣品的Ct值在30以上,則考察其樣品質量或通過重新提取、加大上樣量等方式將Ct值控制在30以內后再檢測該樣品的靶基因計算其表達倍數。通過在整個實驗中分別固定2個基因的閾值,也可以在每個分析批中均加上對照樣品,并且以同一批中對照樣品的檢測結果來計算相對表達倍數;或者建立標準曲線以及質控樣品來調整分析批批間的系統誤差,若采用該種方法須界定標準曲線及質控樣品的接受標準。這3種方法都可以降低試劑、儀器、人為等各種系統誤差引起的數據不可靠的情況。
基于RT-QPCR的分析方法與常規進行基因表達的研究性課題不同,檢測體系中抑制因素的存在、氣溶膠污染是重要的影響因素。QPCR靈敏度極高,容易受到氣溶膠的污染,因此QPCR檢測對環境的要求較高,最好在通風櫥或生物安全柜中配置檢測體系,在檢測過程中一定要設置陰性對照,實驗前后均做好清潔,保證環境中沒有氣溶膠。一般情況下,檢測樣品時每個基因會做2~3個技術重復并根據儀器系統自帶的復孔間Ct值的標準偏差決定是否接受檢測結果(本研究采用的儀器為ABI 7500 fast,儀器自動將SD設為≤0.50)。但低濃度樣品如標準品100拷貝/μL,由于泊松分布原理造成了就單個分析批檢測值來看其Ct值精密度較差,復孔間Ct可能相差1以上,但如果增加低濃度樣品的重復數,從多個分析批結果可以發現低濃度樣品的平均Ct值很接近,STDEV在0.5以內,說明在樣品濃度較低時,在泊松分布、氣溶膠污染、加樣手法等因素下2~3個技術重復很難保證復孔間的SD維持在較小的值,但增加復孔數后,雖然批內SD較大,但批間平均Ct的SD滿足要求(<0.5),更加準確地反應樣品的真實情況。因此,在設計實驗時,需要根據實驗目的、測樣要求、經濟因素等優化實驗方法,確定數據處理原則,做到具體問題具體分析,根據實驗目的設計并優化實驗方法,以期獲得最接近真實水平的結果。
[1]張成偉,周乃康.生物標記物在非小細胞肺癌個體化治療中的研究進展[J].中國醫院用藥評價與分析,2010,10(2):189-192.
[2] Zonta E,Garlan F,Pécuchet N,et al.Multiplex detec?tion of rare mutations by picoliter droplet based digi?talPCR:sensitivity and specificity considerations[J].PLoS One,2016,11(7):e0159094.
[3] Bian C,Li Z,Xu Y,et al.Clinical outcome and ex?pression of mutant P53,P16,and Smad4 in lung ade?nocarcinoma:a prospective study[J].World J Surg On?col,2015,13:128.
[4] Zheng X,Andruska N,Lambrecht M J,et al.Target?ing multidrug-resistant ovarian cancer through estro?gen receptor α dependent ATP depletion caused by hyperactivation of the unfolded protein response[J].On?cotarget,2016,doi:10.18632/oncotarget.10819.
[5] Lindsey J C,Lusher M E,Strathdee G,et al.Epigene?tic inactivation of MCJ(DNAJD1)in malignant paediat?ric brain tumours[J].Int J Cancer,2006,118(2):346-352.
[6] Lin Y,Wang F,Zhang G L.Natural products and their derivatives regulating the janus kinase/signal transducer and activator of transcription pathway[J].J Asian Nat Prod Res,2014,16(7):800-812.
[7] Fan W,Xu L,Ren L,et al.Functional characteriza?tion of canine interferon-lambda[J].J Interferon Cyto?kine Res,2014,34(11):848-857.
[8] MacQuillan G C,Mamotte C,Reed W D,et al.Upreg?ulation of endogenous intrahepatic interferon stimulat?ed genes during chronic hepatitis C virus infection[J].J Med Virol,2003,70(2):219-227.
[9] Sung C,Nardelli B,LaFleur D W,et al.An IFN-be?ta-albumin fusion protein that displays improved phar?macokinetic and pharmacodynamic properties in nonhu?man primates[J].J Interferon Cytokine Res,2003,23(1):25-36.
[10]Hu X,Miller L,Richman S,et al.A novel PEGylat?ed interferon beta-1a formultiple sclerosis:safety,pharmacology,and biology[J].J Clin Pharmacol,2012,52(6):798-808.
[11]Myzithras M,Li H,Bigwarfe T,et al.Development of an ultra-sensitive Simoa assay to enable GDF11 detec?tion:a comparison across bioanalyticalplatforms[J].Bioanalysis,2016,8(6):511-518.
[12]Collet-Brose J,Couble P J,Deehan M R,et al.Evalu?ation of multiple immunoassay technology platforms to selectthe anti-drug antibody assay exhibiting the most appropriate drug and target tolerance[J].J Immu?nol Res,2016,2016:5069678.
[13]Jordan G,Moheysen-Zadeh M,Heinrich J,et al.Plat?form switching from ELISA to Gyrolab:a novel gener?ic reagent omits the need to change critical reagents[J].Bioanalysis,2016,8(8):807-814.
[14]Frankel M,Forberg K,Coller K E,et al.Develop?ment of a high-throughput multiplexed real time RTPCR assay for detection of human pegivirus 1 and 2[J].J Virol Methods,2016,241:34-40.
[15]Bruisson S,Lebel S,Walter B,et al.Comparative de?tection of a large population of grapevine viruses by TaqManRT-qPCR andELISA[J].JVirolMethods,2017,240:73-77.
[16]Otsuji K,Sasaki T,Tanaka A,et al.Use of droplet digital PCR for quantitative and automatic analysis of the HER2 status in breast cancer patients[J].Breast Cancer Res Treat, 2017,162(1):11-18.
[17]Ponnampalam S N,Kamaluddin N R,Zakaria Z,et al.A blood-based gene expression and signaling path?way analysisto differentiate between high and low grade gliomas[J].Oncol Rep,2017,37(1):10-22.

