基于TCGA數據庫的乳腺癌進程關鍵基因系統篩選和鑒定
馮 旭, 趙麗敏, 柯 浩, 張洪磊
(1.昆明理工大學 生命科學與技術學院 云南 昆明 650500; 2.中國科學院昆明動物研究所 遺傳資源與進化國家重點實驗室 云南 昆明 650223; 3.中國科學院大學 昆明生命科學學院 云南 昆明 650500)
DOI: 10.13705/j.issn.1671-6841.2017121
基于TCGA數據庫的乳腺癌進程關鍵基因系統篩選和鑒定
馮 旭1, 趙麗敏2,3, 柯 浩2,3, 張洪磊2
(1.昆明理工大學 生命科學與技術學院 云南 昆明 650500; 2.中國科學院昆明動物研究所 遺傳資源與進化國家重點實驗室 云南 昆明 650223; 3.中國科學院大學 昆明生命科學學院 云南 昆明 650500)
利用 TCGA 數據庫收集的乳腺癌轉錄組數據和病人臨床信息,分析基因表達與乳腺癌臨床病理學參數的相關性及其對預后的影響,同時結合表達差異分析篩選出與乳腺癌進程密切相關的7個候選基因(UBE2T、RRM2、SGOL1、ERCC6L、CCNE1、HMGB3和SPDYC).通過基因功能注釋檢索確定SGOL1作為實驗驗證的候選基因.實驗結果表明在乳腺癌細胞系MDA-MB-231中敲降SGOL1能顯著抑制細胞生長,細胞周期被阻滯在G2/M期.上述結果表明SGOL1在乳腺癌進程中具有重要作用,同時也說明該生物信息結合實驗的篩選方法可以加速腫瘤關鍵基因的篩選和鑒定.
轉錄組; TCGA數據庫;SGOL1
DOI: 10.13705/j.issn.1671-6841.2017121
0 引言
乳腺癌是我國婦女最常見的癌癥,我國新發乳腺癌的病例占全球乳腺癌新發病例的 12.2%[1].作為目前最大的癌癥基因信息的數據庫,TCGA收錄了包括29種原位腫瘤類型,涉及基因組、轉錄組、蛋白質組及表觀遺傳等多層次的腫瘤數據,圍繞TCGA數據庫的研究提高了人們對腫瘤發病分子基礎的科學認識,同時提高我們診斷、治療和預防腫瘤的能力.這里我們結合臨床病理特征、分子分型、生存分析、基因富集分析(GSEA)等生物信息手段,篩選影響乳腺癌進程的重要候選基因.在有絲分裂期間,染色體分離的保真度取決于對姐妹染色單體分離的精確控制,姐妹染色單體內聚的維持可以確保姐妹染色體正確分離[2-3].而有絲分裂的異常與腫瘤的發生存在密切關系[4],SGOL1可以維持著絲粒凝聚素從前期直至后期轉變[5],是染色體穩定的守護者[6].已有研究發現在胰腺癌中SGOL1呈現過度表達[7-8].而在結腸癌中敲降SGOL1可以引起細胞周期阻滯在G2/M期,導致細胞凋亡以及造成染色體不穩定[3,9].在肝癌細胞中SGOL1的異常表達可以導致有絲分裂延遲,最終導致細胞死亡[10].SGOL1是有絲分裂中必不可少的蛋白質,本文通過對TCGA數據庫中乳腺癌數據集的分析,確定了7個與乳腺癌進程密切相關的基因.確定SGOL1作為實驗功能驗證的候選基因,進一步的實驗結果顯示SGOL1在癌組織中高表達,在MBA-MB231中敲降SGOL1能顯著抑制細胞生長,細胞周期被阻滯在G2/M期,上述結果表明SGOL1能在乳腺癌進程中有重要作用.
1 材料與方法
1.1 TCGA數據下載
登陸firehose(http://gdac.broadinstitute.org/)網站下載公開的乳腺癌(BRCA)轉錄組數據(RNASeqV2:https://wiki.nci.nih.gov/display/TCGA/RNASeq+Version+2)和腫瘤樣本臨床數據(數據版本:2016_01_28).其中:癌癥樣本1 093例;正常癌旁組織樣本112例.
1.2 乳腺癌樣本分子分型
利用乳腺癌轉錄組數據集進行分子分型.首先通過Limma軟件包的lowess-fit方法對基因的原始表達量進行歸一化處理[11-12].然后使用Genefu軟件包對乳腺癌樣本進行PAM50分子分型[13-14].
1.3 基因表達與臨床病理學參數的相關性分析
使用survivalROC法將樣本分為兩組,基因表達邊界值為3.255 207,低于該值為低表達組,高于該值為高表達組,結合TCGA的臨床病理學參數(TNM分期)和乳腺癌分子分型結果,使用χ2檢驗檢測基因表達與臨床病理學參數的相關性.
1.4 生存曲線分析
使用survivalROC法將樣本分為兩組,基因表達邊界值為3.255 207,低于該值為低表達組,高于該值為高表達組,使用R語言survival軟件包繪制總體生存曲線.
1.5 GSEA基因富集分析
乳腺癌樣本根據基因表達水平的高低分為兩組.數據處理使用默認參數.利用FDRlt;0.25,Plt;0.01作為標準鑒定顯著富集信號通路.
1.6 基因表達差異分析
使用“RNASeqV2”數據中RSEM輸出的reads count評估基因表達水平,采用Limma軟件包中voom法[15]進行均一化處理,并用貝葉斯模型進行表達差異分析,篩選logFCgt;2的基因為表達差異基因.
1.7 細胞來源、主要試劑及儀器
MDA-MB-231細胞購自中科院昆明動物所細胞庫,siRNA購自廣州瑞博公司,DMEM/F12培養基、胎牛血清(Gibco公司),Trizol總RNA提取試劑盒(Invitrogen公司),脂質體轉染試劑盒Lipfectamine 2000,PrimeScriptTMRT Reagent反轉錄試劑盒(TaKaRa),Annexin V-FITC細胞凋亡檢測試劑盒、FACSAria流式細胞儀(BD公司).
1.8 細胞培養及 siRNA 轉染
細胞使用DMEM/F12完全培養基(DMEM/F12+10%胎牛血清+100-U/ml青霉素/鏈霉素),在37 ℃,5%CO2培養箱中培養.待細胞貼壁生長,取對數生長期細胞,利用脂質體轉染試劑盒Lipfectamine 2000進行轉染處理.
1.9 RT-PCR
采用Trizol法提取總RNA,使用PrimeScriptTMRT Reagent反轉錄試劑盒(TaKaRa)合成cDNA,PCR反應條件為:預變性94 ℃ 10 s;變性94 ℃ 5 s;退火/延伸60 ℃ 34 s;共40個循環;以β-Actin作為相對定量內參,采用2-ΔΔCT法計算基因相對表達量[28].
1.10 細胞生長實驗
采用不同siRNA轉染MDA-MB-231細胞,轉染24 h后用胰蛋白酶將細胞消化并計數,按每孔2×105個細胞接種于6孔板,置于37 ℃培養箱培養.分別在細胞培養36 h、60 h、84 h時采用胰蛋白酶消化細胞并計數.
1.11 凋亡實驗
MDA-MB-231用不同siRNA處理72 h后,胰酶消化細胞,并用PBS洗兩次,用染色緩沖液重懸細胞至每毫升1×106個細胞.取100 μl細胞(1×105),加入5 μl AnnexinV-FITC和5 μl PI室溫下避光孵育15 min左右后,用流式細胞儀檢測細胞凋亡.
1.12 周期實驗
MDA-MB-231用不同siRNA處理72 h后,采用無血清培養基饑餓處理24 h,之后采用完全培養基繼續培養12 h,胰酶消化并收集細胞.加入1 ml預冷的70%乙醇中,吹打使細胞分散成單個細胞,4 ℃固定2 h.1 000 g離心3~5分鐘,沉淀細胞.棄上清,用1 ml預冷PBS洗1遍.PI染色,每個樣品用0.5 ml染色緩沖液重懸,加入25 μl PI染色液(20×)、10 μl RNase A(50×).37 ℃避光孵育30 min后,采用流式細胞儀檢測細胞周期.
2 結果
2.1 篩選影響乳腺癌進程的候選基因
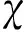

表1 SGOL1與病理學參數相關性
A表示TCGA 數據庫中篩選乳腺癌進程相關基因的流程; B表示GSEA分析顯示SGOL1的差異表達與細胞 周期過程相關; C、D: 生存分析(左圖數據來源于TCGA乳腺癌數據、右圖數據來源于KM-plotter乳腺癌數據)圖1 SGOL1與乳腺癌進程相關Fig.1 SGOL1 is associated with breast cancer progression
2.2 SGOL1在乳腺癌樣本中高表達
首先檢測了SGOL1在人正常永生化的乳腺上皮細胞系(MCF10A)和人乳腺癌細胞系中的表達量,發現在SGOL1乳腺癌細胞系中表達偏高(圖2A).之后檢測了癌組織以及癌旁正常組織中SGOL1的表達量,發現除了7號和13號樣本中SGOL1的表達量略低于正常樣本(圖2B),在其余樣本中均發現SGOL1在癌組織中高表達.這些結果表明SGOL1在乳腺癌進程中很可能有重要作用.

A表示qRT-PCR檢測不同乳腺癌細胞系中SGOL1基因mRNA表達水平; B表示不同例乳腺癌組織與正常乳腺組織SGOL1基因mRNA相對 表達量; C表示在MDA-MB-231乳腺癌細胞系中檢測其敲降效率; D表示不同時間點檢測敲降SGOL1基因對MDA-MB-231細胞系生長的影響圖2 SGOL1在乳腺癌中高表達Fig.2 SGOL1 is highly expressed in breast cancer
2.3 采用siRNA敲降SGOL1
為了檢驗SGOL1與乳腺癌的關系,我們通過siRNA來敲降SGOL1(圖2C),設計了3對siRNA,結果顯示si-SGOL1-2、si-SGOL1-3都有很好的敲降效果.
2.4 敲降SGOL1抑制細胞生長
采用不同siRNA處理MDA-MB-231細胞后,我們發現與si-Control組相比較,除了si-SGOL1-1(無明顯敲降效果),si-SGOL1-2和si-SGOL1-3后,細胞數目都明顯低于si-Control組(圖2D).這些結果證明敲降SGOL1能夠抑制MDA-MB-231細胞生長.
2.5 抑制G2/M細胞周期但不影響細胞凋亡.
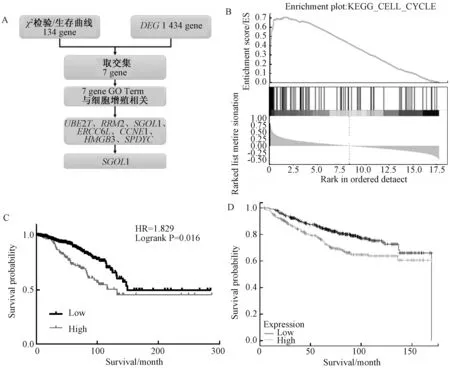
我們前期的實驗結果發現敲降SGOL1能顯著抑制細胞生長,因此我們開始探索敲降SGOL1后通過什么途徑來影響細胞生長.敲降SGOL1后,首先檢測了細胞周期的變化,實驗結果發現,敲降SGOL1可使細胞G2/M期顯著增加(圖3).
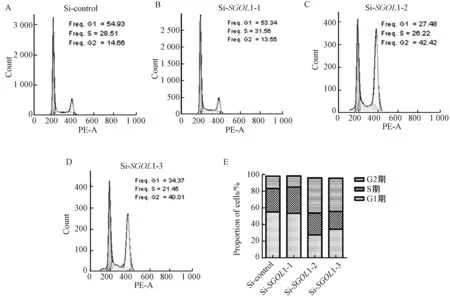
A表示對照組中細胞周期; B、C、D表示siRNA敲降SGOL1的細胞周期; E表示細胞周期的統計結果圖3 敲降SGOL1后用流式細胞儀檢測細胞周期Fig.3 Knock down SGOL1 and then use flow cytometry to detect cell cycle
之后我們采用凋亡試劑盒來檢測細胞凋亡,發現敲降SGOL1凋亡細胞雖然比control組略微有所上升(圖4),但并沒有顯著的差異.因此在乳腺癌中SGOL1很可能主要是通過影響細胞周期來影響細胞增殖的.
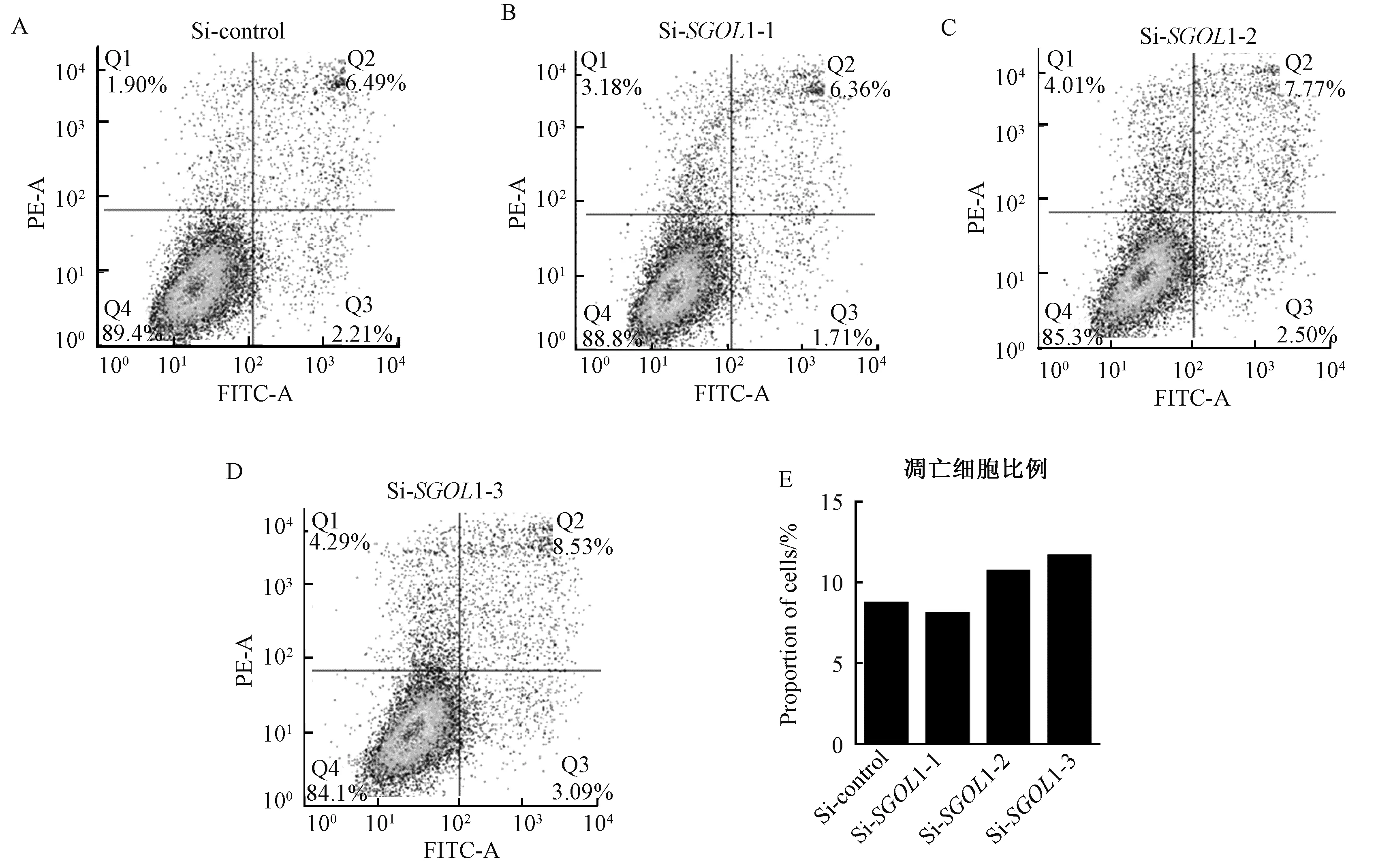
A表示對照組中細胞凋亡結果;B、C、D表示siRNA敲降SGOL1的細胞凋亡結果;E表示細胞凋亡的統計結果圖4 Annexin V/PI雙染法檢測敲降SGOL1對MDA-MB-231細胞凋亡的影響Fig.4 Annexin V/PI double staining method to detect knockdown of SGOL1 on MDA-MB-231 cell apoptosis
3 討論
由TCGA的臨床數據統計可知SGOL1的表達與癌癥大小、遠端轉移、癌癥分級以及癌癥分子分型密切相關.總體生存曲線顯示該基因高表達患者相較于低表達患者生存期顯著縮短.基因共表達網絡分析顯示,在乳腺癌中該基因所在的共表達模塊可能與細胞周期相關,從而影響癌癥的進程,GSEA分析可知SGOL1的表達差異與細胞周期和有絲分裂細胞通路相關.作為進化保守的蛋白質,SGOL1是有絲分裂過程中染色體準確分離所必需的.文獻[23]表明哺乳動物細胞中敲降SGOL1導致染色體分離異常,引起遺傳不穩定和腫瘤轉化.文獻[24]發現通過短肽競爭抑制SGOL1的功能能夠促進癌癥凋亡.結腸癌中該基因的下調使遺傳不穩定進而影響癌癥的發展過程,然而乳腺癌中SOGL1的表達水平卻是上升的,該基因的高表達同樣伴隨大量的染色體不穩定現象[25-26].因此,SOGL1在乳腺癌癥中控制染色體穩定性的分子機制可能不同于其他癌癥類型.已有研究發現SGOL1對細胞凋亡和細胞周期都有影響[10],但是在乳腺癌中,我們發現SGOL1并不影響細胞的凋亡,但是能顯著影響細胞周期.
乳腺癌是最常見的癌癥之一,且近年來的發病人數逐年增加[27].近年來精準治療作為癌癥治療的探索新方向,本項研究發現,乳腺癌中敲低SGOL1能夠有效抑制癌細胞生長、致使細胞周期停滯在G2/M期,這能為癌癥治療提供了新的藥物靶標.
[1] FAN L, STRASSERWEIPPL K, LI J J, et al. Breast cancer in China[J]. Lancet oncology,2014, 15(7): 279-289.
[2] WANG L H, MAYER B, STEMMANN O, et al. Centromere DNA decatenation depends on cohesin removal and is required for mammalian cell division[J]. Journal of cell science,2010, 123(5): 806-813.
[3] YAMADA H Y, YAO Y, WANG X, et al. Haploinsufficiency of SGO1 results in deregulated centrosome dynamics, enhanced chromosomal instability and colon tumorigenesis[J]. Cell cycle,2012, 11(3): 479-488.
[4] FUNK L C, ZASADIL L M, WEAVER B A. Living in CIN: mitotic infidelity and its consequences for tumor promotion and suppression[J]. Developmental cell,2016, 39(6): 638-652.
[5] GIMENEZ-ABIAN J F, DIAZ-MARTINEZ L A, WIRTH K G, et al. Regulated separation of sister centromeres depends on the spindle assembly checkpoint but not on the anaphase promoting complex/cyclosome[J]. Cell cycle,2005, 4(11): 1561-1575.
[6] DIAZ-MARTINEZ L A, YU H. Sgo1 as a guardian of chromosome stability[J]. Cell cycle,2012, 11(4): 650-651.
[7] CUTTS R J, GADALETA E, HAHN S A, et al. The Pancreatic Expression database: 2011 update[J]. Nucleic acids research,2011, 39: D1023-1028.
[8] ANDRE F, MICHIELS S, DESSEN P, et al. Exonic expression profiling of breast cancer and benign lesions: a retrospective analysis[J]. Lancet oncol,2009, 10(4): 381-390.
[9] IWAIZUMI M, SHINMURA K, MORI H, et al. Human Sgo1 downregulation leads to chromosomal instability in colorectal cancer[J]. Gut,2009, 58(2): 249-260.
[10] WANG L H, YEN C J, LI T N, et al. Sgo1 is a potential therapeutic target for hepatocellular carcinoma[J]. Oncotarget,2015, 6(4): 2023-2033.
[11] KATZ Y, LI F, LAMBERT N J, et al. Musashi proteins are post-transcriptional regulators of the epithelial-luminal cell state[J]. Elife,2014, 3: e03915.
[12] RITCHIE M E, PHIPSON B, WU D, et al. Limma powers differential expression analyses for RNA-sequencing and microarray studies[J]. Nucleic acids research,2015, 43(7): e47.
[13] FUMAGALLI D, BLANCHET-COHEN A, BROWN D, et al. Transfer of clinically relevant gene expression signatures in breast cancer: from Affymetrix microarray to Illumina RNA-sequencing technology[J]. BMC genomics,2014, 15(1): 1008.
[14] GENDOO D M, RATANASIRIGULCHAI N, SCHRODER M S, et al. Genefu: an R/Bioconductor package for computation of gene expression-based signatures in breast cancer[J]. Bioinformatics,2016, 32(7): 1097-1099.
[15] LAW C W, CHEN Y, SHI W, et al. Voom: precision weights unlock linear model analysis tools for RNA-seq read counts[J]. Genome biology,2014, 15(2): R29.
[16] HEAGERTY P J, LUMLEY T, PEPE M S. Time-dependent ROC curves for censored survival data and a diagnostic marker[J]. Biometrics,2000, 56(2): 337-344.
[17] UEKI T, PARK J H, NISHIDATE T, et al. Ubiquitination and downregulation of BRCA1 by ubiquitin-conjugating enzyme E2T overexpression in human breast cancer cells[J]. Cancer research,2009, 69(22): 8752-8760.
[18] PUTLURI N, MAITY S, KOMMAGANI R, et al. Pathway-centric integrative analysis identifies RRM2 as a prognostic marker in breast cancer associated with poor survival and tamoxifen resistance[J]. Neoplasia,2014, 16(5): 390-402.
[19] PU S Y, YU Q, WU H, et al. ERCC6L, a DNA helicase, is involved in cell proliferation and associated with survival and progress in breast and kidney cancers[J]. Oncotarget,2017, 8(26): 42116-42124.
[20] LUHTALA S, STAFF S, TANNER M, et al. Cyclin E amplification, over-expression, and relapse-free survival in HER-2-positive primary breast cancer[J]. Tumour biology,2016, 37(7): 9813-9823.
[21] GUO S, WANG Y, GAO Y, et al. Knockdown of high mobility group-box 3 (HMGB3) expression inhibits proliferation, reduces migration, and affects chemosensitivity in gastric cancer cells[J]. Medical science monitor international medical journal of experimental and clinical research,2016, 22: 3951-3960.
[22] CHENG A, SOLOMON M J. Speedy/Ringo C regulates S and G2 phase progression in human cells[J]. Cell cycle,2008, 7(19): 3037-3047.
[23] WANG X, YANG Y, DAI W. Differential subcellular localizations of two human Sgo1 isoforms: implications in regulation of sister chromatid cohesion and microtubule dynamics[J]. Cell cycle,2006, 5(6): 635-640.
[24] YANG Y, WANG X, DAI W. Human Sgo1 is an excellent target for induction of apoptosis of transformed cells[J]. Cell cycle,2006, 5(8): 896-901.
[25] TANNO Y, SUSUMU H, KAWAMURA M, et al. The inner centromere-shugoshin network prevents chromosomal instability[J]. Science,2015, 349(6253): 1237-1240.
[26] RADFORD D M, FAIR K L, PHILLIPS N J, et al. Allelotyping of ductal carcinoma in situ of the breast: deletion of loci on 8p, 13q, 16q, 17p and 17q[J]. Cancer research,1995, 55(15): 3399-3405.
[27] ZUO T T, ZHENG R S, ZENG H M, et al. Female breast cancer incidence and mortality in China, 2013[J]. Thoracis cancer,2017, 8(3): 214-218.
[28] 張芬. MicroRNA-124a對SCI繼發性損傷的影響及相關機制研究[J]. 鄭州大學學報(理學版), 2016, 48(1): 74-80.
(責任編輯:方惠敏)
AStudyofSystematicScreeningandIdentificationofEssentialGenesInvolvedinBreastCancerProgressionBasedonTCGADatabase
FENG Xu1, ZHAO Limin2,3, KE Hao2,3, ZHANG Honglei2
(1.FacultyofLifeScienceandTechnology,KunmingUniversityofScienceandTechnology,Kunming650500,China; 2.StateKeyLaboratoryofGeneticResourcesandEvolution,KunmingInstituteofZoology,ChineseAcademyofSciences,Kunming650223,China; 3.KunmingCollegeofLifeScience,UniversityofChineseAcademyofSciences,Kunming650500,China)
Based on the expression profile data and clinical information of breast cancer cohort in the TCGA database, the correlations between gene expression and clinical parameters and differential expression analysis were carried out. Seven genes (UBE2T,RRM2,SGOL1,ERCC6L,CCNE1,HMGB3 andSPDYC) which were closely related to the breast cancer progression were dentified. After gene function annotation surveySGOL1 was selected as one candidate gene for our experimental validation. Results showed that knockdown ofSGOL1 in breast cancer cell line MDA-MB-231 significantly inhibited cell growth and cell cycle was arrested in G2/M phase. These results suggested thatSGOL1 played an important role in the breast cancer progression and meanwhile the method of combining bioinformatics and experiment provided an efficient way for screening and identification of tumor essential genes.
transcriptome; TCGA database;SGOL1
2017-05-04
科技部重點研發計劃項目(2016YFA0100900);云南省應用基礎研究計劃面上項目(2016FB038).
馮旭(1993—),男,陜西渭南人,主要從事生物信息研究,E-mail:cheerfulloster@protonmail.com;通信作者:張洪磊(1983—),男,山東煙臺人,助理研究員,主要從事表觀遺傳、生物信息研究,E-mail:hlzhang2014@163.com.
R857.3
A
1671-6841(2017)04-0093-07

