大瀧六線魚6個野生群體遺傳多樣性的12S rRNA基因分析
王偉 ,高偉峰 ,張賽賽 ,2,董安然 ,姜欣彤
(1.大連海洋大學農業部北方海水增養殖重點實驗室,遼寧 大連 116023;2.天津現代晨輝科技集團有限公司,天津 301800)
大瀧六線魚6個野生群體遺傳多樣性的12S rRNA基因分析
王偉1,高偉峰1,張賽賽1,2,董安然1,姜欣彤1
(1.大連海洋大學農業部北方海水增養殖重點實驗室,遼寧 大連 116023;2.天津現代晨輝科技集團有限公司,天津 301800)
本文利用PCR技術擴增了6個野生群體大瀧六線魚線粒體12SrRNA基因的部分序列。在獲得的582bp堿基序列中,共檢測到314個變異位點,占總位點數的53.95%。34尾個體共定義了16種單倍型,6個群體的單倍型多樣性指數為0.40~1.00,核苷酸多樣性指數為0.00071~0.10441。中性檢驗Fu's Fs值為1.19782(P<0.01)。分子變異分析表明:Fst=0.88326(P<0.01),群體間和群體內變異分別為88.33%和11.67%。不同個體間的遺傳距離構建的NJ和MP系統樹表明,東港和旅順群體親緣關系較近,構成一個分支,其他群體構成另一分支。
大瀧六線魚;野生群體;線粒體DNA;12SrRNA部分序列
大瀧六線魚Hexagrammos otakii Jordan&Starks也被稱為歐氏六線魚、六線魚等[1,2],屬鲉形目Scorpaeniformes、六線魚科Hexagrammidae、六線魚屬Hexagrammos[2],俗稱黃魚,廣泛分布于我國東部沿海及日本、韓國、朝鮮等國的近海[3,4],在我國主要分布于遼寧和山東等地近海的多巖礁海區。大瀧六線魚味道鮮美,營養豐富,深受人們的青睞,還可用于出口創匯,具有極高的經濟價值,在我國北方的海水養殖經濟魚類中占有重要地位。早在20世紀70年代,日本便對其開展人工繁殖研究,我國也從20世紀80年代開始大瀧六線魚的人工育苗[5-8]、營養成分分析[9,10]、生物學指標[11,12]等研究。然而關于大瀧六線魚的遺傳多樣性的研究較少,目前國內僅見劉奇[13]的遺傳多樣性研究。
魚類種群的種質資源評估對漁業資源的合理開發和利用具有重要的導向作用。在魚類種質資源的相關研究中,檢測線粒體DNA(mtDNA)多態性是一種較為普遍方式。線粒體DNA的核苷酸分子較小,易與核DNA分離和純化,進化速率較快,是其他單拷貝DNA的5~10倍。同一合子來源的個體各組織器官內的mtDNA結構均一,每個魚類個體中所含的mtDNA具有高度的一致性[14]。遺傳中遵循嚴格的母系遺傳規律,通常不會發生重組,容易確定其種質特征,反映群體遺傳特征、種群分化和種屬關系等特點。因此,作為一種較為常用的遺傳分子標記,廣泛應用于系統進化和種群遺傳研究中[15,16]。12SrRNA基因位于線粒體基因組,具有較高的保守進化性,多用于系統發生及分子進化等研究[17,18]。但該基因較少應用于研究魚類群體遺傳多樣性等。本試驗分析6個群體大瀧六線魚線粒體DNA中12S rRNA序列的遺傳結構、遺傳多樣性和遺傳分化等,有助于了解不同大瀧六線魚群體的遺傳多樣性及其遺傳背景,為合理利用漁業資源以及資源的保護和可持續開發,提供一定的理論基礎。
1 材料與方法
1.1 樣本采集
試驗用魚分別采自青島、丹東和大連等6個地區,為保證采樣群體的準確性,樣品均采自各地的采樣點碼頭,樣品規格為(20±1.5)cm。樣本采集相關信息詳見圖1和表1。
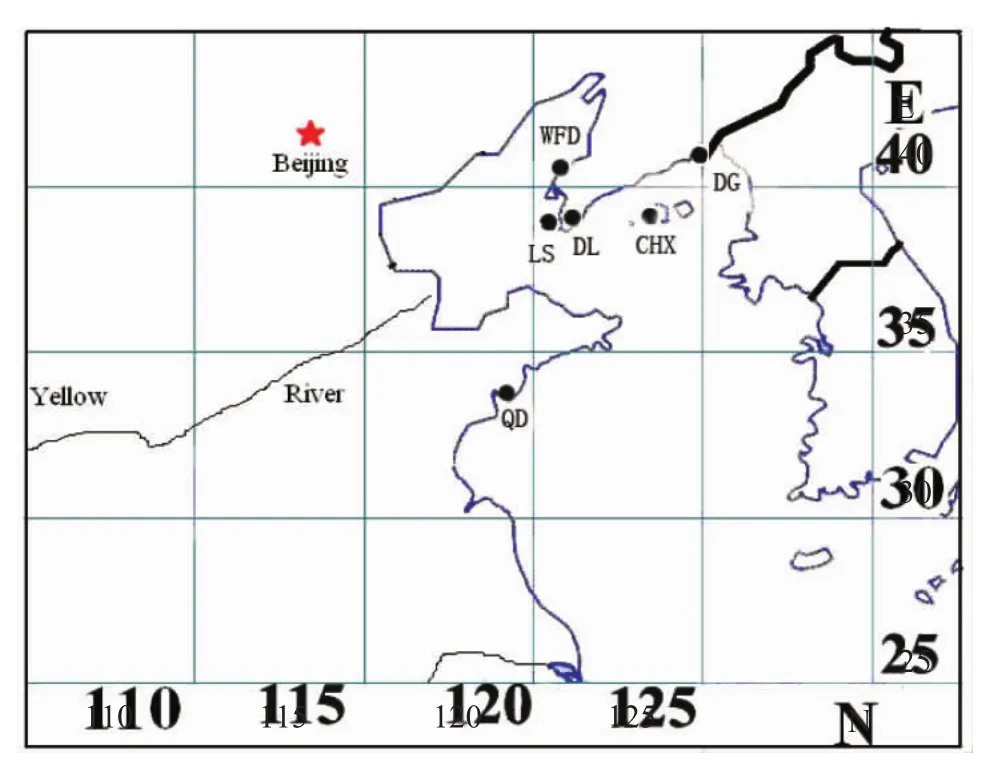
圖1 大瀧六線魚采集地點Fig.1 Sampling sites of fat greenling Hexagrammos otakii

表1 大瀧六線魚樣品信息Tab.1 Sample information of fat greenling Hexagrammos otakii
1.2 基因組DNA提取
DNA提取采用本實驗室常用的一種簡化方法進行。從魚尾部取約50mg肌肉組織樣品,充分剪碎,加入蛋白酶K、勻漿液以及SDS,經過大約2h的完全消化,使用C2H3KO2溶液進行抽提,加入無水乙醇形成沉淀,用75%酒精洗滌沉淀物,再用適量TE溶解,于-20℃下保存備用[19]。
1.3 12S rRNA序列的PCR擴增
從NCBI(National Center for Biotechnology Information)的GenBank中檢索大瀧六線魚12S rRNA基因同源序列,運用Primer Primer 5軟件設計PCR擴增引物、上游引物(5’-CAGAGCGTGGCTAAGATGGA-3’) 和 下 游 引 物(5’-CAGAGCGTGGCTAAGATGGA-3’)。使用約50ng基因組DNA作為PCR反應的模板。反應體系中包含:1.0mmol/LMgCl2、2μL基 因 組 DNA、0.2mmol/L dNTPs、2.5μL 10 ×PCR Buffer、引物各 0.4μmol/L、1U Taq 酶,PCR 反應的總體積為25μL。將擴增體系于95℃下3min預變性,然 后 94℃ 變 性 30s,56℃ 退 火 45s,72℃ 延 伸1min,35個循環結束后,72℃延伸10min。擴增產物采用瓊脂糖凝膠試劑盒進行回收純化。PCR產物委托上海鼎安生物科技有限公司采用雙向測序法進行測序,依據峰圖對測序的結果進行確認。
1.4 數據分析
采用ClustalX1.83軟件對所得的序列進行對位排列,并結合人工校正;采用Mega4.10軟件核對堿基組成及相關的變異位點,計算遺傳距離,構建NJ和MP分子系統樹[20];采用DNASP4.10軟件計算核苷酸多樣性指數(π)、單倍型多樣性指數(Hd)及單倍型數(h);采用Arlequin3.11軟件進行中性檢驗,計算Fu's Fs值推斷大瀧六線魚各個群體在歷史上發生種群擴張的情況[21];最后結合分子變異分析方法(AMOVA)分析種群間的遺傳分化指數Fst,通過排列測驗法(permutation test)檢測Fst的顯著性。
2 結果與分析
2.1 序列特征
PCR擴增所得到的堿基序列長度約為700bp,比對得出34尾大瀧六線魚mtRNA中高度一致的12SrRNA序列,長度約為 582bp。A、T、G、C堿基的平均含量各為30.0%、27.0%、21.0%和22.0%。A+T含量(59.0%)高于G+C含量(41.0%)。旅順群體T+A含量最高(57.4%),長海縣群體最低(56.5%)。G+C含量則相反,長海縣群體最高(43.5%),旅順群體最低(42.6%)。
在34尾大瀧六線魚12S rRNA序列中,共檢測到314個變異位點,多態位點百分率為53.95%。其中單態位點53個,簡約信息位點261個,插入/缺失位點32個。共有轉換位點54個,其中C-T有28個位點,A-G有26個位點。顛換位點62個,四種形式均有,其中A-C有19個位點,T-G有12個位點,A-T有17個位點,G-C有14個位點。平均轉換與顛換之比為0.87。由表2可知,6個群體的34尾大瀧六線魚共定義16種單倍型。2個單倍型為不同群體所共享,僅占單倍型總數的12.5%,14種單倍型均為單個群體所特有,在單倍型總數中占87.5%,僅有1個為4個種群的共有單倍型,1個為2個種群的共有單倍型。
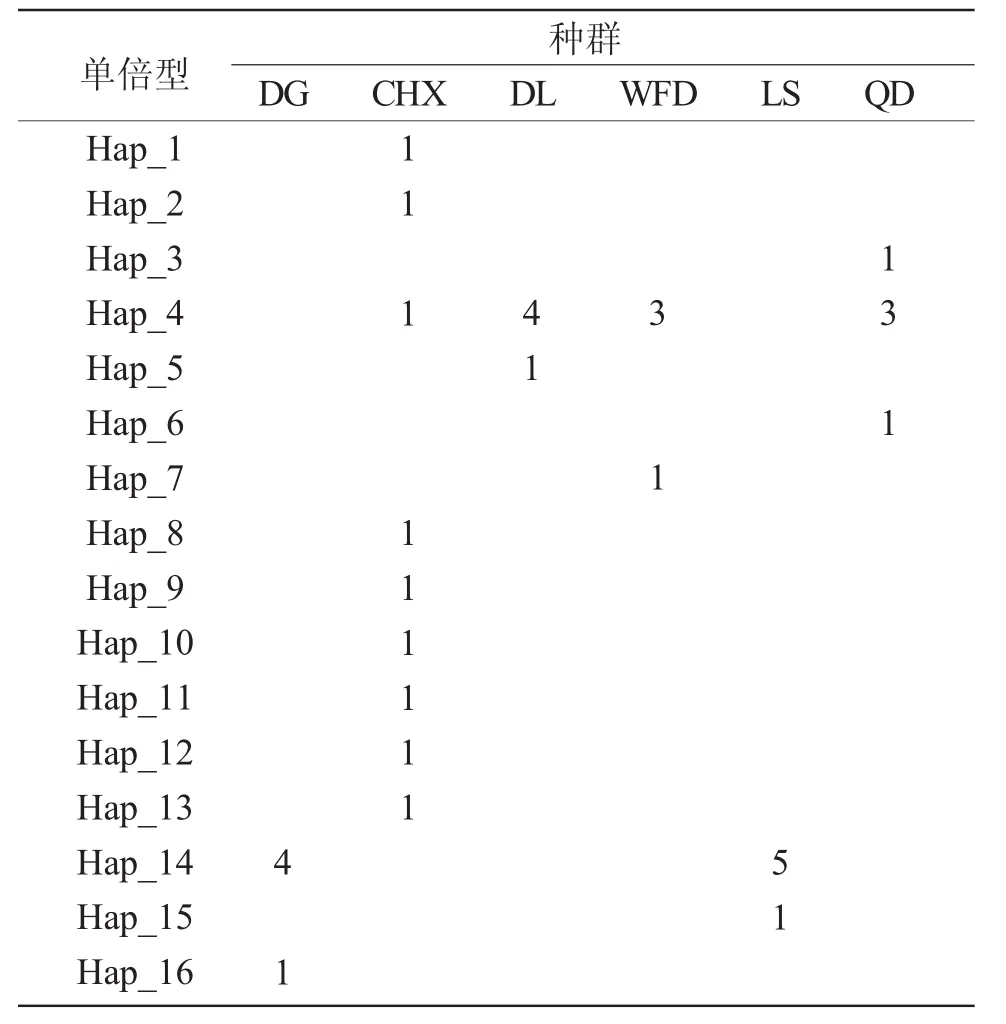
表2 16種單倍型在大瀧六線魚群體中的分布Tab.2 The distribution of the 16 haplotypes in fat greenling Hexagrammos otakii populations
2.2 遺傳多樣性
群體遺傳多樣性參數的統計結果(表3):表明各種群遺傳多樣性差異明顯。長海縣群體的核苷酸多樣性指數(0.10441)和單倍型多樣性指數(1.00)最高,而東港和大連群體的單倍型多樣性指數最低(0.40),大連群體的核苷酸多樣性指數最低(0.00071)。旅順群體序列Fu's Fs值的計算結果為負值,統計學計算的結果具有顯著的差異性,可將此群體的檢驗結果定義為偏離中性模式。
2.3 遺傳結構
Kimura 2-paramter遺傳分化指數Fst和遺傳距離的計算結果(表4)表明,6個群體間的平均遺傳距離為0.00097~0.76765,其中瓦房店群體與東港群體的群體間遺傳距離最高,瓦房店群體與大連群體的群體間遺傳距離最低。6個群體間的遺傳分化指數Fst為0~0.99832,其中大連群體與旅順群體之間的遺傳分化指數最高,東港與旅順群體之間以及青島、瓦房店和大連群體之間的分化可以忽略不計。
由表5可知:分子變異分析(AMOVA)的結果為Fst=0.88326(P<0.01)。群體間變異占變異總量的88.33%,群體內變異僅占11.67%,由此推斷,6個大瀧六線魚群體間發生了顯著的遺傳分化。
2.4 分子系統樹的構建
外群選用斑頭魚Agrammus agrammus的同源序列(登錄號:AB290791.1)進行比對分析,基于Kimura-雙參數計算群體間的遺傳距離,構建NJ和MP分子系統進化樹,通過1 000次bootstrap檢驗對其節點的支持率進行檢驗。由圖2可知,NJ和MP分子系統進化樹具有極高的相似度,6個群體的34尾大瀧六線魚大致呈現出兩個分支。其中旅順和東港群體的親緣關系較為接近,為一條分支,其他群體皆位于另一條分支。
3 討論
遺傳多樣性是物種延續和發展的基礎[22],其豐富的種群在復雜多變的自然環境中表現出更強的適應性。近年來,隨著漁業資源的開發利用力度加大,魚類種群遺傳多樣性減少,對各個魚類種群的延續能力產生不良影響,嚴重時甚至會導致瀕危物種的滅絕速度加快[23]。在本研究中,共測序得出16種單倍型,各個群體的單倍型多樣性指數范圍為0.40~1.00,核苷酸多樣性指數范圍為0.00089~0.10441,各群體具有較為顯著的遺傳多樣性差異。本研究中,旅順群體的變異位點數最高,達到244個,核苷酸多樣性指數(0.10441)最高,說明旅順群體的遺傳多樣性非常豐富。分析旅順的地理位置及其結果發現,旅順東臨黃海,西瀕渤海,南與山東半島隔海相望,中間有水道可與外海相通,對基因的交流提供了極大的便利。而大連群體(0.40,0.00071)與東港群體(0.40,0.00089)的遺傳多樣性相對貧乏,其原因可能與其所處的地理環境有關。Fu[24]認為,Fs中性檢驗的結果顯著偏離中性,P值具有顯著差異且Fs值呈負值,表明該種群在其進化歷程中,可能發生過快速的種群擴張。在本試驗的1 000次的模擬檢測中,旅順群體的Fs值為負值(P<0.05),推斷旅順的大瀧六線魚群體可能在歷史上發生過短時間、大規模的種群擴張,使該地區大瀧六線魚群體的遺傳多樣性較高。

表3 6個大瀧六線魚群體單倍型參數及遺傳多樣性比較Tab.3 The comparison of haplotype diversity and genetic diversity among the 6 populations of fat greening Hexagrammos otakii

表4 各種群之間的遺傳距離(左下角)及其遺傳分化指數Fst(右上角)Tab.4 Genetic distance(below diagonal)and Fst(above diagonal)among different populations of fat greening Hexagrammos otakii

表5 大瀧六線魚群體的AMOVA分析Tab.5 The AMOVA analysis of the populations of fat greening Hexagrammos otakii

圖2 基于DNA 12S rRNA的16個單倍型構建的NJ和MP分子系統樹Fig.2 Neighbor-joining and Maximum Parsimony tree of fat greening Hexagrammos otakii based on the 16 haplotypes in mtDNA 12S rRNA sequences
群體間的遺傳距離以及遺傳分化指數是衡量群體分化程度的重要指標,二者的數值與群體分化程度為線性關系。東港群體以及旅順群體與其他群體間Fst值都大于0.8,接近1;長海縣群體與青島、瓦房店和大連群體之間的值也大于0.3,其他三個群體間的遺傳多樣性分化程度可忽略不計。由于各種群間的親緣關系與其相對遺傳距離呈明顯的正相關[25],因此,青島及旅順群體與其他群體之間的遺傳距離(0.68805~0.76765)明顯高于旅順與東港群體之間的遺傳距離(0.00209)及其他三個群體之間的遺傳距離(0.00097~0.09305),表明青島及旅順群體的親緣關系較為接近。AMOVA分析結果表明,群體間變異占變異總量的88.33%,6個樣點的大瀧六線魚群體間差異遺傳較大,這與程起群等(2007)[26]、李瑩(2012)等[19]研究結果一致。NJ和MP分子系統樹的結果與遺傳分化指數的結果一致,這與張源真等研究結果一致[27]。東港群體和旅順群體為一個分支,長海縣、瓦房店、大連和青島群體共同組成另一大分支。mtDNA屬母系遺傳,由此可推斷,這6個大瀧六線魚群體在進化過程中,曾由共同祖先分化為兩個進化分支,繼而分別展開進一步的分化。
本研究基于線粒體控制區的對比發現,大瀧六線魚的分布與其基因多樣性的地理位置分布存在一定差異。青島群體與大連群體間的親緣關系反而較為接近,這可能基因交流所形成,青島群體形成的時間較短,分化速度較快,而12S rRNA序列比較保守,在短時間內無法形成足夠的有效信息含量[28]。同時也可能是進化樹的構建過程中,受多方面因素影響的結果,例如分析軟件的不同,所選擇的分子標記、物種差異以及計算方法的不同等,目前由于上述因素缺乏統一的標準,都有可能影響分析結果。但是通過本試驗,可以發現除東港群體與旅順群體外,其他群體間依然具有較大的遺傳分化,這種分化的結果與其地理位置的分布具有較高的一致性。因此結合本次針對兩種分子標記方法的研究結果,建議按照不同的地理區域對大瀧六線魚種群資源展開劃區域、多層次的保護,使漁業資源能夠得到更加合理的開發,達到可持續利用的目標。
[1]劉蟬馨,秦克靜.遼寧動物志(魚類)[M].沈陽:遼寧科學技術出版社,1987:393-396.
[2]成慶泰,鄭寶珊.中國魚類系統檢索[M].北京:科學出版社,1987.
[3]遼寧省海洋局.遼寧省海島資源綜合調查研究報告[M].北京:海洋出版社,1996:357.
[4]唐啟升,葉懋中.山東近海漁業資源開發與保護[M].北京:農業出版社,1990:80-102.
[5]Fukuhara O and Fushimi T.Development and early live history of the greenlings Hexagrammos otakii(spices:Hexagrammidae)reared in the laboratory[J].Bulletin of the Japanese SocietyofScientific Fisheries,1983,49(12):1843-1848.
[6]吳立新,秦克靜,姜志強,等.大瀧六線魚(Hexagrammos otakii)人工育苗初步試驗[J].海洋科學,1996(4):32-34.
[7]于鴻仙,莊虔增,徐春華,等.六線魚人工育苗技術研究[J].齊魯漁業,1998,15(5):21-24.
[8]莊虔增,于鴻仙,劉崗,等.六線魚育種生產技術的研究[J].中國水產科學,1999,6(1):103-106.
[9]康斌,武云飛.大瀧六線魚的營養成分分析[J].海洋科學,1999(6):23-25.
[10]Lee S M,Cho S H and Kim K D.Effect of dietary protein and energy levels on growth and body composition of juvenileflounderParalichthys olivaceus[J].WorldAquaSoc,2000,31(3):306-315.
[11]王書磊,姜志強,苗治歐.大連海區大瀧六線魚生物學指標的季節變化[J].水產科學,2005,24(5):1-3.
[12]劉奇,王亮,高天翔,等.北黃海大瀧六線魚主要生物學特征比較研究[J].中國海洋大學學報:自然科學版,2009,39(S1):13-18.
[13]劉奇.大瀧六線魚(Hexagrammos otakii)生物學特征與遺傳多樣性研究[D].青島:中國海洋大學,2010.
[14]鄭光明,朱新平,張躍.魚類種質鑒定技術與漁業管理[J].中國水產科學,1996,6(2):108-111.
[15]Lu G and Li S.Advances in the study and application of fishmitochondrialDNApolymorphism[J].JournalofFisherySciences ofChina,1998,3:94-103.
[16]李春枝,張邦杰,李本旺,等.尖塘鱧屬魚類線粒體12S rRNA 基因序列分析[J].生態科學,2006,25(5):433-436.
[17]Honda M,Otah K,Kobayashi M,et al.Phylogenetic relationships ofAustralian skinks ofthe Mabuya group(Reptilia:Scincidae)inferred from mitochondrial DNA sequences[J].Genes Genet Syst,1999,74(4):135-139.
[18]Medina Mand Walsh P J.Molecular systematics ofthe order Anaspida based on mitochondrial DNA sequences(12S,16S,and COI)[J].Molecular Phylogenetics and Evolution,2000,15(1):41-58.
[19]李瑩,王偉,孟凡平,等.利用線粒體DNA控制區部分序列分析不同地理群體大瀧六線魚遺傳多樣性[J].海洋科學,2012,36(8):40-46.
[20]Kumar S,Tamura K and Nei M.MEGA3:integrated software for molecular evolutionary genetics analysis and sequence alignment[J].Briefings in Bioinformatics,2004(5):150-163.
[21]Rozas J,Sánchez-DelBarrio J C,Messeguer X,et al.DnaSPDNA polymorphismanalyses bythe coalescent and other methods[J].Bioinformatics,2003,19(18):2496-2497.
[22]郭新紅,劉少軍,顏金鵬,等.異源四倍體鯽鯉、三倍體湘云鯽和它們父母本線粒體DNA 12S rRNA基因遺傳變異的分析[J].遺傳,2004,26(6):875-880.
[23]季維智,宿兵.遺傳多樣性研究的原理與方法[M].杭州:浙江科學技術出版社,1999.
[24]Fu Y X.Statistical tests of neutrality of mutations against population growth,hitchhiking and background selection[J].Genetics,1997,147(2):915-925.
[25]任崗,章群.中國沙塘鱧屬魚類線粒體12S rRNA基因序列分析[J].水生生物學報,2007,31(4):473-478.
[26]程起群,馬春艷,繆炯,等.基于線粒體12S rRNA序列研究鳳鱭兩個野生群體的遺傳多樣性[J].大連水產學院學報,2007,22(5):387-391.
[27]張源真,王偉,姜志強,等.基于16S rRNA部分序列探討部分鳚亞目魚類的分子系統進化關系[J].海洋科學,2012,36(9):89-95.
[28]邵愛華,朱江,史全良,等.暗紋東方鲀線粒體12S rRNA基因的克隆及序列分析[J].蘇州科技學院學報:自然科學版,2008,25(4):39-44.
Genetic Diversity Analysis of Six Wild Populations of Fat Greenling Hexagrammos otakii by Mitochondrial DNA 12S rRNA Partial Sequence
WANG Wei1,GAO Wei-feng1,ZHANG Sai-sai1,2,DONG An-ran1,JIANG Xin-tong1
(1.Key Laboratory of Mariculture&Stock Enhancement in North China's Sea,Ministry of Agriculture,Dalian Ocean University,Dalian 116023,China;2.Tianjin Chen Hui Modern Technology Group Co.,Ltd.,Tianjin 301800,China)
Nucleotide sequences of mitochondrial DNA 12S rRNA partial sequence were amplified in six wild populations of fat greenling Hexagrammos otakii using PCR techniques.A total of 314 variable sites were detected among the aligned sequences of 582 bp with mutation rate of 53.95%.Sixteen haplotypes were identified from 34 individuals according to the determined sequences,and the six populations had haplotype diversity of 0.40~1.00,nucleotide diversity of 0.00071~0.10441 and Fu's Fs of 1.19782 in neutrality tests(P<0.01).AMOVA analysis demonstrated that:there were Fst=0.88326(P<0.01),variance of 88.33%among six populations and variance of 11.67%within the six populations.The NJ and MP molecular phylogenetic trees constructed by the distances among different individuals revealed that the phylogenetic trees were all divided into two branches,one in Donggang and Lvshun populations and the others in the other populations.
Hexagrammos otakii;wild population;mitochondrial DNA;12S rRNA partial sequence
S917
A
1005-3832(2017)05-0007-06
2017-04-18
大連市科技計劃項目(2015B11NC072);大連市科技之星計劃項目(2015R081).
王偉(1978-),男,理學博士,研究方向為魚類群體遺傳學.E-mail:wangwei@dlou.edu.cn

