哈薩克斯坦傳統發酵乳制品中乳酸菌的多樣性
(內蒙古農業大學乳品生物技術與工程教育部重點實驗室,呼和浩特010018)
哈薩克斯坦傳統發酵乳制品中乳酸菌的多樣性
劉紅新,呼斯楞,劉文俊,孫天松
(內蒙古農業大學乳品生物技術與工程教育部重點實驗室,呼和浩特010018)
為了闡明哈薩克斯坦傳統發酵乳制品中乳酸菌生物多樣性,采用傳統純培養方法對采集的5份傳統發酵乳制品中的乳酸菌進行分離純化,運用16S rRNA基因序列分析方法和系統發育樹研究進行屬種鑒定。結果表明,從5份傳統乳制品中分離鑒定了49株乳酸菌分屬于4個屬8個種或亞種,乳桿菌屬(Lactobacillus)44株,片球菌屬(Pediococcus)1株、腸球菌屬(Enterococcus)3株,鏈球菌屬(Strep?tococcus)1株。其中乳桿菌屬、片球菌屬、腸球菌屬、鏈球菌屬是哈薩克斯坦地區傳統發酵乳制品中的主要乳酸菌,其中Pediococcus pento?saceus為優勢菌種,占總分離株的22%。
乳酸菌;傳統發酵乳制品;16S rRNA基因序列分析
0 引言
傳統發酵乳制品是以乳為原料,在自然開放的環境中,經微生物發酵后,通過不同工藝制作而成的發酵乳制品。包括:酸牛奶、酸馬奶、酥油、馬奶酒、曲拉、乳酒(以馬奶為主)等[1]。傳統發酵乳相比于鮮乳,不僅延長了食品的保質期,也具有獨特的風味和良好的益生作用,深受廣大消費者所喜愛[2]。近幾年對我國不同地區傳統乳制品中乳酸菌研究均有報道,但對哈薩克斯坦傳統乳制品中乳酸菌的研究甚少,而且不夠深入。
本論文采用實驗室純培養方法對哈薩克斯坦傳統發酵乳制品中的乳酸菌進行分離、純化,應用16S rRNA基因序列分析方法對分離株進行鑒定,主要對哈薩克斯坦傳統發酵乳制品中的乳酸菌多樣性進行分析,更好的了解傳統發酵乳制品中乳酸菌的組成與種類。
1 實驗
1.1 樣品來源
本研究中5份奶制品樣品由內蒙古農業大學乳品生物技術與工程教育部重點實驗室的研究人員于2015年6月16日~2015年6月18日在哈薩克斯坦的不同地區采集。樣品采集后低溫保存,帶回實驗室進行后續實驗。
1.2 培養基與試劑
菌株分離與純化培養基為MRS固體和MRS液體培養基,M17固體和M17液體培養基,提取DNA所用試劑,PCR擴增和電泳檢測所用試劑,酶類,Mark?er。
1.3 樣品采集
本研究樣品來自哈薩克斯坦地區不同加工成品的傳統乳制品,將采集的樣品置于4℃便攜式冰箱中以維持其低溫狀態,帶回實驗室進行乳酸菌的分離純化等實驗。
1.4 乳酸菌的分離純化及保存
乳酸菌計數:采用傾注培養法[3]。將樣品混勻,用濃度為0.85%的無菌生理鹽水以十倍稀釋法[4]對樣品進行梯度稀釋,選取10-5,10-6,10-7稀釋液1 mL于無菌培養皿中(組內做兩個平行),分別傾注20~25 mLMRS和M17固體培養基混勻,置于30℃恒溫培養箱中厭氧培養48~72 h。菌落形成后進行計數,計數即為每毫升樣品的菌落數。
吸取0.2 mL上述不同梯度(10-5,10-6,10-7)的稀釋液分別涂布于加有放線菌酮和多黏菌素(1×10-5)的MRS和M17固體培養基上,涂布后置于37℃恒溫培養箱中厭氧培養48 h,待菌落形成后,觀察并記錄菌落形態(顏色、形狀、大小、邊緣等),隨機挑取形態學特征不同的單個菌落,于MRS或M17固體培養基上劃線分離2~3次,直至鏡檢結果為純的單菌后編號并傳代。選取革蘭氏陽性、過氧化氫陰性的純培養物,加入質量分數為0.1%谷氨酸鈉的脫脂乳保護劑,分裝混勻后置于-80℃保存備用。
1.5 總DNA的提取及純度的檢測
純化好的菌株采用液氮反復凍融-CTAB法提取乳酸菌基因組DNA[5-6]。再用ND-1000型微量紫外分光光度計測其濃度和純度。再用質量分數為1.0%的瓊脂糖凝膠電泳檢測。
1.616 S rRNA基因的PCR擴增
PCR擴增16S rRNA基因的引物采用細菌通用引物[7],引物序列為:
FA-27F:5'-GCAGAGTTCTCGGAGTCACGA AGAGTTTGATCCTGGCTCAG-3′,
RA-1495R:5'-AGCGGATCACTTCACACAG GACTACGGCTACCTTGTTACGA-3′。
擴增體系:5 μL10×EasyTaq Buffer(+Mg2+);4 μL High Pure dNTPs(2.5 mmol/L);1.5 μL引物FA-27F(濃度10 mmol/L);1.5 μL引物RA-1495R(10 mmol/L);0.5 μL EasyTaq DNA polymerase(5 U/μ L);2 μL DNA模板(質量濃度100 ng/μL);35.5 μL ddH2O。
PCR擴增循環參數為:94℃預變性5 min;94℃變性1 min,58℃退火1 min,72℃延伸2 min,30個循環;72℃末端延伸10 min[8]。
擴增完畢后,取約2 μL的PCR擴增產物用質量分數為1.0%的瓊脂糖凝膠電泳檢測,若在1 500 bp處有清晰的擴增條帶且無拖尾、彌散現象,則PCR擴增成功。將PCR擴增產物送往上海美吉生物公司進行純化和DNA測序。
1.7 同源性分析
將測序得到的DNA序列運用DNA Star 5.01軟件中的SepMan模塊,對所測得菌株的兩條16S rRNA基因序列進行整理、拼接、校準,得到1 450 bp左右的有效序列,再利用NCBI中的BLAST(http://blast.ncbi. nlm.nih.gov/Blast.cgi)將拼接好的序列與數據庫中已鑒定的乳酸菌16S rDNA序列進行同源性比對,將待鑒定菌株序列與同源性最高的模式菌株序列應用MEGA 5.0(http://www.megasoftware.net)軟件構建系統發育樹,進行遺傳進化研究。
2 結果分析與討論
2.1 乳酸菌的計數結果
活菌數是乳制品樣品新鮮程度的重要指標。由表1可知,哈薩克斯坦傳統乳制品樣品活菌數在4.95~7.67 mL-1(對數值)之間,平均值為6.34±0.05 mL-1(對數值)。其中駝奶、酸馬奶高于106mL-1,符合新的酸奶國家標準。楊吉霞等人[9]研究了西藏、云南、新疆3個地區奶酪中的乳酸菌活菌數分別為3.74~4.60,3.62~4.98,3.31~4.49 mL-1(對數值)。楊彥榮等[10]在自俄羅斯卡爾梅克共和國奶酪樣品中乳酸菌數為6.64~9.05 mL-1(對數值)。呼斯楞等人[11]對內蒙古呼倫貝爾傳統乳制品進行分析研究乳酸菌活菌數在6.80~9.00 mL-1(對數值)之間。Bao Qiuhua[12]對四川曲拉進行研究,活菌數為(7.18±1.49)mL-1(對數值)。由此可見哈薩克斯坦地區乳制品的新鮮程度比較高。

表1 哈薩克斯坦傳統乳制品中乳酸菌活菌計數結果
2.2 乳酸菌的鑒定
2.2.1 乳酸菌分離株的菌落形態及表型特征
根據乳酸菌的形態學特征[13-14],初步從4份傳統乳制品樣品中共分離出49株疑似乳酸菌的菌株。在MRS和M17固體培養基上菌株的菌落呈圓形,中等大小,凸起,乳白色,濕潤,邊緣整齊,有些菌落表面光滑有些粗糙。經過革蘭氏染色、過氧化氫酶反應,均為革蘭氏陽性,過氧化氫陰性菌。初步將49株菌定為乳酸菌。
2.2.2 菌種DNA提取和16S rRNA擴增結果
DNA濃度和OD260/280的測定:OD260/280值在1.8~2.0間即為純DNA樣品。將其質量濃度稀釋至100 ng/μL后對乳酸菌分離株16S rRNA基因進行擴增。經1.0%瓊脂糖凝膠電泳檢測,可以觀察到在大約1 500 bp的位置處有一條清晰明亮的條帶,并且無彌散現象,無明顯非特異擴增現象。
圖1 部分分離菌16S rRNA基因的PCR擴增產物瓊脂糖凝膠電泳圖
這表明菌株的16S rRNA基因擴增產物可以滿足測序的要求。將16S rRNA基因擴增產物送于上海美吉生物科技有限公司進行測序。所有的16S rD?NA序列都與GenBank數據庫中的模式株有99%及以上的相似度。
2.2.3 系統發育樹的構建和乳酸菌鑒定結果
將測序獲得的16S rRNA基因序列通過NCBI數據庫中BLAST進行同源序列比對分析,以與模式菌株序列同源性大于98%為種的鑒定閾值,將49株分離株鑒定為乳酸菌的4個屬,8個種。部分分離株與模式株系統發育樹如圖2所示。
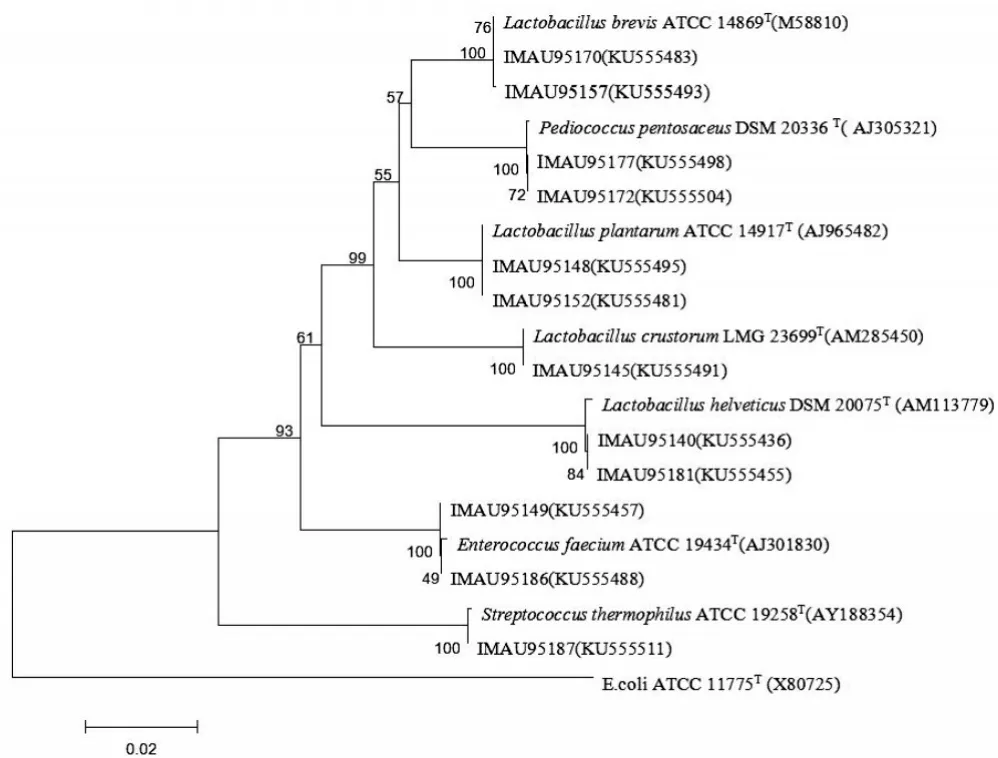
圖2 哈薩克斯坦地區代表性分離株的16S rRNA基因序列的系統發育樹
由圖2可以看出,IMAU95170(KU555483)、IMAU 95157(KU555493)與模式菌株Lactobacillus brevis ATCC 14869聚為一類,且同源性為100%,故將其鑒定為Lac?tobacillus brevis。菌株IMAU95177(KU555498)、IMAU9 5172(KU555504)與模式菌株Pediococcus pentosaceus DSM 20336(T)聚為一類,且同源性為100%,因此可將其鑒定為Pediococcus pentosaceus。菌株IMAU95148 (KU555495)、IMAU95152(KU555481)與模式菌株Lac?tobacillus plantarum ATCC 14917(AJ965482)聚為一類,且同源性為99%,故將其歸為Lactobacillus plantarum。菌株IMAU95145(KU555491)與模式菌株Lactobacillus crustorum LMG 23699T聚為一類,且同源性高達100%,因此可將其歸為Lactobacillus crustorum。菌株IMAU95140(KU555436)、IMAU95181(KU555455)與模式菌株Lactobacillus helveticus(AM113779)聚為一類,且同源性高達100%,故將其鑒定為Lactobacillus helveti?cus。菌株IMAU95149(KU555457)、IMAU95186 (KU555488)與模式菌株Enterococcus faecium 1ATCC 19434聚為一類,且同源性達到99%,因此鑒定為En?terococcus faecium。IMAU95187(KU555511)與模式菌株Streptococcus thermophilus ATCC 19258聚為一類,同源性達到100%,故將其歸為Streptococcus thermophilus。
2.3 優勢菌種分析
哈薩克斯坦地區傳統發酵乳制品中乳酸菌的分離結果見表2。由表2可知,5份傳統發酵乳制品中分離出的49株乳酸菌疑似菌株,經16S rDNA序列測定和同源性分析鑒定,分屬于4個屬8個種或亞種。5份樣品均分離到Pediococcus pentosaceus,共11株,占總分離株的22%,為五份哈薩克斯坦地區的優勢菌種。其中HSK1、HSK2、HSK4樣品分離出Lactobacillus helveticus,且共分離出16株,占總分離株的33%,說明Lactobacil?lus helveticus為哈薩克斯坦地區HSK1、HSK2、HSK4樣品中乳酸菌的優勢菌種。由表2還可以看出,HSK5(奶酪)分離的菌種類別最多,分別為Lactobacillus plan?tarum、Lactobacillus brevis、Lactobacillus crustorum、Pediococcus pentosaceus、Enterococcus faecium。其次為HSK4(奶酪),分別為Lactobacillus helveticus、Lactobacillus plantarum、Lac?tobacillus brevis、Pediococcus pentosaceus。由于本次采樣樣品比較少,而且沒有定點、全面采樣,可能是HSK1(酸駝奶)和HSK2(酸馬奶)分離的菌種類別較少的原因,以后研究進行改善。綜上所述,本研究的5份傳統發酵乳制品中優勢菌種為Pediococcus pentosaceus,占總分離株的22%。
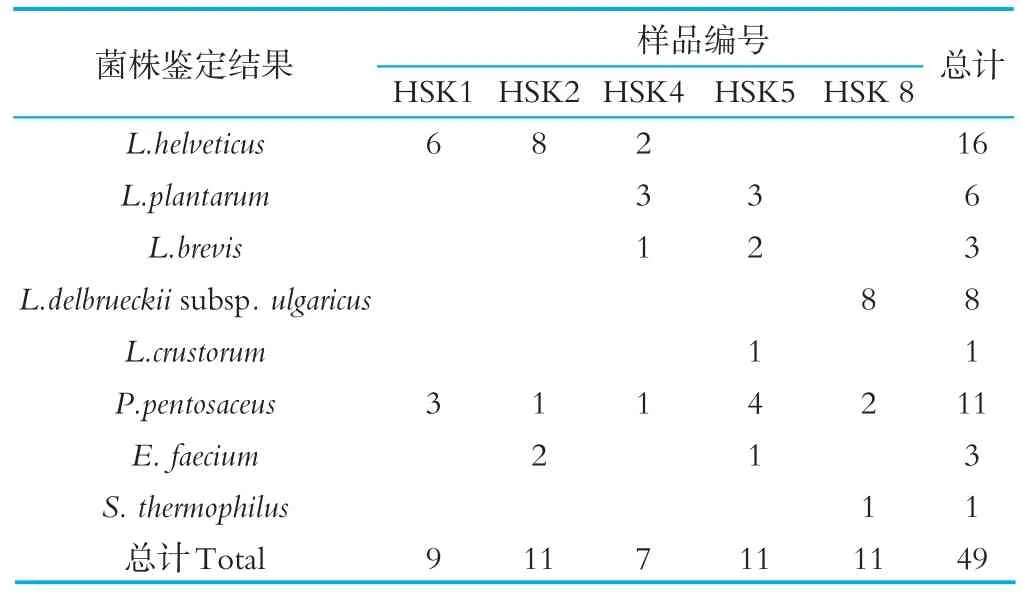
表2 哈薩克斯坦地區傳統乳制品中乳酸菌的分離結果
3 結論
本研究從哈薩克斯坦地區采集的5份傳統發酵乳制品樣品中乳酸菌數為4.95~7.67 mL-1(對數值),乳酸菌含量都比較高。5份乳樣品中共分出49株菌,經鑒定分別歸屬于Streptococcus thermophilus、Enterococcus faecium、Pediococcus pentosaceus、Lactobacillus plantarum、Lac?tobacillus brevis、Lactobacillus crustorum、Lactobacillus helveti?cus、Lactobacillus delbrueckii subsp.Bulgaricus 8個種或亞種。其中Pediococcus pentosaceus為哈薩克斯坦地區傳統發酵乳制品中的優勢菌種,占總分離株的22%。
4 討論
4.1 研究方法的可靠性
16S rRNA基因序列分析方法在乳酸菌分離鑒定中應用較多,近幾年,隨著核酸測序技術的發展,越來越多的16S rRNA基因序列被輸入數據庫,有大量的數據和強大的數據庫作支持。本文采用16S rRNA基因序列分析方法對不同樣品中乳酸菌進行分離鑒定,與傳統生理生化方法相比,此方法更為可靠[15]。
4.2 菌種分析
本研究的5份傳統發酵乳制品中優勢菌種為Pediococcus pentosaceus,而Aydemir等[16]所研究的奶酪中優勢菌為Lactobacillus casei和Lactobacillus plantarum;Watanabe等[17]和Takeda等[18]對內蒙古自然發酵牛乳進行研究,結果表明,德氏乳桿菌保加利亞亞種(Lac?tobacillus delbrueckii subsp.bulgaricus)是蒙古國自然發酵牛乳的優勢乳酸菌菌群。Airidengcaicike等[19]對西藏藏南和藏北地區發酵酸牛乳進行分析研究,分析表明在寒冷的藏北那曲、拉薩地區以發酵乳桿菌(Lactoba?cillus fermentum)和干酪乳桿菌(Lactobacillus casei)為優勢菌群。前人也曾報道,當環境酸度較高時,乳酸球菌的生長受到抑制[20-21],所以戊糖片球菌多數在酸腌菜、泡菜、青貯料等中分離得到,和本文分離的結果存在差異,可能與當地的氣候、環境、發酵溫度和發酵時間以及制作方法等因素有關。本研究對傳統發酵乳制品進行多樣性分析,這對豐富我國乳酸菌菌庫很有幫助,為乳酸菌資源開發與研究奠定良好的基礎。
[1]張和平.自然發酵乳制品中乳酸菌的生物多樣性[J].生命科學, 2015,27(7):837-846.
[2]LIU S,HAN Y,ZHOU Z.Lactic acid bacteria in traditional fermented Chinese foods[J].Food Research International,2011,44(3):643-651.
[3]楊吉霞,張利玲,蔣厚陽,等.眉山泡菜中乳酸菌的分離鑒定[J].食品科學,2015,36(17):158-163.
[4]WU R N,WU Z X,ZHAO C Y,et al.Identification of Lactic acid bac?teria in suancai,a traditional Northeastern Chinese fermented food,and salt response of Lactobacillus paracasei LN-1[J].Annals of Microbiolo?gy.2014,64(3):1325-1332.
[5]ZHOU J,BRUNS M A,TIEDJE J M.DNA recovery from soils of diverse composition[J].Applied and environmental microbiology, 1996,62(2):316-322.
[6]ZHU H,QU F,ZHU L H.Isolation of genomic DNAs from plants, fungi and bacteria using benzyl chloride[J].Nucleic Acids Research, 1993,21(22):5279.
[7]WIJIZES T,BRUGGEMAN M R,NOUT M J,et al.A computer?ised system for the identification of Lactic acid bacteria.[J].Interna tional Journal of Food Microbiology.1997,38(1):65-70.
[8]YU J,GAO W,QING M J,et al.Identification and characterization of Lactic acid bacteria isolated from traditional pickles in Sichuan,China [J].The Journal of General and Applied Microbiology,2012,58(3): 163-172.
[9]楊吉霞,陳芝蘭,楊海燕,等.牦牛奶酪中乳酸菌的分離鑒定及發酵性能分析[J].食品科學,2013,34(9):198-204.
[10]楊彥榮,任艷,張和平,等.傳統奶酪樣品中乳酸菌的分離鑒定[J].中國乳品工業,2015,43(9):19-22.
[11]呼斯楞,劉紅新,于潔,等.內蒙古呼倫貝爾地區傳統發酵乳中乳酸菌的多樣性分析[J].微生物學通報,2016,43(5):984-990.
[12]BAO Q H,YU J,LIU W J,et al.Predominant Lactic acid bacteria in traditional fermented yak milk products in the Sichuan Province of China[J].Dairy Science&Technology,2012,92(3):309-319.
[13]凌代文.乳酸菌分類鑒定及實驗方法[M].北京:中國輕工業出版社, 1999:1-129.
[14]東秀珠,蔡妙英.常見細菌系統鑒定手冊[M].北京:科學出版社, 2001:289-294.
[15]QUAIL M A,KOZAREWA I,SMITH F,et al.A large genome cen?ter's improvements to the Illumina sequencing system[J].Nature Methods,2008,5(12):1005-1010.
[16]AYDEMIR O,HARTH H,WECKX S,et al.Microbial communi?ties involved in Ka§ar cheese ripening[J].Food microbiology,2015, 46:587-595.
[17]WATANABE K,FUJIMOTO J,SASAMOTO M,et al.Diversity of lactic acid bacteria and yeasts in Airag and Tarag,traditional ferment?ed milk products of Mongolia[J].World J Mi-crobiol Biotechnol, 2008,24(8):1313-1325.
[18]TAKEDA S,YAMASAKI K,TAKESHITA M,et al.The investiga?tion of probiotic potential of Lactic acid bacteria isolated from tradition?al Mongolian dairy products.Anim Sci J,2011,82(4):571-579.
[19]AIRIDENGCAICIKE,CHEN X,DU X,et al.Isolation and identif?cation of cultivable Lactic acid bacteria in traditional fermented milk of Tibet in China.Int J Dairy Techol,2010,63(3):437-444.
[20]孫天松,王俊國,張列兵,等.中國新疆地區酸馬奶中乳酸菌生物多樣性研究[J].微生物學通報,2007,34(3):451-454.
[21]魏艷,曾小群,潘道東,等.新疆地區不同酸奶中優勢乳酸菌的分離與鑒定[J].中國食品學報,2012,12(12):161-166.
Survey of diversity of Lactic acid bacteria in traditional fermented milk products from Kazakhstan
LIU Hongxin,HU Sileng,LIU Wenjun,SUN Tiansong
(Key Laboratory of Dairy Biotechnology and Engineering,Ministry of Education,Inner Mongolia Agricultural Universi?ty,Huhhot 010018,China)
The aim of this paper is to research the diversity of Lactic acid bacteria in traditional fermented milk products from Kazakhstan.Lactic acid bacteria were isolated and purified by traditional pure culture form traditional fermented milk products samples,and then all isolates were identified by 16S rRNA gene sequence analysis.A total of 49 strains of Lactic acid bacteria were classified into 4 genera and 8 species or subspe?cies,namely Lactobacillus(44 strains),Pediococcus(1 strains),Enterococcus(3 strains),Streptococcus(1 strain).Pediococcus pentosaceus was regarded as the dominant species,occupied 22%of the total isolates.
Lactic acid bacteria;traditional fermented milk products;16S rDNA sequence analysis
TS252.54,Q939.11+7
:A
:1001-2230(2017)07-0009-04
2016-09-06
國家科技支撐計劃項目(2013BAD18B01);國家自然科學基金課題(31471711)。
劉紅新(1991-),女,碩士研究生,研究方向為微生物生理與發酵工藝。
孫天松

