毛竹生長素反應因子基因的生物信息學分析及差異表達
程占超,侯 丹,馬艷軍,高 健
(國際竹藤中心 國 家林業局竹藤科學與技術重點開放實驗,北京100102)
毛竹生長素反應因子基因的生物信息學分析及差異表達
程占超,侯 丹,馬艷軍,高 健
(國際竹藤中心 國 家林業局竹藤科學與技術重點開放實驗,北京100102)
生長素反應因子(ARF)基因家族在植物的生長發育中起至關重要的作用。關于毛竹Phyllostachys edulis ARF基因家族在花器官中生物信息學分析未見報道。以毛竹花器官為材料,采用生物信息學的方法,對毛竹中ARF基因進行篩選,并對其系統進化關系、保守基序及在花器官中的差異表達模式進行了初步的分析。結果表明:毛竹全基因組中含有44個ARF基因,分為Ⅰ,Ⅱ,Ⅲ類。與擬南芥Arabidopsis thaliana和水稻Oryza sativa分別比較,發現毛竹與水稻存在11個姐妹同源基因對。此外,PheARF13,PheARF14和PheARF35在花芽、雌蕊和幼胚中高量表達,同時PheARF2,PheARF30,PheARF14,PheARF13,PheARF35,PheARF7,PheARF37,PheARF38在雌蕊和幼胚中高量表達,推測這些基因可能在花發育和種子發育中發揮重要的作用。圖3表1參32
林木育種學;毛竹;生長素應答因子;花發育
植物生長激素能夠調節或者影響植物不同的生理過程,比如頂端優勢、側根萌發、導管分化、胚胎發育和芽的伸長,也可以促進細胞的分裂、延伸和分化[1]。在分子水平上,生長素能夠特異性地調節基因的表達[2]。生長素信號轉導相關的3類主要蛋白是Aux/IAAs,ARFs和SCF復合體[3-4]。作為生長素信號途徑一個重要的成員,ARFs基因能夠通過結合生長素啟動子上的AuxRE來調控生長素反應基因的表達[5]。這些ARFs基因調控不同的發育過程,比如頂芽形成,導管組織的形成,胚胎、花和果實的形成[6]。一個經典的ARF蛋白,結構相對簡單,大部分蛋白的分子量為70~130 kD,含有3個保守的結構域,它們分別是N末端DNA結合結構域(DBD),中間區域(MR)和C末端二聚體結構域(CTD)。不同的結構域也決定ARF不同的功能,DBD在信號轉導中起著鑒別作用,CTD可以使ARF之間形成二聚體,而MR則起著轉錄激活或抑制的功能[7]。ULMASOV等[8]鑒定出第1個ARF基因,即AtARF1,先后在擬南芥Arabidopsis thaliana和水稻Oryza sativa中鑒定出23和25個ARF基因[9-10]。ARFs大部分在植物中發現,比如在雙子葉、單子葉、裸子和蕨類植物中都有發現,但是在動物和微生物中至今沒有發現。因此,ARFs是植物中特有的一類轉錄因子[11]。ARF的生物學功能主要來自擬南芥ARF基因功能缺失的表型,到目前為止,已經發現很多ARF基因及其功能,比如ARF3和ARF7[12-14]。研究表明:擬南芥arf3的突變體會出現花蕊基部和頂端的發育不良,說明ARF3在調節花器官發育上起作用;缺失ARF7,導致上胚軸的向光性和下胚軸的向地性功能的消失。這2個ARF基因有著不同的功能,很少在功能上出現冗余現象。毛竹Phyllostachys edulis與水稻、玉米Zeamays等同屬于禾本科Gramineae單子葉植物,但是,毛竹營養生長周期長,開花時期不確定,開花后死亡,導致竹林面積減少,對經濟發展和生態環境造成重大損失和破壞,竹子開花的調控機制一直是竹類植物研究中的難點和熱點。目前,ARF家族基因在模式植物擬南芥、水稻、玉米中已有研究,而毛竹ARF家族基因在花器官和幼胚發育上鮮有報道。本研究通過生物信息學的方法,根據毛竹的基因組,鑒定ARF基因家族,進行進化樹分析、基序分析、基因差異表達模式分析,為研究ARF基因在毛竹花和種子發育過程中的功能奠定基礎。
1 材料與方法
1.1 實驗材料
毛竹開花實驗地位于廣西壯族自治區桂林市南嶺山系的西南部。該毛竹林屬于自然生長狀態,基本無人為干擾。以毛竹的花器官為材料,進行解剖,分離出花芽、苞片、穎片、稃片、雄蕊、雌蕊和幼胚以及未開花的成熟葉片,建立8個樣本進行轉錄組高通量測序。
1.2 方法
1.2.1 毛竹中PheARF家族成員的檢索 從擬南芥基因組數據庫(http://www.arabidopsis.org/)和水稻基因組數據庫(http://rice.plantbiology.msu.edu/index.html)中分別檢索擬南芥和水稻中ARF蛋白序列;毛竹ARF相關蛋白序列從毛竹基因組數據庫(http://www.bamboogdb.org/index.jsp)中獲得。毛竹PheARF家族蛋白的序列號,開放閱讀框的長度、氨基酸數目、分子質量和等電點在表1中提供。毛竹ARF分子量及等電點數據通過ExPASY(http://web.expasy.org/compute_pi/)獲得。
1.2.2 進化樹分析 用ClustalX 1.83(http://www.clustal.org/)[15]軟件對蛋白全長的多序列比對進行分析,進化樹分析前去掉比對序列的差異和不明確序列。用no-rooted neighbor-joining方法通過 MEGA 6.0(http://www.megasoftware.net/mega.html)[16]構建系統進化樹。
1.2.3 基序分析 MEME version 4.11.2(http://meme-suite.org/)[17]在線工具鑒定候選蛋白序列的保守區域,公式為any,maximum number ofmotifs=20,minimum width≥6和maximum width≤200。
1.2.4 表達量分析 將花芽、苞片、穎片、稃片、雄蕊、雌蕊和幼胚以及未開花的成熟葉片的FPKM值輸入到Cluster 3.0,用Java TreeView生成熱點圖[18]。
2 結果
2.1 毛竹PheARF家族序列鑒定
通過23個擬南芥和25個水稻ARF蛋白序列檢索毛竹基因組數據庫,共得到44個ARF蛋白(表1)。從表1可以看出,ARF家族的蛋白的等電點為5.22~9.23,蛋白序列的長度為192~1 257個氨基酸,分子量的大小為31.70~139.07 kD。
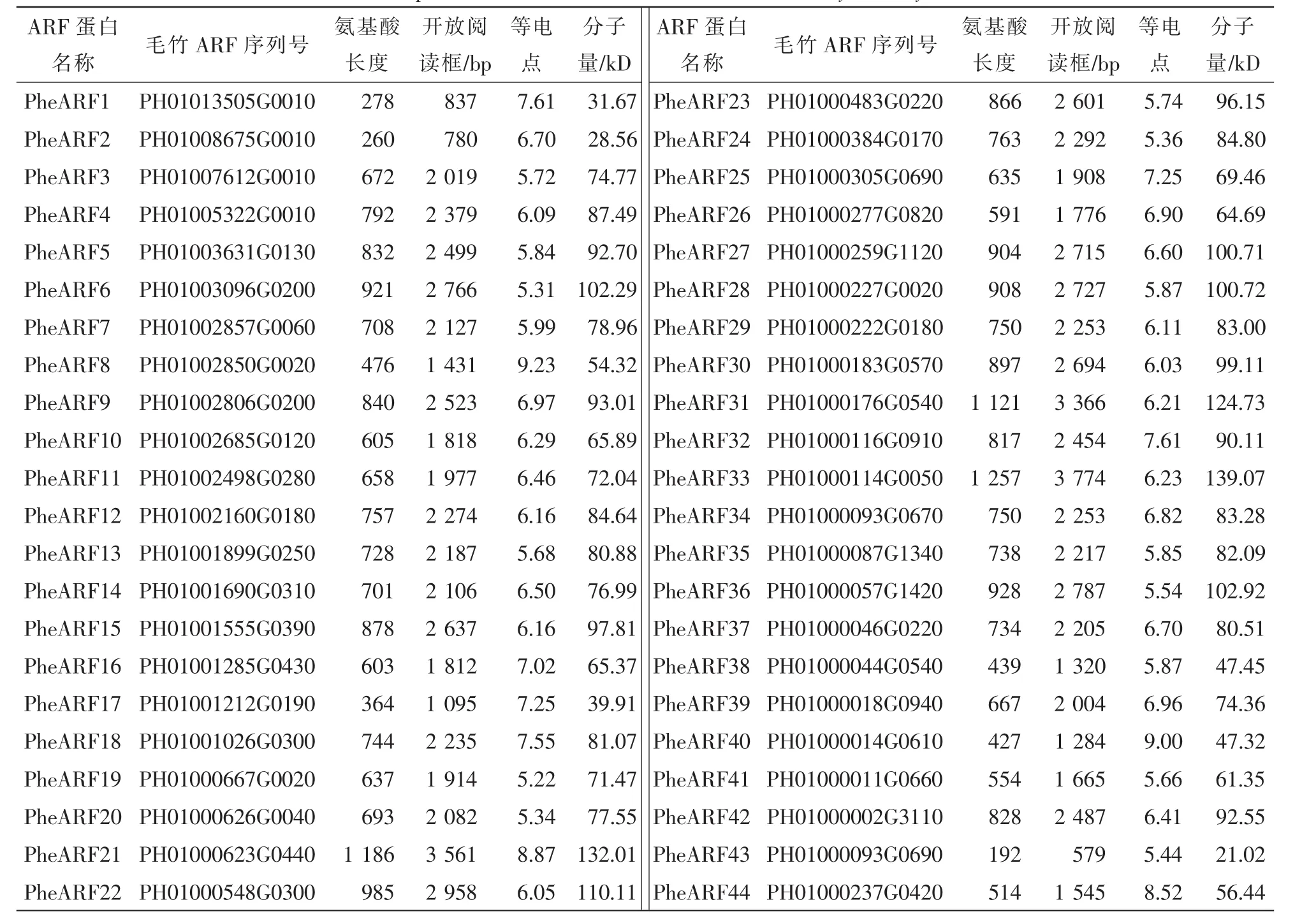
表1 毛竹中ARF成員數量及其屬性Table 1 Properties and numbers of ARF identified from Phyllostachys edulis
2.2 毛竹PheARF家族進化分析
系統進化結果顯示:44個基因之間相似性比較高(圖1)。將44個毛竹的ARF蛋白列同23個擬南芥和25個水稻的蛋白序列同時比對,發現可以將這3個物種的ARF分為三大類,分別為Ⅰ,Ⅱ和Ⅲ。其中有13個基因分布在Ⅰ類,17個基因分布在Ⅱ類,還有14個基因屬于Ⅲ類。大部分的PheARFs包含3個經典的結構域:DBD,結構域Ⅱ和AUX/IAA家族結構域。有11個與水稻的同源關系較近的基因對:OsAR25/PheARF30(Ⅱ),OsARF16/PheARF40(Ⅱ),OsARF19/PheARF31(Ⅱ),OsARF15/PheARF37(Ⅲ),OsARF2/PheARF14(Ⅲ),OsARF14/PheARF39(Ⅲ),OsARF3/PheARF2 6(Ⅲ),OsARF13/ PheARF38(Ⅲ),OsARF8/PheARF16(Ⅲ),OsARF10/PheARF10(Ⅲ)和OsARF18/PheARF18(Ⅲ)。這些基因大部分出現在Ⅱ類和Ⅲ類中,說明這2類的基因相對比較保守。
2.3 毛竹PheARF基序分析
為了進一步了解毛竹ARF保守區域的結構,通過MEME在線工具構建基序分析圖,結果如圖2所示。毛竹ARF成員含有的基序結構不一,大概有20個不同的基序組成。其中每個基因都包含不同種類的基序,大部分的基序為4~17個。大部分的PheARF基因都包含Motif1~8,Motif10,Motif11,Motif14,Motif18和Motif20等,這些基序出現的次數較多,而Motif13,Motif16和Motif17等基序在這些基因中不常見。從進化角度來看,同源關系較近的Ⅰ,Ⅱ,Ⅲ類的基因在基序的長度和種類較為相似,比如Ⅰ類的 PheARF4,PheARF9和 PheARF32等,Ⅱ類的 PheARF22,PheARF31和 PheARF33等,Ⅲ類的PheARF17,PheARF18和PheARF37等。以上結果說明大部分的毛竹ARF家族基因是相當保守的。
2.4 毛竹PheARF基因在花器官中差異表達分析
為了研究毛竹PheARF基因在花器官發育中的作用和調控機制,進一步分析PheARF基因在花芽、苞片、穎片、稃片、雌蕊、雄蕊、幼胚和未開花的葉中表達分析。結果表明:PheARF12,PheARF13,PheARF14,PheARF15,PheARF24,PheARF35,PheARF43,PheARF23和PheARF32在花芽中高量表達,說明這些ARF基因可能在開花初期起著重要的調控作用(圖3)。在雄蕊中,PheARF9,PheARF29,PheARF31, PheARF10, PheARF4, PheARF22, PheARF11, PheARF25, PheARF40, PheARF8,PheARF39,PheARF1,PheARF44,PheARF45,PheARF42和PheARF41高量表達,但是這些基因在幼胚中表達量極低。PheARF2,PheARF30,PheARF14,PheARF13,PheARF35,PheARF7,PheARF37和PheARF38在雌蕊中表達量較高,同時在幼胚中也高量表達,說明這些基因同時調控毛竹雌蕊形成和幼胚的發育。PheARF13,PheARF14和PheARF35同時在花芽、雌蕊和幼胚中高表達,這3個基因可能既調控開花又能控制花的發育。此外,PheARF5,PheARF18,PheARF27,PheARF10和PheARF4在穎片和稃片中都高量表達,可能調節稃片和穎片的發育。
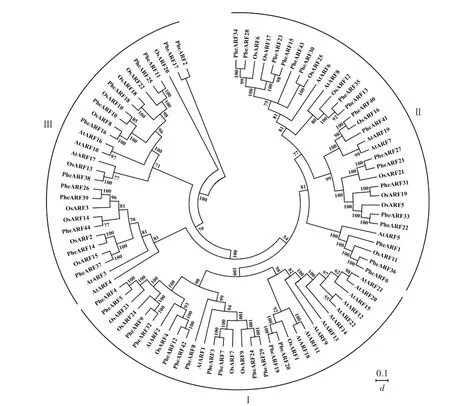
圖1 毛竹、水稻和擬南芥ARF的系統進化樹分析Figure 1 Phylogenetic analyses of the ARF proteins in Phyllostachys edulis,Oryza sativa and Arabidopsis thaliana
3 討論
生長素是植物器官發育和模式形態形成和發育非常重要的信號分子。生長素轉導途徑中最重要的2類家族分別為ARFs和Aux/IAAs[19]。在發育過程中,ARFs能直接地調控下游靶基因的表達[6,20],但大部分的ARF和Aux/IAA都是以家族的形式存在,所以它們在植物體內的調控機制是相當復雜的[21-23]。ARFs基因也參與生殖過程[24]。木瓜Carica papaya和番茄Solanum lycopersicum的ARFs基因家族的分析和鑒定揭示了該家族基因在花和果實的發育過程中具有調控作用[25-26]。
在本研究中,通過生物信息學工具,檢索44個毛竹的ARF基因。在擬南芥和水稻中的ARF基因分別為23和25個。ARF基因家族在不同植物中成員數量不同,這可能與植物體內的基因組復制有關[27],而基因數量多少由該基因復制事件的頻率決定。毛竹基因組為2 G左右[28],大于水稻和擬南芥的基因組。以上分析表明:毛竹ARF基因復制事件的頻繁發生有可能導致毛竹ARF家族成員數量增多。通過系統進化分析,毛竹ARF家族基因大致分為3個亞組(圖1),亞組內的基因相似性比較高,但是亞組之間相似性不高,或許在基因功能上也有差異。毛竹與擬南芥和水稻的ARF家族的系統進化關系表明,毛竹與水稻之間有11對同源基因對,這些基因對都屬于Ⅱ類和Ⅲ類,一方面說明毛竹的ARF家族與水稻有很高的同源性(圖1),另一方面說明毛竹ARF家族Ⅱ類和Ⅲ類的基因保守性很高。

圖2 毛竹ARF保守基序分析Figure 2 Distribution of conserved motifs in Phylloastachys edulis ARF proteins identified using MEME search tool
研究人員對水稻和擬南芥的ARF基因進行了廣泛地研究[29-31]。ARF基因在花和種子發育中起著非常重要的作用,為研究毛竹的開花及種子發育提供了許多有用的信息。據報道,擬南芥的AtARF3和AtARF4參與花的發育,在花中高量表達[32]。與AtARF3和AtARF4同源的毛竹PheARF14和PheARF37在幼胚和雌蕊中都高量表達,說明2個毛竹ARF基因可能與AtARF3和AtARF4基因功能相似,推測它們在毛竹花和種子發育過程中起著關鍵的作用。
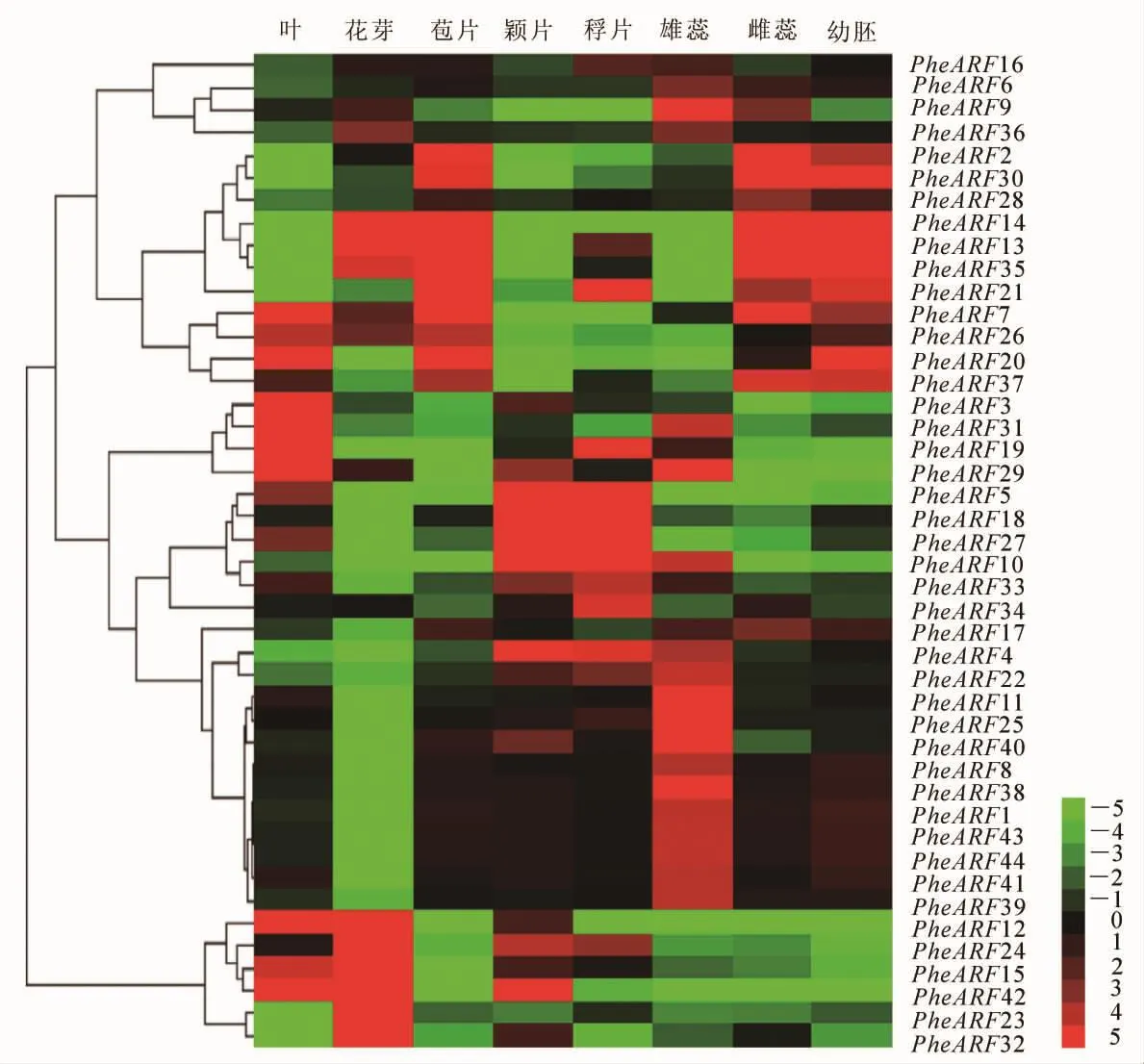
圖3 毛竹PheARF基因在不同花器官中表達模式分析Figure 3 Expression analysis of PheARF genes among different flower organs of Phyllostachys edulis
[1] DAVIES P J.Plant Hormones:Physiology,Biochemistry and Molecular Biology[M].Dordrecht:Kluwer Academic Publishers,2013.
[2] THEOLOGISA.Rapid gene regulation by auxin[J].Ann Rev Plant Physiol,1986,37(1):407-438.
[3] GUILFOYLE T J,ULMASOV T,HAGEN G.The ARF family of transcription factors and their role in plant hormoneresponsive transcription[J].Cell Mol Life Sci,1998,54(7):619-627.
[4] SHEN Chenjia,YUE Runqing,SUN Tao,et al.Genome-wide identification and expression analysis of auxin response factor gene family in Medicago truncatula[J].Front Plant Sci,2015,6(11):3932-3935.
[5] TIWARISB,HAGEN G,GUILFOYLE T.The roles of auxin response factor domains in auxin-responsive transcription[J].Plant Cell,2003,15(2):533-543.
[6] GUILFOYLE T J,HAGEN G.Auxin response factors[J].Curr Opin Plant Biol,2007,10(5):453-460.
[7] RAMOS JA,ZENSER N,LEYSER O,et al.Rapid degradation of auxin/indoleacetic acid proteins requires conserved amino acids of domainⅡand is proteasome dependent[J].Plant Cell,2001,13(10):2349-2360.
[8] ULMASOV T,HAGEN G,GUILFOYLE T J.ARF1,a transcription factor that binds to auxin response elements[J]. Science,1997,276(5320):1865-1868.
[9] HAGEN G,GUILFOYLE T.Auxin-responsive gene expression:genes,promoters and regulatory factors[J].Plant Mol Biol,2002,49(3/4):373-385.
[10] REMINGTON D L,VISION T J,GUILFOYLE T J,et al.Contrasting modes of diversification in the Aux/IAA andARF gene families[J].Plant Physiol,2004,135(3):1738-1752.
[11] LISCUM E,REED JW.Genetics of Aux/IAA and ARF action in plant growth and development[J].Plant Mol Biol, 2002,49(3/4):387-400.
[12] SESSIONSR.Arabidopsis (Brassicaceae)flower development and gynoecium patterning in wild type and ettin mutants[J].Am JBot,1997,84(9):1179-1179.
[13] HARDTKE C S,BERLETH T.The Arabidopsis gene MONOPTEROS encodes a transcription factormediating embryo axis formation and vascular development[J].EMBO J,1998,17(5):1405-1411.
[14] HARPER R M,STOWE-EVANSE L,LUESSE D R,et al.The NPH4 locus encodes the auxin response factor ARF7, a conditional regulator of differential growth in aerial Arabidopsis tissue[J].Plant Cell,2000,12(5):757-770.
[15] THOMPSON JD,GIBSON T J,PLEWNIAK F,et al.The CLUSTAL_X windows interface:flexible strategies formultiple sequence alignment aided by quality analysis tools[J].Nucl Acids Res,1997,25(24):4876-4882.
[16] TAMURA K,STECHER G,PETERSON D,et al.MEGA6:molecular evolutionary genetics analysis version 6.0[J]. Mol Biol Evol,2013,30(12):2725-2729.
[17] BAILEY T L,ELKAN C.The value of prior knowledge in discoveringmotifs with MEME[C].Proc Third Int Conf Intel SystMol Biol,California:AAAIPress,1995:21-29.
[18] SALDANHA A J.Java Treeview-extensible visualization ofmicroarray data[J].Bioinformatics,2004,20(17):3246-3248.
[19] FARCOTE,LAVEDRINE C,VERNOUX T.Amodular analysis of the auxin signalling network[J].PloSOne,2015, 10(3):864-865.
[20] ULMASOV T,MURFETT J,HAGEN G,et al.Aux/IAA proteins repress expression of reporter genes containing natural and highly active synthetic auxin response elements[J].Plant Cell,1997,9(11):1963-1971.
[21] 王壘,陳勁楓,婁麗娜,等.黃瓜果實中 ARF和 Aux/IAA基因對外源激素的應答[J].西北植物學報,2011,31(6):1127-1131.
WANG Lei,CHEN Jinfeng,LOU Lina,et al.Expression analysis about some ARF and Aux/IAA family members in fruits of cucumberwith exogenous hormones[J].Acta Bot Boreal-Occident Sin,2011,31(6):1127-1131.
[22] 史夢雅,李陽,張巍,等.生長素反應因子作用機制研究進展[J].生物技術通報,2012(8):24-28.
SHIMengya,LIYang,ZHANGWei,et al.Progress in mechanism of auxin response factors[J].Biotechnol Bull, 2012(8):24-28.
[23] AUDRAN-DELALANDE C,BASSA C,MILA I,et al.Genome-wide identification,functional analysis and expression profiling of the Aux/IAA gene family in tomato[J].Plant Cell Physiol,2012,53(4):659-672.
[24] CHO H,RYU H,RHO S,et al.A secreted peptide acts on BIN2-mediated phosphorylation of ARFs to potentiate auxin response during lateral root development[J].Nat Cell Biol,2014,16(1):66-76.
[25] PAULL R E,IRIKURA B,WU Pingfang,et al.Fruit development,ripening and quality related genes in the papaya genome[J].Trop Plant Biol,2008,1(3/4):246-277.
[26] KUMAR R,TYAGIA K,SHARMA A K.Genome-wide analysis of auxin response factor(ARF)gene family from tomato and analysis of their role in flower and fruit development[J].Mol Gen Genomics Mgg,2011,285(3):245-260.
[27] OHNO S.Evolution by Gene Duplication[M].Heidelberg:Springer Verlag,2013.
[28] PENG Zhenhua,LU Ying,LILubin,et al.The draft genome of the fast-growing non-timber forest speciesmoso bamboo(Phyllostachys heterocycla)[J].Nat Gen,2013,45(4):456-461.
[29] QIYanhua,WANG Suikang,SHEN Chenjia,et al.OsARF12,a transcription activator on auxin response gene,regulates root elongation and affects iron accumulation in rice(Oryza sativa)[J].New Phytol,2012,193(1):109-120.
[30] COLE M,CHANDLER J,WEIJERS D,et al.DORNR?SCHEN is a direct target of the auxin response factor MONOPTEROS in the Arabidopsis embryo[J].Development,2009,136(10):1643-1651.
[31] LIJisheng,DAIXinhua,ZHAO Yunde.A role for auxin response factor 19 in auxin and ethylene signaling in Arabidopsis[J].Plant Physiol,2006,140(3):899-908.
[32] HUNTER C,WILLMANN M R,WU Gang,et al.Trans-acting siRNA-mediated repression of Ettin and ARF4 regulates heteroblasty in Arabidopsis[J].Development,2006,133(15):2973-2981.
Bioinformatic analysis and differential expression of auxin response factor(ARF)gene in Phyllostachys edulis
CHENG Zhanchao,HOU Dan,MA Yanjun,GAO Jian
(Key Laboratory of Bamboo and Rattan Science and Technology of State Forestry Administration,International Center for Bamboo and Rattan,Beijing 100102,China)
The auxin response factor (ARF)gene family plays a key role in plant growth and developmental processes,such as root and shoot development aswell as flower and fruit development.To provide a theoretical basis for flower and seed development of Phyllostachys edulis,a genome-wide analysis of the previously undocumented ARF gene family for Ph.edulis was conducted.In this study a whole-genome survey of Ph.edulis was performed and a detailed analysis of the gene motif and phylogenetic classification was provided.Results showed 44 ARF geneswhich were classified into three groups.A comparative analysis of the ARF genes among Ph.edulis,rice,and Arabidopsis suggested a total of 11 sister pairs (OsARF-PheARF)providing insights into various orthologous relationships between OsARFs and PheARFs.For ARF expression patterns of diverse floral organs,PheARF13,PheARF14,and PheARF35 showed the highest expression in the flower bud,pistil,and young embryo;whereas,PheARF2,PheARF30,PheARF14,PheARF13,PheARF35,PheARF7,PheARF37,and PheARF38 were highly expressed in the pistil and the young embryo.This study suggested that ARF genesmay play a very critical role during flower and fruit development of Ph.edulis.[Ch,3 fig.1 tab.32 ref.]
forest tree breeding;Phyllostachys edulis;auxin response factors;floral development
S722
A
2095-0756(2017)04-0574-07
10.11833/j.issn.2095-0756.2017.04.002
2016-06-13;
2016-07-14
國家高技術研究發展計劃(“863”計劃)項目(2013AA102607-4);國家自然科學基金資助項目(31570673)
程占超,博士,從事林木遺傳育種研究。E-mail:chengzhan_chao@126.com。通信作者:高健,研究員,博士,博士生導師,從事林木遺傳育種研究。E-mail:gaojian@icbr.ac.cn

