基于RNA-seq分析Eha調控遲緩愛德華菌抵抗酸化的作用
劉 念,李玉紅,2,鄭恩金,高大慶,陸承平
?
基于RNA-seq分析Eha調控遲緩愛德華菌抵抗酸化的作用
劉 念1,李玉紅1,2,鄭恩金1,高大慶1,陸承平3
目的 Eha是一個影響遲緩愛德華菌(Et)胞內生存的轉錄調控因子,本研究有助于揭示其調控Et抵御酸的分子機制。方法 用ATPase 抑制劑洛霉素A1抑制巨噬細胞的酸化,菌落計數法比較酸化對野生株和eha基因缺失株胞內存活數目的影響;比較兩種細菌在酸性應激實驗中存活率的差異;構建pMP220-PehaLacZ質粒,采用β-半乳糖苷酶實驗檢測eha基因的啟動子在不同酸性pH值下和不同培養時間的轉錄活性;選擇Eha轉錄水平最高的一個酸性pH值和培養時間,分別提取兩種細菌RNA,進行RNA-Sequencing;并用qRT-PCR驗證其結果。結果 野生株ET13在巨噬細胞內和不同pH酸環境中的存活率明顯高于缺失株,阻止酸化胞內菌數明顯高于未阻止酸化的胞內菌數(P<0.05)。對數期細菌pH6.3培養基生長2 h,RNA-Sequencing結果表明:eha基因缺失株轉錄水平和野生株相比,147個差異顯著表達的基因(DEGs)(|log2 Ratio|≥1),其中113個上調,34個基因下調,qRT-PCR隨機抽樣,和RNA-Sequencing 表達趨勢呈強相關。147個基因采用GO數據庫進行功能聚類,分成25類,主要涉及細菌加工、定位、代謝、結合、催化、運輸、細胞成份;基于KEGG通路的富集分析,有130個可以富集到55條通路中,包括與氨基酸、核苷酸、脂質代謝及鐵的轉運等路徑,涉及基因較多的有雙組分系統、ABC轉運系統、不同環境中的微生物代謝和次級代謝產物等路徑。結論 在酸性生存環境,Eha對Et的轉錄組呈多途徑、多基因的適應性的全局性調控。
eha基因;RNA-sequencing;E.tarda;酸化
遲緩愛德華菌(Edwardsiellatarda,簡稱Et)屬于腸桿菌科愛德華菌屬,該菌能夠感染多種魚類,是水產養殖業的重要病原體[1]。Et也可感染人,引起人的胃腸炎、腹膜炎、敗血癥和腦膜炎等疾病[2]。Et溶血調控基因(Et haemolysin activator gene,簡稱eha)是Et一個重要的毒力調控基因[3-4],Et菌能夠抵抗巨噬細胞吞噬體內氧化和酸化的殺菌而生存繁殖,是其致病的關鍵[5],eha基因缺失引起Et 在巨噬細胞內繁殖率降低的機制[6],尚未完全研究清楚。高通量測序技術,具有測序通量高、成本低、速度快等特點,廣泛應用于動植物及微生物的基因組學、轉錄組學等領域的研究。
近年來,隨著新一代高通量測序技術(High-throughput sequencing technology)的發展,轉錄組測序(RNA sequencing,RNA-Seq)技術已成為測序技術的重要手段。本研究首先分析Eha在調控細菌抵御巨噬細胞酸化殺菌的作用,采用RNA-Seq比較在酸性條件下,ET-13野生株和eha基因缺失株轉錄組表達差異的基因(Differentially Expressed Genes,DEGs),通過生物信息學對其數據進行定性定量的分析,旨在分子水平識別DEGs的功能及其代謝通路,并利用熒光定量PCR隨機抽樣,驗證DEGs表達趨勢,初步探索Eha在調控Et抵御巨噬細胞酸化殺菌的分子機制。
1 材料與方法
1.1 Et毒力株 ET13為南京農業大學陸承平教授惠贈;ET13的eha基因缺失株為本室保存;鼠源巨噬細胞RAW264.7為上海獸醫研究所王少輝博士惠贈;pMP220-PehalacZ質粒東南大學毛曉華教授惠贈。細菌培養基(Luria Broth,LB):細胞培養液(Dulbecco’s Minimum Essential Medium,DMEM),胎牛血清(Fetal Bovine Serum,FBS)均購自Gibco公司。
1.2 抑制酸化對細菌胞內存活的影響 方法參考文獻[6],實驗組用含1 μmol/L ATPase 抑制劑洛霉素(Bafilomycin,BAF)A1的培養液處理細胞30 min,以阻止細胞吞噬體酸化,對照組用正常細胞培養液。對數期ET13野生株和eha缺失株以10∶1的感染復數 (Multiplicity Of Infection,MOI)分別加入24孔單層細胞。共孵育1 h后,再用含慶大霉素(100 μg/mL)的培養液殺胞外的細菌1 h,此時定為0 h;換用含慶大霉素(10 μg/mL)的培養液繼續培養0 h,2 h,4 h 或 6 h。在預定時間點,加入1 mL 1% Triton X-100裂解細胞10 min,將裂解產物經稀釋,涂平板,培養,菌落記數。以培養時間(h)為橫坐標,活菌數(CFU/mL)為縱坐標,繪制細菌胞內細菌數目和時間曲線。
1.3 酸性環境的應激實驗 方法參考文獻[6],對數期的菌體用PBS(pH7.2)漂洗2次后,菌液 OD600調節為0.5。菌液分為兩等份,一組為實驗組,經不同pH(7.2,6.3,5.5,4.8,4.3)LB處理2 h,另一組作為對照組,正常培養。然后進行兩組培養液系列稀釋,涂平板,計數菌落。菌液濃度=平板菌落數/mL×稀釋倍數,細菌存活率=實驗組菌液濃度(CFU/mL)/對照組菌液濃度(CFU/mL)×100%。
1.4 構建eha基因的啟動子和LacZ融合質粒及β-半乳糖苷酶實驗 以ET13基因組DNA為模板,擴增eha的啟動子區域,將該片段插入 pMP220-LacZ載體中,構建了eha基因的啟動子探針載體pMP220-PehaLacZ,然后將該載體電極導入eha基因缺失株中,檢測eha基因的啟動子在不同酸性pH值下和不同培養時間的轉錄活性,選擇Eha轉錄水平最高的一個酸性pH值和培養時間。
1.5 提取細菌的RNA 野生株與eha基因缺失株分別在pH值7.2的LB培養至對數生長期(OD值0.8),再在pH值6.3的LB培養2 h。分別提取兩種細菌的RNA,并檢測其濃度(μg/μL)和純度(OD260/OD280,OD260/OD230),及RNA完整性計數(RNA Integrity Number,RIN)。
1.6 cDNA文庫的構建與Illumina測序儀測序 根據細菌mRNA純化試劑盒的說明書提供的方法,將提取的總RNA中16S rRNA 與23S rRNA 除去,用帶有Oligo T(dT)的磁珠對mRNA進行富集純化。mRNA打斷成100-500 nt短片段后逆轉錄成cDNA,3′末端加polyA并連接測序接頭,連接產物經PCR擴增得到測序文庫,上機進行 Illumina Hi Seq 2000 測序(深圳華大基因公司完成)。
1.7 轉錄組數據分析 測序所得的原始數據稱為原始標簽(raw read),再進行過濾得到凈標簽(clean read),用于后續分析。該序列信息已提交至NCBI Sequence Read Achieve數據庫,登錄號:SRX 1898774。
1.8 差異表達基因分析 利用短reads比對軟件SOAPaligner/SOAP2 軟件,將測序數據與E.tarda菌ATCC15947參考基因組序列(http://www.ncbi.nlm.nih.gov/ nuccore/)進行比對,統計clean reads 參考基因組及基因序列上的分布情況及覆蓋度;利用 RPKM(Reads Per Kb per Million)法計算基因的表達量,用于比較兩樣品間基因的差異表達情況,以p-value做多重假設檢驗校正,通過控制錯誤發生率(false discovery rate,FDR)來決定P閾值;采用Gene Ontology(GO)數據庫(http://www.geneontology.org/)和Kyoto Encyclopedia of Genes and Genomes (KEGG)數據庫(http://www. genome. jp/kegg/)進行差異表達基因的功能聚類和生物學功能分析。
1.9 實時熒光定量PCR(quantitative real time PCR,qRT-PCR) 分別提取野生株與eha基因缺失株的RNA,隨機選取RNA-seq差異顯著15個基因,根據表1設計引物,操作按試劑盒說明書,反應在ABI7300型熒光定量PCR儀上進行,以16S rRNA基因的表達量作為內標參照,采用2-ΔΔct法計算eha基因缺失株的基因轉錄水平相對野生株的倍數,這些基因的表達趨勢和RNA-seq表達的基因趨勢做相關性分析。
表1 qRT-PCR引物序列,產物大小和變性溫度
Tab.1 Primer sequence product size and annealing temperature for RT-qPCR
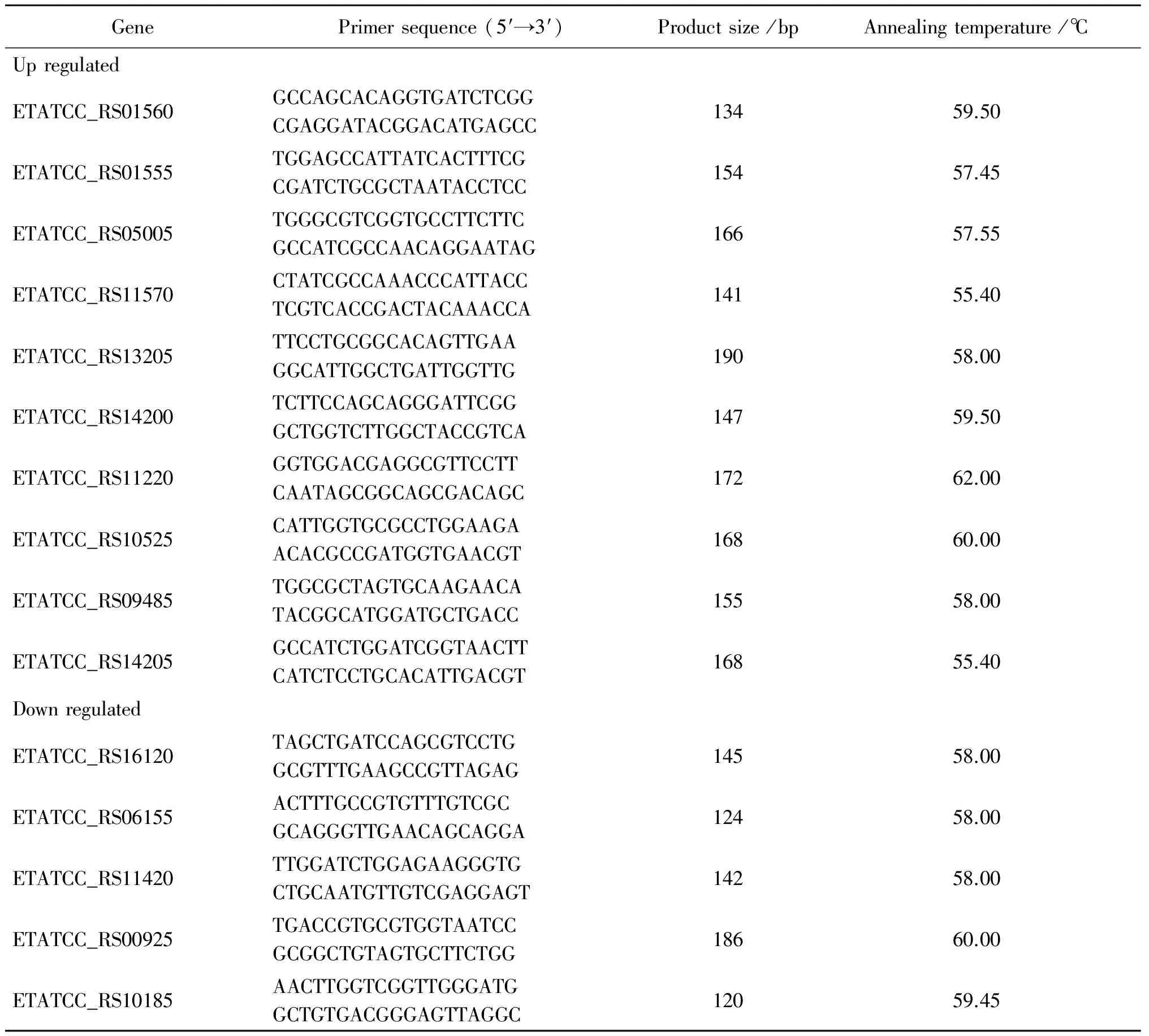
GenePrimersequence(5′→3′)Productsize/bpAnnealingtemperature/℃UpregulatedETATCC_RS01560GCCAGCACAGGTGATCTCGGCGAGGATACGGACATGAGCC13459.50ETATCC_RS01555TGGAGCCATTATCACTTTCGCGATCTGCGCTAATACCTCC15457.45ETATCC_RS05005TGGGCGTCGGTGCCTTCTTCGCCATCGCCAACAGGAATAG16657.55ETATCC_RS11570CTATCGCCAAACCCATTACCTCGTCACCGACTACAAACCA14155.40ETATCC_RS13205TTCCTGCGGCACAGTTGAAGGCATTGGCTGATTGGTTG19058.00ETATCC_RS14200TCTTCCAGCAGGGATTCGGGCTGGTCTTGGCTACCGTCA14759.50ETATCC_RS11220GGTGGACGAGGCGTTCCTTCAATAGCGGCAGCGACAGC17262.00ETATCC_RS10525CATTGGTGCGCCTGGAAGAACACGCCGATGGTGAACGT16860.00ETATCC_RS09485TGGCGCTAGTGCAAGAACATACGGCATGGATGCTGACC15558.00ETATCC_RS14205GCCATCTGGATCGGTAACTTCATCTCCTGCACATTGACGT16855.40DownregulatedETATCC_RS16120TAGCTGATCCAGCGTCCTGGCGTTTGAAGCCGTTAGAG14558.00ETATCC_RS06155ACTTTGCCGTGTTTGTCGCGCAGGGTTGAACAGCAGGA12458.00ETATCC_RS11420TTGGATCTGGAGAAGGGTGCTGCAATGTTGTCGAGGAGT14258.00ETATCC_RS00925TGACCGTGCGTGGTAATCCGCGGCTGTAGTGCTTCTGG18660.00ETATCC_RS10185AACTTGGTCGGTTGGGATGGCTGTGACGGGAGTTAGGC12059.45
1.10 統計學分析 兩組數據之間的比較采用t檢驗,以P<0.05 為差異有統計學意義。qRT-PCR基因的表達趨勢和RNA-seq表達的基因趨勢,用R2做相關性分析,R2>0.75表示強相關性,0.75>R2>0.45表示中等相關性,R2<0.45表示弱相關性。
2 結 果
2.1 比較抑制酸化對ET13野生株和eha基因缺失株在胞內存活的影響 從圖1中可以看出,先用ATPase 抑制劑洛霉素A1處理巨噬細胞,以阻止酸化,再用野生株和eha基因缺失株分別感染RAW264.7細胞1 h,慶大霉素殺胞外菌1 h,繼續培養4 h 或6 h時,它們胞內CFU/mL明顯高于各自未阻止酸化的胞內CFU/mL(P<0.05),表明巨噬細胞的酸化在抑制Et胞內繁殖中起了很大作用,但酸化對野生株和缺失株各自抑制程度明顯不同。繼續培養6 h時,未阻止酸化野生株胞內CFU/mL和未阻止酸化eha基因缺失株胞內CFU/mL差異明顯(P<0.05),這說明eha基因在調控細菌抵御巨噬細胞酸化殺菌中起了重要作用,但同時,阻止酸化野生株胞內CFU/mL和阻止酸化eha基因缺失株胞內CFU/mL差異也明顯(P<0.05),這也說明eha基因在調控細菌抵御巨噬細胞殺菌中其它機制也起作用,如我們以前的研究表明,Eha蛋白能夠調控Et 抵抗Mф 氧化殺菌的作用[6]。
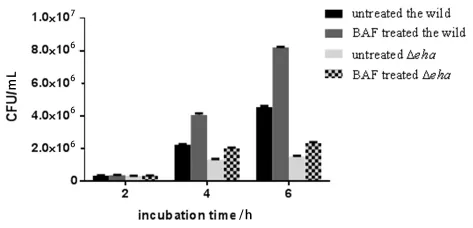
圖1 比較酸對野生株和eha基因缺失株巨噬細胞胞內存活的不同影響Fig.1 Comparison of the differences of the intracellular survival rates of the wild type and its eha mutant in macrophages against acid
2.2 比較經不同pH LB處理,野生株和eha基因缺失株存活率的差異 模擬細菌在巨噬細胞吞噬溶酶體中酸性的環境,對數期細菌經不同pH(7.2,6.3,5.5,4.8,4.3)LB處理細菌2 h后,結果如圖2所示,eha基因缺失株的存活率明顯低于野生株(P<0.05),互補株的存活率介于缺失株和野生株之間。結果表明,eha基因在細菌在體外抵御酸的殺菌機制中起重要作用。
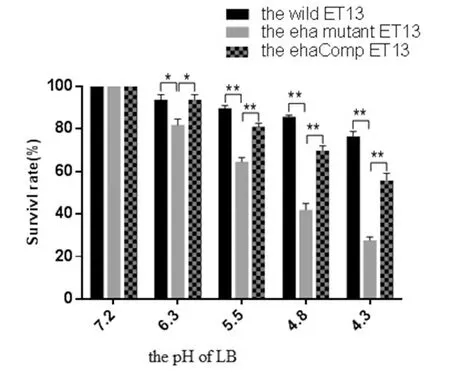
圖2 比較不同pH酸對野生株和eha基因缺失株存活率的影響Fig.2 Comparison of the differences of the survival rates of the wild type and its eha mutant against different pH acid
2.3 檢測eha基因的啟動子在不同環境刺激下的轉錄活性 體外模擬在巨噬細胞酸的生存環境,分別在不同時間和pH值的LB培養Et菌,檢測含pMP220-PehaLacZEt菌的β-半乳糖苷酶活性,結果表明,對數期的Et菌在pH值6.3的LB培養2 h,Eha轉錄水平最高。
2.4 轉錄組的測序數據評估 提取野生株與eha基因缺失株RNA濃度分別為1.905 μg/μL和1.255 μg/μL;純度:OD260/OD280,≥1.9,OD260/OD230≥2.0;RIN:10.0,表明其純度高,滿足建庫要求。為了構建Et菌的cDNA文庫,其mRNA被隨機性打斷。其隨機性評估表明,ET13菌野生株和缺失株轉錄組測序不同的長度的clean reads在基因組中分布均勻。利用軟件將成功比對到基因組上的clean reads與參考基因序列進行比對,進行基因覆蓋度分析,結果表明,野生株和eha基因缺失株的clean reads中覆蓋率在90%以上的基因分別超過92%。
2.5eha基因缺失株和野生株差異表達基因比較和功能聚類分析 利用RPKM法計算兩種細菌基因的表達量,閾值設為:|log2 Ratio|≥1,FDR≤0.001。在ET13野生株中,共有147個基因的轉錄水平與eha基因缺失株相比差異顯著,其中113個基因轉錄水平上調,34個基因轉錄水平下調。采用GO數據庫對147個差異表達的基因進行功能聚類分析,分成25類,這些基因主要涉及細胞加工(68個)、定位(30個)、結合(59個)、催化(83個)、運輸 (27個)、代謝 (78個)、細胞成份(37個)和細胞膜(33個) (圖3),Eha在酸性條件下調節這些基因。
基于KEGG通路的顯著性富集分析,能確定DEGs參與的最主要生化代謝途徑和信號轉導途經。結果顯示,147個DEGs中,有130個基因可以富集到55條通路中,包括10條與氨基酸代謝相關的路徑,16條與碳代謝、脂質、酯酸及能量代謝相關的路徑,4條與核苷酸代謝相關的路徑,3條與硫代謝相關的路徑,以及一些與氮代謝、鐵的轉運等相關的路徑。涉及差異表達基因較多的代謝路徑主要有(|log2 Ratio|≥1) (表2):雙組分系統路徑(20個),ABC轉運系統路徑(12個),不同環境中的微生物代謝路徑(21個)和次代謝的合成路徑(17個)。
2.6 實時熒光定量PCR 隨機選取 15個DEGs進行qRT-PCR,驗證RNA-seq測結果。兩種方法結果表明,14個基因的表達趨勢出現了一致的結果,其相關性為強相關(R2=0.799>0.75),僅ETATCC-RS11570基因在eha基因缺失株和野生株中通過qRT-PCR檢測無明顯差異,與RNA-seq結果不一致(圖4)。
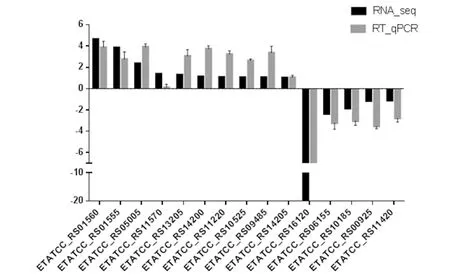
圖4 比較qRT-PCR和RNA-seq發現的差異表達基因Fig.4 Comparison of significantly different expressed genes by qRT-PCR和RNA-seq
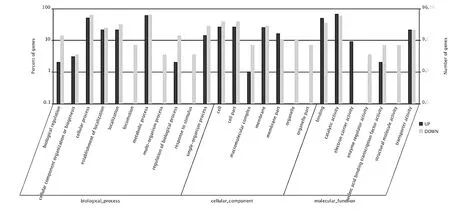
Classification of DEGs between the wild type and the eha mutant,according to GO functional analysis (|log2 Ratio|≥1).圖3 ET 13 eha基因缺失株和野生株差異表達基因比較和聚類分析Fig.3 Classification of significantly different expressed genes between the wild type and its eha mutant of ET 13
3 討 論
本研究首先用洛霉素A1抑制酸化和菌落計數法,比較酸化對ET13株和它的eha基因缺失株胞內存活數目的影響,以及比較兩種細菌在體外酸性應激實驗中存活率的差異,發現了Eha在調控Et菌抵御巨噬細胞酸化殺菌和體外酸性環境中的重要作用。然后,采用β-半乳糖苷酶實驗檢測eha的啟動子在不同酸性pH值下和不同培養時間的轉錄活性;選擇Eha轉錄水平最高的一個酸性pH值和培養時間,分別提取兩種細菌RNA,進行RNA-Seq,比較ET13株和eha基因缺失株的轉錄組表達,發現了147個DEGs,對它們進行GO功能聚類和KEGG通路分析,結果如下:
為了生存和適應外界環境,細菌通過雙組分系統(Two-component system,TCS)等信號轉導途徑來響應環境信號并對刺激作出應答。本研究提示,20個DEGs涉及TCS系統路徑。在酸壓力下,eha基因缺失株中有7個DEGs上調,13個DEGs下調。ETATCC_RS06140、ETATCC_RS06135 和ETATCC _RS06130分別編碼檸檬酸裂合酶CitC的α、β、γ亞基,ETATCC_RS06145和ETATCC_RS06150編碼CitG亞基。檸檬酸裂合酶是參與三羧酸循環的關鍵酶,已經報道,檸檬酸裂合酶基因突變的Et在感染魚的模型中生存力和致病性明顯下降[7]。ETATCC_RS14195和ETATCC_RS00005以及ETATCC_RS12655分別編碼谷氨酰胺酶和絲氨酸脫水酶。幽門螺桿菌產生的谷氨酰胺酶可以催化谷氨酰胺脫氨,中和胃酸,這和該菌在胃的生存能力和致病性有關[8]。ETATCC_RS16530編碼一種外膜蛋白,它負責將莢膜多糖從周質間隙運輸到菌體表面,而莢膜直接參與細菌對環境的抗逆性[9]。Et菌通過Eha調控這些基因的表達量以抵抗酸性環境的刺激,增加其在細胞內的存活能力。
表2 基于KEGG通路分析在酸性條件eha基因調控差異明顯表達的基因
Tab.2 KEGG pathway analysis of theeha-dependent genes differentially expressed in an acid condition
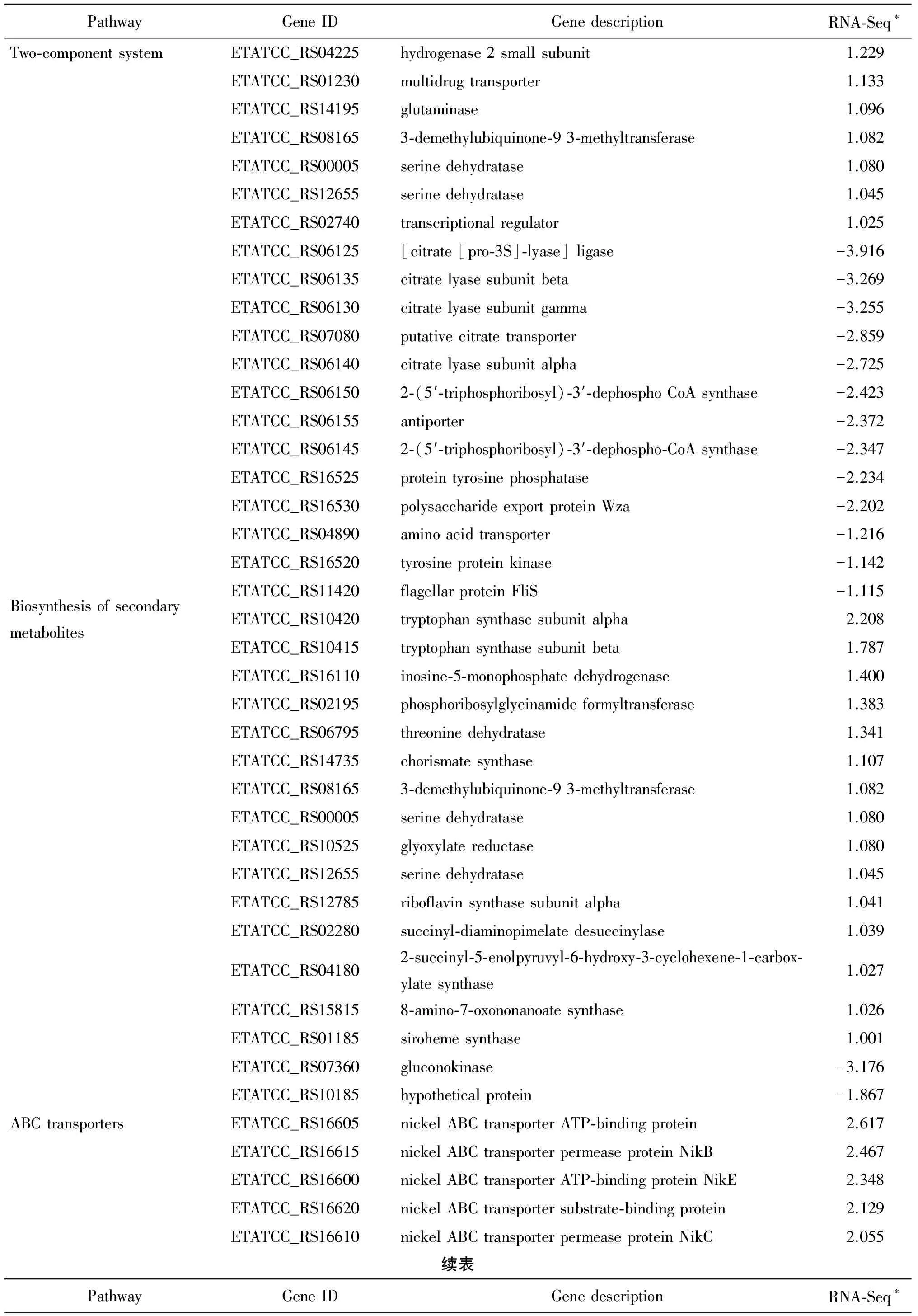
PathwayGeneIDGenedescriptionRNA?Seq?Two?componentsystemETATCC_RS04225hydrogenase2smallsubunit1.229ETATCC_RS01230multidrugtransporter1.133ETATCC_RS14195glutaminase1.096ETATCC_RS081653?demethylubiquinone?93?methyltransferase1.082ETATCC_RS00005serinedehydratase1.080ETATCC_RS12655serinedehydratase1.045ETATCC_RS02740transcriptionalregulator1.025ETATCC_RS06125[citrate[pro?3S]?lyase]ligase-3.916ETATCC_RS06135citratelyasesubunitbeta-3.269ETATCC_RS06130citratelyasesubunitgamma-3.255ETATCC_RS07080putativecitratetransporter-2.859ETATCC_RS06140citratelyasesubunitalpha-2.725ETATCC_RS061502?(5′?triphosphoribosyl)?3′?dephosphoCoAsynthase-2.423ETATCC_RS06155antiporter-2.372ETATCC_RS061452?(5′?triphosphoribosyl)?3′?dephospho?CoAsynthase-2.347ETATCC_RS16525proteintyrosinephosphatase-2.234ETATCC_RS16530polysaccharideexportproteinWza-2.202ETATCC_RS04890aminoacidtransporter-1.216ETATCC_RS16520tyrosineproteinkinase-1.142ETATCC_RS11420flagellarproteinFliS-1.115BiosynthesisofsecondarymetabolitesETATCC_RS10420tryptophansynthasesubunitalpha2.208ETATCC_RS10415tryptophansynthasesubunitbeta1.787ETATCC_RS16110inosine?5?monophosphatedehydrogenase1.400ETATCC_RS02195phosphoribosylglycinamideformyltransferase1.383ETATCC_RS06795threoninedehydratase1.341ETATCC_RS14735chorismatesynthase1.107ETATCC_RS081653?demethylubiquinone?93?methyltransferase1.082ETATCC_RS00005serinedehydratase1.080ETATCC_RS10525glyoxylatereductase1.080ETATCC_RS12655serinedehydratase1.045ETATCC_RS12785riboflavinsynthasesubunitalpha1.041ETATCC_RS02280succinyl?diaminopimelatedesuccinylase1.039ETATCC_RS041802?succinyl?5?enolpyruvyl?6?hydroxy?3?cyclohexene?1?carbox?ylatesynthase1.027ETATCC_RS158158?amino?7?oxononanoatesynthase1.026ETATCC_RS01185sirohemesynthase1.001ETATCC_RS07360gluconokinase-3.176ETATCC_RS10185hypotheticalprotein-1.867ABCtransportersETATCC_RS16605nickelABCtransporterATP?bindingprotein2.617ETATCC_RS16615nickelABCtransporterpermeaseproteinNikB2.467ETATCC_RS16600nickelABCtransporterATP?bindingproteinNikE2.348ETATCC_RS16620nickelABCtransportersubstrate?bindingprotein2.129ETATCC_RS16610nickelABCtransporterpermeaseproteinNikC2.055續表PathwayGeneIDGenedescriptionRNA?Seq?
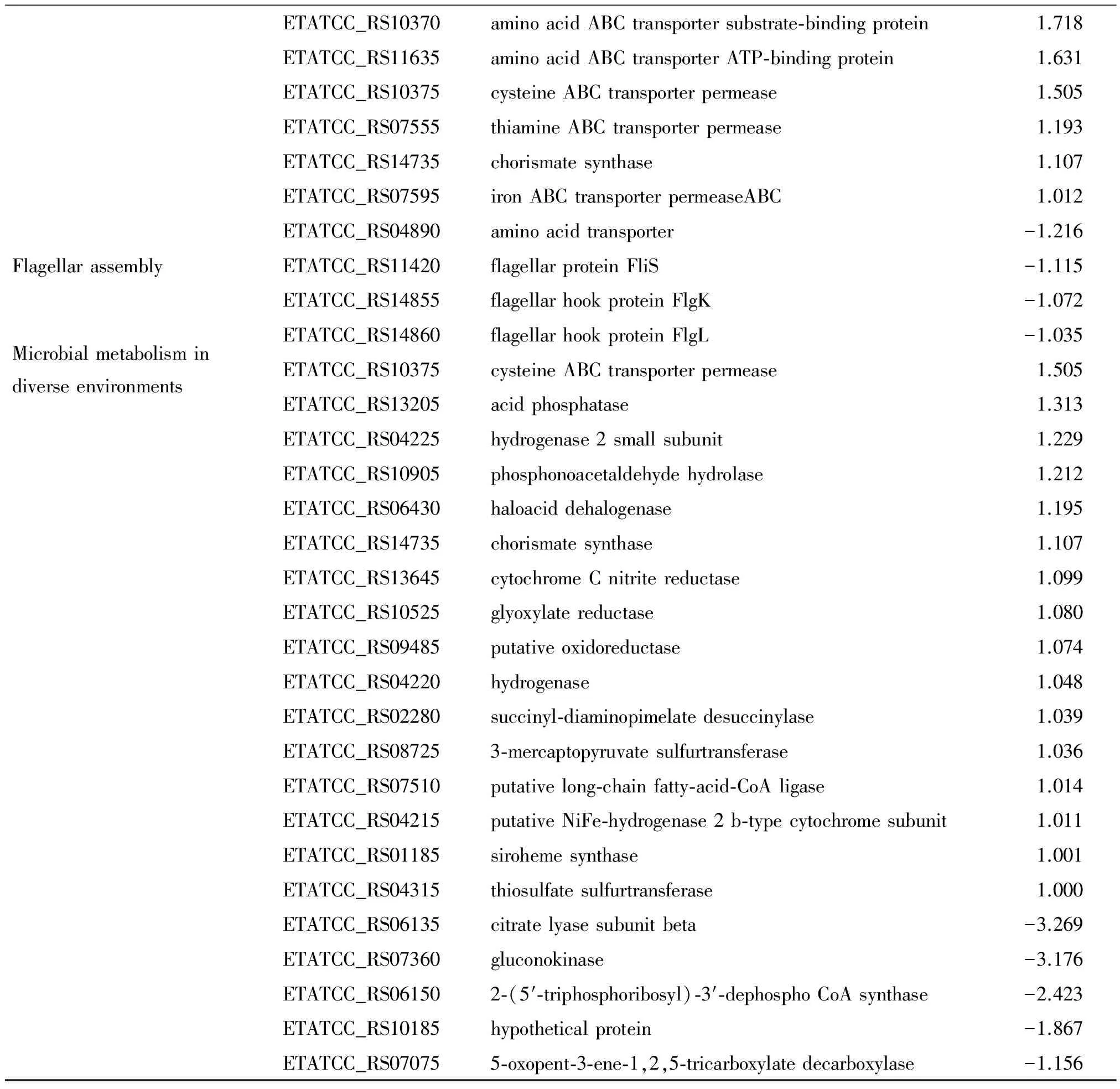
ETATCC_RS10370aminoacidABCtransportersubstrate?bindingprotein1.718ETATCC_RS11635aminoacidABCtransporterATP?bindingprotein1.631ETATCC_RS10375cysteineABCtransporterpermease1.505ETATCC_RS07555thiamineABCtransporterpermease1.193ETATCC_RS14735chorismatesynthase1.107ETATCC_RS07595ironABCtransporterpermeaseABC1.012ETATCC_RS04890aminoacidtransporter-1.216FlagellarassemblyETATCC_RS11420flagellarproteinFliS-1.115ETATCC_RS14855flagellarhookproteinFlgK-1.072ETATCC_RS14860flagellarhookproteinFlgL-1.035MicrobialmetabolismindiverseenvironmentsETATCC_RS10375cysteineABCtransporterpermease1.505ETATCC_RS13205acidphosphatase1.313ETATCC_RS04225hydrogenase2smallsubunit1.229ETATCC_RS10905phosphonoacetaldehydehydrolase1.212ETATCC_RS06430haloaciddehalogenase1.195ETATCC_RS14735chorismatesynthase1.107ETATCC_RS13645cytochromeCnitritereductase1.099ETATCC_RS10525glyoxylatereductase1.080ETATCC_RS09485putativeoxidoreductase1.074ETATCC_RS04220hydrogenase1.048ETATCC_RS02280succinyl?diaminopimelatedesuccinylase1.039ETATCC_RS087253?mercaptopyruvatesulfurtransferase1.036ETATCC_RS07510putativelong?chainfatty?acid?CoAligase1.014ETATCC_RS04215putativeNiFe?hydrogenase2b?typecytochromesubunit1.011ETATCC_RS01185sirohemesynthase1.001ETATCC_RS04315thiosulfatesulfurtransferase1.000ETATCC_RS06135citratelyasesubunitbeta-3.269ETATCC_RS07360gluconokinase-3.176ETATCC_RS061502?(5′?triphosphoribosyl)?3′?dephosphoCoAsynthase-2.423ETATCC_RS10185hypotheticalprotein-1.867ETATCC_RS070755?oxopent?3?ene?1,2,5?tricarboxylatedecarboxylase-1.156
Note:*RNA-seq,llog2 Ratio (eha_mutant_ET13/wild_ET13)l≥1.00
ABC轉運子(ABC transporter)是細菌膜上的一種運輸ATP酶,如寡肽透過酶(Oligopeptide permease,Opp),控制著必需營養物質進出細胞.在本研究中, 12個DEGs涉及該轉運路徑,編碼鎳、氨基酸、硫胺素、鐵等轉運蛋白。金葡菌Opp1ABC轉運子運輸鎳和鈷進入細胞,該突變子降低黏附小鼠膀胱和腎臟的能力,以及在泌尿道感染的模型中小鼠死亡率[10]。Et菌通過Eha調控這些基因的表達量以抵抗酸性環境的刺激,增加其在細胞內的存活能力。
微生物次級代謝產物的合成通常以初級代謝產物為前體,并受其調節;次級代謝產物的合成過程易受環境因素的影響. 本研究發現17個DEGs涉及該路徑,均呈上調趨勢。ETATCC_RS10420和ETATCC_RS10415編碼的色氨酸合酶,沙眼衣原體的色氨酸合酶有助于它在Hela細胞內的存活和對干擾素的抵抗力[11]。
本研究發現ETATCC_RS11420、ETATCC_RS14855和ETATCC_RS14860分別編碼鞭毛蛋白FliC的分子伴侶FliS、鞭毛鉤絲FlgK和FlgL。FlgM-FliA回路在協調細菌鞭毛組裝中起關鍵作用。鞭毛是細菌的運動器官,鞭毛的運動方向受到細菌趨化作用系統的控制,鞭毛對于細菌的環境適應性及致病性至關重要。在假結核耶爾森氏菌中,FliS作為分子伴侶通過調節FlgM的活性,調控后期鞭毛的表達、運動及生物膜形成(biofilm)[12]。生物膜是細菌為適應自然環境,在生長過程中附著于固體表面而形成的特殊存在形式,生物膜的形成顯著增強了細菌對環境的抵抗能力。
不同環境中的微生物代謝路徑涉及多種物質的代謝與降解過程,參與細菌物質和能量代謝網絡。本研究發現20個DEGs直接涉及該路徑,ETATCC_RS13205拷貝的酸性磷酸酶,能夠在酸性條件下水解磷酸酯鍵如酪氨酸磷酸酯酶,它可以作用于許多含酪氨酸磷酸的蛋白,這些蛋白涉及信號傳導。對于細菌來說,該酶和致病菌的毒力因子I和IV莢膜的合成有關,莢膜直接參與細菌對環境的抵抗能力[13]。
綜上所述,我們將RNA-seq技術首次用于Et菌的轉錄組研究。 Eha通過調控147個靶基因,影響Et的能量,代謝和毒力,以適應其酸性環境。因此,在酸性生存環境,Eha對Et菌的轉錄組呈多途徑、多基因的適應性的全局性調控,這些結果有助于探討 Eha調控Et抵抗巨噬細胞酸化的殺菌機制。
[1] Chen AP,Jiang YL,Qian D,et al. Edwardsiellasis[J]. China Fisheries,2011,7: 49-50. (in Chinese)
陳愛平,江育林,錢冬,等. 遲緩愛德華氏菌病 [J].中國水產,2011,7:49-50.
[2] Nelson JJ,Nelson CA,Carter JE. Extraintestinal manifestations ofEdwardsiellatardainfection: a 10-year retrospective review[J]. J La State Med Soc,2009,161 (2): 103-106.
[3] Sheng AK,Li YH,Zhang P,et al.ehagene regulates the virulence ofEdwardsiellatarda[J]. Chin J Zoonoses,2015,31(2): 125-129.DOI:10.3969/j.issn.1002-2694.2015.02.007 (in Chinese)
盛安康,李玉紅,張坡,等.eha基因調控遲緩愛德華菌的毒力[J]. 中國人獸共患病學報,2015,31(2):125-129.
[4] Gao D,Cheng J,Zheng E,et al.Eha,a transcriptional regulator of hemolytic activity ofEdwardsiellatarda[J]. FEMS Microbiol Lett,2014,353(2): 132-140. DOI:10.1111/1574-6968. 12420.
[5] Zhang L,Ni C,Xu W,et al. Intramacrophage infection reinforces the virulence ofEdwardsiellatarda[J]. J Bacteriol,2016,198(10): 1534-1542. DOI: 10.1128/JB.00978-15
[6] Xu ZY,Li YH,Cheng J,et al.Ehagene is required forEdwardsiellatardaoxidative stress resistance in macrophage[J]. Chin J Zoonoses,2015,31(6):497-500. DOI:10.3969/j.issn.1002-2694.2015.06.001(in Chinese)
徐澤炎,李玉紅,成靜,等.eha基因調控遲緩愛德華菌抵抗巨噬細胞氧化殺菌作用[J]. 中國人獸共患病學報,2015,31(6):497-500.
[7] Srinivasa Rao PS,Lim TM,Leung KY. Functional genomics approach to the identification of virulence genes involved inEdwardsiellatardapathogenesis[J]. Infect Immun,2003,71(3): 1343-1351.
[8] Stark RM,Suleiman MS,Hassan IJ,et al. Amino acid utilisation and deamination of glutamine and asparagine byHelicobacterpylori[J]. J Med Microbiol,1997,46(9): 793-800.
[9] Cooper CA,Mainprize IL,Nickerson NN. Genetic,biochemical,and structural analyses of bacterial surface polysaccharides[J]. Adv Exp Med Biol,2015,883: 295-315. DOI: 10.1007 /978-3-319-23603-2_16
[10] Remy L,Carrière M,Derré-Bobillot A,et al. TheStaphylococcusaureusOpp1 ABC transporter imports nickel and cobalt in zinc-depleted conditions and contributes to virulence[J]. Mol Microbiol,2013,87(4): 730-743. DOI: 10.1111/mmi.12126
[11] Muramatsu MK,Brothwell JA,Stein BD,et al. Beyond tryptophan synthase: identification of genes that contribute toChlamydiatrachomatissurvival during Gamma interferon-induced persistence and reactivation[J]. Infect Immun,2016,84(10): 2791-2801. DOI: 10.1128/ IAI.00356-16
[12] Xu S,Peng Z,Cui B,et al. FliS modulates FlgM activity by acting as a non-canonical chaperone to control late flagellar gene expression,motility and biofilm formation in Yersinia pseudotuberculosis[J]. Environ Microbiol,2014,16(4): 1090-1104. DOI: 10.1128 /IAI.00356-16
[13] Caselli A,Paoli P,Santi A,et al. Low molecular weight protein tyrosine phosphatase: Multifaceted functions of an evolutionarily conserved enzyme[J]. Biochim Biophys Acta,2016,1864(10): 1339-1355. DOI: 10.1016/j.bbapap.2016.07.001
Deep sequencing analysis on transcriptomes ofEdwardsiellatardaregulated by Eha following acidification
LIU Nian1,LI Yu-hong1,2,ZHENG En-jin1,GAO Da-qing1,LU Cheng-ping3
(1.SchoolofMedicine,SoutheastUniversity,Nanjing210009,China;2.WuxiMedicineSchool,JiangnanUniversity,Wuxi214122,China;3.CollegeofVeterinaryMedicine,NanjingAgriculturalUniversity,Nanjing210095,China)
Our studies tried to demonstrate Eha (Et haemolysin activator) could regulate the resistance of the bacterium against acidification to survive in the macrophage and explain its underlying molecular mechanism. When the bacteria infected the macrophages at time intervals,intracellular survival rate in bafilomycin-treated macrophages was higher than that with untreated cells,and the rate of wild type ET 13 was higher than that of itsehamutant,respectively (P<0.05). The survival rate of the wild type was higher than that of the mutant under acid treatment (P<0.05). To determine the conditions that induced the highest eha expression,we constructed a pMP220-PehaLacZ plasmid and determined thelacZ expression under different conditions. After exposure of pH6.3 medium for 2 h time,we performed the whole transcriptomic profiles of the wild type and mutant by RNA-sequencing. We identified 147 differentially-expressed genes (|log2 ratio|≥1),113 and 34 of which were significantly up-and down-regulated,respectively in the mutant,comparing with the wild type. These findings were validated by qRT-PCR. GO functional analysis revealed that these genes were divided into 25 categories,including the bacterial catalysis,cellular composition,combination,localization,metabolism,processing,and transportation. Based on the KEGG database,these genes were distributed in 55 pathways,such as two-component system,ABC transporters,and microbial metabolism in diverse environments. Overall,Eha is an important regulator to affect all kinds of target genes and pathways forE.tardato adapt to an acid environment. These results could be helpful for further investigations of the mechanisms by whichE.tardasurvives in macrophages.
ehagene; RNA-sequencing;Edwardsiellatarda; acidification
Gao Da-qing,Email: dgao2@seu.edu.cn
10.3969/j.issn.1002-2694.2017.07.001
國家自然科學基金(No.31570124)資助
高大慶:Email:dgao2@seu.edu.cn
1.東南大學醫學院,南京 210009; 2.江南大學無錫醫學院,無錫 214122; 3.南京農業大學動物醫學院,南京 21009
R378.2
A
1002-2694(2017)07-0575-08
2016-10-01 編輯:張智芳
Supported by the National Natural Science Foundation (No. 31570124)

