西藏不同地區黑木耳多樣性分析
施樹,蔣厚陽,羅章
(1.重慶旅游職業學院,重慶409000;2.西南大學食品科學學院,重慶北碚400700;3.西藏大學農牧學院,西藏林芝860000)
西藏不同地區黑木耳多樣性分析
施樹1,蔣厚陽2,羅章3*
(1.重慶旅游職業學院,重慶409000;2.西南大學食品科學學院,重慶北碚400700;3.西藏大學農牧學院,西藏林芝860000)
運用酯酶同工酶酶譜分析和ISSR分子標記技術對3株西藏野生黑木耳親緣性和遺傳多樣性進行分析。運用丙烯酰胺蛋白電泳技術對野生黑木耳中酯酶同工酶進行電泳;運用PCR-ISSR分子標記技術對野生黑木耳DNA進行隨機擴增,擴增產物進行瓊脂糖電泳。用Ntsys 2.10e和PopGen32軟件對電泳結果進行聚類和遺傳分析。結果表明:3株野生黑木耳中察隅和魯郎地區樣品的親緣性關系較近,樣品間遺傳分化指數高。結論:本實驗為西藏地區野生黑木耳品種分類、資源保護和新品種培育提供了初步依據。
西藏;野生黑木耳;酯酶同工酶;ISSR
西藏位于我國西南邊陲,平均海拔在4000m以上,是青藏高原的主體部分。空氣稀薄、氣壓低、含氧量少、輻射強、日照時間上、日溫差大、全年分為明顯的干季和雨季。這些氣候條件賦予了西藏地區獨特的作物資源。黑木耳在真菌學上的分類屬擔子菌綱,木耳目,木耳科[1],是人們餐桌上的常見食物,具有豐富的營養。目前還沒有人對西藏地區野生黑木耳在基因型上進行研究,運用分子標記技術對物種親緣關系和遺傳多樣性進行分析,可以為雜交育種培養新品種提供依據,從而培育出高營養的優勢黑木耳菌株。
同工酶電泳技術在生物進化研究中得到廣泛地應用,在分子發生、進化、分析標記和遺傳上的研究也越來越重要,主要應用于物種鑒定、酶譜鑒定、遺傳育種分子標記、群體差異統計等[2-3]。ISSR標記技術由Zietkiewicz等創建,是一種建立在PCR反應基礎上的DNA分子標記[4]。廣泛地應用于物種鑒定[5]、基因定位[6]、遺傳作圖[7]、遺傳多樣性[8]等研究。該技術不需要知道檢測物質的基因組序列,操作簡單,成本低,靈敏度高[9]。
本研究將酯酶同工酶酶譜分析和ISSR分子標記技術應用于西藏野生黑木耳的基因型研究中,通過丙烯酰胺蛋白電泳對酯酶同工酶酶譜分析,從4種隨機引物種選擇最佳引物,對野生黑木耳DNA進行隨機擴增,通過瓊脂糖電泳進行檢測。用Ntsys 2.10e,PopGen32軟件對電泳圖譜進行分析,以探討西藏不同地區黑木耳之間的親緣關系,為今后建立野生黑木耳資源庫,培育新品種打下基礎,同時也探討酯酶同工酶酶譜分析和ISSR分子標記技術在黑木耳的分類鑒定方面的應用價值。
1 材料與方法
1.1 材料與試劑
1.1.1 供試菌株。供試野生黑木耳分別采自西藏亞東、魯朗和察隅3個地區,菌株的編號為A-C。
1.1.2 試劑。磷酸氫二鈉、磷酸二氫鈉、蔗糖、溴酚藍 重慶滴水化學藥品責任有限公司;Acrylamide、Bis-acrylamide、過硫酸銨、Glycine TEMED、乙酸-1-萘酯、乙酸-2-萘酯、固蘭RR鹽 上海生工;100bp DNA ladder、Taq酶(5U/uL)、dNTPs(10mmol/L)、10×酶緩沖液、MgCl2(25mM) 寶生物有限公司;1kb DNA ladder、EDTA Na2、Tris-base、Guanidine Thiocyanate、DEPC加拿大BBI;瓊脂糖 西班牙Agarose; Gelred核酸染料 美國BIOTIUM。引物合成 上海生工。
1.2 儀器與設備
HB031金屬恒溫加熱器 上海博彩生物技術有限公司;SIM-F140AY65型制冰機 三洋國際貿易有限公司;1-15PK小型臺式離心機 德國sigma實驗室離心機公司;Biospec-mini型島津DNA/RNA/蛋白質分析裝置,島津制作所;S1000伯樂 1000系列高性能PCR儀,美國BIO-RAD;164-5050伯樂基礎電源電泳儀,美國BIO-RAD;Syngene GeneGenius 凝膠成像系統,基因有限公司;GL-88B渦旋混合器,海門市其林貝爾儀器制造有限公司; SW-CJ-1F型單人雙面凈化工作臺。
1.3 方法
1.3.1 野生黑木耳酯酶同工酶的提取及電泳。黑木耳酯酶同工酶的提取、電泳和染色參照NY/T 1097-2006[10]中的方法。
1.3.2 野生黑木耳基因組DNA的提取。將干的黑木耳用液氮進行充分研磨,然后參照NY/T 1730-2009[11]中的方法提取樣品中的DNA。提取的DNA保存于-20℃冰箱中。測定提取DNA的OD260,OD280及OD260/OD280數值,利用公式c= OD260×50(μg/mL) ×n(稀釋倍數)[12]計算出DNA濃度,為后續實驗提供數據。
1.3.3 ISSR隨機引物的篩選和反應條件的建立。隨機引物的篩選:通過對4條不同隨機擴增引物(表1)進行PCR反應,根據條帶的多態性和亮度選擇出最適引物,進行后續實驗。

表1 隨機引物編號及序列
根據參考文獻[13]設定PCR反應程序為:94℃預變性5min,94℃變性30s;相應褪火溫度(表1)1:00min;72℃延伸1:30min;35個循環,然后72℃延伸10min。PCR反應體系為:50ng模板DNA,10×Taq酶緩沖液(無Mg2+)2.5μL,2.5 mM MgCl22.5μL,10mmol/L dNTPs 0.5μL,10μmol/L引物2μL,5U/μL Taq 酶0.2μL。
1.3.4 ISSR擴增產物檢測。擴增產物經1.2%瓊脂糖凝膠電泳分離。取10μL擴增產物與2μloading buffer混勻,點樣于凝膠孔,用1kb DNA ladder作為分子量標尺,電泳緩沖液為1×TAE,70V恒壓電泳60min,gelred染色后通過凝膠呈像系統拍照。
1.3.5 酯酶同工酶酶譜和ISSR譜帶分析
酯酶同工酶酶譜分析:酯酶同工酶相對遷移率(Rf)[14]:Rf=X2/X1,在式中X1為固定染色前凝膠中指示劑遷移距離,X2為固定染色后凝膠中酶蛋白區帶的遷移距離。根據相對遷移率的結果,特定遷移率上無條帶賦值為0,有條帶賦值為1,用NTsys2.10e制作為“0,1”文件,采用UPGMA(Unweighted Pair Group Method with Arithmetic Mean)法進行聚類分析,并構建各菌株的遺傳距離聚類圖。采用PopGen32軟件分析樣品的遺傳多樣性及香農指數等數值。
ISSR分子標記分析:各菌株的電泳圖譜上特定位點無條帶的賦值為0,有條帶的賦值為1,用NTsys2.10e制作為“0,1”文件,采用UPGMA(Unweighted Pair Group Method with Arithmetic Mean)法進行聚類分析,并構建各菌株的遺傳距離聚類圖。采用PopGen32軟件分析樣品的遺傳多樣性及香農指數等數值。
2 結果與分析
2.1 酯酶同工酶電泳圖
3株西藏野生黑木耳經過PAGE丙烯酰胺電泳后,所有菌株均擴增出明顯的條帶,個菌株間存在明顯的差異性,擴增出的條帶的1~4條,Rf值0.4~0.8,其中察隅和魯郎2個樣品的相似條帶較多,亞東與察隅和魯郎沒有相似條帶。圖1顯示了三株野生黑木耳的電泳圖。
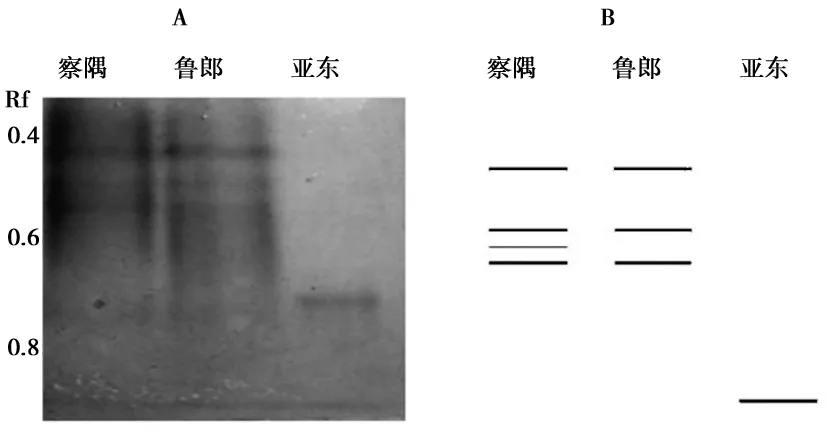
注:A為電泳圖,B為模式圖
圖1 西藏野生黑木耳酯酶同工酶電泳圖
2.2 ISSR分子標記電泳圖
3株西藏野生黑木耳菌株用P4引物擴增后,所有菌株均擴增出明顯的條帶,各個菌株之間也存在特異性,擴增出的條帶數目在3~4條,所獲得的片段分子量大小在250~3000,圖2顯示了3株野生黑木耳擴增后的電泳圖。

注:marker為寶生物有限公司100bp DNA ladder
圖2 西藏野生黑木耳ISSR擴增圖譜
2.3 聚類分析
用NTsys2.10e軟件,對所得擴增圖譜的條帶進行數據統計處理,分別得出3株野生黑木耳酯酶同工酶和ISSR分子標記后的遺傳相似系數矩陣,結果見表2。通過酯酶同工酶分析得到3株野生黑木耳遺傳相似系數0.20~0.80,其中魯郎和察隅之間的相似性系數最大,說明它們之間的親緣性最近。而亞東與魯郎、察隅之間的相似性較小,說明它們之間的親緣性關系較遠。 通過ISSR分子標記得到的結果與酯酶同工酶分析得到的結果相近。3株野生黑木耳遺傳相似系數0.57~0.86,魯郎與察隅之間的相似性系數較大,亞東與魯郎和察隅之間的差異較大。
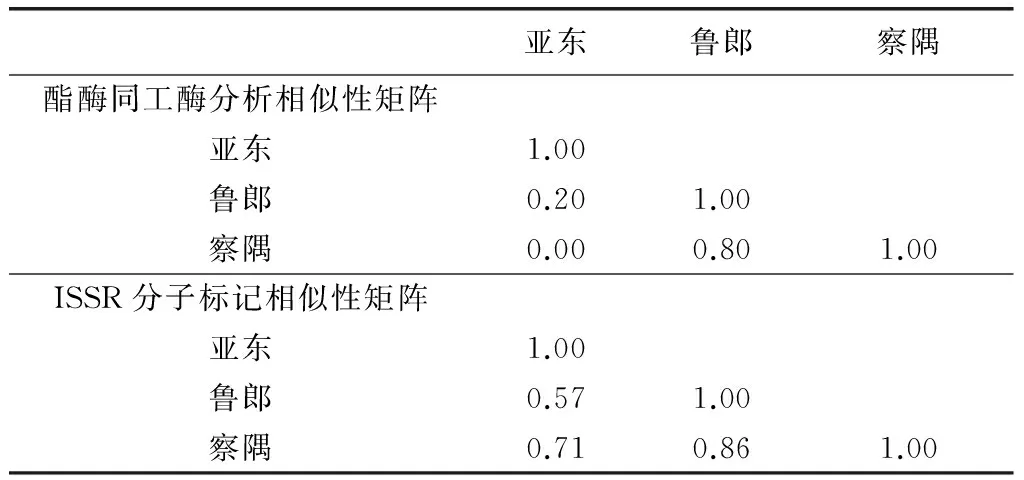
表2 供試菌株間的遺傳相似系數矩陣

圖3 西藏野生黑木耳酯酶同工酶分析親緣關系聚類樹狀圖
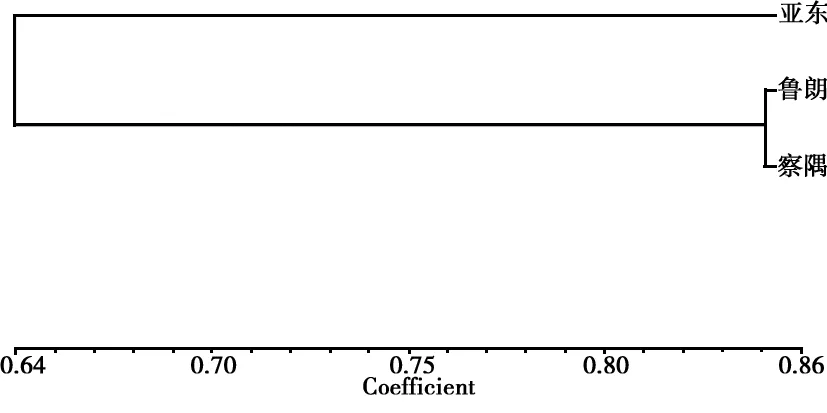
圖4 西藏野生黑木耳ISSR分析親緣關系聚類樹狀圖
用NTsys2.10e軟件,按照UPMGA法,對供試菌株之間的遺傳相似系數矩陣進行聚類分析,得到聚類分析圖3和圖4。由圖3可知,當相似性系數為0.1時,3株野生黑木耳被分為了2組。第一組為亞東地區樣品,第二組包括察隅和魯郎地區樣品。由圖4可知,當相似性系數為0.64時,3株野生黑木耳被分為了2組,分組的結果同酯酶同工酶分析結果相同。
2.4 遺傳多樣性分析
采用PopGen32軟件分析供試野生黑木耳的遺傳多樣性和香濃指數等數值。由表3分析可以看出,酯酶同工酶酶譜分析和ISSR分子標記分析所得到的遺傳多樣性指數和香濃指數都較高,說明西藏野生黑木耳具有很高的遺傳分化價值,可以通過雜交育種培育黑木耳新品種。同時也可以看到樣品間的多態性位點百分率同樣具有很高的數值,這些都表明西藏野生黑木耳間有很高的遺傳分化潛力。

表3 西藏野生黑木耳遺傳多樣性的統計
3 討論
西藏地區具有由于其獨特的氣候和地理條件,具有獨特的作物資源。黑木耳做為日常飲食中的常見食物,具有很高的研究價值,對其親緣性和遺傳性進行分析,對當地培育黑木耳新品種具有很大的借鑒作用。
目前還沒有對于西藏地區野生黑木耳進行分子水平方面研究的報道,所以將酯酶同工酶酶譜分析和ISSR分子標記技術應用于西藏野生黑木耳親緣性和遺傳性分析具有非常重要的意義。國內外很多學者已經將酯酶同工酶酶譜分析和ISSR分子標記技術應用于食用菌分析中。Wang等[15]人運用RAPD和ISSR技術對中國的杏鮑菇基因多樣性進行研究。Du等[16]人運用ISSR技術對中國野生毛木耳菌群基因多樣性進行了研究。Zhang等[17]人研究了培養時間對于黑木耳酯酶同工酶酶譜的影響。趙勇[18]等人將酯酶同工酶和RAPD技術應用于香菇雜交優勢研究中。
本研究探討了酯酶同工酶酶譜分析和ISSR分子標記技術在西藏野生黑木耳親緣性和遺傳性中的應用。用NTsys 2.10e軟件計算出了3株野生黑木耳之間的遺傳相似系數矩陣,并用UPMGA法作聚類分析圖,用PopGen32軟件計算出了3株野生黑木耳之間的遺傳多樣性數據。綜合酯酶同工酶和ISSR分子標記分析,察隅和魯郎2個地區的樣品親緣性關系較近,3個地區的樣品具有很高的遺傳分化指數,為培養新型黑木耳雜交品種提供了理論基礎。
[1]張光亞. 中國常見食用菌圖譜[M]. 北京:農業出版社,1999:19-20.
[2]Mohammadi M, Banishemi M, Hedjaroude G A, et al.Genetic Diversity Among Iranian Isolates of Rhizoctonia solani Kuhu Anastomosis Groupl Subgroups Based on Isozyme Analysis and Total Soluble Protein Pattern[J].Journal of Phytopathology-phytopathologische Zeitschrift, 2003, 151(3):162-170.
[3]胡能書,萬賢國.同工酶技術及其應用[M].長沙:湖南科學技術出版社,1985.
[4]Zietkiewicz E, Rafalski A, Labuda D. Genome fingerprinting by simple sequence repeat(SSR)-anchored polymerase chain-reaction amplification[J]. Genomics, 1994, 20(2):176-183.
[5]Kumar L D, Kathirvel M, Rao G V, et al. DNA profiling of disputed chilli samples(Capsicum annum) using ISSR-PCR and FISSR-PCR marker assays[J]. Forensic Science International, 2001, 116(1):63-68.
[6]Hussain A J, Gupta V, Ali J, et al. Physiological characterization, genetics and molecular mapping of a new source of temperature sensitive genentic male sterility in rice[G]. IRRI. Fourth International Rice Genetics Symposium. Philippines: IRRI, 2000:22-27.
[7]Levi A, Thomas C, Joobeur T, et al. A genetic linkage map for watermelon derived from a testcross population:(Citrullus lanatus var.citroides×C-lanatus var.lanatus) ×Citrullus colocynthis[J]. Theoretical and Applied Genetics, 2002, 105(4):555-563.
[8]聶瓊,劉仁祥. 23份煙草種子遺傳多樣性的SSR和ISSR標記分析[J].西南農業學報,2011,24(1):15-19.
[9]秦蓮花,宋春艷,譚琦,等. 用ITS和ISSR分子標記技術鑒別香菇生產用種[J]. 菌物學報,2006,25(1):94-100.
[10]中華人民共和國農業部. NY/T 1097-2006 食用菌菌種真實性鑒定酯酶同工酶電泳法[S]. 北京:中國標準出版社,2006.
[11]中華人民共和國農業部. NY/T 1730-2009 食用菌菌種真實性鑒定 ISSR法[S]. 北京:中國標準出版社,2009.
[12]林萬明. 細菌分子遺傳學分類鑒定法[M]. 上海: 上海科學技術出版社, 1990: 48-83.
[13]唐利華,肖揚,邊銀丙. 中國黑木耳主要栽培菌株ISSR指紋分析及SCAR標記[J]. 菌物學報,2008, 27(2):243-251.
[14]韓增華,高娃,張丕齊,等. 東北黑木耳栽培菌株的酯酶同工酶分析[J]. 黑龍江科學,2011,2(6):4-9.
[15]Wang S X, Yin Y G, Liu Y, et al. Evaluation of Genetic Diversity Among Chinese Pleurotus eryngii Cultivars by Combined RAPD/ISSR Marker[J]. Current Microbiology, 2012, 65(4):424-431.
[16]Du P, Cui B K, Dai Y C. Assessment of genetic diversity among wild Auricularia polytricha populations in China using ISSR markers[J]. Cryptogamie Mycologie, 2012, 33(2):191-201.
[17]Zhang P Q, Han Z H, Dai X D, et al. Effect of culture time on the zymogram of esterase isozyme of Auricularia auricula[J]. Journal of Fungal Research, 2008, 6(2):106-109.
[18]趙勇,賀東梅,溫亞麗,等. 酯酶同工酶及RAPD技術在香菇雜種優勢研究中的應用[J]. 菌物系統,2003,22(4):549-556.
Analysis of Genetic Diversity and Relationship of Black Fungus from Different Regions of Tibet
Shi Shu1,Jiang Houyang2,Luo Zhang3*
(1. Chongqing Vocational Institute of Tourism,Qianjiang, 409000;2. Southwest University,Beibei, Chongqing 400700;3. Agriculture and Animal Husbandry College of Tibet University,Linzhi, Tibet 860000)
Analysis genetic diversity and relationship of black fungus from different regions of Tibet by method of esterase isozyme enzyme spectrum and ISSR molecular markers. Using acrylamide protein electrophoresis technology on esterase isozyme of wild black fungus for electrophoresis; take a random amplificatioon of wild fungus by PCR-ISSR molecular marker techonlogy. Conduct product electrophoresis in agarose gel. Clustering and inheritance were analyzed by Ntsys 2.10 and PopGen32 software. Result shows: Samples of Lulang and Chayu have a closer relationship among wild fungus, there is a high genetic differentiation index among samples. This study provides a preliminary basis for species classification and hybrid of wild fungus of Tibet.
Tibet; Black fungus; Esterase isozyme; ISSR
2017-03-07
西藏自治區自然基金資助項目
施樹(1978-),男,講師,主要從事食品化學與營養學研究,E-mail:shishu1978@163.com;*通訊作者:羅章(1965-),男,教授,從事農產品加工與貯藏研究。
S646.6
A
DOI.:10.13268/j.cnki.fbsic.2017.03.004

