基于三種分子標記的外伶仃島石珊瑚系統進化關系研究
肖寶華, 廖寶林, 楊小東, 謝子強
?
基于三種分子標記的外伶仃島石珊瑚系統進化關系研究
肖寶華1, 2, 廖寶林2, 楊小東2, 謝子強3
(1. 廣東海洋大學, 廣東 湛江 524025; 2. 廣東海洋大學 深圳研究院, 廣東 深圳 518108; 3. 深圳市碧海藍天海洋科技有限公司, 廣東 深圳 518108)
以廣東省珠海市外伶仃島地區常見的4屬5種石珊瑚為研究對象, 對線粒體COI、16S rRNA和ITS三種基因片段數據進行分析, 并計算了種間遺傳距離。序列分析結果表明5種石珊瑚的COI、16S rRNA基因片段堿基AT含量大于GC含量, 而ITS序列堿基AT含量小于GC含量; 5種石珊瑚種間的三種基因平均遺傳距離分別為0.043、0.134和0.763, 所得遺傳距離結果存在一定差異。將三種基因串聯起來并利用鄰位連接法(NJ)、最大似然法(ML)和最小進化法(ME)分別構建了分子系統樹。在系統發育樹中, 科一級的系統進化關系與形態學分類一致, 但蜂巢珊瑚科內的分子系統分類結果與形態學分類闡述的遺傳進化關系略有差別, 說明石珊瑚傳統分類可能受骨骼可塑性的影響。研究結果為外伶仃島石珊瑚的親緣關系研究提供相關基礎數據, 并為該地區石珊瑚資源保護奠定理論基礎。
石珊瑚; COI; 16S rRNA; ITS; 分子系統進化
石珊瑚分子系統進化研究始于20世紀90年代, 1996年Romano等[1]在世界上首次使用分子標記(線粒體16S rRNA和細胞核28S rRNA)對石珊瑚系統進化關系進行研究, 研究結果表明現有石珊瑚目起源于兩個分支類群(堅實型類群和復合型類群)。目前, 應用于石珊瑚系統進化的分子標記主要有核糖體18S rRNA、5.8S rRNA、內轉錄間隔(Internal transcribed spacers, ITS)和線粒體的12S rRNA基因、16S rRNA基因和細胞色素b(cytochromeb, cyt b)基因、細胞色素c氧化酶亞基I基因(COI)等[2]。經過近20年的研究發展, 石珊瑚的分子系統進化研究已經對石珊瑚較高階元的系統分類學產生了極大的影響, 顛覆了以往通過形態學對石珊瑚系統進化的認識[3]。
鹿角珊瑚科的石珊瑚品種是目前國內外利用分子標記研究系統進化關系最多一類珊瑚。Odorico等[4]利用ITS基因對5種鹿角珊瑚科品種的系統關系進行了初步的探討, 為鹿角珊瑚科分子系統研究打下了堅實的理論基礎。Fukami等[5]利用線粒體的細胞色素b和ATP6兩種基因片段對鹿角珊瑚科4個屬15個品種進行系統進化分析, 分子系統進化樹顯示15種珊瑚可分為三個大的珊瑚類群。Forsman等[6]利用16S rRNA作為分子標記對鹿角珊瑚科的薔薇珊瑚屬()多種薔薇珊瑚的種間遺傳關系進行分析, 構建了薔薇珊瑚屬的系統進化樹。蜂巢珊瑚科的石珊瑚作為石珊瑚種類第二豐富的種類, 使用分子標記進行系統進化研究也在國內外積累了大量的報道。Huang等[7]基于線粒體COI和一個非編碼區序列對新加坡附近海域的41種蜂巢珊瑚科珊瑚系統進化關系進行研究, Arrigoni等[8]采用COI作為分子標記對印度洋海域的蜂巢珊瑚科的珊瑚品種進行系統進化分析。這些學者都發現蜂巢珊瑚科的分子系統進化結果與形態學系統進化結果存在一定的差異, 再次證明石珊瑚中普遍存在并系現象。相比較國外對石珊瑚分子系統進化的報道, 國內有關石珊瑚分子系統進化的研究報道仍較少, 僅有海南[9-10]、廣東徐聞[11-13]等少數地方的石珊瑚系統進化有過相關的報道。
外伶仃島位于萬山群島中部東北端, 珠江口東側。目前, 有關外伶仃島附近海域石珊瑚的研究知之甚少, 僅有黃暉等[15]在2008年開展萬山群島的石珊瑚群落分布研究時對外伶仃島海域石珊瑚情況進行了初步的調查, 調查結果顯示多孔同星珊瑚()和腐蝕刺柄珊瑚()為外伶仃島的優勢種。其他有關外伶仃島石珊瑚的研究還未見報道。
本研究采用線粒體COI、16S rRNA基因以及核糖體ITS基因的部分序列片段作為分子標記, 對外伶仃島海域的5種石珊瑚的系統進化關系進行研究, 為外伶仃島石珊瑚的親緣關系研究提供相關基礎數據, 并為該地區石珊瑚資源保護奠定理論基礎。
1 材料與方法
1.1 樣品采集和形態學鑒定
本研究的石珊瑚樣品采自廣東省珠海市外伶仃島附近海域, 形態學鑒定依照鄒仁林[16]的《中國動物志·腔腸動物門·珊瑚蟲綱·石珊瑚目·石珊瑚》和Veron[17]的六射珊瑚骨骼形態特征分類系統。經形態學鑒定采集的樣品可分為3科4屬5種, 樣品形態學鑒定結果見表1。截取一小段斷枝保存于無水乙醇中以備后續DNA提取實驗使用。
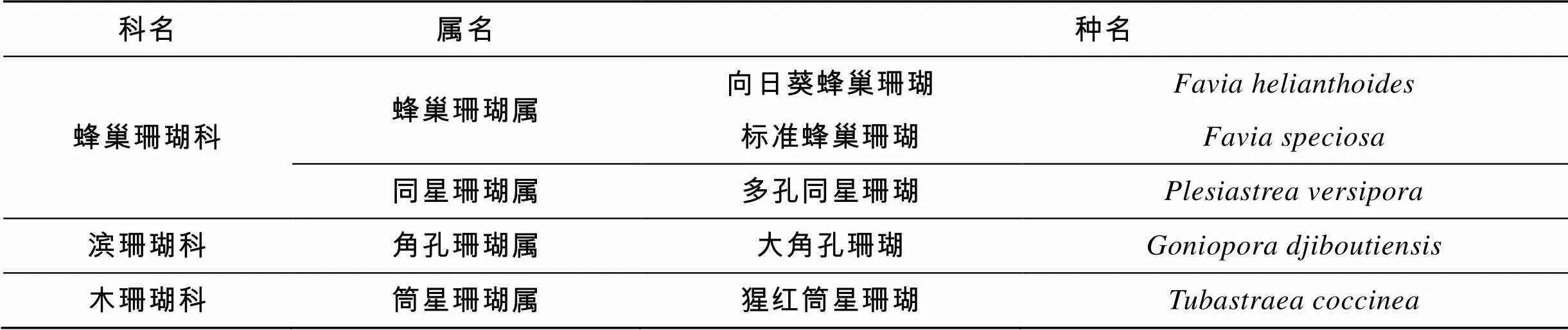
表1 珊瑚樣品
1.2 基因組總DNA的提取
使用北京天根生物有限公司生產的海洋動物組織基因組提取試劑盒提取珊瑚樣品的DNA, 所提取的DNA置于4℃保存備用。
1.3 引物設計合成
COI基因的PCR引物參考李曉娜等[11]的研究報道, 16S rRNA基因的PCR引物參考Forsman等[6]的研究報道, ITS基因的PCR引物參考Odorico等[4]的研究報道。引物由上海生工生物技術服務有限公司合成, 引物信息見表2。
1.4 PCR擴增和DNA測序
PCR反應體系反應總體積25 μL: 10×Buffer 2.5 μL, dNTP 2 μL, 上下游引物各1 μL, , Taq DNA聚合酶1U及DNA模板2 μL; 其余用去離子水補至25 μL。PCR反應條件: 95℃預變性5min, 95℃變性45 , 50℃退火45 s, 72℃延伸1 min 30 s, 共35個循環, 最后72℃延伸10 min。擴增產物在1.0%的瓊脂糖凝膠中檢測, 凝膠成像儀拍照記錄。選取擴增條帶單一、濃度較高的PCR產物送至上海生工生物技術公司進行測序, 獲取各基因片段的序列信息。
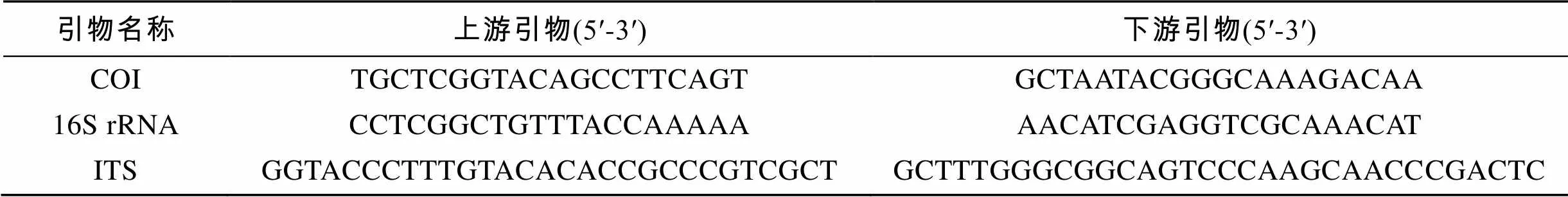
表2 COI、16S rRNA、ITS基因的PCR引物
1.5 序列分析及系統進化樹的構建
測序所得序列用DNASTAR中的Editseq軟件進行編輯、剪切、校正, 再將校正好的序列與NCBI的數據庫進行比對, 確認樣品的分子結果。采用MEGA6.0軟件進行三種基因序列的串聯與分析, 統計出三種線粒體基因組序列長度、堿基組成、簡約信息位點等相關信息。同時根據簡約信息位點, 計算出種間遺傳距離。本研究三種基因的系統進化樹是使用MEGA6.0軟件基于Kimu-ra雙參數模型(Kimura 2-parameter, K2P), 只計算顛換(Transversional substitutions only), 采用鄰位連接法(Neighbor-Joinning, NJ)、最大似然法(Minimum-Likelihood, ML)和最小進化法(Minimum-Evolution, ME)構建出來的。三種系統進化樹中節點的自舉置信度水平由自引導值(Bootstrapvalue) 估計, 重復次數為1 000次。
2 結果與分析
2.1 三種基因序列及堿基組成分析
本研究的5個物種的COI、16S rRNA基因以及ITS基因部分序列長度分別為464~465bp, 470~485bp, 964~988bp。從5種石珊瑚的COI、16S rRNA基因以及ITS基因部分序列的堿基組成比例來看, 兩種線粒體基因(COI、16S rRNA)片段堿基中的A、T、C、G含量相差較大, 序列堿基AT含量大于GC含量, 與目前已知的無脊椎動物mtDNA序列中AT含量大于GC含量的情況相同。而ITS基因序列片段堿基中的A、T、C、G含量相差不大, 除大角孔珊瑚外(), 其余物種的堿基AT含量小于GC含量。三種片段序列基因都處于高度飽和狀態。堿基組成比例見表3。
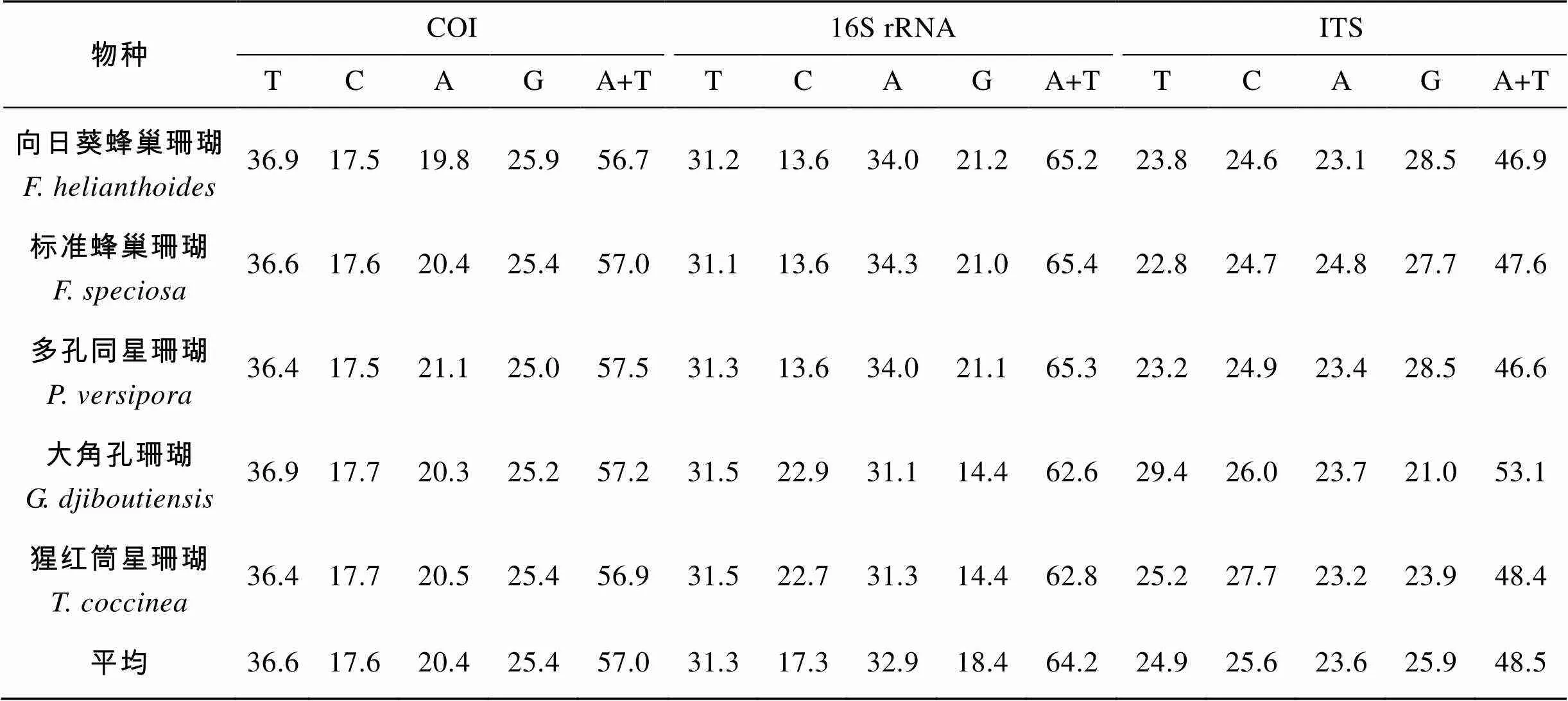
表3 5種石珊瑚COI、16S rRNA以及ITS基因片段的堿基組成比例
2.2 5種石珊瑚種間遺傳距離
通過MEGA6.0計算5種石珊瑚的種間遺傳距離, 得到COI、16S rRNA基因以及ITS序列間的平均距離為0.043、0.134和0.763。在COI序列中, 標準蜂巢珊瑚和大角孔珊瑚的種間遺傳距離最大為0.093, 而標準蜂巢珊瑚和猩紅筒星珊瑚的遺傳距離最小, 為0.004。在16S rRNA基因序列中, 猩紅筒星珊瑚和多孔同星珊瑚以及向日葵蜂巢珊瑚的種間遺傳距離最大, 都為0.224, 而標準蜂巢珊瑚和多孔同星珊瑚的遺傳距離最小, 為0.002。在ITS序列中, 大角孔珊瑚和多孔同星珊瑚的種間遺傳距離最大, 為1.224, 而多孔同星珊瑚和向日葵蜂巢珊瑚的遺傳距離最小, 為0.028。三種序列得出的種間遺傳距離結果存在一定差異, 種間遺傳距離見表4、表5和表6。
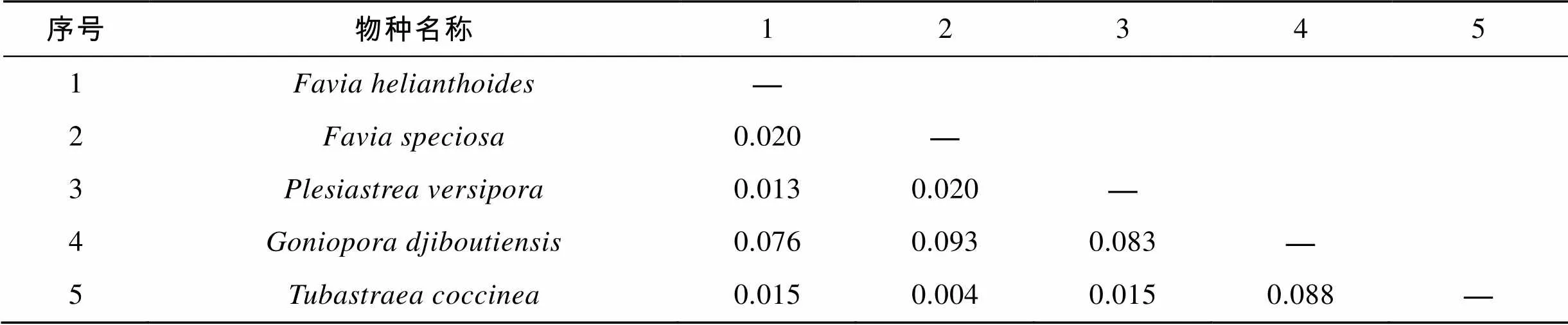
表4 基于COI的5種石珊瑚種間遺傳距離
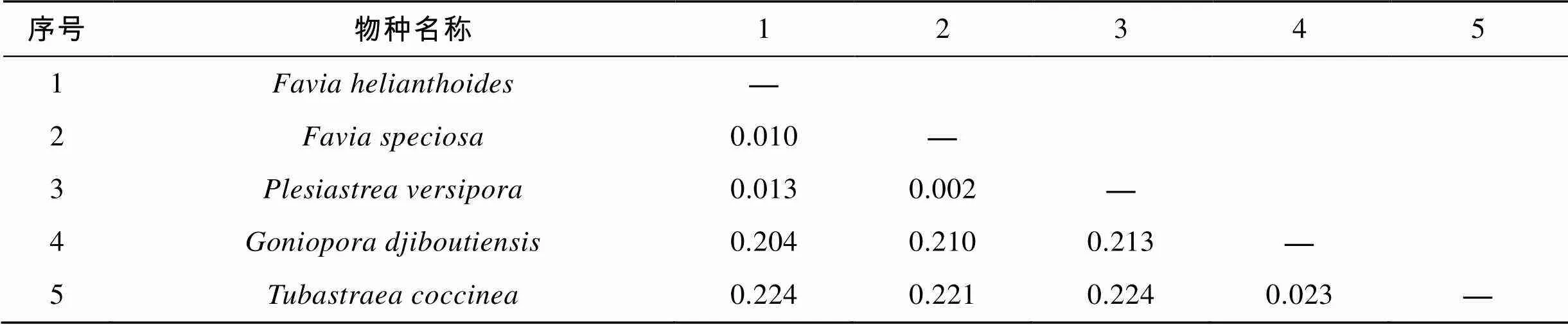
表5 基于16S rRNA基因的5種石珊瑚種間遺傳距離
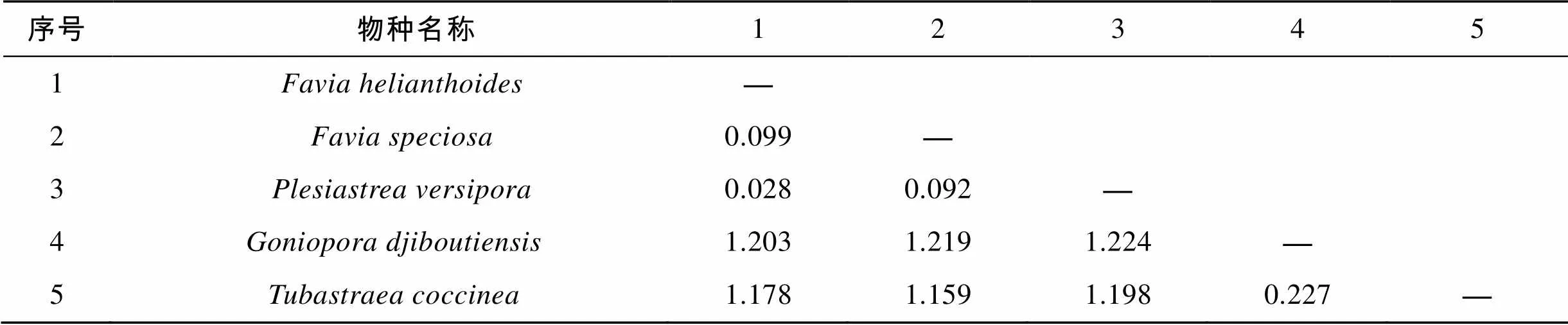
表6 基于ITS的5種石珊瑚種間遺傳距離
2.3 基于三種基因序列對5種石珊瑚的系統進化分析
使用MEGA6.0軟件將每種石珊瑚的COI、16S rRNA基因以及ITS三種基因片段序列串聯起來, 再采用鄰位連接法(Neighbor-Joinning, NJ)、最大似然法(Minimum-Likelihood, ML)和最小進化法(Minimum-Evolution, ME)構建5種石珊瑚的分子系統樹, 結果顯示3種方法構建的系統進化樹拓撲結構完全一致, 蜂巢珊瑚科、濱珊瑚科和木珊瑚科的珊瑚品種各聚為一枝, 但在蜂巢珊瑚科中, 向日葵蜂巢珊瑚與多孔同星珊瑚先聚為一支然后再與標準蜂巢珊瑚再聚為一支, 這與傳統形態學科屬分類結果存在一定差異。三種基因構建的系統進化樹見圖1、圖2和圖3。

圖1 利用三種基因序列數據構建的分子系統進化NJ樹

圖2 利用三種基因序列數據構建的分子系統進化ML樹

圖3 利用三種基因序列數據構建的分子系統進化ME樹
3 討論
3.1 5種石珊瑚COI、16S rRNA基因和ITS基因序列特點
線粒體細胞色素c氧化酶亞基I基因(COI)是動物線粒體DNA的重鏈上參與細胞色素氧化反應的關鍵基因。COI基因擁有長度適宜、進化速率慢及富含系統發育信號等特點, 常被當作動物界物種鑒定的DNA條形碼工具[18]。近年來, 很多研究表明在許多低等動物的分子系統進化研究中COI基因具有更為緩慢的進化速率, 特別是石珊瑚。造成這一現象的原因可能是由于石珊瑚采用無性生殖方式使珊瑚蟲個體可以存活上百年[19]。Hellberg[19]在使用COI基因對橙杯珊瑚()和猩紅筒星珊瑚系統進化的研究中發現其較慢的進化速率導致其較低的種內分化。然而在石珊瑚種間關系的研究中, COI基因作為主要的分子標記得到了廣泛的應用[20-22]。本研究的5種石珊瑚COI序列長度變化不大, 遵循真核生物線粒體基因AT偏向性的規律, 是本研究三種基因中種間序列變化程度最小的基因。
線粒體16S rRNA基因相對于其他石珊瑚的線粒體基因進化速率較慢, 且較為保守, 常用于種以上分子分類的標記[23]。本研究的5種石珊瑚16S rRNA基因序列長度變化較大, 遵循真核生物線粒體基因AT偏向性的規律。國外學者研究發現, 16S rRNA基因在石珊瑚屬以上的分類階元中種間片段長度具有較大的差異, 而屬內差異片段長度差異不大[23-24]。本研究的結果也較為符合這種情況, 兩種復合型珊瑚(大角孔珊瑚和猩紅筒星珊瑚)16S rRNA基因具有較為相似的堿基組成, 而與3種蜂巢珊瑚科珊瑚的16S rRNA基因序列堿基組成則相差較大。從序列的堿基組成、序列變化和遺傳距離上看, 16S rRNA基因比較適宜作為科、屬之間的分子分類標記。
內轉錄間隔序列(ITS)是細胞核中核糖體18S r RNA和5.8S r RNA之間的一段非轉錄區序列, 由于所受的選擇壓力較小、DNA堿基替換速率較快, 加上較大的拷貝數、適中的長度和快速的同步化等特點, 常被用于低等真核生物屬水平的種間關系研究[7]。而在石珊瑚目的鹿角珊瑚、扁腦珊瑚、沙珊瑚、濱珊瑚等類群研究中, 國內外都有相關ITS標記的報道[3, 25]。本研究的5種石珊瑚ITS序列長度變化是本研究三種基因中變化最大的基因。序列堿基組成情況上與16S rRNA基因結果較為一致, 同時由于其較高的堿基變化率, 對蜂巢珊瑚科內不同種石珊瑚遺傳分化情況是三種基因中最為顯著一種。從序列的堿基組成、序列變化和遺傳距離上看, ITS基因比較適宜作為屬間的分子分類標記。
3.2 石珊瑚系統進化分析
早在20世紀90年代, Romano和Palumbi[1]在應用16S rRNA基因對34種石珊瑚的系統進化關系進行研究的過程中, 發現全球范圍內現存的石珊瑚可劃分為兩大分支類群, 并將其命名為堅實型(Robust)和復合型(Complex)。結合已有的形態學研究, Romano認為堅實型珊瑚骨骼鈣化程度較高, 具有堅實的骨骼結構, 珊瑚外觀主要以碟片狀和團塊狀為主, 其代表種類包括蜂巢珊瑚科、石芝珊瑚科、褶葉珊瑚科等。復合型珊瑚骨骼鈣化程度較低, 具有較為復雜多變的生長形態, 其代表種類包括鹿角珊瑚科、菌珊瑚科、木珊瑚科、濱珊瑚科等。經過近20年的石珊瑚分子系統學研究發展, 國內外的許多學者積累的分子數據都極力地支持了這一學說。Fukami等[26]使用多種分子標記對17科75屬127種石珊瑚進行了系統進化分析, 通過構建的系統進化樹將127種石珊瑚細分成21個類群, 其中9個類群屬于復合型珊瑚, 12個類群屬于堅實型珊瑚。Kitahara等[22]對現有324種石珊瑚COI基因的系統進化關系進行了探討, 引入外源種進行分析后, 將堅實型分支的位置放在了復合型分支輻射范圍之內。相對來說, 復合型珊瑚類群的分子系統進化關系與傳統分類學的系統關系較為一致。而堅實型珊瑚類群的分子系統進化關系則與傳統分類學存在很多不一致的地方, 部分科屬之間關系較為混亂。其中最為混亂的第17個分類群被稱為“Bigmessidae”, 里面包含了4個石珊瑚科: 蜂巢珊瑚科、裸肋珊瑚科、梳狀珊瑚科、腦珊瑚科。這個類群在分子系統進化關系上存在相互并系現象, 不同科屬之間的物種重新聚為新的分支[27]。蜂巢珊瑚科作為堅實型珊瑚的代表, 同時也是最為多源的系統進化種類, 在不同的分子系統進化研究中都分散在7種堅實型珊瑚分類群中, 在系統進化樹上與裸肋珊瑚科、褶葉珊瑚科、梳狀珊瑚科、鐵星珊瑚科等珊瑚種類交雜在一起, 成為并系情況最為復雜的一類珊瑚。這些分子證據與傳統形態學系統進化存在許多不協調和不統一的地方, 暗示著傳統形態學可能受珊瑚骨骼生長變異性限制, 對石珊瑚的系統進化關系分析中存在錯誤, 尤其是堅實型珊瑚類型。
本研究采用鄰位連接法(NJ)、最大似然法(ML)和最小進化法(ME)對5種石珊瑚三種基因片段的串聯序列構建了系統發育樹, 從科一級單元來看, 串聯基因構建的系統進化關系符合Romano的兩大分支類群學說。蜂巢珊瑚科的珊瑚品種屬于堅實型珊瑚類群分支、而濱珊瑚科和木珊瑚科品種屬于復合型珊瑚類群分支。同時, 蜂巢珊瑚科科內多源系統進化的現象也在本研究體現出來, 傳統形態學分類中同屬的向日葵蜂巢珊瑚和標準蜂巢珊瑚并沒有聚為一支, 反而向日葵蜂巢珊瑚與不同屬的多孔同星珊瑚聚率先聚為一支。
近年來, 許多學者通過顯微掃描技術對石珊瑚骨骼的精細結構進行觀察, 結合石珊瑚分子系統進化數據, 綜合全面的分析其系統進化關系, 對Veron傳統形態學研究結果進行了校正和補充[28-29]。很多精細的石珊瑚骨骼數據正逐漸改變我們對石珊瑚傳統分類的看法, 同時也對分子系統進化研究結果進行了有力的佐證。在當今的石珊瑚系統進化研究中, 無論是傳統的形態學方法還是利用分子技術手段, 兩者都必須相互利用和補充。
[1] Romano S L, Palumbi S R. Evolution of scleractinian corals inferred from molecular systematics[J]. Science, 1996, 271(5249): 640.
[2] 尤豐, 黃暉, 董志軍, 等. 造礁石珊瑚的分子系統學研究進展[J]. 海洋通報, 2010, 29(4): 459-465. You Feng, Huang Hui, Dong Zhijun, et al. Progress in taxonomy and genetic diversity of Scleratinia corals[J]. Marine Science Bulletin, 2010, 29(4): 459-465.
[3] Budd A F, Romano S L, Smith N D, et al. Rethinking the phylogeny of scleractinian corals: a review of morphological and molecular data[J]. Integrative and Comparative Biology, 2010, 50(3): 411-427.
[4] Odorico D M, Miller D J.Variation in the ribosomal internal transcribed spacers and 5.8S r DNA among five species of(Cnidaria; Sclerac-tinia): Patterns of variation consistent with reticulate evolution[J]. Molecular Biology and Evolution, 1997, 14: 465-473.
[5] Fukami H, Omori M, Hatta M. Phylogenetic relationships in the coral family Acroporidae, reassessed by inference from mitochondrial genes[J]. Zoological science, 2000, 17(5): 689-696.
[6] Forsman Z H, Concepcion G T, Haverkort R D, et al. Genetic and morphological characterization of a coral species of concern montipora dilatata in Kaneohe Bay, Oahu, Hawaii[J]. Pacific Islands Region Species of Concern, 2008: 1-17.
[7] Huang D, Meier R, Todd P A, et al. More evidence for pervasive paraphyly in scleractinian corals: Systematic study of Southeast Asian Faviidae (Cnidaria; Scleractinia) based on molecular and morphological data[J]. Molecular Phylogenetics and Evolution, 2009, 50(1): 102-116.
[8] Arrigoni R, Stefani F, Pichon M, et al. Molecular phylogeny of the robust clade (Faviidae, Mussidae, Merulinidae, and Pectiniidae): an Indian Ocean perspective[J]. Molecular Phylogenetics and Evolution, 2012, 65(1): 183-193.
[9] 齊文同, 柯葉艷, 顧紅雅. 石珊瑚演化的18S rDNA分子生物學和形態學研究[J]. 北京大學學報(自然科學版), 2008, 44(2): 171-176. Qi Wentong, Ke Yeyan, Gu Hongya. Molecular systematics of the 18S rDNA within the scleractinian corals[J]. Acta Scientiarum Naturalium Universitatis Pekinensis, 2008, 44(2): 171-176.
[10] 程希婷. 石珊瑚的 DNA 條形碼研究及其系統發育分析[D]. 海南: 海南大學, 2012. Cheng Xiting. DNA barcoding and molecular phylogeny of the scleractinia[D].Hainan: Hainan University, 2012.
[11] 李曉娜, 劉麗, 劉楚吾. 運用線粒體COI基因分析8種石珊瑚系統發育關系[J]. 漁業科學進展, 2012, 33(4): 83-88. Li Xiaona, Liu Li, Liu Chuwu. Phylogenetic relationship in 8 species of Scleractinian coral based on COI sequences[J]. Progress in Fishery Sciences, 2012, 33(4): 83-88.
[12] 劉麗, 李曉娜, 陳育盛, 等. 基于線粒體基因的石珊瑚分子系統學研究[J]. 海洋與湖沼, 2012, 43(4): 814-820. Liu Li, Li Xiaona, Chen Yusheng, et al. Molecular systematics study of scleractinian based on mitchondrial genes[J]. Oceanologia and Limnologia Sinica, 2012, 43(4): 814-820.
[13] 劉麗, 陳育盛, 李曉娜, 等. 基于線粒體Cytb基因的10種石珊瑚的系統發育關系[J]. 廣東海洋大學學報, 2011, 31(1): 6-11. Liu Li, Chen Yusheng, Li Xiaona, et al. Phylogenetic Relationships in 10 Species of Scleractinian Coral Based on Cyt b Sequences[J]. Journal of Guangdong Ocean University, 2011, 31(1): 6-11.
[14] 廖偉群, 張加恭, 黃慧萍. 萬山群島的旅游資源及其開發利用研究[J]. 熱帶地理, 2000, 20(2): 134-138, 143.Liao Weiqun, Zhang Jiagong, Huang Huiping. Development of the tourism resources of the wanshan islands[J]. Tropical Geography, 2000, 20(2): 134-138, 143.
[15] 黃暉, 尤豐, 練健生, 等. 珠江口萬山群島海域造礁石珊瑚群落分布與保護[J]. 海洋通報, 2012, 31(2): 189-197. Huang Hui, You Feng, Lian Jianshen, et al. Status and conservation strategies of the scleractinian coral community in the Wanshan Islands at Pearl River Estuary[J]. Marine Science Bulletin, 2012, 31(2): 189-197.
[16] 鄒仁林. 中國動物志—造礁石珊瑚[M]. 北京: 科學出版杜, 2001. Zou Renlin. Fauna Sinica: Coelenterata: Anthozoa, Scleractinia, Hermatypic coral[M]. Beijing: Science Press, 2001.
[17] Veron J E N. Corals in space and time: the biogeography and evolution of the Scleractinia[M]. Ithaca, New York: Comstock/Cornell, 1995.
[18] Yamauchi M M, Miya M U, Nishida M. Complete mitochondrial DNA sequence of the swimming crab, Portunus trituberculatus (Crustacea: Decapoda: Brachyura)[J]. Gene, 2003, 311: 129-135.
[19] Hellberg M E. No variation and low synonymous substitution rates in coral mtDNA despite high nuclear variation[J]. BMC evolutionary biology, 2006, 6(1): 24.
[20] Arrigoni R, Kitano Y F, Stolarski J, et al. A phylogeny reconstruction of the Dendrophylliidae (Cnidaria, Scleractinia) based on molecular and micromorphological criteria, and its ecological implications[J]. Zoologica Scripta, 2014, 43(6): 661-688.
[21] Gittenberger A, Reijnen B T, Hoeksema B W. A molecularly based phylogeny reconstruction of mushroom corals (Scleractinia: Fungiidae) with taxonomic consequences and evolutionary implications for life history traits[J]. Contributions to Zoology, 2011, 80(2): 107-132.
[22] Kitahara M V, Cairns S D, Stolarski J, et al. A comprehensive phylogenetic analysis of the Scleractinia (Cnidaria, Anthozoa) based on mitochondrial CO1 sequence data[J]. PloS One, 2010, 5(7): e11490.
[23] Fukami H, Knowlton N. Analysis of complete mitochondrial DNA sequences of three members of thecoral species complex (Cnidaria, Anthozoa, Scleractinia)[J]. Coral Reefs, 2005, 24(3): 410-417.
[24] Romano S L, Cairns S D. Molecular phylogenetic hypotheses for the evolution of scleractinian corals[J]. Bulletin of Marine Science, 2000, 67(3): 1043-1068.
[25] 李文娟, 劉楚吾, 劉麗, 等. 利用ITS基因研究徐聞濱珊瑚的系統發生關系[J]. 海洋科學, 2012, 36(7): 56-60. Li Wenjuan, Liu Chuwu, Liu Li, et al. Phylogeny analysis of Porites link in Xuwen using its gene[J]. Marine Sciences, 2012, 36(7): 56-60.
[26] Fukami H, Chen C A, Budd A F, et al. Mitochondrial and nuclear genes suggest that stony corals are monophyletic but most families of stony corals are not (Order Scleractinia, Class Anthozoa, Phylum Cnidaria)[J]. PloS one, 2008, 3(9): e3222.
[27] Huang D, Licuanan W Y, Baird A H, et al. Cleaning up the'Bigmessidae': Molecular phylogeny of scleractinian corals from Faviidae, Merulinidae, Pectiniidae and Trachyphylliidae[J]. BMC Evolutionary Biology, 2011, 11(1): 1.
[28] Stolarski J, Kitahara M V, Miller D J, et al. The ancient evolutionary origins of Scleractinia revealed by azooxanthellate corals[J]. BMC Evolutionary Biology, 2011, 11(1): 316.
[29] Huang D, Benzoni F, Fukami H, et al. Taxonomic classification of the reef coral families Merulinidae, Montastraeidae, and Diploastraeidae (Cnidaria: Anthozoa: Scleractinia)[J]. Zoological Journal of the Linnean Society, 2014, 171(2): 277-355.
(本文編輯: 梁德海)
Study of phylogenetic relationships of scleractinian from Wailing-ding Island based on three standard molecular markers
XIAO Bao-hua1, 2, LIAO Bao-lin2, YANG Xiao-dong2, XIE Zi-qiang3
(1. Guangdong Ocean University, Zhanjiang 524025, China; 2. Guangdong Ocean University, Shenzhen Institute, Shenzhen 518108, China; 3. Shenzhen Bihai Blue Sky Marine Technology Co, LTD, Shenzhen 518108, China)
The mitochondrial cytochrome oxidase subunit I (CO I), 16S rRNA, and nuclear ITS genes have been routinely used as standard molecular markers. A total of five species pertaining to four genera of scleractinian, collected from Wailingding Island, Zhuhai City, Guangdong Province, were analyzed by partial sequences, and the genetic distances of these three genes were calculated. The results indicated that the average AT contents of CO I and 16S rRNA gene fragments were obviously higher than the GC contents. However, the average AT content of ITS fragments was obviously less than the GC content. The average genetic distances of the three genes were 0.043, 0.134, and 0.763 from the five species, respectively. The neighbor-joining, maximum-likelihood, and minimum-evolution trees of combined partial sequences of these three genes revealed that the phylogenetic relationships were consistent with the morphological classification in the families. However, the phylogenetic relationships were slightly different from the morphological classification in Faviidae. This result suggested that morphological classification may be limited by coral skeleton plasticity. These findings provide basic data for studying the genetic relationship of Wailingding Island corals and a theoretical basis for resource protection of stone corals in this area.
Scleractinian; COI; 16S RNA; ITS; Molecular phylogenetics
Dec. 22, 2016
肖寶華(1978-), 男, 福建長樂人, 高級工程師, 碩士, 主要從事海洋生態學, 特別是珊瑚生態系統方面研究, 電話: 0759- 2396216, E-mail: gdouxxhpaper@126.com
Q75
A
1000-3096(2017)09-0001-08
10.11759/hykx20160414001
2016-12-12;
2017-02-22
廣東省漁港建設和漁業產業發展專項(A201708D06); 大鵬新區產業發展專項(KY20160107)
Guangdong Ocean and Fishery Administration Project, No. A201708D06; Dapeng New District Industry Development Fund, No. KY20160107]

