藍藻的蛋白質翻譯后修飾研究進展
楊明坤 林小煌, 馬炎炎, 王 炎, 葛 峰
(1. 中國科學院水生生物研究所,藻類生物學重點實驗室,武漢 430072; 2. 中國科學院大學,北京 100049)
藍藻的蛋白質翻譯后修飾研究進展
楊明坤1林小煌1,2馬炎炎1,2王炎1,2葛峰1
(1. 中國科學院水生生物研究所,藻類生物學重點實驗室,武漢 430072; 2. 中國科學院大學,北京 100049)
蛋白質翻譯后修飾系統幾乎參與了細胞所有的正常生命活動過程,并發揮著重要的調控作用。目前,基于生物質譜技術進行蛋白質翻譯后修飾的規模化分析鑒定,已經成為蛋白質組學研究的核心內容之一。近年來的研究表明,藍藻細胞中存在著復雜的蛋白質翻譯后修飾系統,如磷酸化,乙酰化,甲基化,糖基化,氧化等,這些翻譯后修飾在藍藻細胞的代謝過程中可能發揮著重要的調控作用。本文主要針對藍藻細胞中蛋白質翻譯后修飾的發現與鑒定,以及翻譯后修飾潛在的生物學功能展開簡要綜述。
藍藻;蛋白質組學;翻譯后修飾
藍藻(Cyanobacteria),具有革蘭氏陰性細菌相似的細胞結構,其細胞內含有大量葉綠素a和藻藍素使得細胞呈藍綠色,因此,藍藻又被稱為藍細菌或藍綠藻。藍藻在地球上分布廣泛,是最早出現的光合放氧生物,對地球表面從無氧環境變為有氧的大氣環境發揮了關鍵性作用[1]。藍藻結構簡單,沒有分化真正意義上的細胞核、線粒體、葉綠體等細胞器,卻具有類似于真核藻類和高等植物的光合作用系統,能夠進行光解水放氧。藍藻還具有固氮,氫代謝以及基于光合作用生[2]產生物燃料的能力,同時,在遺傳學上又具有操作性強,構建突變體簡單且周期短等優點,藍藻已經成為研究光合作用和生物能源的重要模式生物之一。近年來的研究發現,藍藻細胞中存在多種不同類型的蛋白質翻譯后修飾,且這些修飾在藍藻細胞的代謝過程中可能發揮著重要的調控作用。本文針對藍藻細胞中蛋白質翻譯后修飾的分析鑒定及其潛在的生物學功能展開簡要綜述,將有助于深入了解藍藻的生長以及對環境變化的適應等,可為研究光合作用與開發藻類生物能源提供重要的理論依據。
1 蛋白質翻譯后修飾
蛋白質是執行細胞功能的基本單元,其表達受基因組,表觀遺傳學和翻譯后修飾等多個層次的調控。其中蛋白質翻譯后修飾作為蛋白質功能調節的一種重要方式,在調控不同生物體的眾多生物學過程中都發揮著重要的作用[2,3]。它通過在一個或多個氨基酸殘基加上修飾基團,可以改變蛋白質的物理、化學性質,進而影響蛋白質的空間構象、活性狀態、亞細胞定位以及蛋白質-蛋白質相互作用等。翻譯后修飾使蛋白質的結構更為復雜,功能更為完善,調節更為精細,作用更為專一。據估計,人體內50%—90%的蛋白質發生了不同類型的翻譯后修飾,包括肽鏈骨架的剪接,特定氨基酸側鏈上添加新的基團以及對已有基團進行化學修飾等[4]。目前,記錄在冊的翻譯后修飾達到幾千種(http://www. unimod.org/modifications_list.php?)(http://www. abrf.org/index.cfm/dm.home),仍有新的翻譯后修飾不斷被發現。根據蛋白質翻譯后修飾的專業數據庫PhosphoSitePlus(http://www.phosphosite.org)統計結果顯示,研究較多的蛋白質翻譯后修飾包括磷酸化、乙酰化、甲基化、糖基化和泛素化等[5]。由于蛋白質翻譯后修飾發生在轉錄之后,因此基因組學方法無法實現翻譯后修飾類型以及結構的檢測。
近年來,隨著生物質譜技術的進步和高效分離方法的發展,蛋白質組學技術為蛋白質翻譯后修飾的系統分析提供了前所未有的發展契機,由此也派生出一門新的學科:修飾蛋白質組學[2,6],它主要利用蛋白質組學分析技術對修飾蛋白質及其位點進行規模化分析鑒定,對修飾結構進行確認以及對不同生理狀態下翻譯后修飾的變化情況進行定量分析。然而,大多數發生翻譯后修飾的蛋白質通常具有豐度低、動態范圍寬以及結構復雜等特點; 同時,翻譯后修飾存在“時空特異性”,即蛋白質的修飾類型與修飾程度隨生物生存環境及內在狀態的變化會表現出極大的差別,有的修飾甚至是轉瞬即逝的,所以,蛋白質翻譯后修飾的程度以及其生物學功能的挖掘還存在著極大的空間,對蛋白質翻譯后修飾進行規模化分析也面臨著巨大的挑戰。
2 藍藻的蛋白質翻譯后修飾研究
近年來,越來越多的研究發現,蛋白質翻譯后修飾,如磷酸化,乙酰化,甲基化,糖基化,生物素化,氧化等,可能參與藍藻的脅迫響應,能量代謝以及光合作用等一系列的細胞進程; 甚至在藍藻的次級代謝產物中(如各種多肽),也發現存在糖基化,甲基化,酰基化等修飾,這些修飾可能具有調控次級代謝產物的功能,從而在抗菌、抗癌以及免疫活性抑制等方面發揮重要作用[7]。然而,目前對于藍藻蛋白質翻譯后修飾的功能研究仍然較少,尤其是翻譯后修飾在藍藻細胞中的分子調控機制仍有待于進一步的深入研究,以下將對目前藍藻細胞中的蛋白質翻譯后修飾研究進展進行討論。
2.1磷酸化修飾
藍藻能夠耐受高低溫、紫外線輻射、干旱和鹽等脅迫,其細胞內復雜的信號轉導系統起了非常重要的調控作用。它能夠使藍藻非常迅速的感受外界環境的信號變化,通過調控基因的表達,使細胞對外界環境作出積極快速的響應,從而使藍藻在各種極端環境中得以生存下來。目前,藍藻的信號轉導系統主要有二元信號傳導系統,類似于真核的絲氨酸/蘇氨酸激酶系統(Serine/threonine protein kinase,STK)以及近年來發現的酪氨酸激酶/磷酸酶(Tyrosine kinase/tyrosine phosphatase)系統[8]。隨著藍藻全基因組測序工作的完成,藍藻細胞中磷酸化修飾介導的信號轉導系統也受到越來越多的關注。通過基因組分析發現,在集胞藻PCC 6803和聚球藻PCC 7002中均含有12個預測的STK序列,集胞藻PCC 6803含有7個相關的磷酸酶,聚球藻PCC 7002中含有6個相關的磷酸酶[9—11],而在魚腥藻PCC 7120中則含有多達52個預測的STK序列和12個相關的磷酸酶[12]。此外,還在藍藻中發現有少量預測的酪氨酸激酶/磷酸酶序列[8]。這些激酶/磷酸酶介導的可逆磷酸化修飾系統,在細胞感受外界刺激以及作出適應的應答過程中起著至關重要的調控作用[13]。發現并鑒定這些激酶/磷酸酶的作用底物以及驗證其在信號網絡中的作用,將有助于更加深入全面的理解磷酸化修飾介導的藍藻信號轉導網絡。
早在1984年,Schuster等[14]就運用32P標記技術,首次在藻細胞Calothrix sp. PCC 7601中發現了磷酸化修飾蛋白質。基于該方法,在Synechococcas sp. PCC 6301[15],Anabaena sp. PCC 7120[16],Synechocystis sp. PCC 6803[17,18],Synecbococcus sp. PCC 7942[19]等藻株中均鑒定到磷酸化修飾蛋白質的存在。進一步研究表明,藻膽蛋白復合物磷酸化修飾有助于藻膽體的組裝,而在高光與缺氮條件下,藻膽蛋白復合物會發生去磷酸化修飾,從而導致藻膽體發生解聚,使細胞免受光損傷[20,21]。研究發現,KaiC蛋白則可以通過自磷酸化與去磷酸作用調控其自身活性,從而調節藍藻細胞的生物鐘[22]。此外,磷酸化修飾還可能參與了藍藻的碳源/氮源利用調控,高光適應,狀態轉換以及鹽脅迫等一系列的過程[8,20]。雖然在藍藻中已經發現一些磷酸化蛋白可能參與多種代謝調控途徑,但仍然只有較少的磷酸化蛋白被鑒定,且具體的磷酸化修飾位點也仍然未知,這嚴重阻礙了藍藻細胞中磷酸化修飾的分子調控作用機制的研究。
隨著修飾蛋白質組學技術的日趨成熟,使利用高通量質譜技術鑒定藍藻細胞中磷酸化修飾蛋白及其位點成為可能。作者首次利用目前比較成熟的磷酸化肽段富集技術-TiO2富集技術,對一株極端耐鹽、耐高光的模式藍藻-聚球藻PCC 7002進行磷酸化肽段富集,并結合高通量質譜技術,鑒定到245個磷酸化蛋白,其中較多磷酸化蛋白參與了二元信號轉導通路以及光合作用通路,包括光合系統Ⅰ,光合系統Ⅱ,藻膽體以及細胞色素復合物等[23]。而在另一株模式藍藻中-集胞藻PCC 6803,同樣鑒定到較多的磷酸化蛋白。Mikkat等[24]首次結合雙向電泳技術與MALDI-TOF MS/MS質譜技術,從高鹽和低鹽培養的集胞藻PCC 6803中鑒定到32個磷酸化蛋白。由于磷酸化蛋白及其修飾位點鑒定數目仍然較少,Sp?t等[25]則結合磷酸化肽段富集與高通量質譜技術,系統地分析了集胞藻PCC 6803中的磷酸化蛋白,共鑒定到202個磷酸化蛋白,并通過二甲基化定量標記方法,對藻細胞在缺氮條件下的蛋白表達水平進行了分析,最終定量到了2111個蛋白,其中包含148個磷酸化蛋白; 研究結果顯示,藻細胞缺氮條件處理24h后,細胞內磷酸化修飾水平顯著提高,包括PII信號蛋白,碳/氮代謝途徑以及光合作用途徑中的相關蛋白,表明磷酸化修飾在藻細胞應對環境刺激方面具有重要的調控作用。同年,Lee等[26]也報道了集胞藻PCC 6803中的磷酸化蛋白,不同的是,該研究對膜蛋白的磷酸化修飾進行了分析,共鑒定到33個磷酸化蛋白,其中11個為具有跨膜結構的磷酸化膜蛋白。作者通過對集胞藻PCC 6803中磷酸化蛋白研究發現,較多蛋白參與了能量代謝與光合作用途徑,進一步功能研究顯示,磷酸化修飾參與調控了藍藻的光能吸收與傳遞過程[27]。
磷酸化修飾作為一種快速的應激調控方式,在調控藍藻細胞適應環境脅迫方面起著重要作用。越來越多磷酸化蛋白及其修飾位點的發現,將為全面深入研究磷酸化修飾調控藍藻細胞脅迫響應的分子作用機制提供新的理論依據與研究思路。
2.2乙酰化修飾
蛋白質的賴氨酸乙酰化修飾與磷酸化修飾一樣,在生物體內廣泛存在,是一種可逆、保守且高度調控的翻譯后修飾,參與調控代謝、轉錄活性、蛋白質穩定性、信號通路及病原微生物的感染等眾多重要的生理功能[28—30]。乙酰化修飾能夠中和蛋白分子帶正電荷的賴氨酸殘基,從而改變蛋白的物理、化學性質,實現對蛋白功能的調節[31,32]。最初的研究主要集中在染色體組蛋白的乙酰化修飾上,它能中和組蛋白賴氨酸殘基的正電荷,使其與DNA 的結合松散而利于基因的轉錄,調控基因表達水平[33]。因此,乙酰化修飾對表觀遺傳學有著深遠影響。近年來隨著研究的深入,發現很多非組蛋白也可進行乙酰化修飾調節,由此開拓出以非組蛋白為底物的蛋白質乙酰化修飾研究領域。隨后的研究表明,細胞內幾乎所有的代謝酶都可以被乙酰化修飾,該修飾廣泛參與到整個細胞最基礎的生命代謝活動中,對細胞內的各類代謝通路進行精確的調節與控制(圖1)[34,35]。
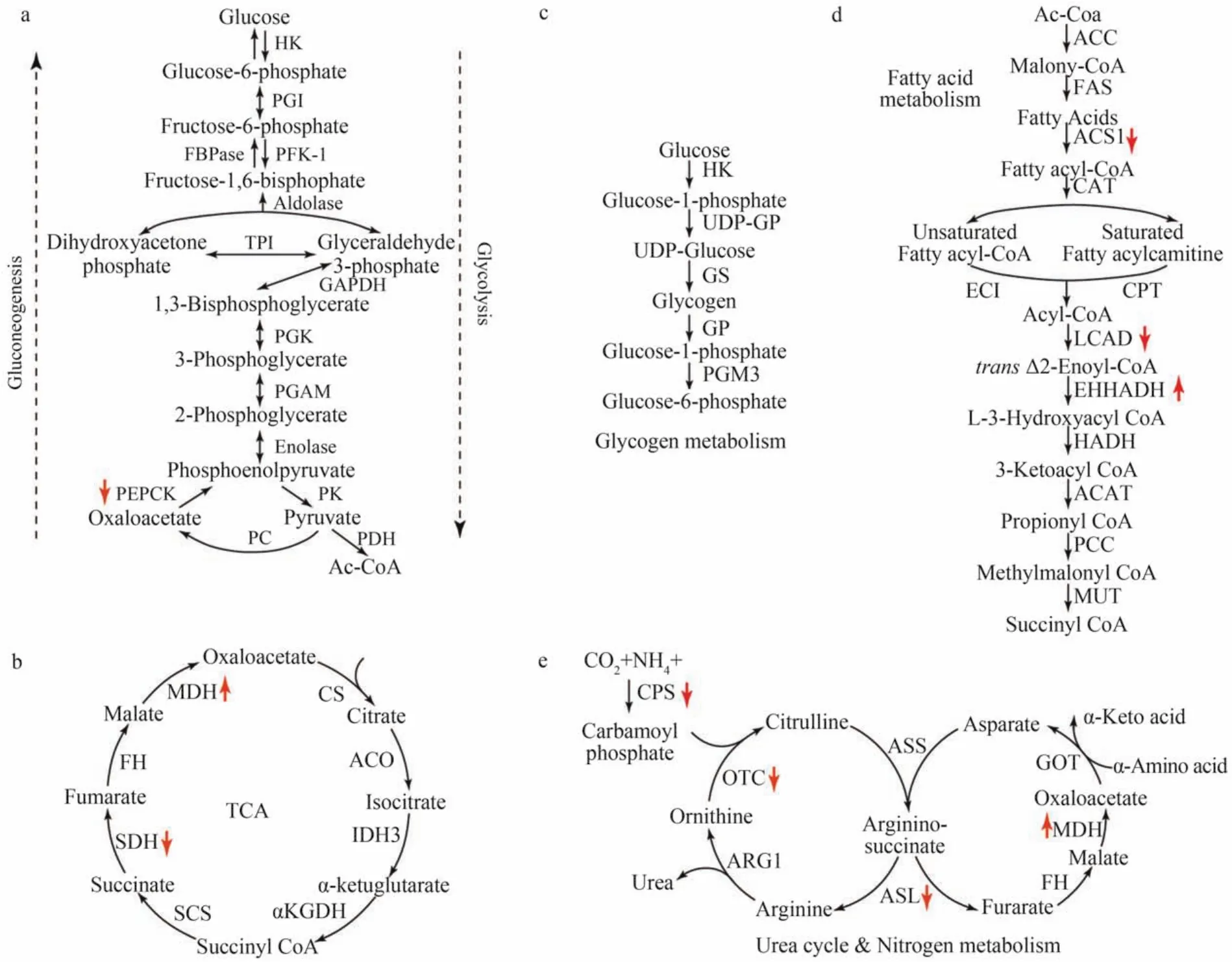
圖1 乙酰化修飾廣泛調控細胞代謝過程[35]Fig. 1 Acetylation regulation of metabolic processes
在原核生物中,最早由Zhang等[36]利用乙酰化肽段富集技術,結合高通量的nano-HPLC/MS/MS技術,在大腸桿菌中首次大規模鑒定到91個乙酰化蛋白。高通量乙酰化蛋白質組學的關鍵就在于乙酰化肽段富集技術,通常只有親和性較高的泛乙酰化修飾抗體,才能有效的富集到細胞中含量較低的乙酰化修飾肽段。由于高質量抗體的缺乏,嚴重阻礙了乙酰化蛋白質組學的發展。2013年,Zhang等[37]改進了乙酰化修飾抗體的制備策略,獲得了高效的乙酰化修飾抗體,最終在大腸桿菌中鑒定到了349個乙酰化蛋白的1070個乙酰化修飾位點。之后,在枯草芽孢桿菌[38],鼠傷寒沙門氏菌[39],熾熱芽孢桿菌[40]以及結核分枝桿菌[41]等一系列原核生物中,均鑒定到較多乙酰化蛋白及其修飾位點。隨著更多乙酰化蛋白的鑒定,該修飾的調控作用也逐漸被揭示,其中最主要的作用就是參與代謝調控,如糖酵解,TCA循環,磷酸戊糖途徑、氨基酸代謝以及脂肪酸代謝等。研究發現,乙酰化修飾甚至可以調控致病相關蛋白的免疫原性,影響病原菌的致病性。這些研究揭示了乙酰化修飾在調控能量代謝過程以及影響病原菌致病性等方面的重要作用。
在藍藻的光合系統復合物中,已經發現鐵硫蛋白Pet,細胞色素蛋白PsbF以及光系統Ⅱ蛋白PsbJ都存在乙酰化修飾[42,43]。此外,在藍藻的次級代謝產物中,同樣發現部分肽段或是藻毒素被乙酰化修飾[44—46]。作者結合乙酰化肽段富集與高通量nano-HPLC/MS/MS技術,首次系統地分析了藍藻細胞中的乙酰化修飾水平,在集胞藻PCC 6803中鑒定到513個乙酰化蛋白及其776個修飾位點,生物信息學分析結果顯示,大多數乙酰化蛋白參與了藻細胞的能量代謝過程,并首次發現了31個被乙酰化修飾的光合作用相關蛋白與16個碳固定途徑相關蛋白,該研究揭示了乙酰化修飾可能參與調控藍藻的光合作用途徑[47]。在另外一株藍藻-聚球藻PCC 7002中,作者同樣鑒定到較多乙酰化蛋白,進一步的功能研究結果顯示,可逆的乙酰化修飾不僅能夠調控光能吸收與傳遞效率,還能通過影響光合作用相關蛋白結構,進而影響光合作用復合物的組裝,顯著降低光合作用效率(未發表)。綜上結果,乙酰化修飾不僅能夠調控代謝酶活性,還能通過改變蛋白結構,影響蛋白的功能,從而參與藍藻細胞內的光合作用調控過程。
蛋白質乙酰化與去乙酰化修飾是一種精細的調節過程,由乙酰轉移酶和去乙酰化酶對蛋白質賴氨酸上的一個或多個位點進行乙酰化和去乙酰化修飾,從而改變蛋白的結構與功能,對細胞內眾多代謝相關通路進行精確的調節與控制。隨著蛋白質乙酰化修飾研究的深入,其參與調控的代謝途徑及其重要性也倍受關注。在真核生物中,乙酰轉移酶與去乙酰化酶的功能研究較多,兩種酶介導的乙酰化和去乙酰化反應,能夠影響細胞的多種代謝網絡[48—51]。在原核生物中,Starai等[52]在沙門氏菌中首次發現了乙酰轉移酶(Protein acetyltransferase,簡稱Pat),它能夠調節中心代謝酶-乙酰輔酶A合成酶活性,進而調控細胞的代謝網絡。而具有去乙酰化酶活性的蛋白CobB(NAD+-dependent deacetylase,簡稱CobB)最早被用于ADP-核糖基轉移酶的功能研究[53]。2002年,Starai等[54]在鼠傷寒沙門氏菌中發現乙酰輔酶A合成酶的609位賴氨酸殘基的乙酰化被CobB去掉后,能夠激活其酶活性,從而首次報道了原核生物的去乙酰化酶。隨后以Pat、CobB作為乙酰轉移酶和去乙酰化酶展開了一系列的研究,揭示了兩種酶催化底物的作用機制[55—57]。作者通過比對分析發現,在聚球藻PCC 7002中含有16個預測的乙酰轉移酶基因以及4個去乙酰化酶基因,在集胞藻PCC 6803中也含有10個預測的乙酰轉移酶基因以及2個去乙酰化酶基因,這些乙酰轉移酶和去乙酰化酶可能通過改變底物蛋白的乙酰化修飾水平,從而對代謝以及光合作用途徑進行精細調控。然而,迄今為止,在藍藻中還未見到乙酰轉移酶和去乙酰化酶的相關研究報道,其所催化的底物蛋白在光合代謝中的調控機理也還沒有被揭示,均有待于進一步的深入研究。
2.3其他修飾類型
在藍藻細胞中,還存在許多其他不同種類的蛋白質翻譯后修飾,對藻細胞的生長,代謝等過程進行精細調控,如氧化修飾,糖基化修飾等。
光合生物能夠適應光合放氧與呼吸作用所產生的活性氧,主要依賴于其胞內復雜的抗氧化防御系統,而在大多數生物中,還原型谷胱甘肽介導的抗氧化起著具有非常重要的作用[58]。谷胱甘肽通過巰基與蛋白中半胱氨酸的自由巰基結合,形成蛋白的谷胱甘肽修飾[59,60],當細胞受到氧化脅迫或亞硝化脅迫時,可逆的谷胱甘肽修飾能夠起到保護蛋白以及調控蛋白活性的作用[59,61]。在聚球藻PCC 6803中,也發現谷胱甘肽修飾能夠調控蛋白活性[62,63]。Chardonnet等[64]就利用高通量蛋白質組學方法,在集胞藻PCC 6803中發現了383個谷胱甘肽修飾的蛋白質,這些蛋白廣泛參了到碳/氮代謝,細胞分裂,脅迫響應以及H2代謝等過程。除了谷胱甘肽修飾,在蛋白的半胱氨酸上還存在其他重要的翻譯后修飾,如巰基氧化修飾,Guo等[65]通過化學方法標記已發生氧化修飾的半胱氨酸,再針對標記的化學基團進行特異性的富集,最終,鑒定并定量到1350個半胱氨酸的巰基氧化修飾。
糖基化修飾也是較早被發現的翻譯后修飾之一,一直認為只有真核生物中才存在,直到在嗜鹽桿菌Halobacterium salinarium的膜組分中也發現糖基化修飾[66],該修飾在原核生物(尤其是病原菌)中的作用也相繼被揭示,而糖基化修飾的蛋白大多數屬于纖毛蛋白,在調控細胞毒力以及細菌與宿主細胞黏著方面起著重要作用[67—71]。2004年,Kim等[72]首次在藍藻中發現了纖毛蛋白可能存在不同類型的翻譯后修飾,這些修飾可能影響到纖毛的組裝,其中就包括纖毛蛋白中的O連接的糖基化修飾,該修飾可能與藻細胞的趨向運動有關[73]。此外,Kim等[74]還發現了另外一種重要纖毛蛋白修飾-三甲基化修飾,他們利用超高分辨率質譜(FTICR)MS,成功鑒定到纖毛蛋白PiliA的三甲基化修飾以及二甲基化、單甲基化修飾。已知三甲基化修飾(ΔM=42.0106 Da)是一種分子量與乙酰化修飾(ΔM=42.0470 Da)非常接近的翻譯后修飾,對于分辨率較低的質譜,往往無法區分0.03 Da的誤差。Kim等[74]不僅鑒定到纖毛蛋白三甲基化修飾,還通過修飾位點的定點突變實驗分析發現,三甲基化修飾能夠通過調控纖毛蛋白的功能,進而影響細胞的趨向運動。
此外,在藍藻細胞中,還發現存在甲基化[75,76]、過硫化[77,78]、ADP-核糖基化[79]、脂酰基化[80]、異戊二烯化[81]、棕櫚酰化[82]等不同的翻譯后修飾,這些修飾可能在藍藻細胞的代謝與光合作用等過程中可能發揮著不同的調控作用。
3 藍藻蛋白質翻譯后修飾的大規模發現
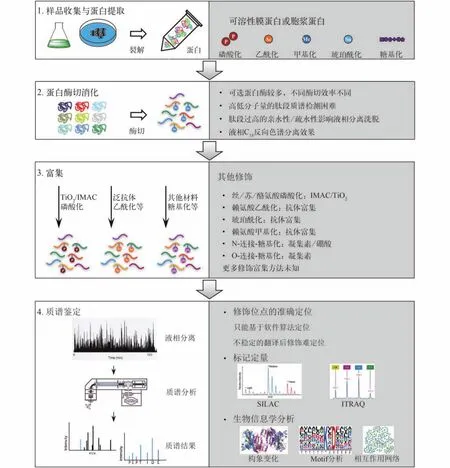
圖2 蛋白質翻譯后修飾分析策略Fig. 2 Strategies of proteomic analysis of post-translational modifications
雖然利用高通量蛋白質組學技術,已經發現了一些不同類型的蛋白質翻譯后修飾,但由于修飾蛋白質的不均一性以及相對豐度低的特性,使得蛋白質翻譯后修飾的大規模分析鑒定仍存在困難。目前,對復雜生物樣品中修飾蛋白質和肽段主要采用選擇性富集的策略,來實現修飾蛋白質組學的規模化鑒定(圖2),如技術路線最成熟的富集策略-基于TiO2等化學材料富集的磷酸化蛋白質組學和基于乙酰化修飾抗體富集的乙酰化蛋白質組學。然而,還存在許多不同類型的翻譯后修飾并沒有很好的富集策略,無法實現大規模鑒定。作者研究團隊利用近年來興起的一個新的技術手段-蛋白質基因組學(蛋基組學,Proteogenomics)技術,不經過樣品富集,在聚球藻PCC 7002中,首次大規模鑒定到23種不同的蛋白質翻譯后修飾。
蛋基組學,就是利用蛋白質組學數據,結合基因組與轉錄組數據對基因組進行注釋。最先由Jaffe等[83]于2004年首次提出,作者采用高通量質譜數據匹配基因組直接翻譯得到蛋白序列的方法,在僅有810 kb大小的細菌基因組上直接鑒定開放閱讀框(open reading frame,ORF),通過此方法,作者對原有基因組信息進行了驗證與補充,并修訂了約10%的ORF。隨著蛋白質組學技術手段日益成熟,高靈敏度、高精度的質譜儀使得完全覆蓋蛋白質組也成為可能,如人類蛋白質組數據中84%的蛋白已被鑒定到[84,85],因此,蛋白質組數據不僅可以實現對基因組序列的重新注釋、發現新基因,還能系統發現蛋白質特有的翻譯后事件(如翻譯后修飾和信號肽等)。
作者利用蛋基組學方法,對集胞藻PCC 7002進行了基因組的重注釋以及翻譯后修飾的大規模鑒定。收集了不同生長時期與代謝狀態的藻細胞,提取總蛋白并分離出膜組分與胞漿組分,采用Trypsin/ Glu-C蛋白酶進行酶切以獲得較高覆蓋度的肽段序列; 再利用C18填料的固相萃取柱或陽離子交換柱對獲得的肽段進行分離,降低肽段的復雜程度; 最后使用超高分辨率的質譜LTQ-Orbitrap Elite對肽段進行分析。雖然樣品中翻譯后修飾蛋白豐度較低,但采用了多重方法對樣品進行了處理,很大程度降低了樣品的復雜程度,即使在未經過任何形式的翻譯后修飾富集的情況下,仍能鑒定到較多翻譯后修飾蛋白質。利用非限制性檢索算法-即數據庫檢索時不設定任何修飾類型,對樣品中的翻譯后修飾進行非限制性檢索,主要采用非限制性蛋白質翻譯后修飾檢索引擎-MODa(MODaification via alignment)對樣品進行分析,最終鑒定到690個蛋白含有不同的翻譯后修飾。為了更精確定位翻譯后修飾及其對應的修飾位點,采用了MaxQuant算法,對感興趣的翻譯后修飾進一步驗證,最終大規模鑒定到了23種不同的翻譯后修飾,其中絕大多數修飾是首次在藍藻中發現。進一步研究表明,參與藍藻光合作用的大多數蛋白具有復雜的翻譯后修飾,在不同的生長和處理條件下會發生動態變化,提示這些修飾在光合系統中起著重要的調控作用(圖3)[86]。
4 結論與展望
研究藍藻中蛋白質翻譯后修飾的種類、結構、機制以及生物學功能,將使我們對藍藻蛋白質各種特性有進一步的了解,并為我們全面深入理解藍藻細胞生命活動的調控機制提供必要的信息。然而,蛋白質翻譯后修飾的大規模發現及其功能研究仍存在較多挑戰,如新發現的蛋白翻譯后修飾的富集鑒定方法,翻譯后修飾的定量分析以及翻譯后修飾的生物學功能等問題都是該領域目前遇到的且亟待解決的難題。
為了解新發現的蛋白質翻譯后修飾在全細胞水平的修飾狀態,往往需要借助高通量蛋白質組學手段實現其大規模鑒定,而這其中最重要的關鍵技術就是富集策略,目前大多采用的富集策略是用相應的泛抗體進行富集。對于不同類型的翻譯后修飾,如何選擇合適的富集方法以及對應的富集策略,將直接影響修飾蛋白及其位點的鑒定。
在生物體內,蛋白質的修飾種類不僅繁多,還具有可逆性以及時空特異性等特點,使蛋白質的修飾成為一個動態變化的過程,因此,對不同生長、分化以及代謝狀態下細胞內蛋白質修飾水平的定量具有重要的意義。目前,已有的定量蛋白質組學技術手段主要分為兩類,一類是穩定同位素標記方法,另一類是非標記定量方法。穩定同位素標記技術是蛋白質組學相對定量的經典方法,樣品在穩定同位素標記后、質譜分析前混合,一次分析實現差異定量,有效消除了色譜和質譜分析過程中的不穩定性,最大程度減小了定量誤差。常見的標記方法分為體內標記方法,如SILAC與15N標記。Mann等[87]采用同位素標記的甲硫氨酸標記細胞中的甲基基團,首次實現了甲基化翻譯后修飾蛋白質組學規模上的定量分析; SILAC技術同樣可以用于磷酸化與乙酰化修飾蛋白組學的規模化定量分析[29,88,89]; 由于SILAC標記方法只適用于不能自我合成氨基酸的哺乳動物細胞株,所以對于大多數原核生物并不適用,而15N標記則不受此限制[90,91],并在藍藻中得到了很好的應用[92—94],因此該標記技術用于藍藻翻譯后修飾定量具有廣闊的前景。而體外標記方法,如二甲基標記[95]、TMT標記[96]以及iTRAQ標記[97]等,幾乎全部標記賴氨酸的活性氨基,對于許多賴氨酸上的翻譯后修飾,體外標記卻并不適用。另一類重要的定量技術——非標記定量蛋白質組學技術,則不需要任何昂貴的標記試劑與復雜繁瑣的樣品處理過程,所以適合所有類型細胞的翻譯后修飾定量,如基于蛋白變性膠的非標記定量磷酸化蛋白組與非標記定量乙酰化蛋白組等[50,98]以及基于超高通量質譜的SWATH非標記定量方法[99]。但是,非標記定量蛋白質組學技術仍然不夠成熟,一方面是對nano-LC-MS/MS有很大的依賴性,需要實驗具有較高的重現性; 另一方面不論是常規的基于一級質譜的定量方法,還是最新建立的基于二級質譜的SWATH定量方法,對于后續數據處理過程中肽段定量的算法仍有待于進一步提高,才能更準確的定量肽段以及蛋白。從長遠來看,相比于標記定量技術,非標記定量方法仍具有更大的優勢和前景,而對于藍藻的翻譯后修飾定量組學,非標記定量方法則更為適用。
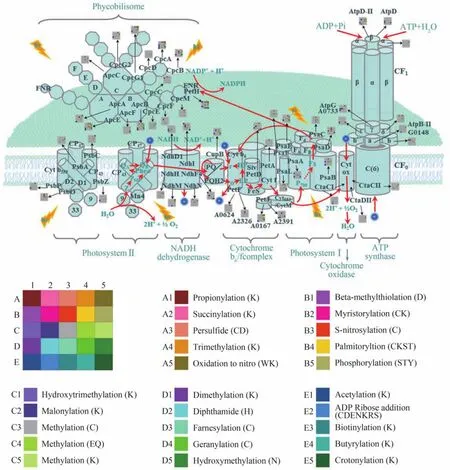
圖3 藍藻Synechococcus sp. PCC 7002光合作用系統中的蛋白質翻譯后修飾[86]Fig. 3 Post-translational modification events involved in the photosynthesis in Synechococcus sp. PCC 7002
蛋白質翻譯后修飾往往不是孤立存在的,同一生理或病理過程的發生,需要各種修飾的蛋白質共同作用,同一個蛋白質又可以同時擁有一種以上的蛋白質修飾,彼此之間相互影響、相互協調。單純的單一蛋白質、單一修飾的研究難以闡明代謝調控機制,只有運用組學手段對不同狀態下的蛋白質修飾動態網絡研究,才能更好的闡明不同翻譯后修飾的功能以及修飾之間的相互調控。在藍藻的光合作用途徑中,已經發現許多蛋白具有不同類型的翻譯后修飾,如磷酸化、乙酰化、甲基化等等(圖3);而不同翻譯后修飾之間相互影響,對蛋白功能進行多重調控,在細胞的整個生命活動中具有非常重要的作用。不同翻譯后修飾對蛋白乃至整個細胞的功能而言,可以是協同或是相反的作用,而這種情況發生的一個重要基礎就是存在于各種蛋白質翻譯后修飾之間的相互作用關系,因此,研究蛋白質翻譯后修飾之間相互調控作用關系,將有助于揭示藍藻細胞內蛋白質翻譯后修飾介導的復雜的蛋白功能調控網絡。
基于高通量蛋白質組學的翻譯后修飾鑒定數據正在快速增長,盡管這些數據證明了許多蛋白質翻譯后修飾是普遍存在的現象,但并沒有揭示出修飾的功能。雖然,發現并鑒定新的翻譯后修飾對理解生物眾多生理功能具有十分重要的意義,但深入理解修飾在整個細胞中的調控作用機制則更能深入闡明翻譯后修飾的重要性。目前大多數基于高通量蛋白質組學鑒定翻譯后修飾的研究,也都結合分子生物學或生物化學等技術手段,挑選若干重要的翻譯后修飾蛋白,試圖去解釋翻譯后修飾的調控作用。但相比于龐大的高通量數據,仍然只有很少翻譯后修飾位點的生物學功能被闡明,對于更多的修飾位點的功能,則只能通過推測討論或生物信息學分析獲得[100]。因此,細胞內各種代謝通路中的翻譯后修飾作用具有怎么樣的調控功能,均有待于進一步研究。
到目前為止,還沒有完整的實驗證據揭示蛋白質翻譯后修飾在藍藻細胞內具有怎樣的調控功能。但可以預期的是,隨著藍藻細胞內越來越多的蛋白質翻譯后修飾被大規模發現,這些修飾在細胞的整個生命活動中的生物學功能也將被逐漸闡明,并將開辟出藍藻的蛋白質翻譯后修飾組學這一新的研究領域。
[1]Bryant D A. The Molecular Biology of Cyanobacteria [M]. Kluwer Academic Publishers Dordrecht. 1994,139—216
[2]Mann M,Jensen O N. Proteomic analysis of post-translational modifications [J]. Nature Biotechnology,2003,21:255—261
[3]Hart G W,Ball L E. Post-translational modifications:a major focus for the future of proteomics [J]. Molecular & Cellular Proteomics,2013,12:3443—3443
[4]Doyle H A,Mamula M J. Post-translational protein modifications in antigen recognition and autoimmunity [J]. Trends in Immunology,2001,22(8):443—449
[5]Khoury G A,Baliban R C,Floudas C A. Proteome-wide post-translational modification statistics:frequency analysis and curation of the swiss-prot database [J]. Scientific Reports,2011,1:90
[6]Witze E S,Old W M,Resing K A,et al. Mapping protein post-translational modifications with mass spectrometry [J]. Nature Methods,2007,4(10):798—806
[7]Welker M,von Dohren H. Cyanobacterial peptides -nature's own combinatorial biosynthesis [J]. Fems Microbiology Reviews,2006,30(4):530—563
[8]Zhang C C,Jang J,Sakr S,et al. Protein phosphorylation on Ser,Thr and Tyr residues in cyanobacteria [J]. Journal of Molecular Microbiology and Biotechnology,2005,9(3—4):154—166
[9]Zhang C C,Gonzalez L,Phalip V. Survey,analysis and genetic organization of genes encoding eukaryotic-like signaling proteins on a cyanobacterial genome [J]. Nucleic Acids Research,1998,26(16):3619—3625
[10]Ludwig M,Bryant D A. Transcription profiling of the model cyanobacterium Synechococcus sp strain PCC 7002 by Next-Gen(SOLiD(TM)) sequencing of cDNA[J]. Frontiers in Microbiology,2011,2
[11]Kaneko T,Sato S,Kotani H,et al. Sequence analysis of the genome of the unicellular cyanobacterium Synechocystis sp. strain PCC6803. Ⅱ. Sequence determination of the entire genome and assignment of potential protein-coding regions [J]. DNA Research,1996,3:109—136
[12]Ohmori M,Ikeuchi M,Sato N,et al. Characterization of genes encoding multi-domain proteins in the genome of the filamentous nitrogen-fixing Cyanobacterium anabaena sp strain PCC 7120 [J]. DNA Research,2001,8:271—284
[13]Liu Z X,Li H C,Wei Y P,et al. Signal transduction pathways in Synechocystis sp. PCC 6803 and biotechnological implications under abiotic stress [J]. Critical Reviews in Biotechnology,2013,35(2):1—12
[14]Schuster G,Owens G C,Cohen Y,et al. Thylakoid polypeptide composition and light-independent phosphorylation of the chlorophyll-a,B-protein in Prochloron,a Prokaryote exhibiting oxygenic photosynthesis [J]. Biochimica Et Biophysica Acta,1984,767:596—605
[15]Allen J F,Sanders C E,Holmes N G. Correlation of membrane-protein phosphorylation with excitation energy distribution in the Cyanobacterium synechococcus-6301 [J]. Febs Letters,1985,193:271—275
[16]Mann N H,Rippka R,Herdman M. Regulation of protein-phosphorylation in the cyanobacterium Anabaena strain Pcc-7120 [J]. Journal of General Microbiology,1991,137:331—339
[17]Bloye S A,Silman N J,Mann N H,et al. Bicarbonate concentration by Synechocystis Pcc6803 - modulation of protein-phosphorylation and inorganic carbon transport by glucose [J]. Plant Physiology,1992,99:601—606
[18]Hagemann M,Golldack D,Biggins J,et al. Salt-dependent protein-phosphorylation in the cyanobacterium Synechocystis PCC-6803 [J]. Fems Microbiology Letters,1993,113:205—210
[19]Forchhammer K,Demarsac N T. The P-Ii protein in the cyanobacterium Synechococcus sp strain Pcc-7942 is modified by serine phosphorylation and signals the cellular N-status [J]. Journal of Bacteriology,1994,176:84—91
[20]Mann N H. Protein-phosphorylation in cyanobacteria [J]. Microbiology-Uk,1994,140:3207—3215
[21]Piven I,Ajlani G,Sokolenko A. Phycobilisome linker proteins are phosphorylated in Synechocystis sp. PCC 6803 [J]. Journal of Biological Chemistry,2005,280:21667—21672
[22]Nakajima M,Imai K,Ito H,et al. Reconstitution of circadian oscillation of cyanobacterial KaiC phosphorylation in vitro [J]. Science,2005,308:414—415
[23]Yang M K,Qiao Z X,Zhang W Y,et al. Global phosphoproteomic analysis reveals diverse functions of serine/threonine/tyrosine phosphorylation in the model cyanobacterium Synechococcus sp. strain PCC 7002 [J]. Journal of Proteome Research,2013,12:1909—1923
[24]Mikkat S,Fulda S,Hagemann M. A 2D gel electrophoresis-based snapshot of the phosphoproteome in the cyanobacterium Synechocystis sp. strain PCC 6803 [J]. Microbiology,2014,160:296—306
[25]Spat P,Macek B,Forchhammer K. Phosphoproteome of the cyanobacterium Synechocystis sp PCC 6803 and its dynamics during nitrogen starvation [J]. Frontiers in Microbiology,2015,6
[26]Lee D G,Kwon J,Eom C Y,et al. Directed analysis of cyanobacterial membrane phosphoproteome using stained phosphoproteins and titanium-enriched phosphopeptides [J]. Journal of Microbiology,2015,53:279—287
[27]Zhuo C,Jiao Z,Ying C,et al. Effects of phosphorylation of β subunits of phycocyanins on state transition in the model cyanobacterium Synechocystis sp. PCC 6803[J]. Plant and Cell Physiology,2015,56:1997—2013
[28]Wang Q,Zhang Y,Yang C,et al. Acetylation of metabolic enzymes coordinates carbon source utilization and metabolic flux [J]. Science,2010,327:1004—1007
[29]Choudhary C,Kumar C,Gnad F,et al. Lysine acetylation targets protein complexes and co-regulates major cellular functions [J]. Science,2009,325:834—840
[30]Lundby A,Lage K,Weinert B T,et al. Proteomic analysis of lysine acetylation sites in rat tissues reveals organ specificity and subcellular patterns [J]. Cell Reports,2012,2:419—431
[31]Kouzarides T. Acetylation:a regulatory modification to rival phosphorylation [J]? EMBO Journal,2000,19:1176—1179
[32]Yang X J. Lysine acetylation and the bromodomain:a new partnership for signaling [J]. Bioessays,2004,26:1076—1087
[33]Allfrey V,Faulkner R,Mirsky A. Acetylation and methylation of histones and their possible role in the regulation of RNA synthesis [J]. Proceedings of the National Academy of Sciences of the United States of America,1964,51:786
[34]Choudhary C,Weinert B T,Nishida Y,et al. The growing landscape of lysine acetylation links metabolism and cell signalling [J]. Nature Reviews Molecular Cell Biology,2014,15:536—550
[35]Guan K L,Xiong Y. Regulation of intermediary metabolism by protein acetylation [J]. Trends in Biochemical Sciences,2011,36:108—116
[36]Zhang J,Sprung R,Pei J,et al. Lysine acetylation is a highly abundant and evolutionarily conserved modification in Escherichia coli [J]. Molecular & Cellular Proteomics,2009,8:215—225
[37]Zhang K,Zheng S,Yang J S,et al. Comprehensive profiling of protein lysine acetylation in Escherichia coli [J]. Journal of Proteome Research,2013,12:844—851
[38]Kim D,Yu B J,Kim J A,et al. The acetylproteome of Gram-positive model bacterium Bacillus subtilis [J]. Proteomics,2013,13:1726—1736
[39]Wang Q J,Zhang Y K,Yang C,et al. Acetylation of metabolic enzymes coordinates carbon source utilization and metabolic flux [J]. Science,2010,327:1004—1007
[40]Lee D W,Kim D,Lee Y J,et al. Proteomic analysis of acetylation in thermophilic Geobacillus kaustophilus [J]. Proteomics,2013,13:2278—2282
[41]Liu F,Yang M,Wang X,et al. Acetylome analysis reveals diverse functions of lysine acetylation in Mycobacterium tuberculosis [J]. Molecular & Cellular Proteomics,2014,13:3352—3366
[42]Baniulis D,Yamashita E,Whitelegge J P,et al. Structure-function,stability,and chemical modification of the cyanobacterial cytochrome b(6)f complex from Nostoc sp PCC 7120 [J]. Journal of Biological Chemistry,2009,284:9861—9869
[43]Guskov A,Kern J,Gabdulkhakov A,et al. Cyanobacterial photosystem Ⅱ at 2.9-? resolution and the role of quinones,lipids,channels and chloride [J]. Nature Structural & Molecular Biology,2009,16:334—342
[44]Philmus B,Christiansen G,Yoshida W Y,et al. Posttranslational modification in microviridin biosynthesis [J]. Chembiochem,2008,9:3066—3073
[45]Shalev-Malul G,Lieman-Hurwitz J,Viner-Mozzini Y,et al. An AbrB-like protein might be involved in the regulation of cylindrospermopsin production by Aphanizomenon ovalisporum [J]. Environmental Microbiology,2008,10:988—999
[46]Fewer D P,Wahlsten M,Osterholm J,et al. The Genetic basis for O-acetylation of the microcystin toxin in Cyanobacteria [J]. Chemistry & Biology,2013,20:861—869
[47]Mo R,Yang M,Chen Z,et al. Acetylome analysis reveals the involvement of lysine acetylation in photosynthesis and carbon metabolism in the model cyanobacterium Synechocystis sp. PCC 6803 [J]. Journal of Proteome Research,2015,14:1275—1286
[48]Carrozza M J,Utley R T,Workman J L,et al. The diverse functions of histone acetyltransferase complexes [J]. Trends in Genetics,2003,19:321—329
[49]DoiM,Hirayama J,Sassone-Corsi P. Circadian regulator CLOCK is a histone acetyltransferase [J]. Cell,2006,125:497—508
[50]Rardin M J,Newman J C,Held J M,et al. Label-freequantitative proteomics of the lysine acetylome in mitochondria identifies substrates of SIRT3 in metabolic pathways [J]. Proceedings of the National Academy of Sciences of the United States of America,2013,110:6601—6606
[51]Hallows W C,Lee S,Denu J M. Sirtuins deacetylate and activate mammalian acetyl-CoA synthetases [J]. Proceedings of the National Academy of Sciences of the United States of America,2006,103:10230—10235
[52]Starai V J,Escalante-Semerena J C. Identification of the protein acetyltransferase(Pat) enzyme that acetylates acetyl-CoA synthetase in Salmonella enterica [J]. Journal of Molecular Biology,2004,340:1005—1012
[53]Tsang A W,Escalante-Semerena J C. CobB,a new member of the SIR2 family of eucaryotic regulatory proteins,is required to compensate for the lack of nicotinate mononucleotide:5,6-dimethylbenzimidazole phosphoribosyltransferase activity in cobT mutants during cobalamin biosynthesis in Salmonella typhimurium LT2 [J]. Journal of Biological Chemistry,1998,273:31788—31794
[54]Starai V J,Celic I,Cole R N,et al. Sir2-dependent activation of acetyl-CoA synthetase by deacetylation of active lysine [J]. Science,2002,298:2390—2392
[55]Li R,Gu J,Chen Y Y,et al. CobB regulates Escherichia coli chemotaxis by deacetylating the response regulator CheY [J]. Molecular Microbiology,2010,76:1162—1174
[56]Xu H,Hegde S S,Blanchard J S. Reversible acetylation and inactivation of Mycobacterium tuberculosis Acetyl-CoA synthetase is dependent on cAMP [J]. Biochemistry,2011,50:5883—5892
[57]Crosby H A,Rank K C,Rayment I,et al. Structural insights into the substrate specificity of the Rhodopseudomonas palustris protein Acetyltransferase RpPat identification of a loop critical for recognition by RpPat [J]. Journal of Biological Chemistry,2012,287:41392—41404
[58]Foyer C H,Noctor G. Ascorbate and glutathione:the heart of the redox hub [J]. Plant Physiology,2011,155:2—18
[59]Dalle-Donne I,Rossi R,Colombo G,et al. Protein S-glutathionylation:a regulatory device from bacteria to humans [J]. Trends in Biochemical Sciences,2009,34:85—96
[60]Zaffagnini M,Bedhomme M,Marchand C H,et al. Redox regulation in photosynthetic organisms:focus on glutathionylation [J]. Antioxidants & Redox Signaling,2012,16:567—586
[61]Grek C L,Zhang J,Manevich Y,et al. Causes and consequences of cysteine S-Glutathionylation [J]. Journal of Biological Chemistry,2013,288:26497—26504
[62]Marteyn B,Sakr S,Farci S,et al. The Synechocystis PCC6803 MerA-Like enzyme operates in the reduction of both mercury and uranium under the control of the Glutaredoxin 1 enzyme [J]. Journal of Bacteriology,2013,195:4138—4145
[63]Sakr S,Dutheil J,Saenkham P,et al. The activity of the Synechocystis PCC6803 AbrB2 regulator of hydrogen production can be post-translationally controlled through glutathionylation [J]. International Journal of Hydrogen Energy,2013,38:13547—13555
[64]Chardonnet S,Sakr S,Cassier-Chauvat C,et al. First proteomic study of S-Glutathionylation in cyanobacteria[J]. Journal of Proteome Research,2015,14:59—71
[65]Guo J,Nguyen A Y,Dai Z Y,et al. Proteome-wide light/dark modulation of thiol oxidation in cyanobacteria revealed by quantitative site-specific redox proteomics [J]. Molecular & Cellular Proteomics,2014,13:3270—3285
[66]Mescher M F,Strominger J L. Purification and characterization of a prokaryotic glucoprotein from the cell envelope of Halobacterium salinarium [J]. Journal of Biological Chemistry,1976,251:2005—2014
[67]Aas F E,Vik A,Vedde J,et al. Neisseria gonorrhoeae O-linked pilin glycosylation:functional analyses define both the biosynthetic pathway and glycan structure [J]. Molecular Microbiology,2007,65:607—624
[68]Linton D,Dorrell N,Hitchen P G,et al. Functional analysis of the Campylobacter jejuni N-linked protein glycosylation pathway [J]. Molecular Microbiology,2005,55:1695—1703
[69]Voisin S,Kus J V,Houliston S,et al. Glycosylation of Pseudomonas aeruginosa strain Pa5196 type IV pilins with mycobacterium-like alpha-1,5-linked D-araf oligosaccharides [J]. Journal of Bacteriology,2007,189:151—159
[70]Smedley J G,Jewell E,Roguskie J,et al. Influence of pilin glycosylation on Pseudomonas aeruginosa 1244 pilus function [J]. Infection and Immunity,2005,73:7922—7931
[71]Banerjee A,Ghosh S K. The role of pilin glycan in neisserial pathogenesis [J]. Molecular and Cellular Biochemistry,2003,253:179—190
[72]Kim Y H,Park Y M,Kim S J,et al. The role of Slr1443 in pilus biogenesis in Synechocystis sp PCC 6803:involvement in post-translational modification of pilins [J]. Biochemical and Biophysical Research Communications,2004,315:179—186
[73]Kim Y H,Kim J Y,Kim S Y,et al. Alteration in the glycan pattern of pilin in a nonmotile mutant of Synechocystis sp PCC 6803 [J]. Proteomics,2009,9:1075—1086
[74]Kim Y H,Park K H,Kim S Y,et al. Identification of trimethylation at C-terminal lysine of pilin in the cyanobacterium Synechocystis PCC 6803 [J]. Biochemical and Biophysical Research Communications,2011,404:587—592
[75]Klotz A V,Glazer A N. Gamma-N-Methylasparagine in phycobiliproteins - occurrence,location,and biosynthesis [J]. Journal of Biological Chemistry,1987,262:17350—17355
[76]Swanson R V,Glazer A N. Phycobiliprotein methylation - effect of the Gamma-N-Methylasparagine residue on energy-transfer in phycocyanin and the phycobilisome [J]. Journal of Molecular Biology,1990,214:787—796
[77]Behshad E,Parkin S E,Bollinger J M. Mechanism of cysteine desulfurase Slr0387 from Synechocystis sp PCC 6803:Kinetic analysis of cleavage of the persulfide intermediate by chemical reductants [J]. Biochemistry,2004,43:12220—12226
[78]Campanini B,Schiaretti F,Abbruzzetti S,et al. Sulfur mobilization in cyanobacteria:The catalytic mecahnism of L-cystine C-S lyase(C-DES) from synechocystis [J]. Journal of Biological Chemistry,2006,281:38769—38780
[79]Silman N J,Carr N G,Mann N H. Adp-ribosylation of glutamine-synthetase in the cyanobacterium Synechocystis sp strain Pcc-6803 [J]. Journal of Bacteriology,1995,177:3527—3533
[80]Fagerlund R D,Eaton-Rye J J. The lipoproteins of cyanobacterial photosystem Ⅱ [J]. Journal of Photochemistry and Photobiology B-Biology,2011,104:191—203
[81]McIntosh J A,Donia M S,Nair S K,et al. Enzymatic basis of ribosomal peptide prenylation in cyanobacteria[J]. Journal of the American Chemical Society,2011,133:13698—13705
[82]Gallon J R,Cheng J J,Dougherty L J,et al. A novel covalent modification of nitrogenase in a cyanobacterium [J]. FEBS Letters,2000,468:231—233
[83]Church G M. Proteogenomic mapping as a complementary method to perform genome annotation [J]. Proteomics,2004,4:59—77
[84]Kim M S,Pinto S M,Getnet D,et al. A draft map of the human proteome [J]. Nature,2014,509:575—581
[85]Zhang B,Wang J,Wang X,et al. Proteogenomic characterization of human colon and rectal cancer [J]. Nature,2014,513:382—387
[86]Yang M K,Yang Y H,Chen Z,et al. Proteogenomic analysis and global discovery of posttranslational modifications in prokaryotes [J]. Proceedings of the National Academy of Sciences of the United States of America,2014,111:E5633—5642
[87]Ong S E,Mittler G,Mann M. Identifying and quantifying in vivo methylation sites by heavy methyl SILAC [J]. Nature Methods,2004,1:119—126
[88]Molden R C,Goya J,Khan Z,et al. Stable Isotope Labeling of Phosphoproteins for Large-scale Phosphorylation Rate Determination [J]. Molecular & Cellular Proteomics,2014,13:1106—1118
[89]Chen Y,Colak G,Zhao Y. SILAC-Based Quantification of Sirt1-Responsive Lysine Acetylome [M]. Sirtuins. Humana Press,2013:105—120
[90]Engelsberger W R,Erban A,Kopka J,et al. Metabolic labeling of plant cell cultures with K15NO3as a tool for quantitative analysis of proteins and metabolites [J]. Plant methods,2006,2:14
[91]Huttlin E L,Hegeman A D,Harms A C,et al. Comparison of full versus partial metabolic labeling for quantitative proteomics analysis in Arabidopsis thaliana [J]. Molecular & Cellular Proteomics,2007,6:860—881
[92]Pandhal J,Ow S Y,Wright P C,et al. Comparative proteomics study of salt tolerance between a nonsequenced extremely halotolerant cyanobacterium and its mildly halotolerant relative using in vivo metabo-lic labeling and in vitro isobaric labeling [J]. Journal of Proteome Research,2009,8:818—828
[93]Aryal U K,Stockel J,Welsh E A,et al. Dynamic proteome analysis of Cyanothece sp ATCC 51142 under constant light [J]. Journal of Proteome Research,2012,11:609—619
[94]Aryal U K,Stockel J,Krovvidi R K,et al. Dynamic proteomic profiling of a unicellular cyanobacterium Cyanothece ATCC51142 across light-dark diurnal cycles [J]. Bmc Systems Biology,2011,5:194
[95]Wang F J,Cheng K,Wei X L,et al. A six-plex proteome quantification strategy reveals the dynamics of protein turnover [J]. Scientific Reports,2013,3
[96]Thompson A,Schafer J,Kuhn K,et al. Tandem mass tags:A novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS [J]. Analytical Chemistry,2003,75:1895—1904
[97]Ross P L,Huang Y L N,Marchese J N,et al. Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents [J]. Molecular & Cellular Proteomics,2004,3:1154—1169
[98]Soderblom E J,Philipp M,Thompson J W,et al. Quantitative Label-free phosphoproteomics strategy for multifaceted experimental designs [J]. Analytical Chemistry,2011,83:3758—3764
[99]Sidoli S,Lin S,Xiong L,et al. SWATH analysis for characterization and quantification of histone post-translational modifications [J]. Molecular & Cellular Proteomics,2015,mcp. O114. 046102
[100]Doll S,Burlingame A L. Mass spectrometry-based detection and assignment of protein posttranslational modifications [J]. Acs Chemical Biology,2015,10:63—71
PROGRESS ON PROTEIN POST-TRANSLATIONAL MODIFICATION IN CYANOBACTERIA
YANG Ming-Kun1,LIN Xiao-Huang1,2,MA Yan-Yan1,2,WANG Yan1,2and GE Feng1
(1. Key Laboratory of Algal Biology,Institute of Hydrobiology,Chinese Academy of Sciences,Wuhan 430072,China; 2. University of Chinese Academy of Sciences,Beijing 100049,China)
Post-translational modifications(PTMs) are vital cellular control mechanisms modulating diverse protein properties. Proteomic analysis of post-translational modifications based on the mass spectrometry has been the key research area. Recently,it has been shown that protein post-translational modifications,such as phosphorylation,acetylation,methylation,glycosylation and oxidation,play crucial regulatory role in various pathways of cyanobacteria. In the present article,we reviewed the recent progress on the functional studies of PTMs in cyanobacteria.
Cyanobacteria; Proteomics; Post translational modifications(PTMs)
Q344+.3
A
1000-3207(2016)05-1056-12
10.7541/2016.137
2015-08-28;
2016-01-11
國家自然科學基金(31570829)資助 [Supported by the National Natural Science Foundation of China(31570829)]
楊明坤(1985—),男,湖北襄陽人; 碩士; 主要從事蛋白質翻譯后修飾組學及其功能研究。E-mail:yangmingkun@ihb.ac.cn
葛峰(1971—),男,研究員; 研究方向為功能蛋白質組學。E-mail:gefeng@ihb.ac.cn

