改良CTAB法用于水環境中超微型浮游生物總DNA的提取
賀云彥,李 詩,劉科賽,侯建軍,劉細霞,李運濤
(湖北師范大學 食用野生植物保育與利用湖北省重點實驗室,湖北 黃石 435002)
改良CTAB法用于水環境中超微型浮游生物總DNA的提取
賀云彥,李 詩,劉科賽,侯建軍,劉細霞,李運濤*
(湖北師范大學 食用野生植物保育與利用湖北省重點實驗室,湖北 黃石 435002)
水環境中超微型浮游生物的研究要求有高質量的總DNA。通過對常規的CTAB法進行改良,對提取過程中的幾個關鍵步驟進行優化,包括聯合使用CTAB和SDS,利用5×CTAB去除雜質等,建立一種可以高效提取水環境中超微型浮游生物總DNA的方法,并與常規CTAB法以及蛋白酶K法進行比較。改良后的CTAB法提取的DNA的產量、純度均最高,也最適合后續的實驗研究。
水環境;超微型浮游生物;改良CTAB法;DNA提取
近年來,很多學者致力于水環境中的浮游生物研究,其中超微型浮游生物的研究受到越來越多的的重視[1]。研究超微型浮游生物通常采用分子生物學方法,因而獲得高質量的總DNA就成了后續研究的重要基礎。超微型浮游生物因為粒徑小,水樣需提前過濾,導致樣品起始量少,且樣品細胞含有較多的環境雜質,給超微型浮游生物總DNA的提取帶來了一定困難[2]。目前雖然已有一些水樣及藻類總DNA的提取方法[3] [4],但這些方法不太適用于超微型浮游生物的研究,提取DNA的效果不佳。因此迫切需要一種更簡便、高效、經濟的提取方法。
1 材料與方法
1.1 材料
先用篩絹過濾除去水樣中的泥沙等大型顆粒雜物,再用5μm Isopore濾膜進行初次過濾,然后用GF/F濾膜將初次過濾的水樣進行二次過濾。
1.2 主要儀器與試劑
5430R臺式高速冷凍離心機(Eppendorf ),BioPhotometer D30 核酸蛋白測定儀(Eppendorf )。CTAB緩沖液(pH8.0,100 mmol/L Tris-HCl ,20mmol/L EDTA,1.4M NaCl,5%CTAB,0.1%PVP,2%β-巰基乙醇),裂解緩沖液(pH8.0,1.0mol/L NaCl,50mmol/L EDTA,50 mmol/L Tris,1% PVP),5×CTAB (pH 8.0,5% CTAB,1.0mol/L NaCl,50mmol/L EDTA,250 mmol/L Tris )。
1.3 DNA提取方法
1.3.1 方法1(CTAB法)
將膜樣品剪碎成粉末狀,加入65℃預熱的800μL CTAB緩沖液,于65℃水浴30min。水浴結束后加入等體積的酚:氯仿:異戊醇(25:24:1),4℃ 6000r/min離心10min。取上清液,加入等體積氯仿:異戊醇(24:1),4℃ 6000r/min離心10min。取上清,加入1/10體積3M醋酸鈉,混勻,加2倍體積-20℃預冷的無水乙醇,于-20℃放置1h到2h。12000r/min離心10min使DNA沉淀,去上清。加入70%乙醇1ml,4℃12000g離心5min,去上清。干燥直至無乙醇殘留,加入20μL TE。
1.3.2 方法2(改良CTAB法)
將膜樣品剪碎成粉末狀,加入65℃預熱的800μL 裂解緩沖液、2%SDS,65℃水浴30min。然后12000g離心5min,取上清,加1/5倍體積的65℃預熱的5×CTAB,混勻后65℃繼續水浴10min。水浴結束后加入等體積的酚:氯仿:異戊醇(25:24:1),4℃ 8000g離心10min。取上清,加入等體積氯仿:異戊醇(24:1),4℃ 8000g離心10min。取上清,加入1/10體積3M醋酸鈉,混勻,加2倍體積-20℃預冷的無水乙醇,于-20℃放置1h到2h。12000g離心10min,去上清。加入1ml 70%乙醇,4℃ 12000g離心5min,去上清,干燥直至無乙醇殘留,加入20μL TE。
1.3.3 方法3(蛋白酶K法)
將膜樣品剪碎成粉末狀,加入65℃預熱的600μL 裂解緩沖液,加入33.5μL 10㎎/mL的蛋白酶K,先37℃水浴50min,再55℃水浴20分鐘。4℃,8000g離心10min后,取上清,加入等體積酚:氯仿:異戊醇(25:24:1)。4℃,10000g離心5分鐘。取上清,加入等體積的氯仿:異戊醇(24:1),8000g離心10min。取上清,加入等體積的異丙醇和0.4倍體積7.5mol/L的醋酸銨,室溫靜置10min后,4℃,10000g離心10min,棄上清。加入1mL 70%的乙醇洗滌,4℃,10000g離心5min。棄上清,干燥直至無乙醇殘留,加入20μL TE。
1.4 DNA提取效率及質量評價
1.4.1 電泳檢測
提取的總DNA用0.8%的瓊脂糖凝膠電泳檢測,觀察DNA產物的完整性和片段分布。
1.4.2 DNA的純度和濃度
通過測定樣品在260和280 nm下的吸光度值,并分別計算A260/280 比值來評價DNA的純度;DNA濃度按下列公式計算:DNA濃度(μg / μl): OD260×稀釋倍數×50/1000。
1.4.3 酶切及PCR擴增
選擇真核18S rRNA基因進行PCR擴增,觀察PCR擴增結果以評價提取的DNA質量。引物序列為[5]:
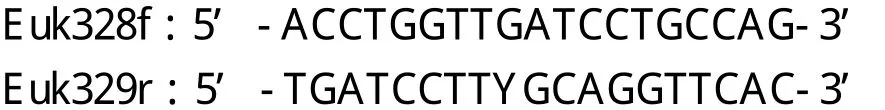
PCR擴增采用25μl體系,PCR條件為:94℃預變性4 min,接著進人35個循環(94℃1 min,55℃1 min, 72℃2 min ),最后72℃延伸8 min。目的產物大小約為1800bp,PCR產物經1%的瓊脂糖凝膠電泳檢測。
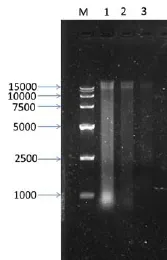
圖1 提取的總DNA(M:marker;泳道1、2、3:方法1、2、3提取的DNA。)
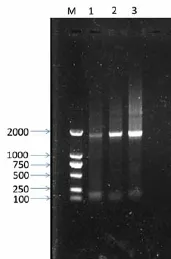
圖2 提取的DNA PCR凝膠電泳結果(M:marker;泳道1、2、3:以方法1、2、3提取的DNA為模板的PCR產物。)
2 結果分析
2.1 DNA產率
三種DNA提取方法所用細胞裂解方法和后續步驟各有不同, 不同的DNA提取方法產率不同。從DNA電泳條帶亮度可以大致判斷DNA產率的高低。本文所用的3種方法中,方法1提取的DNA電泳條帶最亮,其次為方法2和方法3(圖1)。但方法1提取的DNA拖帶嚴重,提示其質量較低。通過計算,三種方法提取的總DNA的濃度分別為0.545、0.972、0.465μg / μl,方法2的產量最高。
2.2 DNA質量
從瓊脂糖凝膠電泳檢測結果可以看出(圖1),3種方法都能夠提取相對完整的DNA。但綜合來看,方法2提取的DNA質量較好。根據經驗,高純度DNA A260/280 比值應在1.8-2.0之間。本研究不同方法提取的DNA在純度上有一定差異(表1)。從A 260/280檢測值可以看出,方法2提取的DNA質量較好。其中,方法1和3的比值稍高,提示可能有RNA殘留。
分別以三種方法提取的總DNA為模板,使用18s rDNA特異引物進行擴增。從擴增結果可以看出(圖2),方法2提取的DNA的擴增效果最好,PCR產物亮度很高,且拖尾很少;而以方法1提取的DNA為模板的PCR產物雖然亮度更高,但有明顯拖帶。故綜合考慮,方法2提取的總DNA質量最高,更適合后續的實驗。
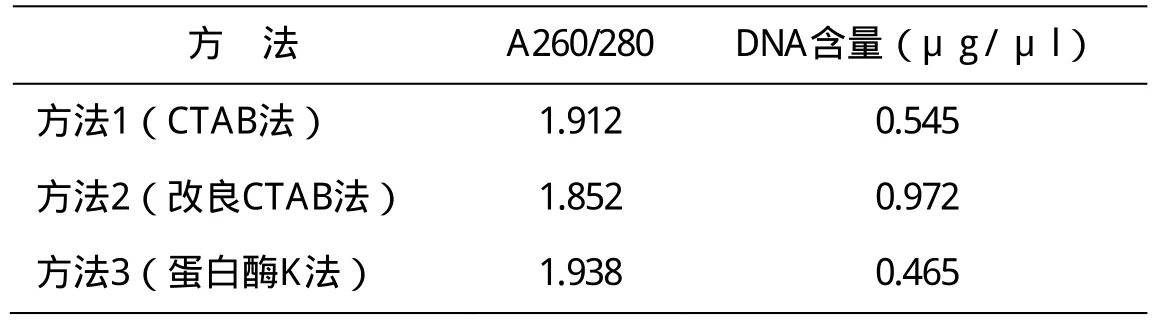
表1 提取的DNA的濃度、純度
3 討論
本文比較了三種不同的超微型浮游生物總DNA提取方法。常規CTAB法提取的總DNA條帶最亮,但其產量不如改良CTAB法,說明常規CTAB法提取的DNA雜質較多,這從其PCR產物拖帶嚴重也能得到證明。針對環境樣品的特點,本研究對常規CTAB法進行了改良,在裂解步驟中,加入了SDS,以增強裂解效果;后續使用5×CTAB以利于雜質的去除[6]。從結果看,改良后的CTAB法提取的DNA產量較高,純度也很高,以該方法提取的DNA為模板的PCR產物條帶明亮,只有輕微拖帶,說明提取的DNA適合后續的實驗研究。而蛋白酶K法提取的DNA,產量、純度均最低,以該方法提取的DNA為模板的PCR產物條帶最暗。綜合得率、純度、對后續實驗的適合度,改良CTAB法更適合于提取現場水環境中的超微型浮游生物總DNA。
A260/280 比值可以作為DNA純度的參考,該比值大于2.0時, 說明DNA樣品中RNA的含量較高;比值小于1.8時,DNA 樣品中可能存在蛋白質污染[7],三種方法提取的DNAA260/280 比值均大于1.8小于2.0,說明三種方法提取DNA的蛋白質污染是主要問題。因為提取時沒有使用RNA酶,故提取的DNA中均有RNA殘留。本研究還探索了裂解液與膜樣品的比例,發現800μL的裂解液提取1/3的膜樣品(膜直徑為3cm)效果比較好。總之,通過對常規CTAB的改良,獲得了高質量高產量的DNA樣品,在檢驗實驗中發現提取的DNA能滿足后續實驗的要求,可用于大量提取水環境中的超微型浮游生物總DNA,為水環境中超微型浮游生物的研究奠定了基礎。
[1] Simon M, Jardillier L, Deschamps P, et al. Complex communities of small protists and unexpected occurrence of typical marine lineages in shallow freshwater systems[J]. Environ Microbiol, 2015, 17: 3610-3627.
[2] Porteous LA, Amstrong JL. A simple mini-method to extract DNA directly from soil for use with polymerase chain reaction amplification[J].Curr Microbial, 1993, 27: 115-118.
[3] 賴樹錦,郭佳,陶新園,等. 四種水體微生物總DNA 提取方法的比較[J].湖北農業科學,2014,53:2440-2442.
[4] 包慧,楊忠委,王華然,等. 3種真核藻類DNA提取方法比較[J].環境與鍵康雜志,2012,29:451-453.
[5] Romari K, Vaulot D.Composition and temporal variability of picoeukaryote communities at a coastal site of the English Channel from 18S rDNA sequences[J].Limnol Oceanogr, 2004, 49: 784-798.
[6] 孫璐宏, 魯周民, 張麗.植物基因組DNA提取與純化研究進展[J].西北林學院學報,2010,25:102-106.
[7] 李金璐,王碩,于婧,等.一種改良的植物DNA提取方法[J].植物學報,2013, 48:72-78.
A Modified CTAB Protocol for DNA Extraction of Picoplankton in Water Environment
HE Yun-yan, LI Shi, LIU Ke-sai, HOU Jian-jun, LIU Xi-xia, LI Yun-tao
(Hubei Key Laboratory of Edible Wild Plants Conservation and Utilization, Hubei Normal University, Huangshi Hubei 435002, China)
The research of picoplankton in water environment requires high quality of total DNA. Through the modification of conventional CTAB method and the optimization of the several key steps in the process of extraction, which included the combining of CTAB and SDS, the removal of impurities by 5× CTAB etc, the method for efficiently extracting total DNA of picoplankton in water environment was established. Furthermore the conventional CTAB method and proteinase K method were compared with modified CTAB method. The yield and purity of DNA extracted by the modified CTAB method were the highest, and it was the most suitable for subsequent experiments.
water environment; picoplankton; modified CTAB method; DNA extraction
Q948.8
A
2095-414X(2016)03-0068-03
李運濤(1979-),男,副教授,研究方向:水域生態學.
國家自然科學基金項目(41171045),湖北師范大學研究生創新科研基金項目(2015024),國家級大學生創新創業訓練計劃項目(201510513021),湖北師范大學優秀創新團隊項目(T201504).

