印度尼西亞卡利安達島近岸熱泉微生物和氫酶基因的多樣性
何培青, Dewi Seswita Zilda, 李江, 張學雷, 崔菁菁, 白亞之, Gintung Patantis, Ekowati Chasanah
(1.國家海洋局第一海洋研究所 海洋生態研究中心, 山東 青島 266061; 2. 海洋與漁業局 海洋與漁業研究與發展司 海洋和漁業產品加工和生物技術研究中心,印度尼西亞 雅加達 40115; 3. 國家海洋局第一海洋研究所 海洋沉積與環境地質國家海洋局重點實驗室,山東 青島 266061)
印度尼西亞卡利安達島近岸熱泉微生物和氫酶基因的多樣性
何培青1, Dewi Seswita Zilda2, 李江1, 張學雷1, 崔菁菁3, 白亞之3, Gintung Patantis2, Ekowati Chasanah2
(1.國家海洋局第一海洋研究所 海洋生態研究中心, 山東 青島 266061; 2. 海洋與漁業局 海洋與漁業研究與發展司 海洋和漁業產品加工和生物技術研究中心,印度尼西亞 雅加達 40115; 3. 國家海洋局第一海洋研究所 海洋沉積與環境地質國家海洋局重點實驗室,山東 青島 266061)
摘要:研究對2011年7月采自印度尼西亞卡利安達島近岸熱泉的樣品,進行了細菌和古菌16S rRNA基因和細菌氫酶基因克隆文庫的構建、序列測定和多樣性分析。結果表明:熱泉系統的細菌和古菌由16個門組成,Proteobacteria為熱液流體和沉積物的優勢類群,豐度分別為59.5%和73.3%,Cyanobacteria為菌苔的優勢類群(豐度,56.5%);具有氨氧化作用的Crenarchaeota為古菌的優勢類群。熱泉系統中,70%以上的種類與海洋細菌的親緣關系最近,說明該系統具有海洋特性;約20%的種類與陸地熱泉中嗜熱或中等嗜熱細菌的親緣關系最近,這些種類可能來源于高溫地層內部,并參與了鐵氧化還原、氫氧化、硫氧化和硝酸還原等過程。熱泉系統中,NAD(P)-關聯的雙向NiFe-氫酶基因和FeFe-氫酶基因的組成分布也受到溫度和鹽度分布變化的影響。研究為揭示淺海熱液系統提供理論參考。
關鍵詞:印度尼西亞卡利安達島;近岸熱泉;16S rRNA;氫酶基因
1引言
深海熱液系統自20世紀70年代首次發現以來,一直是國際關注的焦點,而淺海熱液系統及其微生物的研究在近期才得到重視。淺海熱液系統一般位于近海,水深在0~200 m之間,在地形上屬于深海熱液系統和陸地熱泉系統的過渡,陸地和海水中的流體及物質都會對熱液活動產生影響。淺海熱液系統常位于火山海面下翼部和海底火山頂部,火山活動為其提供熱源并驅動熱液循環。熱液流體來源于大氣降水、海水、原生水和巖漿水,其不同的組成比例制約著熱液系統的溫度、鹽度、酸堿度、礦物等地球化學特征[1—3]。由于水深較淺,熱液流體還會同時影響到底部至表面水層,從而影響到人類活動。由于可以接受到光照,微生物的初級生產不但可以通過化能自養途徑還可以通過光能營養途徑[1—3]。目前,國際上對微生物的研究主要集中在高鹽度淺海熱液系統[4—9],其多樣性豐富,在生物礦化、碳、氮、硫等元素循環中扮演重要的角色;其組成和分布還受到地理位置、溫度、pH和流體組成等物理化學因子的影響。大氣降水為熱液流體主要來源的低鹽度淺海熱液系統中,微生物還經歷了鹽度的變化,而目前對其研究較少[10]。
印度洋卡利安達島位于印度尼西亞,是受到喀拉喀托火山影響的區域之一,東海岸分布著一組近岸熱泉(5°44′46″N, 105°35′12″E),距離陸地數十米。泉眼以單個或成雙分布于相距數米的礁石上,在高潮時沒于海水中,低潮露出海面,熱泉池直徑約1 m,池外周可見綠色菌苔。泉眼水溫在58.5~68.5℃之間,鹽度接近于淡水,氣泡伴隨熱液流體溢出。本研究選取其中一個熱泉,通過PCR擴增熱液流體、熱泉池沉積物和菌苔的細菌和古菌16S rRNA基因、NAD(P)-關聯的雙向NiFe-氫酶基因和FeFe-氫酶基因的部分序列并構建克隆文庫,進一步測定基因序列并分析其多樣性。研究為揭示淺海熱液系統微生物的生態功能及其與環境的相互關系提供理論參考。
2材料與方法
2.1樣品采集
2011年7月,在印度尼西亞卡利安達島東海沿岸,在低潮期采集一個熱泉的熱液流體、表層沉積物和表層菌苔樣品,于冰上低溫運輸至實驗室。1 L熱液流體經醋酸纖維濾膜(0.22 μm)過濾后,與沉積物和菌苔于-80℃保存,用于DNA提取。現場泉眼水溫為65℃,pH為6.0~6.5,鹽度約為4,周圍海水鹽度約為30。沉積物中常量金屬元素、總有機碳(TOC)、總氮(TN)和總碳(TC)的含量由國家海洋局第一海洋研究所海洋地質綜合實驗室完成測試。
2.2基因擴增和基因文庫的構建
稱取沉積物和菌苔樣品各0.5 g,取1 L 熱液流體的過濾菌膜,采用FastDNA?土壤DNA提取試劑盒(MPbio公司,美國)提取基因組。細菌16S rRNA基因引物采用Bac27F/Uni1492R[11],古菌16S rRNA基因引物采用Arch20F/Arch958R[12],NAD(P)-關聯的雙向NiFe-氫酶基因引物采用HoxH-F/HoxH-R[13];FeFe-氫酶基因引物采用FeFe-272F/FeFe-427R[14]。PCR反應采用TransStartTMTaq DNA聚合酶(全式金,北京),PCR反應體系(50 μL)包括:2 μL DNA(約10 ng),各1 μL 引物(0.2 μmol/L 終濃度),5 μL 緩沖液,4 μL dNTP,1 μL DNA 聚合酶(2.5 U),36 μL H2O。細菌16S rRNA基因擴增條件:94℃ 5 min;94℃ 10 s,55℃退火10 s,72℃延伸1.5 min,30個循環;72℃終延伸10 min。古菌16S rRNA基因、NAD(P)-關聯的雙向NiFe-氫酶基因和FeFe-氫酶基因的退火溫度分別為55℃、48℃和52℃,延伸時間為1 min,40個循環,其他條件同細菌16S rRNA基因。
分別取2個重復的PCR產物(100 μL),經1%的瓊脂糖電泳后,切下與目的基因分子量大小相近的條帶,采用瓊脂糖回收試劑盒(天根,北京)進行純化,pBS-T載體連接后,轉化到E.coliDH5α感受態細胞(天根,北京)構建克隆文庫,挑取單克隆在LB液體培養基(氨芐濃度50 μg/mL)中,于37℃過夜培養,并采用M13通用引物進行陽性驗證后,由上海桑尼公司完成測序。
2.3基因序列的生物信息學分析
分別對細菌的16S rRNA基因擴增片段(正向 800 bp)和古菌的16S rRNA基因擴增片段采用CodonCode Aligner 軟件 (CodonCode Corporation,Dedham,Massachusetts,USA)[15]進行比對,相似度高于97%的序列作為一個操作分類單元(OTU)。多樣性指數采用Primer 5軟件[16]計算。NAD(P)-關聯的雙向NiFe-氫酶和FeFe-氫酶的基因序列去除載體和引物序列,并翻譯成氨基酸序列后,采用DOTUR軟件[17],在大于等于99%相似性水平上進行操作分類單元(OTUs)分析,并計算多樣性指數。覆蓋率計算公式為:C=[1-(n/N)]×100%,其中n為總的OTU數,N為總的序列數。采用BioEdit[18]軟件中的Clustal W[19]分別對16S rRNA基因序列和氫酶氨基酸序列與GenBank中近緣序列進行多重比對,并采用MEGA4.0軟件[20]的相鄰連接法(Neighbor-Joining)構建系統發育樹,自舉值設為1 000。
2.4基因序列
將獲得的基因序列提交到GenBank數據庫,細菌16S rRNA基因序列號為:KT239166-KT239341;古菌16S rRNA基因序列號為:KT225015-KT225020;NAD(P)-關聯的雙向NiFe-氫酶基因序列號為:KT225007-KT2250014;FeFe-氫酶基因序列號為:KT224971-KT225006。
3結果與分析
3.1沉積物的主要化學元素組成
熱泉沉積物的化學元素組成如表1所示,其中Fe和P含量高于大陸地殼,而Al、K、Mg和Ti含量低于大陸地殼數據。
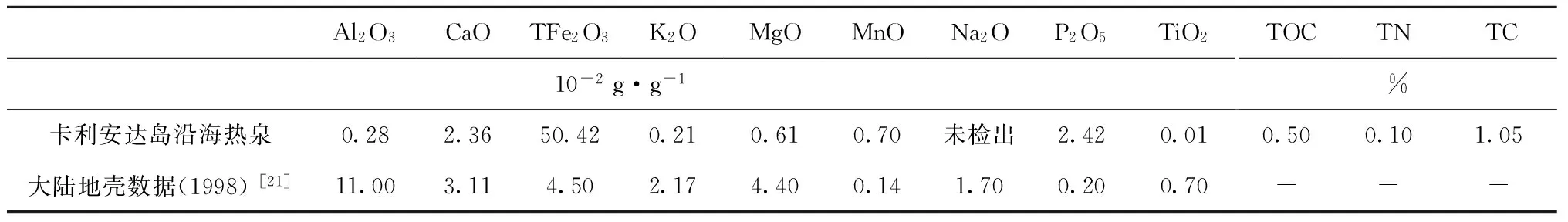
表1 印度尼西亞卡利安達島近岸熱泉沉積物化學元素組成
注:“-”表示未提供數據。
3.2細菌的α-多樣性
共獲得641個含有細菌16S rRNA基因片段的克隆(表2)。熱液流體、沉積物和菌苔分別獲得90、100和52個OTUs。在沉積物和熱液流體中,細菌16S rRNA基因的香農-韋弗多樣性指數和辛普森多樣性指數差別不大,且均高于菌苔。不同生態類型中的細菌均勻度和物種豐富度的變化趨勢與多樣性指數一致。
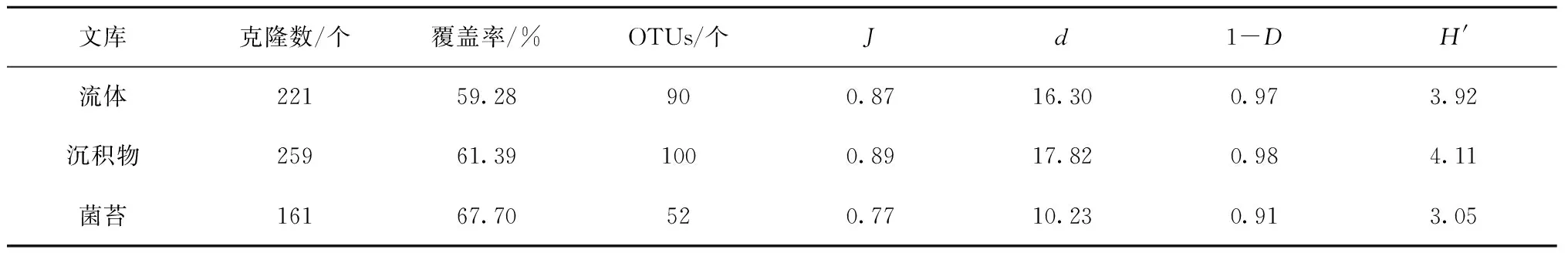
表2 印度尼西亞卡利安達島近岸熱泉細菌16S rRNA基因的α-多樣性
注:OTUs:操作分類單元;J:Pielou 均一度指數;d:物種豐富度;1-D:辛普森多樣性指數;H′:香農-韋弗多樣性指數。
3.3細菌組成
研究共獲得14個門(表3)。在熱液流體和沉積物中,Proteobacteria的豐度最高,分別為59.5%和73.3%,其中Gammaproteobacteria的豐度分別為43.0%和23.2%。菌苔中Cyanobacteria的豐度最高(56.5%),其次為Bacteroidetes(19.9%)和Proteobacteria(13.0%)。菌苔中Bacteroidetes、Acidobacteria和Chloroflexi的豐度高于熱液流體和沉積物。沉積物中Deferribacteres的豐度高于熱液流體和菌苔。
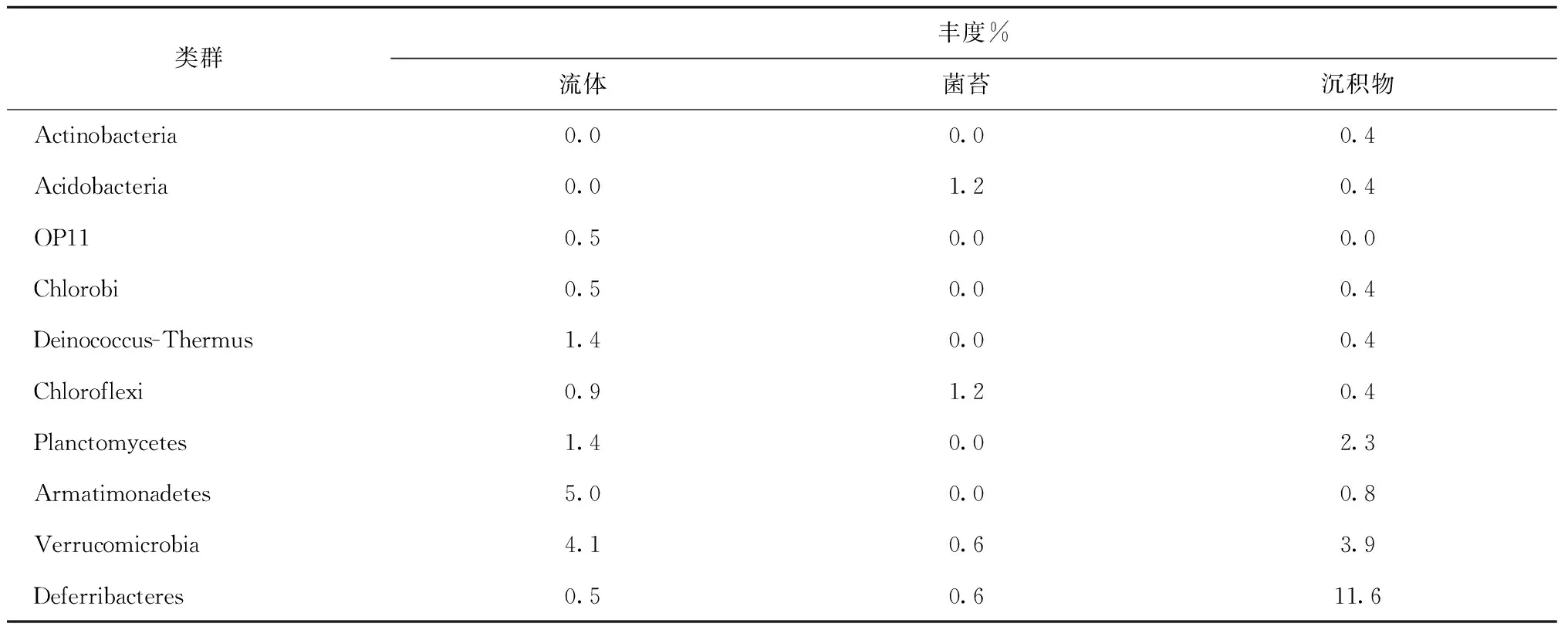
表3 印度尼西亞卡利安達島近岸熱泉的細菌組成
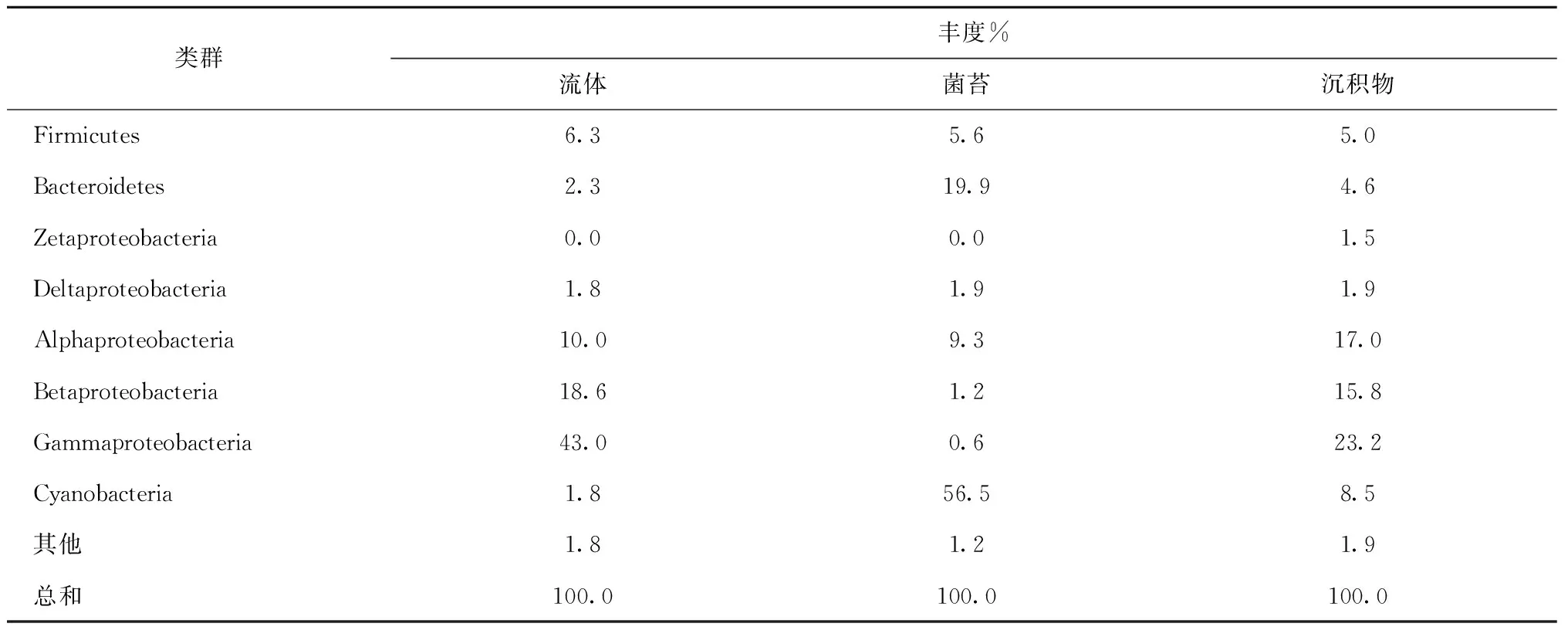
續表3
(1) Gammaproteobacteria
獲得Gammaproteobacteria的35個OTUs。熱液流體中豐度最高的3個OTUs(總豐度,18.0%)與海洋細菌Acinetobacter的相似度高于97%。沉積物中,豐度最高1個OTU(豐度,5.4%)與反硝化海洋細菌Pseudomonasstutzeri相似度為99.5%[22]。熱液流體中獲得的1個克隆,與嗜熱細菌Thermomonashydrothermalis親緣關系最近(96.2%),還獲得2個克隆,與深層地熱環境克隆的相似度高于97%。
(2) Betaproteobacteria
獲得Betaproteobacteria的13個OTUs,其中5個OTUs(約占Betaproteobacteria總豐度的50.0%)與熱泉環境中硫氧化、鐵氧化、氫氧化和甲基氧化細菌的親緣關系最近。熱液流體中,豐度最高的1個OTU(豐度,9.5%)與陸地熱泉化能自養的鐵氧化細菌Sideroxydanslithotrophicus的相似度為94.6%[23],該OTU在沉積物的豐度為3.6%。1個OTU(豐度,0.9%)與陸地熱泉Hydrogenophilushirschii的相似度為97.6%。沉積物中,1個OTU(豐度,0.4%)與熱泉中耐熱的甲基營養細菌Methyloversatilisthermotolerans的相似度為98.5%[24],2個OTUs(總豐度,0.3%)與高溫地下水層化能自養的嗜熱硫氧化細菌Thiobactersubterraneus的相似度高于97%[25]。
(3) Alphaproteobacteria
獲得Alphaproteobacteria的37個OTUs,其中熱液流體中豐度最高的1個OTU(22.0%)與海洋細菌Brevundimonashalotolerans的相似度為99.7%,沉積物中豐度較高的11個OTUs與不產氧光合細菌Rhodospirillales目、固氮細菌Rhizobiales和Brevundimonas屬的親緣關系最近。熱液流體中還獲得1個OTU,與陸地熱泉中等嗜熱的不產氧光合細菌Albidovulumxiamenense的相似度為99.0%。
(4) Deltaproteobacteria
獲得Deltaproteobacteria的10個OTUs(12個克隆)。熱液流體中,1個OTU(1個克隆)與熱泉環境克隆的相似度為99.7%;1個OTU(3個克隆)與熱泉嗜熱氫氧化細菌Hydrogenophilusislandicus[26]的相似度為97.0%,沉積物中也獲得該OTU(1個克隆)。沉積物中,1個OTU(1個克隆)與深海熱液口嗜熱鐵還原細菌Deferrisomacamini的相似度為98.5%,1個OTU(2個克隆)與堿性熱泉環境克隆的相似度為97.0%。
(5) Cyanobacteria
Cyanobacteria是菌苔的優勢類群(豐度,56.5%),其中的28.6%與潮間帶Lyngbyaaestuarii的相似度為99.0%,38.5%與淡水環境中Stanieriacyanosphaera相似度約為91.5%。
(6) Bacteroidetes
獲得Bacteroidetes的21個OTUs,主要包括Flavobacteriia、Bacteroidia和Sphingobacteria3個目。
(7) Firmicutes
獲得Firmicutes的24個OTUs,菌苔和熱液流體中各含有1個OTU(共5個克隆),與深海熱液區中等嗜熱菌Exiguobacteriumprofundum相似度為99.5%。沉積物中有1個OTU(1個克隆)與中等嗜熱Bacillusthermoamylovorans的相似度為99.0%,1個OTU(1個克隆)與嗜熱Desulfotomaculumthermobenzoicum的相似度為91.4%。
(8) Deferribacteres
Deferribacteres具有鐵和硝酸鹽還原作用[27],研究獲得4個OTUs(共32個克隆),均與陸地熱泉嗜熱Calditerrivibrionitroreducens[28]親緣關系最近。沉積物中獲得3個OTUs,其中1個(豐度,10.8%)與Calditerrivibrionitroreducens的相似度為97.8%。
稀有門(豐度小于等于1.0%)中,從熱液流體和沉積物中各獲得Chlorobi的1個克隆,均與陸地熱泉中等嗜熱的綠硫細菌Ignavibacteriumalbum的親緣關系最近。獲得Chloroflexi的2個OTUs(共5個克隆),分別與高溫深部蓄水層嗜熱細菌Caldilineatarbellica和淺海熱液區鐵和硝酸還原細菌Ardenticatenamaritima的親緣關系最近。熱液流體中獲得Deinococcus-Thermus的5個克隆,其中1個OTU(2個克隆)與微嗜熱的Meiothermuscerbereus的相似度為97.6%。菌苔中獲得Acidobacteria的3個克隆,其中1個OTU(2個克隆)與熱泉Thermoanaerobaculumaquaticus相似度達92.3%。主要OTUs的系統進化樹如圖1所示。
3.4古菌組成
古菌僅在沉積物和菌苔中分別獲得3個OTUs和5個OTUs。這些OTUs絕大多數與陸地熱泉中Crenarchaeota氨氧化細菌的親緣關系最近(相似度,92.3%~99.1%)。菌苔中還獲得2個OTUs(豐度,6.1%),與淺海熱液中參與砷和鐵循環的Thaumarchaeota的親緣關系最近(相似度,91.0%和99.2%)。AM3和AM4與CandidatusNitrosocaldusyellowstonii親緣關系最近。AM4在菌苔(豐度,50.0%)和沉積物中(豐度,60.5%)均為優勢類群,AM3在菌苔中的豐度(41.3%)遠高于在沉積物中的(4.0%)。AS36與CandidatusNitrososphaeragargensis的親緣關系最近,為沉積物特有類群(豐度,36.0%)。古菌的系統進化樹分析,如圖2所示。
3.5氫酶基因的α-多樣性
如表4所示,NAD(P)-關聯的雙向NiFe-氫酶基因和FeFe-氫酶基因僅在沉積物和菌苔中獲得。FeFe-氫酶基因編碼的氨基酸序列的多樣性高于NiFe-氫酶的。菌苔中這兩種氫酶的氨基酸序列的多樣性均高于沉積物的。
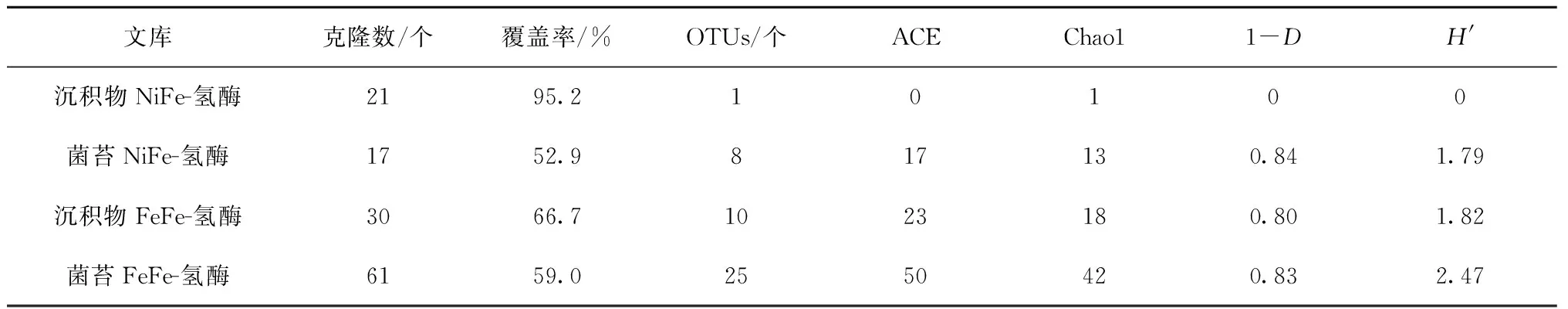
表4 印度尼西亞卡利安達島沿海熱泉NAD(P)-關聯的NiFe-氫酶基因和FeFe-氫酶基因編碼的氨基酸序列的α-多樣性
注:OTUs:操作分類單元;ACE:ACE豐富度指數;Chao1:Chao1豐富度指數;1-D:辛普森多樣性指數;H′:香農-韋弗多樣性指數。
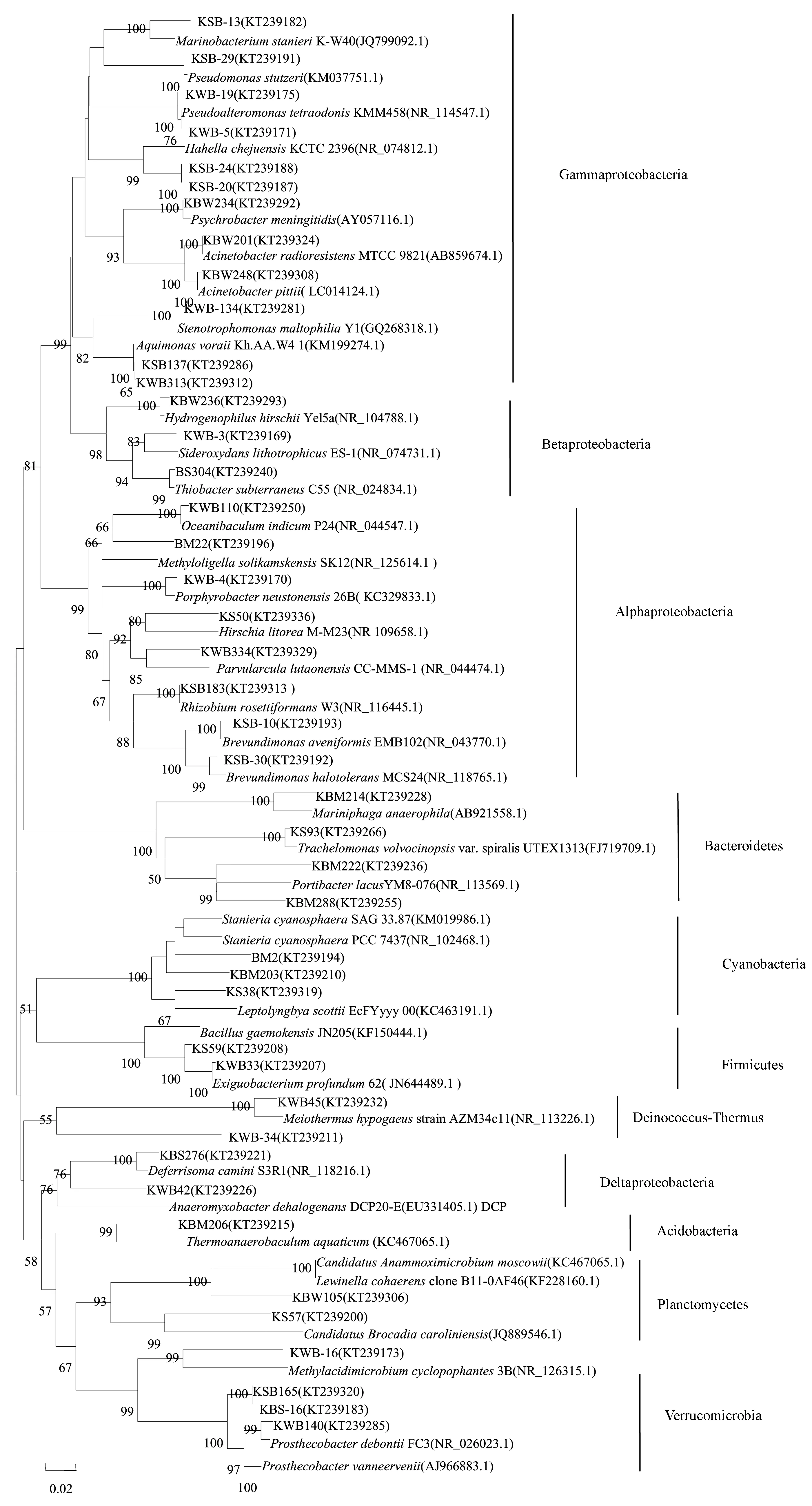
圖1 印度尼西亞卡利安達島近岸熱泉細菌16S rRNA基因序列的系統進化樹Fig.1 Phylogenetic tree of Bacterial 16S rRNA genes sequences from a coastal hot spring of Kalianda, Indonesia

圖2 印度尼西亞卡利安達島近岸熱泉古菌16S rRNA基因序列的系統進化樹Fig.2 Phylogenetic tree of archaea 16S rRNA gene sequences from a coastal hot spring of Kalianda, Indonesia
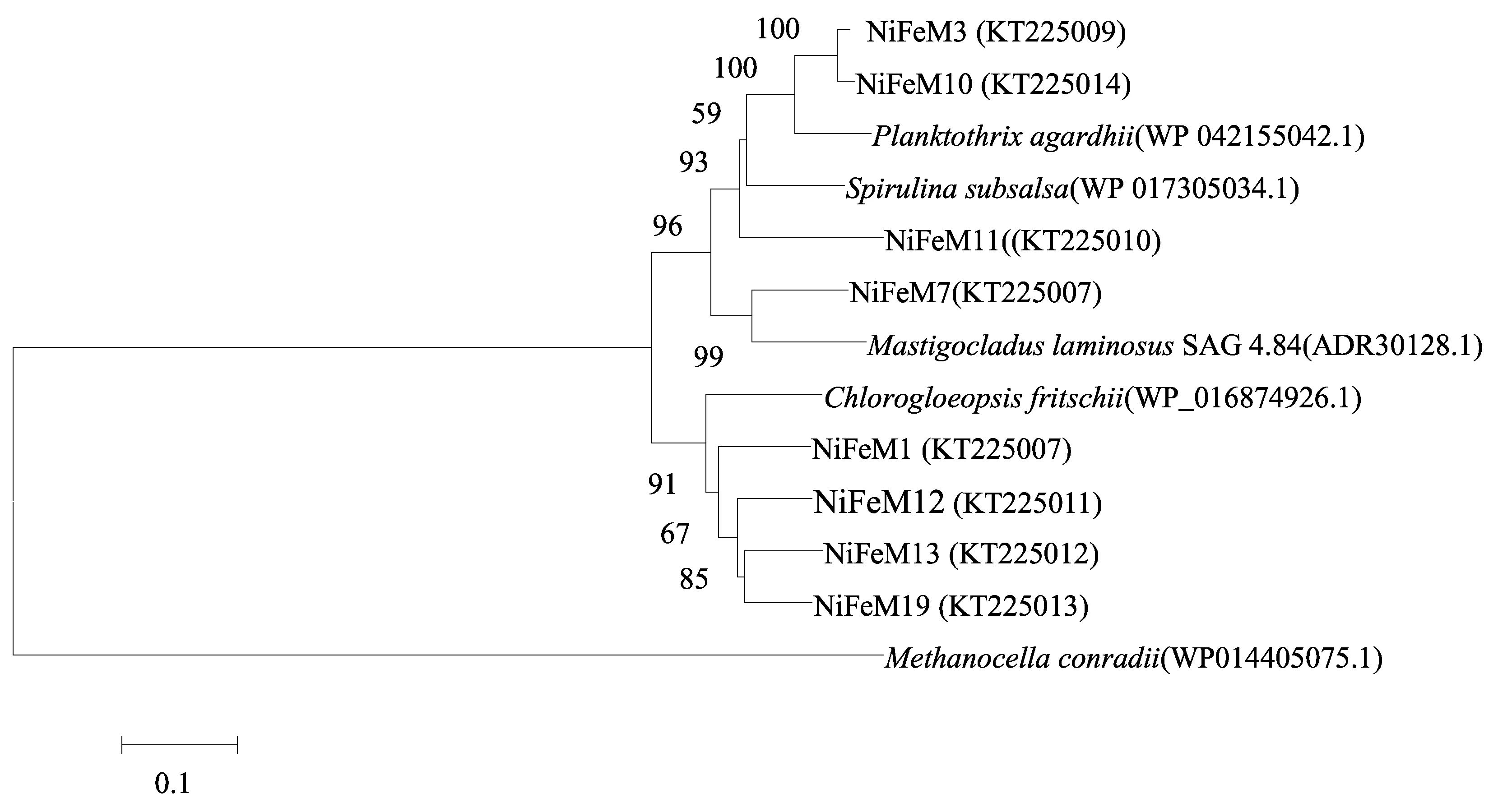
圖3 印度尼西亞卡利安達島近岸熱泉NAD(P)-關聯的雙向NiFe-氫酶基因編碼的氨基酸序列的系統進化樹Fig. 3 Phylogenetic tree of deduced amino acid sequences of NAD(P)-linked NiFe-hydrogease genes from a coastal hot spring of Kalianda, Indonesia
3.6NAD(P)-關聯的雙向NiFe-氫酶基因的組成
NAD(P)-關聯的雙向NiFe-氫酶基因編碼的氨基酸序列包括8個OTUs,其中沉積物中僅含有1個OTU,且與菌苔中的NiFeM12的相似度為99.2%。NiFe-氫酶均屬于Cyanobacteria(相似度,80.3%~90.2%),其中5個OTUs(總豐度,77.5%)與Stigonematales的相似度最高;3個OTUs(總豐度,22.5%)與Oscillatoriophycideae的相似度最高。NiFe-氫酶氨基酸序列的系統進化樹如圖3所示。
3.7FeFe-氫酶基因的組成
沉積物中,FeFe-氫酶基因編碼的氨基酸序列包括4個門,10個OTUs。其中Firmicutes的1個OTU,以及所有的Spirochaetes(3個OTUs),Ignavibacteriae(2個OTUs)和Thermotogae(1個OTUs)的OTUs均與熱泉、油田等高溫環境中FeFe-氫酶氨基酸序列的相似度最高。FeS10(豐度,40.0%)與熱泉中FervidobacteriumpennivoransFeFe-氫酶氨基酸序列的相似度達97.0%,FeS3(豐度,20.1%)與嗜熱SpirochaetathermophilaFeFe-氫酶氨基酸序列相似度達71.2%。這說明熱泉中沉積物的產氫菌群具有嗜熱特性。
菌苔FeFe-氫酶氨基酸序列與沉積物的完全不同,包括25個OTUs,屬于Caldithrix、Spirochaetes、Bacteroidetes、Ignavibacteriae、Firmicute、Alphaproteobacteria和Gammaproteobacteria。其中Bacteroidetes的FeFe-氫酶豐度最高,包括9個OTUs,與海洋或淡水環境的Draconibacteriumorientale(相似度,85.2%~91.5%),Marinilabiliasalmonicolor(相似度,71.0%)和Spirochaetasmaragdinae(相似度,72.0%)的親緣關系最近。還有一些FeFe-氫酶氨基酸序列與熱泉等高溫環境的Caldithrix(豐度2.9%)、Ignavibacteriae(豐度11.6%)和Thioclavapacifica(豐度1.5%)的氫酶氨基酸序列相似度最高。FeFe-氫酶氨基酸序列的系統進化樹如圖4所示。
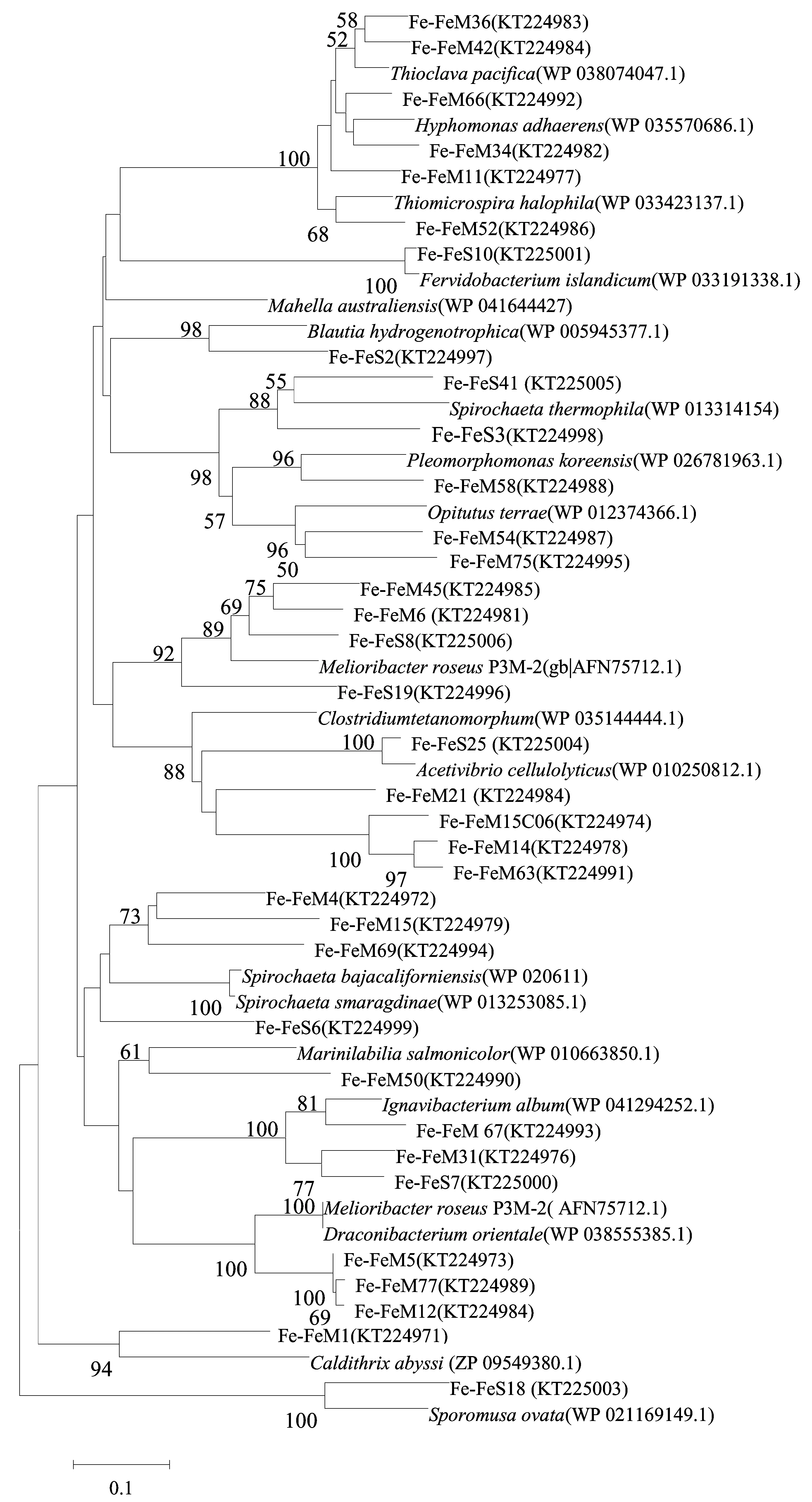
圖4 印度尼西亞卡利安達島近岸熱泉FeFe-氫酶基因編碼的氨基酸序列的系統進化樹Fig.4 Phylogenetic tree of deduced amino acid sequences of FeFe-hydrogease from a coastal hot spring of Kalianda, Indonesia
4討論
印度洋卡利安達島近岸熱泉發育多樣性豐富的微生物,與已知淺海熱液系統硫氧化還原細菌占優勢不同[4-9],該系統在鐵元素和氮元素循環中具有重要作用。如沉積物中與鐵還原相關的嗜熱細菌的豐度高于10%,這可能與沉積物中鐵含量高(約為大陸地殼含量的12倍)有關。古菌包括6個種類,基本由具有氨氧化作用的Crenarchaeota組成。古菌的這種組成特征與阿爾比斯山地下熱泉古菌的組成相似[29]。根據親緣關系最近的種類的生態功能還可以推斷,該系統在氫氧化、甲基營養、硫氧化等生態過程也具有重要作用。與16s rRNA結果一致,熱泉FeFe-氫酶氨基酸序列也與Firmicutes、Bacteroidetes、Alphaproteobacteria和Gammaproteobacteria相關;但占優勢的FeFe-氫酶序列與嗜熱的Thermotogae、Spirochaetes和Ignavibacteriae相關,說明熱泉中通過FeFe-氫酶產氫的菌群為豐度較小的類群。這些產氫菌群可能為鐵還原和硝酸還原等過程提供電子供體和能量。熱泉系統中的雙向NiFe-氫酶均屬于Cyanobacteria,這些Cyanobacteria種類在白天進行光合作用時,通過雙向NiFe-氫酶吸收氫氣并釋放過剩的電子[30—31],在夜晚則通過該酶的可逆反應厭氧產氫,從而使菌體保持穩定的氧化還原態[32]。
溫度和鹽度是熱泉系統微生物組成和分布的控制因素。研究中,豐度高的細菌種類主要與海洋細菌的親緣關系最近,包括Gammaproteobacteria、Alphaproteobacteria和Bacteroidetes等[33]。這些種類可能來源于海洋環境,在地層內部的熱液流體上行時隨海水混入熱泉系統。古菌、Betaproteobacteria、Deltaproteobacteria和Firmicutes中豐度高的種類,所有Deferribacteres種類,以及Chlorobi、Chloroflexi、Deinococcus-Thermus和Acidobacteria等稀有門的大部分種類,與高溫環境微生物的親緣關系最近,這些種類可能來源于地層內部。這種主要由海洋微生物組成且含有少量嗜熱微生物的沿海熱泉,不同于Hobel等[10]報道的,溫度相對高的潮間帶熱泉(大于80℃)主要由陸地嗜熱細菌組成;溫度相對低的熱泉(小于等于70℃)主要由中等嗜熱的細菌組成,同時存在中溫海洋和陸地細菌。這種差異可能由于熱泉系統分布于潮下帶,且溫度相對較低所造成的(65℃)。此外,與嗜熱相關的種類主要來源于陸地熱泉以及高溫地下水層,少量來源于深海熱液區,這可能與熱泉鹽度為4有關。結合淡水屬性的潮間帶熱泉種類與陸地嗜熱微生物或陸地熱泉微生物的親緣關系最近[10],而鹽度接近海水的淺海熱液系統,其種類與深海熱液微生物的親緣關系最近[4—9]的結果,本研究結果也支持了鹽度對淺海熱液系統微生物組成影響的結論。
研究中不同氫酶氨基酸序列的組成和分布特征,也證實了溫度和鹽度對功能微生物類群的影響。沉積物中FeFe-氫酶的氨基酸序列主要與嗜熱微生物FeFe-氫酶的氨基酸序列相似度最高,這可能與沉積物溫度較高有關。Bacteroidetes是近岸優勢類群之一[33],菌苔中Bacteroidetes的16s rRNA 基因和FeFe-氫酶氨基酸序列豐度均為最高,說明菌苔中的這些優勢細菌和產氫類群可能來源于海洋環境。此外,菌苔中至少有22.5% 的NiFe-氫酶氨基酸序列與海洋或淡水環境Cyanobacteria的NiFe-氫酶氨基酸序列的相似度最高,也說明這些Cyanobacteria可能來源于海水或陸地環境。菌苔細菌的中溫屬性可能與其更易于與海水混合有關。
參考文獻:
[1]Dando P R, Hughes J A, Leahy Y, et al. Gas venting rates from submarine hydrothermal areas around the island of Milos, Hellenic Volcanic Arc[J]. Cont Shelf Res, 1995, 15(8): 913-929.
[2]Hoaki T, Nishijima M, Miyashita H, et al. Dense community of hyperthermophilic sulfur-dependent heterotrophs in a geothermally heated shallow submarine biotope near Kodakara-jima Island, Kagoshima, Japan[J]. Appl Environ Microbiol, 1995, 61(5): 1931-1937.
[3]Tarasov V G, Gebruk A V, Mironov A N, et al. Deep-sea and shallow-water hydrothermal vent communities: two different phenomena?[J]. Chem Geol, 2005, 224(1/3): 5-39
[4]Maugeri T L, Lentini V, Gugliandolo C, et al. Bacterial and archaeal populations at two shallow hydrothermal vents off Panarea Island (Eolian Islands, Italy)[J]. Extremophiles, 2009, 13(1): 199-212.
[5]Zhang Yao, Zhao Zihao, Chen C T A, et al. Sulfur metabolizing microbes dominate microbial communities in andesite-hosted shallow-sea hydrothermal systems[J]. PLoS One, 2012, 7(9): e44593.
[6]Tang Kai, Liu Keshao, Jiao Nianzhi, et al. Functional metagenomic investigations of microbial communities in a shallow-sea hydrothermal system[J]. PLoS One, 2013, 8(8): e72958.
[7]Lentini V, Gugliandolo C, Bunk B, et al. Diversity of prokaryotic community at a shallow marine hydrothermal site elucidated by illumina sequencing technology[J]. Curr Microbiol, 2014, 69(4): 457-466.
[8]Price R E, Lesniewski R, Nitzsche K S, et al. Archaeal and bacterial diversity in an arsenic-rich shallow-sea hydrothermal system undergoing phase separation[J]. Front Microbiol, 2013, 4: 158.
[9]Hirayama H, Sunamura M, Takai K, et al. Culture-dependent and-independent characterization of microbial communities associated with a shallow submarine hydrothermal system occurring within a coral reef off Taketomi island, Japan[J]. Appl Environ Microbiol, 2007, 73(23): 7642-7656.
[10]Hobel C F V, Marteinsson V T, Hreggvidsson G, et al. Investigation of the microbial ecology of intertidal hot springs by using diversity analysis of 16S rRNA and chitinase genes[J]. Appl Environ Microbiol, 2005, 71(2): 2771-2776.
[11]Lane D J. 16S/23S sequencing[M]//Stackbrandt E, Goodfellow M. Nucleic Acid Techniques in Bacterial Systematics. New York: John Wiley & Sons, 1991: 115-176.
[12]DeLong E F. Archaea in coastal marine environments[J]. Proc Natl Acad Sci U S A, 1992, 89(12): 5685-5689.
[13]Barz M, Beimgraben C, Staller T, et al. Distribution analysis of hydrogenases in surface waters of marine and freshwater environments[J]. PLoS One, 2010, 5(11): e13846.
[14]Schmidt O, Drake H L, Horn M A. Hitherto unknown [Fe-Fe]-hydrogenase gene diversity in anaerobes and anoxic enrichments from a moderately acidic fen[J]. Appl Environ Microbiol, 2010, 76(6): 2027-2031.
[15]Codon Code Corporation (2011) Codon code aligner[EB/OL]. http://www.codoncode.com/aligner/.
[16]RIMER-E, multivariate statistics for ecologists[EB/OL]. http://www.primer-e.com/.
[17]Schloss P D, Handelsman J. Introducing DOTUR, a computer program for defining operational taxonomic units and estimating species richness[J]. Appl Environ Microbiol, 2005, 71(3): 1501-1506.
[18]Hall T A. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symp Ser, 1999, 41(2): 95-98.
[19]Thompson J D, Higgins D G, Gibson T J. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice[J]. Nucleic Acids Res, 1994, 22(22): 4673-4680.
[20]Kumar S, Tamura K, Jakobsen I B, et al. MEGA2: molecular evolutionary genetics analysis software[J]. Bioinformatics, 2001, 17(12): 1244-1245.
[21]Staudigel H, Albarède F, Blichert-Toft J, et al. Geochemical Earth Reference Model (GERM): description of the initiative[J]. Chem Geol, 1998, 145(3/4): 153-159.
[22]Chen Ming, Yan Yongliang, Zhang Wei, et al. Complete genome sequence of the type strainPseudomonasstutzeriCGMCC 1.1803[J]. J Bacteriol, 2011, 193(21): 6095.
[23]Beckwith C R, Edwards M J, Lawes M, et al. Characterization of MtoD fromSideroxydanslithotrophicus: a cytochrome c electron shuttle used in lithoautotrophic growth[J]. Front Microbiol, 2015, 6: 332.
[24]Doronina N V, Kaparullina E N, Trotsenko Y A.Methyloversatilisthermotoleranssp. nov., a novel thermotolerant facultative methylotroph isolated from a hot spring[J]. Int J Syst Evol Microbiol, 2014, 64(1): 158-164.
[25]Hirayama H, Takai K, Inagaki F, et al.Thiobactersubterraneusgen. nov., sp. nov., an obligately chemolithoautotrophic, thermophilic, sulfur-oxidizing bacterium from a subsurface hot aquifer[J]. Int J Syst Evol Microbiol, 2005, 55(1): 467-472.
[26]Vésteinsdóttir H, Reynisdóttir D B, ?rlygsson J.Hydrogenophilusislandicussp. nov., a thermophilic hydrogen-oxidizing bacterium isolated from an Icelandic hot spring[J]. Int J Syst Evol Microbiol, 2011, 61(2): 290-294.
[27]Slobodkina G B, Reysenbach A L, Panteleeva A N, et al.Deferrisomacaminigen. nov., sp. nov., a moderately thermophilic, dissimilatory iron(III)-reducing bacterium from a deep-sea hydrothermal vent that forms a distinct phylogenetic branch in theDeltaproteobacteria[J]. Int J Syst Evol Microbiol, 2012, 62(10): 2463-2468.
[28]Iino T, Nakagawa T, Mori K, et al.Calditerrivibrionitroreducensgen. nov., sp. nov., a thermophilic, nitrate-reducing bacterium isolated from a terrestrial hot spring in Japan[J]. Int J Syst Evol Microbiol, 2008, 58(7): 1675-1679.
[29]Weidler G W, Gerbl F W, Stan-Lotter H.Crenarchaeotaand their role in the nitrogen cycle in a subsurface radioactive thermal spring in the Austrian Central Alps[J]. Appl Environ Microbiol, 2008, 74(19): 5934-5942.
[30]Vignais P M, Billoud B. Occurrence, classification, and biological function of hydrogenases: an overview[J]. Chem Rev, 2007, 107(10): 4206-4272.
[31]Appel J, Phunpruch S, Steinmüller K, et al. The bidirectional hydrogenase ofSynechocystissp. PCC 6803 works as an electron valve during photosynthesis[J]. Arch Microbiol, 2000, 173(5/6): 333-338.
[32]Troshina O, Serebryakova L, Sheremetieva M, et al. Production of H2by the unicellular cyanobacteriumGloeocapsaalpicolaCALU 743 during fermentation[J]. Int J Hydr Energ, 2002, 27(11/12): 1283-1289.
[33]Zinger L, Amaral-Zettler L A, Fuhrman J A, et al. Global patterns of bacterial beta-diversity in seafloor and seawater ecosystems[J]. PLoS One, 2011, 6(9): e24570.
Diversity of microbe and hydrogenase genes from a coastal hot spring of Kalianda, Indonesia
He Peiqing1, Dewi Seswita Zilda2, Li Jiang1, Zhang Xuelei1, Cui Jingjing3,Bai Yazhi3, Gintung Patantis2, Ekowati Chasanah2
(1.MarineEcologyResearchCenter,TheFirstInstituteofOceanography,StateOceanicAdministration,Qingdao266061,China; 2.ResearchCenterforMarineandFisheriesProductProcessingandBiotechnology,AgencyforMarineandFisheriesResearchandDevelopment,MinistryofMarineandFisheriesAffairs,Jakarta40115,Indonesia; 3.KeylaboratoryofStateOceanicAdministrationforMarineSedimentology&EnvironmentalGeology,TheFirstInstituteofOceanography,StateOceanicAdministration,Qingdao266061,China)
Abstract:During July, 2011, samples were collected from a coastal hot spring of Kalianda, Indonesia. Clone libraries of 16S rRNA genes of bacteria and archaea, and hydrogenase genes were constructed. The sequences were determined, and the diversity were also analyzed. The results demonstrated that the coastal hot spring hosted 16 phyla. Proteobacteria was dominant in hydrothermal fluid and sediment, accounting for 59.5% and 73.3%, respectively. Cyanobacteria was the most abundant in mat with abundance of 56.5%. Ammonia oxidizing Crenarchaeota was predominant in archaea. More than 70% of the species were most related to marine microbes, indicating the marine property of this system. About 20% of the species were most related to the thermophilic or moderate thermophilic microbes from terrestrial hot springs. These species might originate from the hyperthermal subfloor and involve in the process of iron oxidation and reduction, hydrogen oxidation, sulfur oxidation and nitrate reduction. The composition and distribution of NAD(P)-linked bidirectional NiFe-hydrogenase and FeFe-hydrogenase genes were also shaped by temperature and salinity. Our research provided an insight into the shallow-sea hydrothermal system.
Key words:Kalianda Indonesia;coastal hot spring;16S rRNA genes;hydrogenase;diversity
收稿日期:2015-07-07;
修訂日期:2016-03-24。
基金項目:中央級公益性科研院所基本科研業務費專項資金項目(2011T04);國家海洋局海洋生物活性物質與現代分析技術重點實驗室開放課題(MBSMAT-2015-06,MBSMAT-2011-03,MBSMAT-2012-2)。
作者簡介:何培青(1972-),女,山東省濟南市人,博士,從事海洋微生物研究。E-mail:hepeiqing@fio.org.cn
中圖分類號:Q938
文獻標志碼:A
文章編號:0253-4193(2016)06-0119-11
何培青, Dewi Seswita Zilda, 李江,等. 印度尼西亞卡利安達島近岸熱泉微生物和氫酶基因的多樣性[J].海洋學報,2016,38(6):119—129, doi:10.3969/j.issn.0253-4193.2016.06.013
He Peiqing, Dewi Seswita Zilda, Li Jiang, et al. Diversity of microbe and hydrogenase genes from a coastal hot spring of Kalianda, Indonesia[J]. Haiyang Xuebao,2016,38(6):119—129, doi:10.3969/j.issn.0253-4193.2016.06.013

