泰國近海習見有毒立方水母和缽水母的遺傳分析
劉瑞娟,肖潔,張學雷*,Charatsee Aungtonya
(1.國家海洋局第一海洋研究所,山東 青島 266061;2. 海洋生態環境科學與工程國家海洋局重點實驗室,山東 青島 266061;3. Phuket Marine Biological Center, Thailand Phuket 83000)
泰國近海習見有毒立方水母和缽水母的遺傳分析
劉瑞娟1,2,肖潔1,2,張學雷1,2*,Charatsee Aungtonya3
(1.國家海洋局第一海洋研究所,山東 青島 266061;2. 海洋生態環境科學與工程國家海洋局重點實驗室,山東 青島 266061;3. Phuket Marine Biological Center, Thailand Phuket 83000)
摘要:本研究利用線粒體16S rDNA和核基因18S rDNA片段,對泰國沿海常見的有毒水母進行遺傳分析,并比較了2個基因片段作為通用分子標記,在研究水母類多個綱的遺傳多樣性中的應用。研究發現,泰國近海的有毒水母存在較高的遺傳多樣性,所獲得的32個樣品可以分為9個種,包括4種缽水母、4種立方水母和1種水螅水母。然而,完全確定各種的分類地位,還需要更多的形態、生活史等方面信息。兩個基因片段均能明確區分各種類,但核基因18S序列比線粒體基因片段更為保守。根據16S基因片段序列計算水母種內和種間的K2P(Kimura 2-parameter)遺傳距離,發現所研究的9個水母種類,種內遺傳距離在0~0.050之間,其中94%的種內遺傳距離小于0.040,同綱種間的遺傳距離為0.204~0.474,其中91%的種間遺傳距離大于0.250;而利用18S基因,種內距離在0~0.002之間,同綱種間距離為0.008~0.066(平均為0.038,SE=0.006)。16S的AT堿基含量明顯高于核基因18S,且16S的堿基含量在不同綱之間有顯著差異,進一步表明水母線粒體16S基因的突變率相對較高,適合研究水母較低分類階元以及種下的遺傳差異。
關鍵詞:缽水母;立方水母;遺傳差異;核糖體基因;泰國
1引言
水母種類多、數量大、分布廣,在浮游生物群落中占有很重要的地位。近年來,受人類活動和氣候環境變化的影響,一些海域水母數量劇增,乃至暴發的事件頻繁發生[1],從而改變了當地海洋生態系統的結構和功能,嚴重影響了海洋漁業資源以及海洋漁業生產[2]。另外,盡管一些水母種類(例如:海蜇Rhopilemaesculentum)具有重要的經濟和藥用價值,但是許多水母種類(例如:立方水母Cubozoa)的刺絲囊含有毒素,人被蜇傷后可出現皮膚劃痕、紅腫、腹痛、痙攣、麻痹、心臟及呼吸衰竭甚至死亡等癥狀,嚴重危害沿海居民和游客的健康安全[3—4]。因此,鑒別水母種類并掌握其種群動態具有重要意義。
近年來,在泰國沿海,游客被劇毒立方水母蜇傷致死的事故頻繁出現。例如:1999年,一位26歲英國男性游客在泰國灣南部蘇梅島(Koh Samui)查汶海灘(Chaweng Beach)游泳時被蜇傷,并于數分鐘后死亡[5];2001、2002年和2008年,又有數次類似事故發生在帕安島(Koh PhaNgan Island)和蘭塔島(Koh lanta)等地;另外,還有數十次非致命的蜇傷事件[6—7]。有毒水母的出現嚴重威脅泰國沿海當地居民和游客的生命財產安全,引起了泰國政府部門的高度重視。
然而,識別和鑒定不同的有毒水母種類存在一定的困難。目前初步調查研究表明泰國海域存在的有毒水母有10多種(Aungtonya and Chanachon, 泰國普吉生物研究中心,未發表數據),大多屬于立方水母和缽水母兩綱,然而準確鑒定并區分種類存在諸多困難。首先,一些種類的系統分類學地位仍存在爭議。例如:Chiropsoides是泰國海域常見的一類水母,然而其形態上(尤其是基板pedalia)與該屬的已知種類C.quadrigatus(Haeckel, 1880)和C.buitendijki(Horst, 1907)存在明顯區別[8]。另外,泰國西南沿海和安達曼海沿岸存在3種不同形態的金黃水母(Chrysaoraspp.;Aungtonya and Chanachon, 泰國普吉生物研究中心,未發表數據),其是否為不同種類抑或同種的不同變型,還有待于進一步研究。
其次,水母不同時期的形態特征變異較大;形態可塑性高,不同海域的地理群體可能存在形態差異[9];另外,水母含水量多,部分組織器官采集過程中容易被破壞,樣品采集固定后容易變形失真等。這些因素都加大了實際調查中對水母形態學鑒定的難度。利用分子生物學手段,對有毒水母的基因型進行鑒定識別,可以克服上述困難,檢測水母從浮浪幼體蟲到成體的全生活階段,并為一些疑難種類的系統分類地位提供幫助[10—11]。
本研究利用線粒體16S和核基因18S核糖體基因片段,對泰國海域常見有毒水母的系統分類地位進行分析研究,并評估這兩個基因片段作為常用分子標記鑒定這些常見水母種類的可行性、靈敏度,及存在問題,以期為后續有毒水母種類的生活史、監測和生態學等方面的研究提供有效的手段。
2材料與方法
2.1樣品的采集與保存
本研究所用的樣本于2014—2015年在泰國安達曼海沿岸采集,其中8個樣本采自拉廊府(Ranong),其余24個樣本采于攀牙府(Phangnga),詳細信息見表1。水母樣品清洗干凈后,取小塊傘部組織,95%酒精固定,4℃保存。其余用終濃度5%的甲醛固定保存,用于形態鑒定。
2.2基因組DNA的提取及目標片段擴增
切取約3 mg固定的組織樣本,用蒸餾水沖洗3次,并用無菌濾紙汲取多余的水分和固定液。然后用手術刀片將組織切碎,按照E.Z.N.A.TM動物組織DNA提取試劑盒(Omega Bio-Tek, USA)說明步驟提取樣本基因組DNA。
16S反應體系為25 μL,包括2.5 μL 10×buffer,1.5 μL Mg2+(25 mmol/L),0.5 μL dNTP(10 mmol/L),0.04 μL每條引物(100 umol/L),1 U Taq酶(Takara),0.25 μL BSA(10 mg/mL,牛血清白蛋白),其余用超純水補足;18S反應體系為25 μL,包括2.5 μL 10×buffer,3 μL Mg2+(25 mmol/L),0.5 μL dNTP(10 mmol/L),0.062 5 μL每條引物(100 umol/L),1 U Taq酶(Takara),0.25 μL BSA,其余用超純水補足。反應引物見下表(表2)。PCR擴增在PCR儀(Biore,杭州)上進行,16S、18S反應程序均為:94℃預變性2 min,然后38個循環包括94℃變性45 s,48℃退火60 s,72℃延伸2 min,最后72℃延伸10 min。
2.3PCR產物純化及測序
擴增產物經電泳檢測后,用E.Z.N.A.TM割膠回收試劑盒(Omega Bio-Tek, USA)進行回收純化,具體操作參照廠家說明。純化產物直接送上海美吉生物醫藥科技有限公司使用PCR引物雙向測序。部分測序困難的樣品采用pMDTM19-T(Takara,中國大連)連接,并轉化至感受態大腸桿菌DH5α,采用麥康凱選擇性培養基篩選陽性克隆,經PCR檢測后,送往上海美吉生物醫藥科技有限公司進行測序。
2.4數據處理
首先使用DNAstar軟件包(DNAstar Inc.,美國)中的Seqman程序處理原始測序峰圖文件,進行序列拼接,最后進行人工校正,將引物和兩端測序質量差的序列刪除。將得到的序列利用Sequin軟件處理后批量提交到NCBI中,獲得序列注冊號。使用MEGA6.0[14]內置的ClustalW在默認參數下對核苷酸序列進行比對。本研究共獲得水母類16S和18S片段各32條(表1),結合從GenBank數據庫中下載的相關代表性序列(表3),包括18條16S片段和17條18S序列,對所得樣品進行系統發育研究。根據最大似然距離構建NJ(Neighbor-joining)樹[15]和ML(Maximum Likelihood)樹,用bootstrap方法檢驗系統發育樹各節點的可信度,各設1 000次重復。
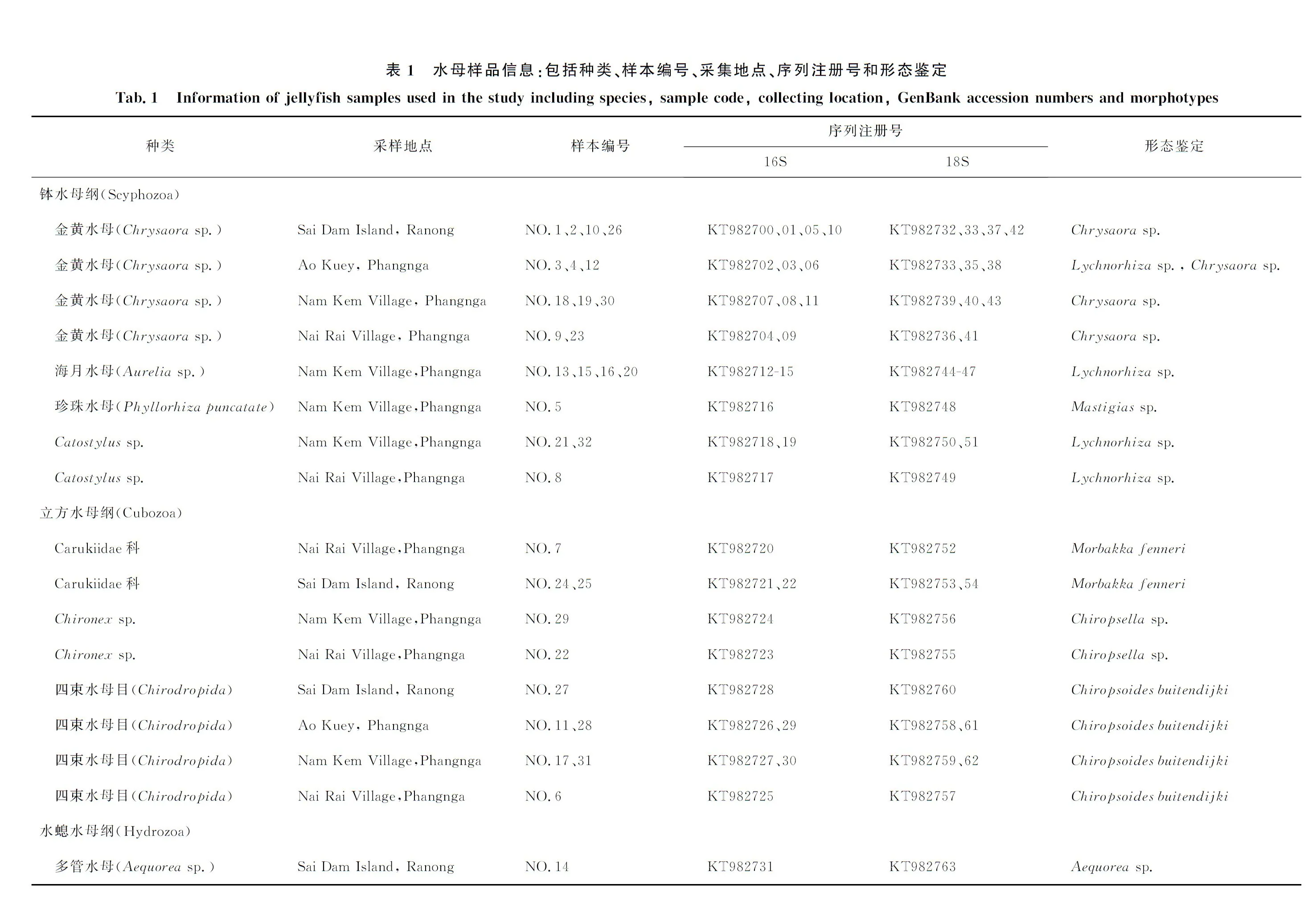
表2 本研究所用引物序列及參考文獻
Tab.2 Sequences and references of the primers used in the study

擴增片段引物名稱及序列(5'-3')參考文獻16SBRDGP-1:TCGACTGTTTACCAAAAACATAGCBRDGP-2:ACGGAATGAACTCAAATCATGTAAGBayha[12]16S16S-F:GCGGTAACYYTGACCGTGMT16S-R:GGTCGAACAGACCWACCYTY本研究18SL18S:CGGAAGGGCACCACCAGGAG18Sb:GATCCTTCTGCAGGTTCACCTACBayha等[13]
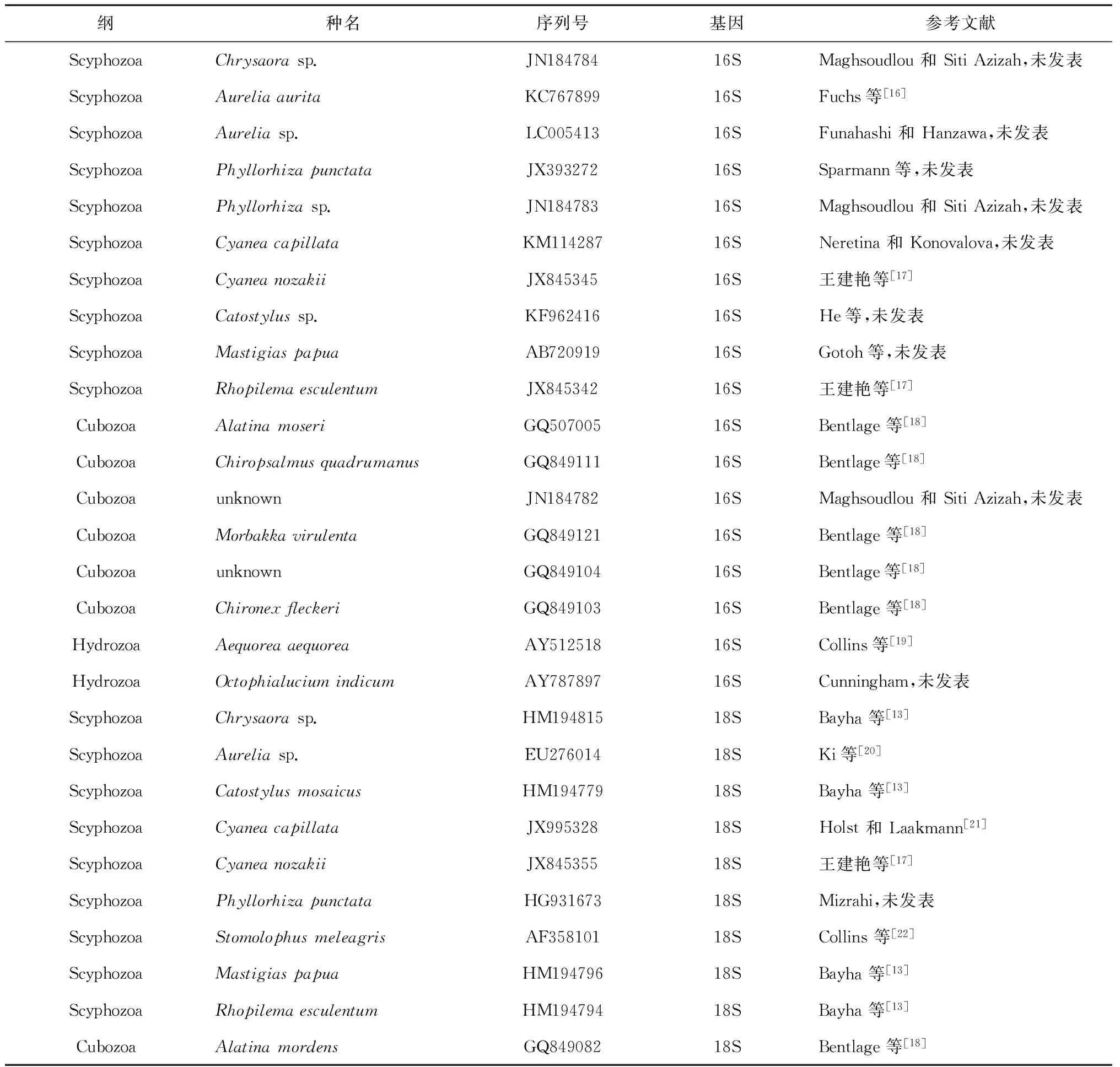
表3 從Genebank下載的序列信息,包括序列所屬基因、序列號、種類和參考文獻
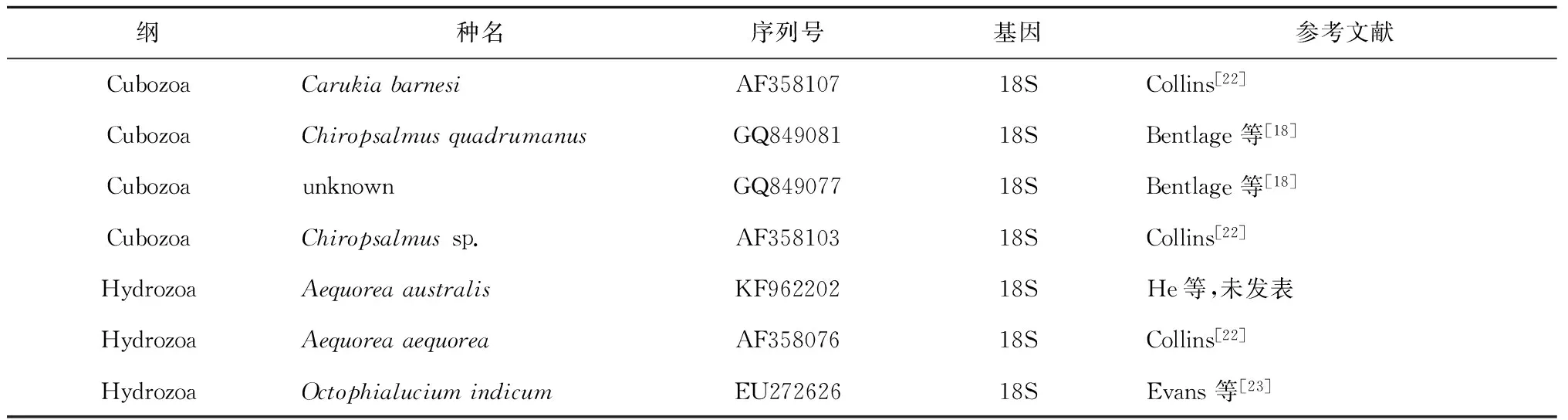
續表3
根據上述系統發育樹,將32個樣品按其種屬歸類,使用MEGA6.0軟件包計算樣品(不包括下載序列片段)間相對遺傳距離K2P(Kimura 2-parameter),并按各樣品的分類組別分別統計和比較種內、種間和綱間遺傳距離的變化[24]。
2.5統計方法
利用SPSS16.0(SPSS Inc.)統計軟件對所得序列的堿基含量差異進行檢驗,并作圖。先用配對t檢驗(paired t-test)分析所有樣品的16S和18S基因AT堿基含量之間是否有明顯差異,然后用單因素方差分析(one-way ANOVA)檢驗缽水母和立方水母綱之間各基因的堿基含量是否有顯著區別,并用事后多重比較(post hoc Turkey HSD)兩兩檢驗各組之間的差異。由于水螅水母綱只有一個樣本,未參加多重比較。
3結果與分析
3.1序列特征
使用16S特異引物BRDGP-1、BRDGP-2的PCR反應擴增片段長度約為610 bp(不包括引物),使用16S特異引物16S-F、16S-R的PCR反應擴增片段長度約為500 bp,聯合來自GenBank的其他18條序列比對后共獲得長為495 bp的共有序列進行分析;使用18S特異引物的PCR反應擴增片段長度約為620 bp,聯合來自GenBank的其他17條序列比對后共獲得長為608 bp的共有序列進行分析。所有樣品線粒體基因16S序列的AT堿基含量在57.9%~73.6%之間,明顯高于核基因的18S序列(50.1%~54.2%,paired t-test,P<0.01),符合后生動物的線粒體基因組AT含量普遍偏高的特點[25]。進一步分析來自于不同綱的16S和18S序列,發現3個綱(缽水母、立方水母、水螅水母)的樣品間,其序列堿基組成差異較大。如圖1所示:3個綱16S序列的AT堿基含量(%)分別為59.5%(SE=0.56)、66.5%(SE=0.65)和73.6%,差異顯著(one-way ANOVA, F=32.2,P<0.01);18S序列的AT%分別為52.0%(SE=0.07)、50.5%(SE=0.10)和54.2%。多重檢驗立方水母綱和缽水母綱的16S、18S基因AT堿基含量,發現缽水母綱16S基因AT含量明顯高于立方水母,兩者又明顯高于18S基因的AT堿基含量,而兩個綱的18S堿基含量差異不明顯(圖1)。水螅水母的兩個基因片段AT堿基含量是否高于其他兩綱,還有待于進一步研究。
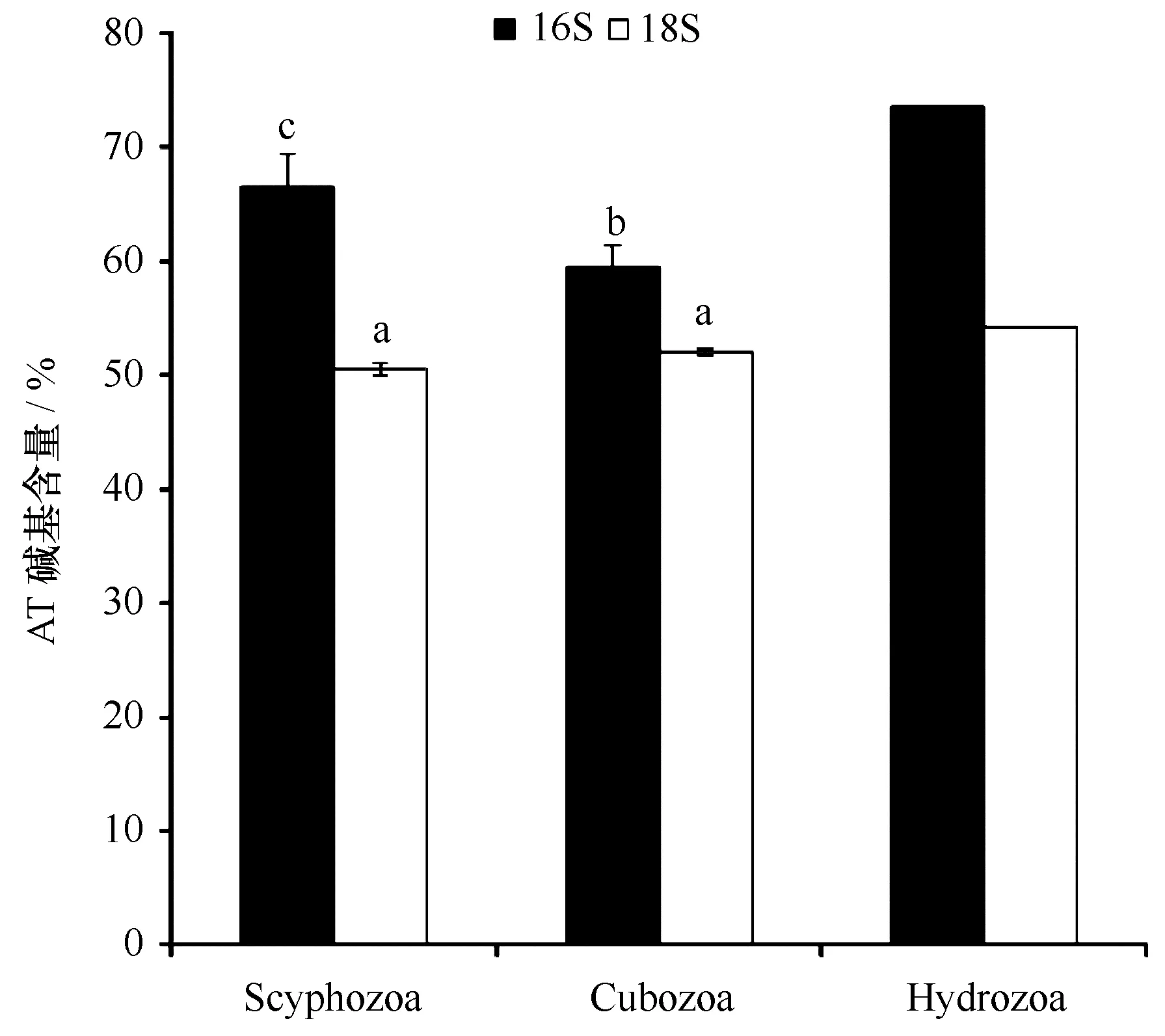
圖1 水母16S、18S序列的AT堿基含量Fig.1 The A and T nucleotide composition of 16S and 18S genes for the samples sequenced in this study字母a、b、c代表顯著性差異,P<0.05Letters a, b,c represent significant difference,P<0.05
3.2基于16S和18S序列的系統樹
3.2.116S和18S序列的相似度分析
將本研究所得的32個樣品序列進行BLAST比對,結果表明,所有樣品分別屬于刺胞動物門的3個綱(缽水母綱,立方水母綱和水螅水母綱)。缽水母綱中,樣品No.1~4、9、10、12、18、19、23、26、30這12個樣品的序列差異不大,18S基因相似度為100%,16S基因只有2個單堿基的突變,相似度為99.8%,應為同一種,屬于金黃水母屬(Chrysaora)。樣品NO.13、15、16、20的18S基因相似度為99.8%,16S基因完全相同(100%),屬于海月水母屬(Aurelia)。樣品NO.5的兩個基因片段序列與珍珠水母(Phyllorhizapunctata)的序列一致(100%)。樣品NO.8、21、32的兩個基因序列100%相同,18S基因與Stomolophusmeleagris的相關序列(AF358101)[22]相差一個堿基,16S基因與中國近海發現的Catostylussp.樣本(KF962416,未發表數據)100%一致。由于缺少Stomolophusmeleagris的16S基因,無法明確這3個樣品屬于哪個屬;然而進一步比較COI基因(數據未顯示),發現這3個樣品與中國近海發現的Catostylussp.相近,應為同種。
立方水母綱中,樣品NO.7、24、25的18S基因100%一致,與立方水母Carukiabarnesi(AF358107)[22]的相似度為98.5%;NO.24和25的16S基因相同,而與NO.7的相似度僅為95.1%。樣品NO.22和29的16S基因片段存在6個堿基的差異,相似度為98.7%,18S基因片段完全相同,與其最相近的為一采自于帕勞(Palau)的未知立方水母種[18],16S 基因(GQ849104)的相似度僅為約93.0%,而18S基因(GQ849077)與NO.22和29一致。結合立方水母綱其他所有序列分析,樣品NO.22和29可能為Chironex屬一未知新種。NO.6、11、17、28、31和27的18S基因相似度在99.8%~100%之間,16S基因相似度為96.0%~100%,其在立方水母綱的四束水母科(Chirodropidae)中形成單系源分支。
水螅水母綱中的一個樣品NO.14的16S基因與印度八擬杯水母(Octophialuciumindicum, AY787897, Cunningham未發表數據)比較相近,但相似度僅為92.6%;而18S基因與多管水母(Aequoreasp.,AF358076)[22]相近,相似度為99.0%,結合COⅠ基因信息等確定NO.14為一種多管水母。
32個樣本的種屬地位見表1,其中20個屬于缽水母綱,11個屬于立方水母,1個屬于水螅水母。缽水母中,12個(NO.1、2、3、4、9、10、12、18、19、26、23、30)為金黃水母(Chrysaorasp.),4個(NO.13、15、16、20)為海月水母(Aureliasp.),1個(NO.5)為珍珠水母(Phyllorhizapuncatate)。11個立方水母分別為一種未知Chironexsp.(NO.22、27、29),Carukiidae科的兩個相近種(NO.7和 NO.24、25),和四束水母目中的一種(NO.6、11、17、28、31和27)。另外,NO.14為水螅水母綱的一種多管水母(Aequoreasp.)。
3.2.216S和18S系統進化樹分析
用NJ法和ML法對本研究所得32條16S序列(KT982700-31)及18條GenBank數據庫中與所得序列最相近的16S序列(表3)構建系統進化樹,發現兩種方法所構建的系統進化樹分支結果基本一致(圖中分支上括號內的數字代表ML法所得的可信度),結果顯示(圖2):50條序列可以分為3大組,序列KT982731與下載序列AY787897及AY512518成為一支,屬于水螅水母綱(Hydrozoa);另有11條序列與6條立方水母綱下載序列(JN184782、GQ849121、GQ507005、GQ849104、GQ849103、GQ849111)組成一支(立方水母綱,Cubozoa);而其余20條本研究所得序列和10條下載序列聚合在一起,屬于缽水母綱(Scyphozoa)。其中,缽水母綱中20條本研究所得序列可進一步分為4個獨立分支(圖2),立方水母包含的本研究所得11條序列亦可分成4個分支。
對本研究所得32條18S序列(KT982732-63)及17條GenBank數據庫下載序列(表3)進行分析,所構建的系統進化樹(圖3)結構與16S基因(圖2)基本一致。
3.3遺傳差異
16S序列不同分類階元水平的序列差異分布如圖4所示。在種內,16S序列的差異在 0~0.050之間,均值為0.009(SE=0.003),其中94%的個體種內差異小于0.040。同綱種間,序列差異在0.204至0.474之間,平均為0.306(SE=0.030),其中91%的同綱遺傳差異大于0.250。不同的綱間遺傳差異在0.477到0.644之間,平均值為0.540(SE=0.046),其中85%的個體綱間遺傳差異大于0.500。
18S序列不同分類階元水平的遺傳差異分布如圖5所示。水母18S的種內差異在0~0.002 之間,均值為0.000 4,(SE=0.003)。同綱種間的遺傳差異從0.008至0.066(平均為0.038,SE=0.006)。不同的綱間遺傳差異在0.084到0.174之間,平均值為0.106(SE=0.010)其中53%的綱間遺傳差異大于0.100。顯而易見,16S序列的遺傳差異遠高于18S基因,約為18S的5倍。
4討論
本研究利用線粒體基因16S和核基因18S rDNA對泰國沿海常見的缽水母和立方水母進行遺傳分析研究。基于16S和18S基因片段構建的系統發育樹,所有同種序列都能形成具有較高支持度的單分支,說明兩個片段均能很好地區分這些物種(圖2,圖3)。然而,16S基因片段在檢測種或種下水平的遺傳差異顯示出更高的靈敏度。本研究中,16S rDNA在不同分類階元的遺傳差異相差較大(32個樣品遺傳距離范圍在0~0.644之間),而相同樣品的18S rDNA不同分類階元的遺傳距離則相對較小(0~0.174);這可能是因為線粒體16S基因在物種演化過程中,積累了較多變異而形成的。該結果與其他關于水母類系統進化的研究一致[12,26]。另外,不同綱之間,16S基因的AT堿基含量出現了明顯的差異(圖1),而18S基因沒有明顯差異,進一步說明該門類(刺胞動物門)的物種線粒體16S基因相對具有較高的進化速率。一般認為,核rDNA的小亞基序列(18S rDNA)相對比較保守,適合研究較高階元的系統發生[27];而與核DNA相比,線粒體DNA具有結構簡單、一級結構的堿基突變率高、進化速度快等特點,更適于較低水平的進化關系和遺傳差異分析研究[28]。
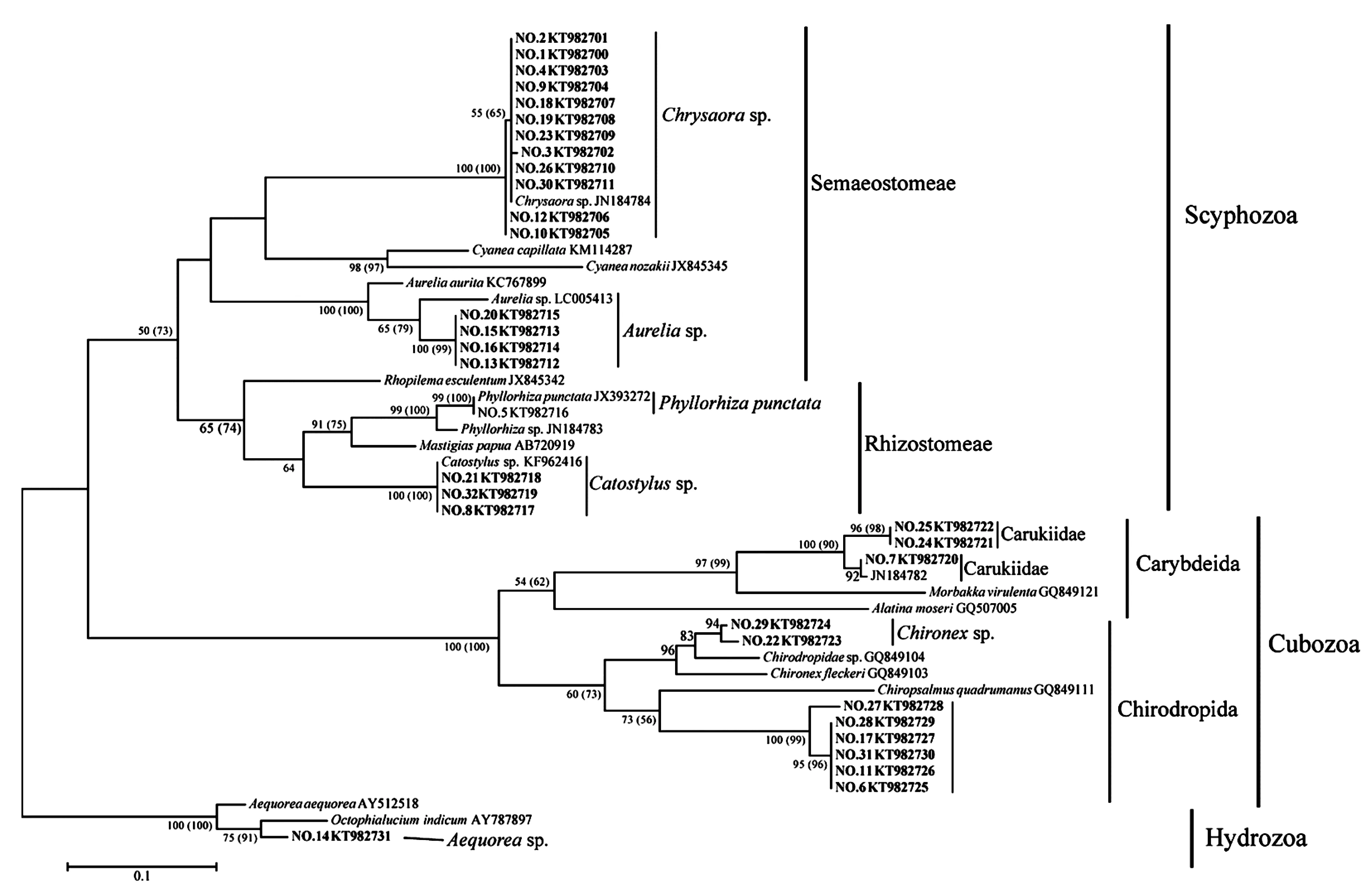
圖2 基于16S序列的系統樹Fig.2 Phylogenetic tree based on 16S rDNA sequences
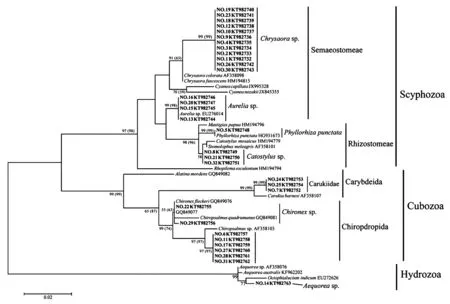
圖3 基于18S序列的系統樹Fig.3 Phylogenetic tree based on the 18S rDNA sequences
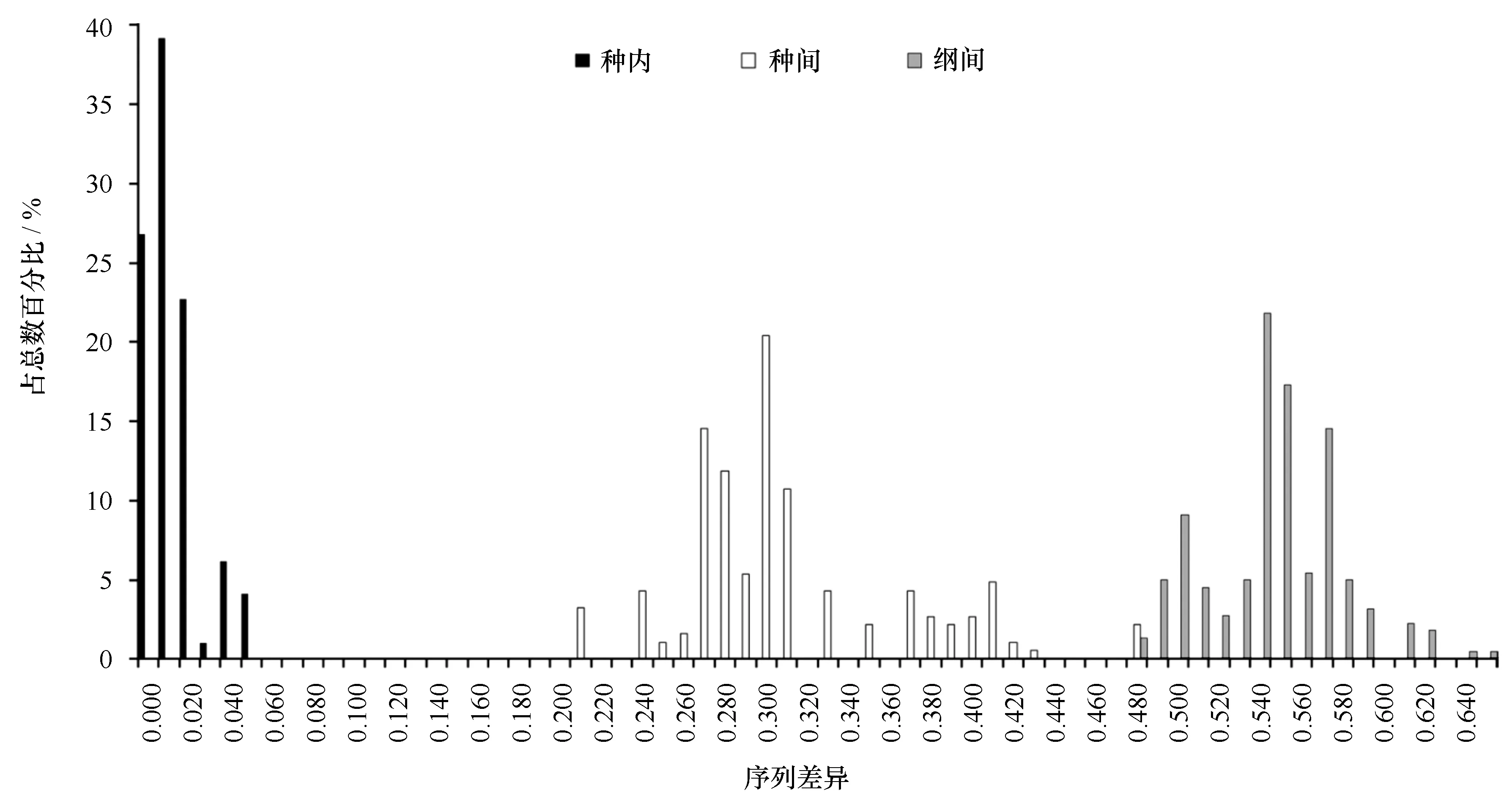
圖4 水母16S序列不同分類階元水平的遺傳差異分布Fig.4 Frequency of pairwised genetic distances at the levels of intra-, inter-species and among classes based on 16S rDNA
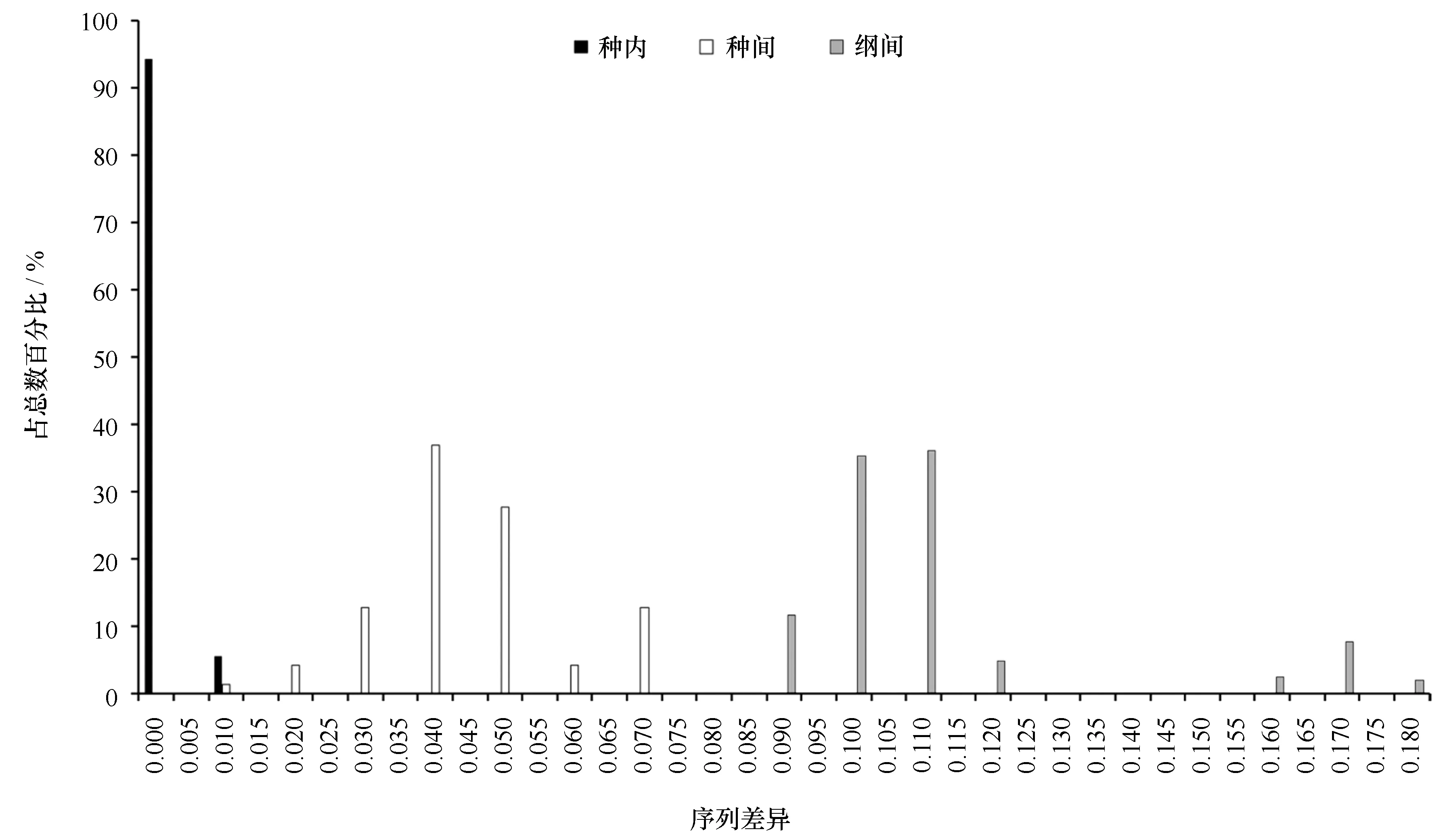
圖5 水母18S序列不同分類階元水平的遺傳差異分布Fig.5 Frequency of pairwised genetic distances at the levels of intra-, inter-species and among classes based on 18S rDNA
水母的分類始終是一難點。由于水母組織含水量多,樣品易碎、固定后易變形,且生活史復雜、不同生活時期形態變異較大,使得單純靠形態學進行水母種類鑒定十分困難[28—29]。分子生物學方法以較為穩定的遺傳物質為研究對象可以較好地克服上述缺點,從而成為水母分類學研究中的有力輔助工具[11,30]。本研究對泰國近海常見的缽水母和立方水母種類進行分析研究,發現泰國沿海水母多樣性較高,涉及3個綱、8科、9種,其中有毒立方水母4種,缽水母4種,水螅水母1種。一些水母樣本的形態學和分子生物學鑒定結果出現分歧(表1),例如:樣品NO.13、15、16、20與NO.21、32、8被鑒定為同一種(Lychnorhizasp.),然而,該樣品16S和18S的基因序列具有明顯區別,可分為兩種,分別為海月水母(Aureliasp.)和Catostylussp.。立方水母綱中,NO.7、24和25鑒定為Morbakkafenneri,但其基因序列與Morbakka屬的已知種M.virulenta相去甚遠,而在Carukiidae科中形成獨立分支。同樣,樣品NO.22和29的相關基因也與Chiropsella屬中的已知種C.bronzie[18]不同,卻與Chironexspp.[18,31]聚成一支;其他基因(如COⅠ)也不支持NO.22和29屬于Chiropsella屬,而應屬于Chironex屬中的一種(劉瑞娟等,未發表)。
另外,目前可利用的基因數據還不夠完整,使得相當一部分樣本僅依賴于基因數據不能確定其種屬分類地位。相較于形態學所描述的眾多缽水母和立方水母物種,基因數據庫(如 http://ncbi.nlm.nih.gov)中有基因數據的物種數量遠少于這些,許多種類僅有形態描述,而沒有基因數據。以刺胞動物門中最小的綱——立方水母綱為例,目前有形態描述的有效種約有36種[32],而NCBI數據庫中有基因數據的只有約20種;而缽水母綱,已知有約200多種[32],而目前最為完整的關于該綱系統發育進化關系的研究,收錄有基因數據的物種數量只占總數的14%,屬的覆蓋率也只有21%[33]。這使得利用基因小片段,對這些綱內的種類進行基因條形碼研究十分困難。為了便于比較,本研究下載并比對了NCBI基因數據庫中與我們所測得的序列比較相近的基因序列,然而仍有相當部分樣本,其基因序列與已知種的的序列差異較大,例如:No.6、11、17、27、28和31的基因數據與四束水母目中的其他種屬存在較大差異,16S基因與Chiropsalmusquadrumanus、Chiropsellabronzie、Chironexfleckeri的相似度僅為73.9%~76.7%,與這3個屬間的相似度相當(73.6%~77.7%),其是未知屬抑或是尚未進行分子生物學研究的已知種類,還需要結合其完整的形態結構、生活史以及其他種屬的標本描述,進行系統的研究。
如前所述,不同形態的金黃水母(Chrysaoraspp.)在泰國和馬來西亞沿海都有發現,一直困擾著當地的分類學家(Aungtonya和Chanachon, 泰國普吉生物研究中心; Maghsoudlou和Siti Azizah, Universiti Sains Malaysia, 未發表數據)。本研究對12個泰國沿海的金黃水母樣本進行分析研究,發現這些樣本的18S基因完全一致,僅NO.4的16S基因有單堿基突變。結合Maghsoudlou等提供的4種金黃水母形態型(Morphotype)的16S序列(JN184784-JN184787)進一步分析,發現共有4種基因型,相似度在99.4%~99.8%之間;然而,這4種基因型與形態型并沒有呈現一一對應的關系。因此,這些金黃水母的不同表型形態,是否由不同的環境條件,抑或不同生活史引起,還值得進一步深入研究。
本研究從分子生物學角度揭示了泰國沿海有毒水母的多樣性,初步描述了一些有毒立方水母和缽水母的分類地位,這對泰國沿海有毒水母的預警和后續生態學等的研究有著重要意義。然而,由于本研究采樣范圍和物種數的局限性,未能明確這些有毒種類的分布范圍,以及是否存在種下的隱種分化等。而且,東南亞沿海地區關于水母的研究起步較晚,分子生物學方面的研究更是比較匱乏,加之基因數據庫的不完善等,使得一些疑難種未能完全明確其分類地位。加強和加深與當地以及世界水母分類學家的合作,通過傳統形態學與分子系統學相結合的方法,對缽水母和立方水母綱展開系統的研究,有望解決上述問題。
參考文獻:
[1]張芳, 孫松, 李超倫. 海洋水母類生態學研究進展[J]. 自然科學進展, 2009, 19(2): 121-130.
Zhang Fang, Sun Song, Li Chaolun. Research advances in marine jellyfish ecology[J]. Process in Natural Science, 2009, 19(2): 121-130.
[2]吳穎, 李惠玉, 李圣法, 等. 大型水母的研究現狀及展望[J]. 海洋漁業, 2008, 30(1): 80-87.
Wu Ying, Li Huiyu, Li Shengfa, et al. Review on the current situation of macro-jellyfish research and expectations[J]. Marine Fisheries, 2008, 30(1): 80-87.
[3]于華華, 劉希光, 劉松, 等. 水母毒素的研究現狀[J]. 海洋科學, 2003, 27(11): 27-29.
Yu Huahua, Liu Xiguang, Liu Song, et al. The research status of jellyfish toxin[J]. Marine Sciences, 2003, 27(11): 27-29.
[4]肖良, 張黎明. 水母毒素的研究進展[J]. 中國海洋藥物, 2008, 26(6): 40-43.
Xiao Liang, Zhang Liming. The research progress of jellyfish toxins[J]. Chinese Journal of Marine Drugs, 2008, 26(6): 40-43.
[5]Suntrarachun S, Roselieb M, Wilde H, et al. A fatal jellyfish encounter in the Gulf of Siam[J]. Journal of Travel Medicine, 2001, 8(3): 150-151.
[6]De Pender A M G, Winkel K D, Ligthelm R J. A probable case of Irukandji Syndrome in Thailand[J]. Journal of Travel Medicine, 2006, 13(4): 240-243.
[7]Fenner P J, Lippmann J, Gershwin L A. Fatal and nonfatal severe jellyfish stings in Thai waters[J]. Journal of Travel Medicine, 2010, 17(2): 133-138.
[8]Gershwin L A. Comments onChiropsalmus(Cnidaria: Cubozoa: Chirodropida): a preliminary revision of the Chiropsalmidae, with descriptions of two new genera and two new species[J]. Zootaxa, 2006, 1231: 1-42.
[9]Cornelius P F S. EuropeanObelia(Cnidaria, Hydroida): systematics and identification[J]. Journal of Natural History, 1990, 24(3): 535-578.
[10]王仁誠. 中國近海常見水母類和蟲戎類的DNA條形碼分析及系統發生研究[D]. 青島: 中國科學院海洋研究所, 2010.
Wang Rencheng. DNA barcoding and phylogenetic of Medusa and Hyperiid in Chinese Costal Region[D]. Qingdao: Institute of Oceanology, Chinese Academy of Sciences, 2010.
[11]程方平, 王敏曉, 王彥濤, 等. 中國北方習見水母類的DNA條形碼分析[J]. 海洋與湖沼, 2012, 43(3): 451-459.
Cheng Fangping, Wang Minxiao, Wang Yantao, et al. DNA barcoding of common Medusozoa in Northern China based on mtCOI sequence[J]. Oceanologia et Limnologia Sinica, 2012, 43(3): 451-459.
[12]Bayha K M. The molecular systematics and population genetics of four coastal ctenophores and scyphozoan jellyfish of the US Atlantic and Gulf of Mexico[D]. Newark: The University of Delaware, 2005.
[13]Bayha K M, Dawson M N, Collins A G, et al. Evolutionary relationships among scyphozoan jellyfish families based on complete taxon sampling and phylogenetic analyses of 18S and 28S ribosomal DNA[J]. Integrative and Comparative Biology, 2010, 50(3): 436-455.
[14]Tamura K, Stecher G, Peterson D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729.
[15]Tamura K, Dudley J, Nei M, et al. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24(8): 1596-1599.
[16]Fuchs B, Wang Wei, Graspeuntner S, et al. Regulation of polyp-to-jellyfish transition inAureliaaurita[J]. Current Biology, 2014, 24(3): 263-273.
[17]王建艷, 甄毓, 王國善, 等. 基于mt-16S rDNA和mt-COI基因的海月水母分子生物學鑒定方法和檢測技術[J]. 應用生態學報, 2013, 24(3): 847-852.
Wang Jianyan, Zhen Yu, Wang Guoshan, et al. Molecular identification and detection of moon jellyfish (Aurelia sp.) based on partial sequencing of mitochondrial 16S rDNA and COI[J]. Chinese Journal of Applied Ecology, 2013, 24(3): 847-852.
[18]Bentlage B, Cartwright P, Yanagihara A A, et al. Evolution of box jellyfish (Cnidaria: Cubozoa), a group of highly toxic invertebrates[J]. Proceedings of the Royal Society of London Series B, 2010, 277(1680): 493-501.
[19]Collins A G, Winkelmann S, Hadrys H, et al. Phylogeny of Capitata and Corynidae (Cnidaria, Hydrozoa) in light of mitochondrial 16S rDNA data[J]. Zoologica Scripta, 2005, 34(1): 91-99.
[20]Ki J S, Hwang D S, Shin K, et al. Recent moon jelly (Aureliasp. 1) blooms in Korean coastal waters suggest global expansion: examples inferred from mitochondrial COI and nuclear ITS-5.8 S rDNA sequences[J]. ICES Journal of Marine Science, 2008, 65(3): 443-452.
[21]Holst S, Laakmann S. Morphological and molecular discrimination of two closely related jellyfish species,CyaneacapillataandC.lamarckii(Cnidaria, Scyphozoa), from the northeast Atlantic[J]. Journal of Plankton Research, 2013, 36(1): 48-63.
[22]Collins A G. Phylogeny of Medusozoa and the evolution of cnidarian life cycles[J]. Journal of Evolutionary Biology, 2002, 15(3): 418-432.
[23]Evans N M, Lindner A, Raikova E V, et al. Phylogenetic placement of the enigmatic parasite,Polypodiumhydriforme, within the Phylum Cnidaria[J]. BMC Evolutionary Biology, 2008, 8(1): 139.
[24]張珰妮, 鄭連明, 何勁儒, 等. 基于線粒體COI和16S片段序列的北部灣北部水螅水母DNA條形碼分析[J]. 生物多樣性, 2015, 23(1): 50-60.
Zhang Dangni, Zheng Lianming, He Jinru, et al. DNA barcoding of Hydromedusae in northern Beibu Gulf for species identification[J]. Biodiversity Science, 2015, 23(1): 50-60.
[25]Beard C B, Hamm D, Collins F H. The mitochondrial genome of the mosquitoAnophelesgambiae: DNA sequence, genome organization, and comparisons with mitochondrial sequences of other insects[J]. Insect Molecular Biology, 1993, 2(2): 103-124.
[26]Cartwright P, Evans N M, Dunn C W, et al. Phylogenetics of Hydroidolina (Hydrozoa: Cnidaria)[J]. Journal of the Marine Biological Association of the United Kingdom 2008, 88(8): 1663-1672.
[27]Hwang U W, Kim W. General properties and phylogenetic utilities of nuclear ribosomal DNA and mitochondrial DNA commonly used in molecular systematics[J]. The Korean Journal of Parasitology, 1999, 37(4): 215-228.
[28]張芳. 黃東海膠質浮游動物水母類研究[D]. 青島: 中國科學院海洋研究所, 2008.
Zhang Fang. Zooplanktivorous gelatinous taxa: medusas in the Yellow Sea and East China Sea[D]. Qingdao: Institute of Oceanology, Chinese Academy of Sciences, 2008.
[29]Myers A G. Medusae of the world: the Scyphomedusae[M]. Washington DC: Carnegie Inst., 1910.
[30]Ortman B D, Bucklin A, Pagès F, et al. DNA barcoding the medusozoa using mtCOI[J]. Deep-Sea Research Ⅱ: Topical Studies in Oceanography, 2010, 57(24/26): 2148-2156.
[31]Lewis C, Bentlage B. Clarifying the identity of the Japanese Habu-kurage,Chironexyamaguchii, sp. nov. (Cnidaria: Cubozoa: Chirodropida)[J]. Zootaxa, 2009, 2030: 59-65.
[32]Daly M, Brugler M R, Cartwright P, et al. The phylum Cnidaria: a review of phylogenetic patterns and diversity 300 years after Linnaeus[J]. Zootaxa, 2007, 1668: 127-182.
[33]Dawson M N. Some implications of molecular phylogenetics for understanding biodiversity in jellyfishes, with emphasis on Scyphozoa[J]. Hydrobiologia, 2004, 530(1):249-260.
Genetic analysis of common venomous Cubozoa and Scyphozoa in Thailand waters
Liu Ruijuan1,2,Xiao Jie1,2,Zhang Xuelei1,2,Charatsee Aungtonya3
(1.TheFirstInstituteofOceanography,StateOceanicAdministration,Qingdao266061,China;2.KeyLaboratoryofScienceandEngineeringforMarineEcologyandEnvironment,StateOceanicAdministration,Qingdao266061,China;3.PhuketMarineBiologicalCenter,Phuket83000,Thailand)
Abstract:The genetic diversity of common venomous Cubozoa and Scyphozoa jellyfish in the coastal waters of Thailand was investigated using the nuclear 18S and mitochondrial 16S rDNA genes. It was also compared about the capability of these two molecular markers to discriminate those common jellyfish species. There were 9 genetically distinctive species among the 32 samples collected from Thai waters, including 4 Cubozoa species, 4 Scyphozoans and 1 hydrozoan. However, it needs further information on the morphology and life cycle to determine the true taxonomic status of most species in this research. The K2P (Kimura 2-parameter) genetic distance was evaluated within and among the species based on the sequences of these two genes. The results showed much higher genetic divergence based on 16S gene (0-0.050 and 0.204-0.474 for intra-and inter-species genetic distances, respectively) compared with those using 18S gene (0-0.002 and 0.008-0.066 for intra-and inter-species genetic distances, respectively). At the same time, the A and T nucleotide composition of 16S gene was obviously higher than 18S gene. And significant variation of nucleotide composition was detected between two jellyfish classes using 16S gene, but not 18S gene. These results further indicated the relatively higher mutation rate of 16S gene, which could be applied for the genetic diversity studies among jellyfish species with close genetic relationship and the intra-species genetic population studies.
Key words:Scyphozoa; Cubozoa; genetic diversity; ribosomal RNA gene (rDNA); Thailand
收稿日期:2015-11-16;
修訂日期:2016-03-16。
基金項目:中國-東盟海上合作基金項目“瀕危海洋物種合作研究”;國家自然科學基金(41206162)。
作者簡介:劉瑞娟(1989—),女,山東省安丘市人,海洋生物學專業,主要從事水母分子生態學研究。E-mail:liurj1224@fio.org.cn *通信作者:張學雷,男,研究員,從事海洋生物、環境科學和生態學研究。E-mail:zhangxl@fio.org.cn
中圖分類號:Q953; Q958.8
文獻標志碼:A
文章編號:0253-4193(2016)06-0051-11
劉瑞娟,肖潔,張學雷,等. 泰國近海習見有毒立方水母和缽水母的遺傳分析[J].海洋學報,2016,38(6):51—61, doi:10.3969/j.issn.0253-4193.2016.06.006
Liu Ruijuan,Xiao Jie,Zhang Xuelei, et al. Genetic analysis of common venomous Cubozoa and Scyphozoa in Thailand waters [J]. Haiyang Xuebao,2016,38(6):51—61, doi:10.3969/j.issn.0253-4193.2016.06.006

