草酸脫羧酶的性質及應用研究進展
賀俊斌,林日輝,韋成昱,龍 寒,梁宇薇,陳陽陽,高 華,杜侶佩
(廣西民族大學海洋與生物技術學院,化學與生物轉化過程新技術廣西高校重點實驗室,廣西 南寧 530006)
草酸脫羧酶的性質及應用研究進展
賀俊斌,林日輝*,韋成昱,龍 寒,梁宇薇,陳陽陽,高 華,杜侶佩
(廣西民族大學海洋與生物技術學院,化學與生物轉化過程新技術廣西高校重點實驗室,廣西 南寧 530006)
草酸脫羧酶(oxalate decarboxylase,Oxdc)屬Cupin蛋白超家族,是一種包含Mn2+的均一聚合酶,能夠在沒有輔因子的條件下催化草酸轉化為甲酸和CO2,是植物、微生物中促使草酸代謝降解的主要酶之一。該酶已在農業、食品、工業生產、醫療和生物監測等領域得到了廣泛的應用。本文綜述近年來在Oxdc來源、結構、作用以及實際應用方面的研究,著重討論其在泌尿系統草酸鹽結石病癥方面的作用,為有效預防治療泌尿系統結石癥的研究提供理論參考。
草酸;草酸脫羧酶;Cupin蛋白超家族;泌尿系統
草酸(oxalic acid,OA)是自然界中酸性最強的有機二元羧酸,其分子式是HO2C-C O2H,它能溶于水和乙醇,不溶于乙醚,通常由植物、微生物通過水解草酰乙酸或氧化乙醛酸、抗壞血酸產生[1]。草酸是一種惰性的代謝終產物,雖然結構極其簡單,但其既可作為質子供體,又可作為電子供體,對金屬陽離子有非常強的螯合性[2],這些特性使之對動植物、微生物以及人類的生產生活產生了不同程度的危害,如造成糧食的減產[3]、蔬菜水果的營養缺失、造紙和啤酒工業管道堵塞[4-5]。同時,草酸在體內的蓄積可產生各種泌尿系統疾病,如腎結石、尿道結石和高草酸尿癥[6]等。因此,研究抑制草酸的產生、加速草酸的分解使其水平降低具有重要的科學和現實意義。
目前生物界降解草酸的酶包括草酸氧化酶(EC 1.2.3.4)、草酸脫羧酶(EC 4.1.1.2)和草酰輔酶A脫羧酶(EC 4.1.1.8),見圖1。這3 種降解草酸的酶中,由于草酸氧化酶和草酰輔酶A脫羧酶的提取條件苛刻、周期長,對酶保藏的穩定性有較高的要求,因此不適用于實驗室操作[7]。而Oxdc有廣泛的微生物來源,提取和反應條件要求簡單,所以得到了廣泛應用。
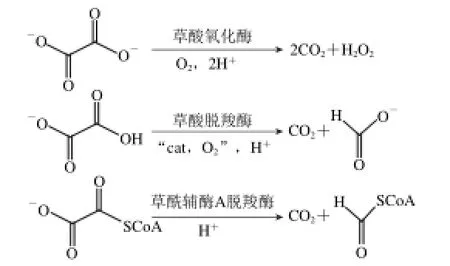
圖1 3種降解草酸的酶Fig.1 Three oxalate-degrading enzymes
草酸脫羧酶(oxalate decarboxylase,Oxdc,EC 4.1.1.2)是一種包含Mn2+的均一聚合酶,屬Cupin蛋白超家族;它可以在沒有輔因子的條件下催化草酸轉化為甲酸和CO2,是植物、微生物中草酸代謝降解的主要催化酶之一[8]。該酶最早由Shimazono[9]發現于白腐菌中,它主要來源于黑曲霉、核盤菌、金針菇和褐腐菌等真菌[10-11],一些細菌中也發現了該酶,但動物中只在豚鼠(天竺鼠)的肝臟中發現該酶[12]。枯草芽孢桿菌在低pH值下也能誘導合成Oxdc[13]。基于Oxdc對底物的高度特異性和酶促反應的高效性等特點,該酶已被成功地應用于農業、食品、工業生產、醫療等領域中。本文綜述近年來Oxdc的研究進展,重點討論了其在泌尿系統草酸鹽結石病癥方面的應用前景和意義。
1 草酸脫羧酶的性質
1.1 理化性質
目前為止已經分離和提純了10多種不同微生物來源的Oxdc[11],并初步研究了它們的生化性質。不同來源的Oxdc蛋白的分子質量有較大的差異,但一般都處于55~264 kD范圍內。不同微生物來源的Oxdc的最適反應pH值也有較大的差異,但一般都在1.1~3.0之間[14]。研究表明Oxdc的N端結構域主要介導草酸的分解,而C端結構域是否具有催化功能目前還不清楚,但定點突變及高場電子順磁共振光譜方面的證據表明C端結構域與酶活性的維持密切相關[15-16]。此外,來自Collybia velutipes的Oxdc具有較強的熱穩定性和抗去污劑的能力;該酶雖能特異地分解草酸,但對檸檬酸、醋酸、草乙酸、琥珀酸、蟻酸等都不產生作用[17-18]。
1.2 結構性質
隨著生物化學與分子生物學技術的發展,以及對Oxdc通過核磁共振、電子順磁共振(electron paramagnetic resonance,EPR)光譜、晶體衍射和蛋白序列測定等的研究,對Oxdc的空間結構特性有了較為深入的研究。Oxdc屬Cupin蛋白超家族的Bicupin亞族,Cupin蛋白具有相同的蛋白質前體和三級結構。Kesarwani等[19]通過5′-cDNA末端快速擴增(5′-rapid-amplification of cDNA ends,5′-RACE)技術和聚合酶鏈反應(polymerase chain reaction,PCR)技術首次克隆出oxdc基因的cDNA全長序列,并通過cDNA序列在其基因組文庫中篩選出該酶的基因組克隆,基因序列研究表明,該酶基因大小2.4 kb,與cDNA序列的比對分析發現,該基因含18 個外顯子和17 個內含子。所有內含子均含保守的5′和3′剪接點,大小為40~60 bp;外顯子為18~300 nt。
目前,來源于枯草芽孢桿菌的Oxdc已被克隆表達,對其空間結構及蛋白序列測定等的研究較為深入,并獲得了一些高分辨率的晶體結構X射線衍射圖。Anand等[20]通過對來源于Bacillus subtilis的Oxdc的氨基酸序列進行測定和分析,表明該酶大約由379 個氨基酸殘基組成,是一個由3 個Cupin二聚體單體組成的六聚體結構(圖2B);每個亞基分子質量為43 kD,每個單體包含兩個結合Mn2+的結構域(圖2A),該酶的N端和C端Cupin結構域具有27%的同源性,是由保守的6 股β-折疊形成的桶狀結構,具有相同的Mn2+結合殘基(由3 個組氨酸殘基及1 個谷氨酸殘基構成八面體環境)。其中結構域Ⅰ包含56~233 個氨基酸殘基,在Mn2+周圍除結合一分子H2O2和一分子甲酸外還結合4 個保守的氨基酸His95、His97、His140和Glu101;結構域Ⅱ包含234~379 個氨基酸殘基,在N端還有一段8~55 個的氨基酸序列,Mn2+周圍除結合兩分子H2O2外還結合4 個氨基酸 His273、His275、His319和Glu280。兩個結構域中的Mn2+相距大約26 ?[16,20]。之前的研究中Anand等[20]認為DomainⅡ是催化活性位點,因為只有該位點的Glu333是質子供體。而運用分子動力學模擬,研究Oxdc催化產生的CO2是如何從反應中心轉移到酶分子表面的結果表明DomainⅠ很可能是該酶的活性位點[21]。此外,研究證明在N端結構域上,由161~165氨基酸殘基構成一個稱為蓋子(lid)的五肽環。蓋子結構決定了催化反應過程酶活性中心的“開”、“閉”構型,這種蛋白構型的轉換使Glu-162接近活性中心的Mn2+,Glu-162可能是脫羧反應的質子供體[22]。蓋子結構還與酶促反應的特異性有關,對構成蓋子的氨基酸殘基進行定點突變,可以使酶的脫羧酶活性轉變為草酸氧化酶(oxalateoxidase,OXO)活性[1,15]。最近的研究表明,N末端和C末端結構域都可能具有催化去碳酸基反應的能力[23]。
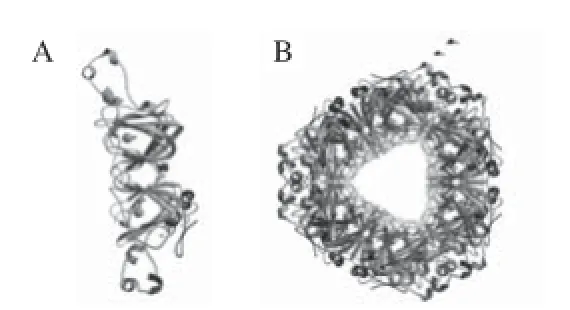
圖2 枯草芽孢桿菌草酸脫羧酶的空間結構預測圖Fig.2 Structural prediction of Bacillus subtilis Oxdc
2 草酸脫羧酶的催化機理
根據Oxdc結構特點,Anand[20]和Svedru?i?[24]等推測Oxdc催化脫羧反應歷程為:單質子化的草酸根及氧分子結合到酶的活性中心上,氧化活性中心的Mn2+轉變為Mn3+,同時伴隨形成Mn3+結合的草酸超氧化合物;活性中心谷氨酸殘基介導質子偶聯電子的轉移,使起始復合物形成Mn2+結合的草酸陰離子自由基;受到金屬離子及活性中心谷氨酸殘基及精氨酸殘基的影響,草酸分子C-C鍵異裂,發生脫羧反應,產生Mn2+結合的甲酸陰離子自由基;活性中心的谷氨酸殘基質子化甲酸陰離子自由基,使電子再次由金屬發生轉移,產生Mn3+結合的甲酸根,甲酸根和氧分子解離后,活性中心恢復為Mn2+。為研究Oxdc的結構域中Mn2+中心的電子性質,Campomanes等[25]采用定點突變及密度泛函理論/分子力學方法評估了Mn2+中心的EPR精細結構參數,建立的方法為運用量子力學法研究與蛋白質結合的金屬離子中心的電子結構特性提供了重要依據。草酸脫羧反應屬于非氧化脫羧反應,但分子氧對于酶的脫羧活性是必需的[26],目前運用膜進樣質譜法研究在分子氧消耗的條件下NO對Oxdc催化活性的作用,發現NO可逆的抑制Oxdc的催化活性,而X頻帶電子順磁共振波譜(X-band EPR)測定結果未能直接證明NO與Oxdc中Mn2+中心的相互作用,Mario等[27]認為該酶中可能存在一個隔離的分子氧結合位點。對于Oxdc催化過程中分子氧的作用,最近研究表明分子氧可能促進質子耦合電子的轉移[28]。
3 草酸脫羧酶的應用
3.1 Oxdc在農業生產中的應用
Oxdc在農業生產中的應用主要是控制由草酸毒素引起的植物病害,其中以控制由菌核病菌(Sclerotinia sclerotiorum)侵染植物的研究備受矚目。Oxdc可以催化草酸轉化為無毒的甲酸,因此在植物中引入oxdc基因對抗高草酸分泌積累造成的植物病害是目前重要的研究方向。隨著基因工程技術的不斷發展,越來越多的植物體基因組中轉入了oxdc基因,從而提高了植株對菌核病的抗性。Kesarwani等[19]運用5′-RACE技術從F. velutipes中擴增出oxdc基因,并將此基因轉入煙草和番茄中,經接種實驗鑒定,發現表型正常且能夠穩定遺傳的轉基因植株對菌核病具有明顯的抗性。Walz等[29]將來自云芝(Trametes versicolor)的oxdc基因轉入到煙草中后,發現菌核菌致病過程中草酸的含量減少,并且減緩了菌核病的擴散速率。Dias等[30]運用農桿菌介導法在萵苣(Lactuca sativa)中轉入來自Flammulina sp.的oxdc基因后,通過離體葉片接種菌核病菌進行檢測發現轉基因植株對S. sclerotiorum表現出抗性。Jin Zhaoxia等[31]用表達Oxdc的Pandorea sp. OXJ-11菌液處理油菜(Brassica napus)的離體葉片,結果后者表現出抑制菌核病擴散的現象。此外,陳曉婷等[32]從已測序菌株枯草芽孢桿菌中克隆得到oxdc基因Yvrk,并構建其植物表達載體,通過農桿菌浸花轉化擬南芥,為緩解真菌病害提供了有益的借鑒。最近Cunha等[33]將Flammulina sp.的oxdc基因轉入到大豆中,T2代自花授粉所獲得的轉基因系通過檢測,發現對菌核病表現出高抗性;離體葉片接種表明,轉基因植株相對于對照組都明顯表現出延遲菌核病斑擴散的現象;反轉錄聚合酶鏈式反應(reverse transcription polymerase chain reaction,RT-PCR)實驗結果顯示oxdc基因的表達與抗菌核病的能力有關,轉基因植株抗性水平與基因表達水平成正相關。通過從Flammulina velutipes中克隆出oxdc基因(FvOXDC)轉入到煙草中,對轉基因煙草中的草酸含量進行測定發現,較對照組轉基因煙草中草酸的含量減少約18%~75%;并對離體葉片觀察發現FvOXDC對由草酸和NLP誘發的植物程序性死亡表現出明顯的抗性[34]。而舒佳賓[35]從金針菇菌中克隆出oxdc基因,通過農桿菌介導法和植物組織培養技術將該基因轉入甘藍型油菜,對成功的轉基因苗運用葉片離體菌核菌技術接種后發現,轉基因植株具有明顯的延遲和抵御菌核病侵染的能力。最近,Chakraborty等[36]將來自Flammulina velutipes的oxdc基因轉入到番茄后,通過對轉基因番茄中的草酸含量及營養成分如VC、蘋果酸等分析發現,與野生型番茄相比,轉基因番茄中草酸的減少量高達90%,而營養成分含量也有所增加,從而為提高農作物營養價值的研究提供了重要依據。
3.2 Oxdc在工業生產中的應用
草酸的積累形成草酸鹽沉淀而帶來危害性的影響是工業生產中的一個常見問題。尤其是在造紙工業中,由于其過程是在封閉系統中進行,木質纖維素被強氧化劑氧化最終分解為草酸與廢水中的Ca2+結合造成草酸鈣的沉淀積累,堵塞系統管道,阻塞濾網孔,引起熱能的損失以及循環水流動不暢等問題[4-5]。為此,從真菌中提取的Oxdc被廣泛地用于去除造紙過程中產生的草酸。2011年瑞典制漿造紙研究所Nilvebrant等[37]利用Oxdc處理造紙過程中的草酸鈣沉積取得了良好的效果。Cassland等[5]發現從黑曲霉(A. niger)中提取的Oxdc在處理二氧化氯漂白紙漿流程中產生的草酸時,與植物中提取的草酸氧化酶相比,效果更明顯且不受氯離子的抑制。最近研究發現來源于Trametes versicolor的Oxdc在工業漂白濾過中的作用較其他草酸降解酶,尤其是較來源于A. niger的效果更明顯[38]。此外,在啤酒工業中,利用Oxdc可去除生產過程中草酸鹽積累產生的沉淀廢物,如啤酒石,從而改善啤酒生產的工藝[39]。
3.3 Oxdc在生物監測中的應用
基于Oxdc對底物的高度特異性,其被廣泛應用于測定各種材料中草酸的含量,Haas等[40]利用來自F. velutipes中的Oxdc首次成功檢測了麥芽汁和啤酒中的草酸含量。隨著人類草酸尿和泌尿系統草酸鹽結石發病率的增加,研究運用Oxdc檢測尿液和血液中草酸含量的方法受到關注。Vadgama等[41]利用丙烯酰胺凝膠包埋Oxdc與CO2電極結合研制了Oxdc電極,成功應用于檢測尿液中草酸的含量。Bishop等[42]通過火焰電離等技術檢測Oxdc分解草酸后產生的CO2濃度來測定尿液中草酸的濃度,在臨床上得到了應用。運用酶/生物熒光檢測技術結合來自F. velutipes中的Oxdc檢測血漿中草酸的濃度也得到良好的效果[43]。此外,結合Oxdc和甲酸脫氫酶,聯合分光光度分析法測定尿液和血液中草酸的濃度,并且結合草酸濃度自動檢測分析技術,在臨床上得到廣泛應用[44-46]。隨著研究的深入,Oxdc酶試劑盒及生物傳感器不斷出現,在臨床上得以應用,但由于該酶的成本高,阻礙了其推廣和發展。有研究利用原核表達系統對Oxdc進行誘導表達,每升發酵液收獲Oxdc活力1 640 U(約12.33 mg酶蛋白),遠高于野生菌的誘導表達量[47]。然而,由于該酶的獲得受發酵條件如培養基成分、溫度、pH值等的影響,存在產量低、品質差等問題。因此,在提高Oxdc酶的產量和從新物種中挖掘新的oxdc基因等方面需進一步深入研究。
3.4 Oxdc在預防治療泌尿系統結石癥方面的應用
泌尿系統結石是一種常見的異常生物礦化現象,根據地理環境,生活環境,遺傳和飲食條件的不同,結石發病率在3%~14%之間。此外,治療泌尿系結石存在的另一個重要問題是復發率高,患者在被檢出患泌尿系結石并經過治療后,短則半個月,長至6 年,60%~80%的病人都會出現復發[48]。泌尿系統結石形成的主要原因是人們攝入的飲食中含有可形成結石的相關成分(草酸)過多,如葡萄、茶葉、橘子、番茄、菠菜、草莓等都具有較高含量的草酸。研究表明,泌尿系統結石中大約有70%~80%為草酸鈣結石[6]。目前雖然這類疾病可通過物理或化學的方法治療,并取得了一定療效,但結石的復發率高、治療費用昂貴,且物理化學方法對人體副作用較大。導致草酸鈣結石的一個重要原因就是人體內缺乏降解草酸的代謝途徑,而生物酶具有作用高效、專一和溫和等特點,因此,采用Oxdc對人體內草酸進行降解已成為預防治療草酸鈣結石癥的重要研究方向。國外的阿爾特斯制藥公司運用交聯酶晶體法(cross-linked enzyme crystals,CLECs)制成Oxdc酶制劑用于治療草酸鹽結石癥,取得了一定的療效[49]。Sidhu等[50]運用噴霧干燥法制成重組Oxdc顆粒用于減少胃腸吸收的草酸含量,取得良好效果,可用于預防和治療高草酸尿癥等。Mufarrij等[51]利用體外實驗設置不同pH值模擬消化道環境,研究了Oxdc制劑Oxazyme對菠菜中草酸的降解能力,與對照組相比,Oxazyme能顯著降低菠菜中草酸的含量。Grujic等[52]使用基因敲除小鼠為模型,通過口服交聯Oxdc晶體(Oxdc-CLEC),有效地防止了小鼠腎鈣質沉著癥及尿石癥的發生。Jeong等[53]模擬高草酸尿癥模型,通過給小鼠口服來源于B. subtilis的重組Oxdc,有效的減少了尿液中草酸的水平。Cowley等[54]以大鼠及狗為實驗動物,口服酶制劑Oxazyme(OC4)連續飼喂14 d,評價了這類用于降解動物消化道中草酸的酶制劑的可能毒性,實驗表明均無可見有害作用水平。此外,在乳酸菌NC8中異源表達的Oxdc以及在重組L. plantarum WCFS1中利用同源信號肽誘導異源表達的Oxdc可被作為益生菌降解腸道內飲食中的草酸,以預防高草酸尿癥的發生[55-57]。以上這些研究表明,使用Oxdc制劑降解消化道內草酸,減少消化系統對草酸鹽的吸收,從而降低草酸鈣結石風險的方法是可行的。
但Oxdc用于胃腸道內降解草酸時,容易受胃酸、蛋白酶消化的影響而失去活性,因此,提高該酶在應用過程中的穩定性,改善其使用性能是亟待解決的問題。為解決這一問題,對該酶進行固定化或化學修飾是一重要研究方向。林日輝等[58-59]提供了一種硫酸銨促進Eupergit C250固定化Oxdc的方法,其制備的固定化酶具有較好的操作性能、易于回收重復使用,可應用于食品或造紙等工業過程中草酸的降解。運用無載體固定化方法制備交聯Oxdc聚集體,對其酶學性質研究表明交聯Oxdc聚集體的耐酸性、耐熱性、耐胰蛋白酶降解能力較游離酶均有提高[60]。梁躍[61]、韋成昱[62]等研究了右旋糖酐對Oxdc的化學修飾,并對修飾Oxdc的熱穩定性、pH值穩定性及對胰蛋白酶抗性進行研究發現,修飾后酶的性質明顯優于修飾前。此外,在活體應用中,利用Oxdc降解泌尿結石時面臨酶蛋白易被稀釋流失的問題,對此本實驗組正在研究對Oxdc進行修飾改性,使之具有對草酸鈣晶體特異性或較強烈的吸附能力,有效吸附固定于草酸鈣結晶物上,從而較長時間地維持草酸鈣結石部位酶制劑的濃度,為Oxdc作為預防、治療結石的酶制劑的開發應用提供重要參考。
4 結 語
目前,草酸脫羧酶(Oxdc)基于其對底物的高度特異性和酶促反應的高效性等特點,已被成功地應用于農作物抗病研究、工業生產中草酸鹽沉淀的去除以及醫療等領域中,但對其研究尚存在許多不足和問題。如對Oxdc的三維空間結構、生理特性等生化性質的研究主要是建立在來源于B. subtilis的Oxdc基礎之上,對其他物種中的Oxdc研究并不深入;另外,該酶在商業化應用中存在產量低、成本高等缺點。因此,通過分子生物學技術等從新物種中挖掘oxdc基因并建立該酶的異源表達系統,挖掘具備不同生化性質的Oxdc,以及通過生物工程技術等優化產酶條件、提高酶的產量和品質以降低成本等方面,還需要更深入的研究。
[1] JUST V J, STEVENSON C E M, BOWATER L, et al. A closed conformation of Bacillus subtilis oxalate decarboxylase OxdC provides evidence for the true identity of the active site[J]. Journal of Biological Chemistry, 2004, 279(19): 19867-19874.
[2] MUNIR E, YOON J J, TOKIMATSU T, et al. A physiological role for oxalic acid biosynthesis in the wood-rotting basidiomycete Fomitopsis palustris[J]. Proceedings of the National Academy of Sciences, 2001, 98(20): 11126-11130.
[3] DUTTON M V, EVANS C S. Oxalate production by fungi: its role in pathogenicity and ecology in the soil environment[J]. Canadian Journal of Microbiology, 1996, 42(9): 881-895.
[4] SJ?DE A, WINESTRAND S, NILVEBRANT N O, et al. Enzymebased control of oxalic acid in the pulp and paper industry[J]. Enzyme and Microbial Technology, 2008, 43(2): 78-83.
[5] CASSLAND P, SJ?DE A, WINESTRAND S, et al. Evaluation of oxalate decarboxylase and oxalate oxidase for industrial applications[J]. Applied Biochemistry and Biotechnology, 2010, 161(1/8): 255-263.
[6] COE F L, EVAN A, WORCESTER E. Kidney stone disease[J]. Journal of Clinical Investigation, 2005, 115(10): 2598-2608.
[7] KOTSIRA V P, CLONIS Y D. Oxalate oxidase from barley roots: purification to homogeneity and study of some molecular, catalytic, and binding properties[J]. Archives of Biochemistry and Biophysics, 1997, 340(2): 239-249.
[8] 趙樹田, 張士青. 草酸代謝酶的研究進展[J]. 上海交通大學學報: 醫學版, 2007, 27(10): 1274-1277.
[9] SHIMAZONO H. Oxalic acid decarboxylase, a new enzyme from the mycelium of wood destroying fungi[J]. Journal of Biochemistry, 1955, 42(3): 321-340.
[10] CHAKRABORTY S, CHAKRABORTY N, JAIN D, et al. Active site geometry of oxalate decarboxylase from Flammulina velutipes: role of histidine-coordinated manganese in substrate recognition[J]. Protein Science, 2002, 11(9): 2138-2147.
[11] M?KEL? M R, HILDéN K, LUNDELL T K. Oxalate decarboxylase: biotechnological update and prevalence of the enzyme in filamentous fungi[J]. Applied Microbiology and Biotechnology, 2010, 87(3): 801-814. [12] MURTHY M S R, TALWAR H S, NATH R, et al. Oxalate decarboxylase from guinea-pig liver[J]. Ircs Medical Science-Biochemistry, 1981, 9(8): 683-684.
[13] TANNER A, BORNEMANN S. Bacillus subtilis YvrK is an acidinduced oxalate decarboxylase[J]. Journal of Bacteriology, 2000, 182(18): 5271-5273.
[14] 曹茂新, 洪楓, 朱利民. 草酸脫羧酶及其應用[J]. 中國生物工程雜志, 2005(增刊1): 170-175.
[15] BURRELL M R, JUST V J, BOWATER L, et al. Oxalate decarboxylase and oxalate oxidase activities can be interchanged with a specifi city switch of up to 282 000 by mutating an active site lid[J]. Biochemistry, 2007, 46(43): 12327-12336.
[16] MOOMAW E W, ANGERHOFER A, MOUSSATCHE P, et al. Metal dependence of oxalate decarboxylase activity[J]. Biochemistry, 2009, 48(26): 6116-6125.
[17] MEHTA A, DATTA A. Oxalate decarboxylase from Collybia velutipes. Purifi cation, characterization, and cDNA cloning[J]. Journal of Biological Chemistry, 1991, 266(35): 23548-23553.
[18] 舒佳賓, 官春云. 草酸脫羧酶的性質及應用[J]. 中國油料作物學報, 2012, 34(1): 109-113.
[19] KESARWANI M, AZAM M, NATARAJAN K, et al. Oxalate decarboxylase from Collybia velutipes molecular cloning and its overexpression to confer resistance to fungal infection in transgenic tobacco and tomato[J]. Journal of Biological Chemistry, 2000, 275(10): 7230-7238.
[20] ANAND R, DORRESTEIN P C, KINSLAND C, et al. Structure of oxalate decarboxylase from Bacillus subtilis at 1.75 ? resolution[J]. Biochemistry, 2002, 41(24): 7659-7669.
[21] KARMAKAR T, PERIYASAMY G, BALASUBRAMANIAN S. CO2Migration pathways in oxalate decarboxylase and clues about its active site[J]. The Journal of Physical Chemistry B, 2013, 117(41): 12451-12460.
[22] MUTHUSAMY M, BURRELL M R, THORNELEY R N F, et al. Real-time monitoring of the oxalate decarboxylase reaction and probing hydron exchange in the product, formate, using Fourier transform infrared spectroscopy[J]. Biochemistry, 2006, 45(35): 10667-10673.
[23] TABARES L C, GA¨TJENS J, HUREAU C, et al. pH-dependent structures of the manganese binding sites in oxalate decarboxylase as revealed by high-fi eld electron paramagnetic resonance[J]. The Journal of Physical Chemistry B, 2009, 113(26): 9016-9025.
[24] SVEDRU?I? D, JóNSSON S, TOYOTA C G, et al. The enzymes of oxalate metabolism: unexpected structures and mechanisms[J]. Archives of Biochemistry and Biophysics, 2005, 433(1): 176-192.
[25] CAMPOMANES P, KELLETT W F, EASTHON L M, et al. Assigning the EPR Fine Structure parameters of the Mn (Ⅱ) centers in Bacillus subtilis oxalate decarboxylase by site-directed mutagenesis and DFT/MM calculations[J]. Journal of the American Chemical Society, 2014, 136(6): 2313-2323.
[26] TANNER A, BOWATER L, FAIRHURST S A, et al. Oxalate decarboxylase requires manganese and dioxygen for activity overexpression and characterization of Bacillus subtilis YvrK and YoaN[J]. Journal of Biological Chemistry, 2001, 276(47): 43627-43634.
[27] MARIO E G, NIGEL G J. Nitric oxide reversibly inhibits Bacillus subtilis oxalate decarboxylase[J]. Chemical Communications, 2011, 47(11): 3111-3113.
[28] SAYLOR B T, REINHARDT L A, LU Z, et al. A structural element that facilitates proton-coupled electron transfer in oxalate decarboxylase[J]. Biochemistry, 2012, 51(13): 2911-2920.
[29] WALZ A, ZINGEN-SELL I, THEISEN S, et al. Reactive oxygen intermediates and oxalic acid in the pathogenesis of the necrotrophic fungus Sclerotinia sclerotiorum[J]. European Journal of Plant Pathology, 2008, 120(4): 317-330.
[30] DIAS B B A, CUNHA W G, MORAIS L S, et al. Expression of an oxalate decarboxylase gene from Flammulina sp. in transgenic lettuce (Lactuca sativa) plants and resistance to Sclerotinia sclerotiorum[J]. Plant Pathology, 2006, 55(2): 187-193.
[31] JIN Zhaoxia, WANG Changhai, CHEN Wenfu, et al. Induction of oxalate decarboxylase by oxalate in a newly isolated Pandoraea sp. OXJ-11 and its ability to protect against Sclerotinia sclerotiorum infection[J]. Canadian Journal of Microbiology, 2007, 53(12): 1316-1322.
[32] 陳曉婷, 陳芳芳, 鄭麟, 等. 枯草芽孢桿菌草酸脫羧酶轉化擬南芥研究初報[J]. 中國農學通報, 2008, 24(2): 53-58.
[33] CUNHA W G, TINOCO M L P, PANCOTI H L, et al. High resistance to Sclerotinia sclerotiorum in transgenic soybean plants transformed to express an oxalate decarboxylase gene[J]. Plant Pathology, 2010, 59(4): 654-660.
[34] da SILVA L F, DIAS C V, CIDADE L C, et al. Expression of an oxalate decarboxylase impairs the necrotic effect induced by Nep1-like protein (NLP) of Moniliophthora perniciosa in transgenic tobacco[J]. Molecular Plant-Microbe Interactions, 2011, 24(7): 839-848.
[35] 舒佳賓. 草酸脫羧酶對油菜抗菌核病的研究[D]. 長沙: 湖南農業大學, 2012.
[36] CHAKRABORTY N, GHOSH R, GHOSH S, et al. Reduction of oxalate levels in tomato fruit and consequent metabolic remodeling following overexpression of a fungal oxalate decarboxylase[J]. Plant Physiology, 2013, 162(1): 364-378.
[37] NILVEBRANT N O, REIMANN A, de SOUSA F, et al. Enzymatic degradation of oxalic acid for prevention of scaling[J]. Progress in Biotechnology, 2002, 21: 231-238.
[38] WINESTRAND S, GANDLA M L, HONG F, et al. Oxalate decarboxylase of Trametes versicolor: biochemical characterization and performance in bleaching filtrates from the pulp and paper industry[J]. Journal of Chemical Technology and Biotechnology, 2012, 87(11): 1600-1606.
[39] HIATT W R, OWADES J L. Oxalic acid removal in beer production: US, 4652452 A[P/OL]. 1987-03-24[2014-01-08]. http://www.google. co.uk/patents/US4652452.
[40] HAAS G J, FLEISCHMAN A I. Oxalate in beer, the rapid enzymatic determination of oxalate in wort and beer[J]. Journal of Agricultural and Food Chemistry, 1961, 9(6): 451-452.
[41] VADGAMA P, SHELDON W, GUY J M, et al. Simplified urinary oxalate determination using an enzyme electrode[J]. Clinica Chimica Acta, 1984, 142(2): 193-201.
[42] BISHOP M, FREUDIGER H, LARGIADER U, et al. Conductivometric determination of urinary oxalate with oxalate decarboxylase[J]. Urological Research, 1982, 10(4): 191-194.
[43] PARKINSON I S, KEALEY T, LAKER M F. The determination of plasma oxalate concentrations using an enzyme/bioluminescent assay[J]. Clinica Chimica Acta, 1985, 152(3): 335-345.
[44] COSTELLO J, HATCH M, BOURKE E. An enzymic method for the spectrophotometric determination of oxalic acid[J]. The Journal of Laboratory and Clinical Medicine, 1976, 87(5): 903-908.
[45] HATCH M, BOURKE E, COSTELLO J. New enzymic method for serum oxalate determination[J]. Clinical Chemistry, 1977, 23(1): 76-78.
[46] LANGMAN L J, ALLEN L C. An enzymatic method for oxalate automated using the Hitachi 911 analyzer[J]. Clinical Biochemistry, 1998, 31(5): 429-432.
[47] 林日輝, 許麗莉, 農勉, 等. 重組草酸脫羧酶的表達及酶學性質研究[J].食品與發酵工業, 2011, 37(2): 57-61.
[48] 代海濤, 陳志強, 葉章群. 草酸, 草酸鈣晶體-上皮細胞相互作用與腎結石[J]. 國際泌尿系統雜志, 2006, 26(2): 254-257.
[49] SHENOY B C, CACHERO T G, SHIN J, et al. Crystallized oxalate decarboxylase and methods of use[P/OL]. 2012-09-04 [2014-01-15]. http://www.google.co.uk/patents/CN103272225A?cl=en&hl=zh-CN.
[50] SIDHU H, COWLEY A B, GOLANDER C, et al. Purification and isolation of recombinant oxalate degrading enzymes and spray-dried particles containing oxalate degrading enzymes[P/OL]. 2013-08-22 [2014-01-17]. http://www.faqs.org/patents/app/20130216515.
[51] MUFARRIJ P W, LANGE J N, KNIGHT J, et al. Second place: the effects of oxazyme on oxalate degradation: results and implications of in vitro experiments[J]. Journal of Endourology, 2013, 27(3): 284-287.
[52] GRUJIC D, SALIDO E C, SHENOY B C, et al. Hyperoxaluria is reduced and nephrocalcinosis prevented with an oxalate-degrading enzyme in mice with hyperoxaluria[J]. American Journal of Nephrology, 2008, 29(2): 86-93.
[53] JEONG B C, HAN D H, SEO S I, et al. Yvrk gene recombinant E. coli reduce the concentration of urine oxalate in transient Hyperoxaluria rat model[J]. The Journal of Urology, 2009, 181(4): 660.
[54] COWLEY A B, POAGE D W, DEAN R R, et al. 14-day repeat-dose oral toxicity evaluation of oxazyme in rats and dogs[J]. International Journal of Toxicology, 2010, 29(1): 20-31.
[55] KOLANDASWAMY A, GEORGE L, SADASIVAM S. Heterologous expression of oxalate decarboxylase in Lactobacillus plantarum NC8[J]. Current Microbiology, 2009, 58(2): 117-121.
[56] ANBAZHAGAN K, SASIKUMAR P, GOMATHI S, et al. in vitro degradation of oxalate by recombinant Lactobacillus plantarum expressing heterologous oxalate decarboxylase[J]. Journal of Applied Microbiology, 2013, 115(3): 880-887.
[57] SASIKUMAR P, GOMATHI S, ANBAZHAGAN K, et al. Secretion of biologically active heterologous oxalate decarboxylase (OxdC) in Lactobacillus plantarum WCFS1 using homologous signal peptides[J]. BioMed Research International, 2013: 280432. doi: 10.1155/2013/280432.
[58] 林日輝. 一種硫酸銨促進Eupergit C 250固定化草酸脫羧酶的方法:中國, 102174502A[P]. 2011-09-07.
[59] LIN Rihui, WU Ruchun, HUANG Xinlin, et al. Immobilization of oxalate decarboxylase to eupergit and properties of the immobilized enzyme[J]. Preparative Biochemistry & Biotechnology, 2011, 41(2): 154-165.
[60] 梁躍, 林日輝, 黃文勤, 等. 交聯草酸脫羧酶聚集體的制備及其性質[J].食品科學, 2013, 34(1): 215-219.
[61] 梁躍. 草酸脫羧酶的無載體固定化及化學修飾的研究[D]. 南寧: 廣西民族大學, 2012.
[62] 韋成昱, 林日輝, 龍寒, 等. 右旋糖酐對草酸脫羧酶的修飾研究[J].食品工業科技, 2014, 35(13): 195-199.
Properties and Applications of Oxalate Decarboxylase
HE Junbin, LIN Rihui*, WEI Chengyu, LONG Han, LIANG Yuwei, CHEN Yangyang, GAO Hua, DU Lüpei
(Key Laboratory of New Techniques for Chemical and Biological Conversion Process, School of Marine Sciences and Biotechnology, Guangxi University for Nationalities, Nanning 530006, China)
Oxalate decarboxylase (Oxdc) is a homogenous polymerase with manganese ion and belongs to the Cupin protein superfamily. In plants and microorganisms, Oxdc is one of the main enzymes which can catalyze the decomposition of oxalate into formic acid and CO2without cofactors. It has been applied widely in agriculture, food, industrial process, medical, biological monitoring and other fi elds. In this paper, the source, structure, function and practical applications of Oxdc are reviewed, with particular discussion on the role and advantages of Oxdc in urinary oxalate stones. This will provide important theoretical reference for the effective prevention and treatment of urinary tract stones.
oxalic acid; oxalate decarboxylase; Cupin protein superfamily; urinary system
Q55
A
1002-6630(2015)01-0262-06
10.7506/spkx1002-6630-201501050
2014-03-12
教育部留學回國人員科研啟動基金項目(教外司留[2012]1707號);廣西民族大學科研項目(2013MDYB031)
賀俊斌(1992—),男,碩士研究生,研究方向為酶工程。E-mail:hejunbin556298@163.com
*通信作者:林日輝(1972—),男,研究員,博士,研究方向為發酵工程、酶工程。E-mail:rihuilin@aliyun.com

