DNA 條形碼在紫斑谷螟幼蟲鑒定中的應用
郭鳳娟,陽建春,胡詩佳,蘇棟棟,李詠施
(廣東省昆蟲研究所華南野生動物物種鑒定中心,廣東省野生動物保護與利用公共實驗室,廣東省農業害蟲綜合治理重點實驗室,廣州 510260)
紫斑谷螟Pyralis farinalis 又名粉螟、粉縞螟、大斑粉螟、縞螟蛾,屬鱗翅目Lepidoptera 螟蛾科Pyralidae 螟蛾亞科Pyralinae 螟蛾屬Pyralis 的昆蟲,全世界廣泛分布(王平遠,1980;王永衛,1982;尚小麗,2011)。紫斑谷螟作為主要的倉庫害蟲之一(趙養昌,1982),它主要為害倉庫中的小麥、稻米、高粱、玉米、黃豆、麩皮等,造成嚴重的經濟損失。
倉庫害蟲種類繁多,為了有效控制倉庫害蟲的發生,首先要對其物種做出正確鑒別。利用傳統的形態學方法進行物種鑒定具有一定的局限性,因為不僅要求蟲體外形完整,而且對不同生長期的蟲體進行正確鑒定需要較強的專業知識和豐富的經驗。DNA 條形碼的產生,為物種鑒定提供了新的研究方法和手段(Frézal,2008)。2003年Hebert 首次將DNA 條形碼技術應用在約200個親緣關系較近的鱗翅目昆蟲的分類鑒定中,結果成功地鑒別出了這些鱗翅目昆蟲(李青青,2010)。目前,專為鱗翅目昆蟲而建的DNA 條形碼網站已收錄了22175個物種的510897 條DNA 條形碼序列,為科研工作者提供了大量的科研素材。在過去的研究中,紫斑谷螟僅被作為倉庫害蟲加以記錄。近年來有研究者發現,紫斑谷螟是生產蟲茶的主要昆蟲種類,并對其形態特征和生物學特性進行了詳細的觀察(尚小麗,2011)。目前,尚未有研究者通過分子生物學方法來研究紫斑谷螟及其幼蟲。本文旨在利用DNA 條形碼技術鑒定殘缺的紫斑谷螟幼蟲,為倉庫害蟲的防治提供理論依據。
1 材料與方法
1.1 材料與方法
1 份殘缺的昆蟲幼體來自于廣州市某動物食品公司倉庫。
1.2 DNA 提取
將殘缺的昆蟲幼體置于滅菌的1.5 mL EP 管內,用PBS 緩沖液清洗樣品,去除污垢,離心去上清,用滅菌的眼科剪將沉淀剪成小碎塊,然后使用Biomage 公司的基因組DNA 小量提取試劑盒進行操作,提取樣品組織中的DNA。
1.3 PCR 擴增
以DNA 條形碼通用引物LCO1490/HCO 2198(上游引物LCO1490 堿基序列為5'-GGTCAAC AAATCATAAAGATATTGG-3'下游引物HCO2198堿基序列為5'-TAAACTTCAGGGTGACCAAAAAA TCA-3')(Hebert,2003)進行PCR 擴增。反應體系采用50 μL,其中10 μM/mL 上下游引物各2 μL,Premix Taq 25 μL,DNA 模板5 μL,最后用滅菌水補至50 μL。擴增體系為94℃預變性1 min;94℃變性1 min,45℃復性1 min 30 s,72℃延伸1 min 30 s,循環次數為5;第一次循環結束后進入第二次循環:94℃變性1 min,50℃復性1 min 30 s,72℃延伸1 min,循環次數為35,最后72℃延伸7 min。擴增產物經1.0%瓊脂糖凝膠電泳,在凝膠成像系統上分析電泳結果。PCR 產物送上海英濰捷基公司進行純化并測序。
1.4 序列比對及分析
用BioEdit 軟件打開測序獲得的序列,觀察序列峰圖,將兩端的低信號序列和出現雙峰的堿基棄掉,得到約660 bp 的樣品序列,輸入NCBI 數據庫中進行序列比對。然后將樣品序列與從GenBank數據庫中下載的螟蛾屬5個種的14 條已知序列(見表1)導入BioEdit 軟件中,對已知序列和樣品序列進行整合,以ClustalW Multiple Alignment 進行對齊處理。用MEGE 6.05 分析序列的變異位點、堿基組成,以Kimura 雙參數模型計算種內及種間遺傳距離。利用MEGE 6.05 軟件中的鄰接法(Neighbor-Joining)構建系統發育樹,對各分支置信度(bootstrap)進行1000 次以上的重復檢驗。
2 結果與分析
2.1 PCR 擴增結果
以殘缺昆蟲幼蟲的基因組DNA 為模板,利用DNA 條形碼通用引物LCO1490/HCO 2198 擴增線粒體COⅠ基因片段,PCR 產物在1.0%瓊脂糖凝膠中的電泳結果如圖1 所示,從送檢樣品中能擴增到目的基因的條帶。經純化、測序后得到約660 bp長度的序列。

圖1 COⅠ通用引物PCR 擴增產物電泳圖譜Fig.1 Agarose electrophoresis pattern of universal primer-directed PCR
2.2 序列比對分析
樣品序列經與NCBI 數據庫中的序列比對發現,樣品序列與紫斑谷螟的序列相似性最高。然后將樣品序列與下載的14 條序列經軟件分析后,得到639 bp 的序列,其中保守位點504個,可變位點135個,簡約位點134個,單個突變位點1個。樣品序列的堿基含量為40.1%T,18.6%C,26.1%A,15.2%G,A+T 含量為66.2%,C+G含量為33.8%,存在明顯的A、T 堿基偏好性。
不同種序列之間比對結果發現(見表2),5種螟蛾的種間遺傳距離為0.066-0.162,平均為0.127,種內遺傳距離為0.000-0.003,平均為0.001。種間遺傳距離遠遠大于種內遺傳距離,COⅠ基因能夠有效的區別該類物種。樣品序列與紫斑谷螟的序列相似性最高,達到99.8%,遺傳距離最小,為0.002-0.003。
2.3 構建系統發育樹
基于K2P 模型,使用鄰接法構建系統發育樹,并對各分支置信度(bootstrap)進行1000 次以上的重復檢驗。結果顯示(見圖2),殘缺的幼蟲樣品與紫斑谷螟聚為一支。
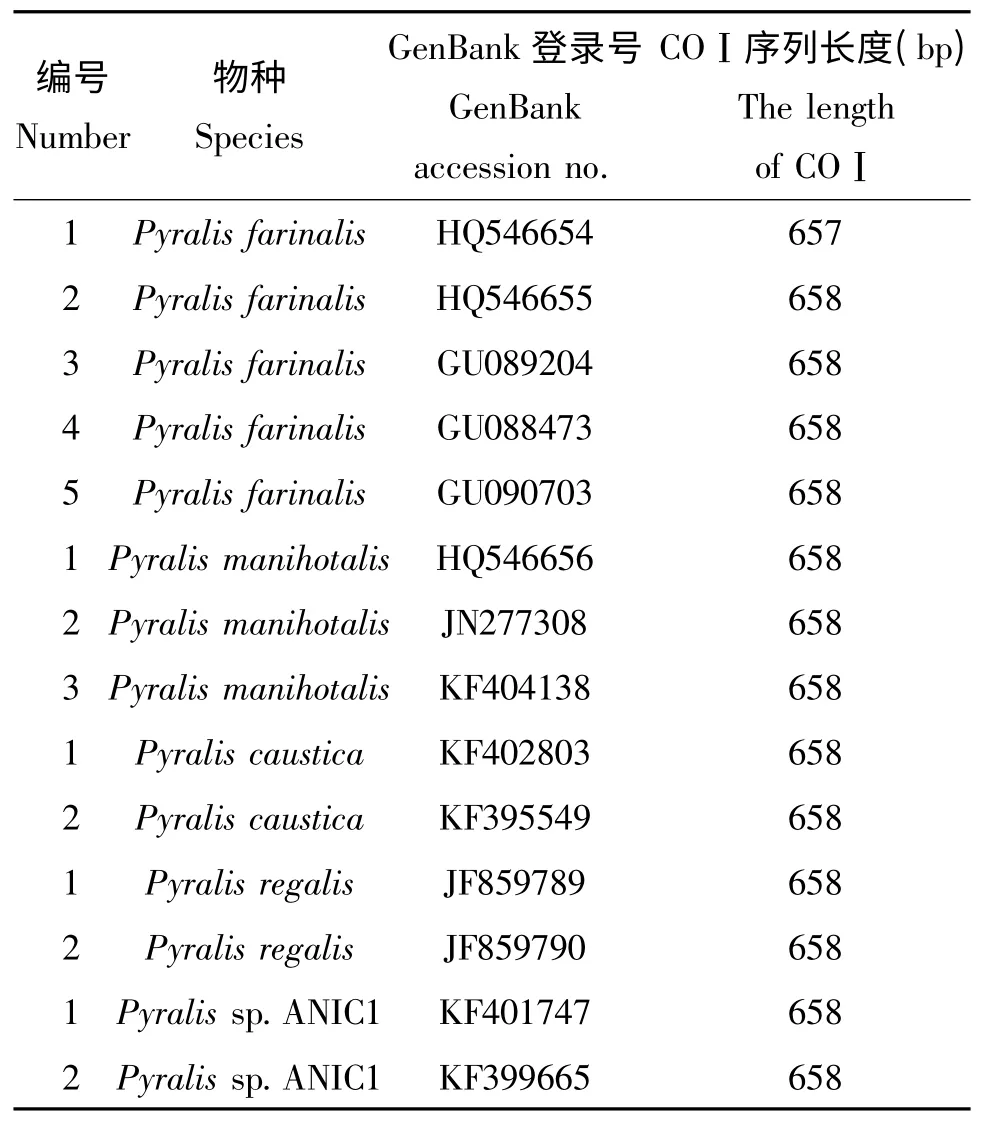
表1 下載的GenBank 數據庫中5種螟蛾COⅠ基因片段相關信息Table1 The COⅠgene information of five species of Pyralis from GenBank database
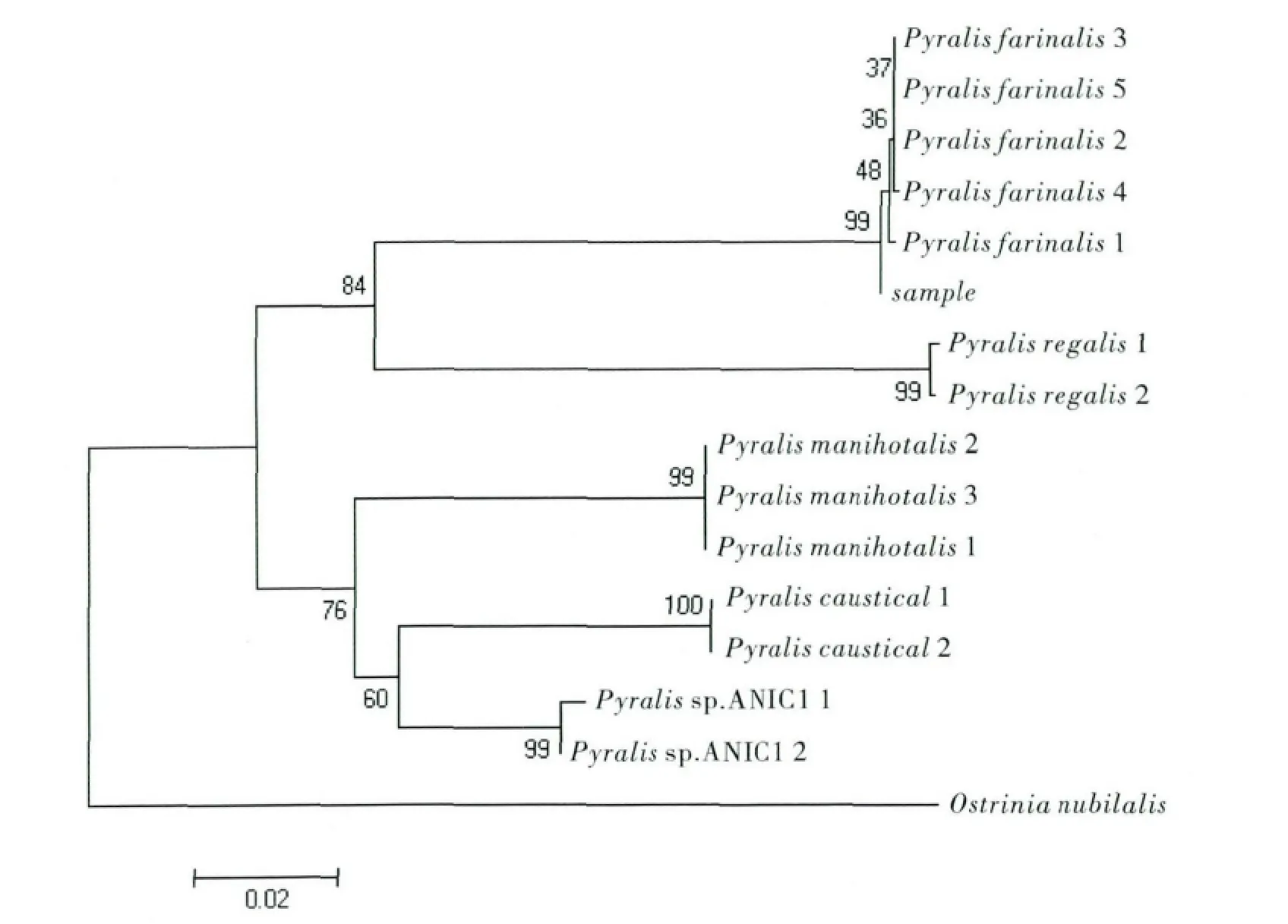
圖2 基于COⅠ基因部分序列以鄰接法構建的5種螟蛾屬昆蟲系統發育樹(以玉米螟為外群)Fig.2 The phylogenic tree of five Pyralis constructed with Neighbor-Joining method based on COⅠpartial sequence(Ostrinia nubilalis as outgroup)

3 結論與討論
DNA 條形碼自提出以來,已經在多個類群的研究中得到應用。包括鳥類、獸類、兩棲爬行類、植物(Kress and Erickson,2007)、真菌(Seifert et al.,2007)、魚類(Ward et al.,2005)、昆蟲等。近年來,也有大量關于鱗翅目昆蟲DNA 條形碼的研究。Hebert 等首次將DNA 條形碼技術應用于約200個鱗翅目近緣種的分類鑒別中,并成功的鑒別了這些昆蟲。Hajibabaei 等利用DNA 條形碼對哥達加斯加西北地區的521種已通過形態學鑒定的鱗翅目昆蟲進行物種鑒定,發現97.9%的物種可以用COⅠ基因進行區別鑒定(2006)。濮佳明(2009)研究表明DNA 條形碼可用于鱗翅目昆蟲柳蠶種質資源的分子鑒定,且適用于柳蠶各發育階段材料的分析。
DNA 條形碼作為物種鑒定和分類的有利工具,不僅極大的簡化了物種分類和鑒定工作,同時也彌補了形態學分類方法的不足。在DNA 條形碼廣泛應用的同時,部分學者也提出了質疑。他們認為DNA 條形碼作為物種分類鑒定的依據,其基因片段單一,應適當增加一些其他的基因序列信息,另外,由于核基因組中的線粒體假基因的存在會對PCR 擴增線粒體基因片段有影響,故采用多個方法或基因進行物種鑒定會更加科學和準確。
本研究以未知昆蟲的殘缺幼蟲作為樣本,利用DNA 條形碼對該樣本進行了快速而準確的鑒定。通過將未知樣品序列與5種螟蛾的14 條序列比對發現,未知樣品序列與紫斑谷螟的序列相似性最高,達到99.8%。5種螟蛾的種間遺傳距離為0.066-0.162,平均為0.127,種內遺傳距離為0.000-0.003,平均為0.001,種間遺傳距離遠遠大于種內遺傳距離。Hebert 等根據對鱗翅目部分種類的研究結果發現,種內遺傳距離一般都低于1%,同屬種間遺傳距離平均為6.8%,本研究中螟蛾屬5個種的種內遺傳距離均低于1%,平均種間遺傳距離則比6.8%大,表明COⅠ基因能夠用于該類物種的分類鑒定。
倉庫害蟲種類繁多,分布廣、危害大、對環境的適應性強,往往造成嚴重的經濟損失。我國是世界上倉儲技術發展最早的國家之一,在倉庫害蟲的研究和防治上具有悠久的歷史,積累了豐富的經驗(李隆術和趙志模,2000)。準確的物種鑒定為倉庫害蟲的防治提供了可靠的理論基礎,也為研究人員研制更有效的防治方法提供了素材。
References)
Frézal L and Leblois R.Four years of DNA barcoding:current advances and prospects[J].Infect Genet.Evol.,2008,8:727-736.
Hajibabaei M,et al.DNA barcodes distinguish species of tropical Lepidoptera[J].Proc.Natl.Acad.Sci.USA,2006,103:968-971.
Hebert PDN,et al.Barcoding animal life:cytochrome coxidase subunitⅠdivergences among closely related species[J].Proc.Biol.Sci.,2003,270:S96-S99.
Kress WJ and Erickson DL.A two-locus global DNA barcode for land plants:the coding rbcL gene complements the non-coding trnHpsbA spacer region[J].PLoS ONE,2007,2(6):e508.
Li LX and Zhao ZM.Retrospect and prospect of our storage insect research and prevention[J].Chinese Bulletin of Entomology,2000,(2):84-88.[李隆術,趙志模.我國倉儲昆蟲研究和防治的回顧與展望[J].昆蟲知識,2000,(2):84-88]
Li QQ,Li DY,Duan YQ,et al.Application of DNA barcoding in lepidopteran insects[J].Chinese Bulletin of Life Sciences,2010,22(4):307-312.[李青青,李地艷,段焰青,等.DNA 條形碼在鱗翅目昆蟲中的應用[J].生命科學,2010,22(4):307-312]
Pu JM,Wu S,Huo XM,et al.DNA barcode and phylogenetic analysis of the moon silkworm,Actias selene[J].Science of Sericulture,2009,35(1):154-159.[濮佳明,武松,霍錫敏,等.珍稀絹絲昆蟲柳蠶的DNA 條形編碼與系統進化初步分析[J].蠶業科學,2009,35(1):154-159]
Seifert KA,et al.Prospects for fungus identifcation using COⅠDNA barcodes,with Penicillium as a test case[J].Proc Natl Acad Sci USA,2007,104:3901-3906.
Shang XL,Yang MF,Huang L,et al.Biological characteristics of Pyralis farinalis Linnaeus a newly reported insect producing the“insect-tea”[J].Chinese Journal of Applied Entomology,2011,48(3):652-656.[尚小麗,楊茂發,黃麗,等.一種新的產蟲茶昆蟲—紫斑谷螟的生物學特性研究[J].應用昆蟲學報,2011,48(3):652-656]
Wang PY.China Economic Insect Fauna[M].Beijing:Science Press,1980,95.[王平遠.中國經濟昆蟲志[M].北京:科學出版社,1980,95]
Wang YW.The preliminary research on the Pyralis farinalis[J].Xinjiang Agricultural Sciences,1982,4:16-17.[王永衛.紫斑谷螟的初步研究[J].新疆農業科學,1982,4:16-17]
Ward RD,et al.DNA barcoding Australia's fish species[J].Philos.Trans.Soc.Lond B Biol.Sci.,2005,360:1847-1857.
Zhao YC.China Warehouse Pests Fauna Investigation[M].Beijing:Agriculture Press,1982,128-130.[趙養昌.中國倉庫害蟲區系調查[M].北京:農業出版社,1982,128-130]

