梅毒螺旋體TprF蛋白B細胞表位的預測與鑒定
曹龍古,凌 暉,蔡恒玲,趙飛駿,歐陽丹明,陳蘇芳,吳移謀,曾鐵兵
梅毒螺旋體TprF蛋白B細胞表位的預測與鑒定
曹龍古1,2,凌 暉3,蔡恒玲1,趙飛駿1,歐陽丹明2,陳蘇芳2,吳移謀1,曾鐵兵1
目的 預測和鑒定梅毒螺旋體(Tp) 膜蛋白TprF氨基端保守區(TprFN)的優勢B細胞表位,為深入研究梅毒多價表位疫苗提供依據。方法 從GenBank獲取TprFN的氨基酸序列,采用Mobyle、ABCpred和IEDB在線軟件綜合分析預測TprFN的B細胞表位并人工合成多肽;表達重組蛋白TprFN并經Western blot鑒定后免疫兔,獲取血清并測定抗體效價;以TprFN免疫兔血清、梅毒患者血清(設正常人血清和正常兔血清為陰性對照),間接ELISA測定預測的7條人工合成的B細胞表位多肽的免疫反應性和特異性。結果 軟件綜合預測TprFN的P1 (43-62AA)、P2(57-71AA)、P3(81-88AA)、P4(89-103AA)、P5(125-138AA)、P6(231-251AA)、P7(268-279AA)可能為B細胞表位;表達一可溶性蛋白,WB鑒定為目的蛋白,其免疫抗體效價為1∶12 800以上;ELISA結果顯示,預測表位P1、P3與TprFN免疫兔血清及梅毒患者血清均呈陽性反應,而與對照血清均不反應。結論 P1、P3為TprF潛在的優勢B細胞表位。
梅毒螺旋體;TpF;B細胞表位
Supported by the National Natural Science Foundation of China (Nos. 81273322 and 81373230), the Scientific Research Fund of Hunan Provincial Education Department (No. 13C876), the Chenzhou Municipal Science and Technology Bureau (No. 2012CJ126), and the Hunan Provincial Key Laboratory for Special Pathogens Prevention and Control Foundation (No. 2014-5) Corresponding author:Zeng Tie-bing,Email:nhdxztb@126.com
梅毒是由梅毒螺旋體(Treponemapallidum, Tp) 引起的一種嚴重危害成人和新生兒健康的性傳播疾病(STD),并可明顯促進艾滋病傳播[1]。近年我國梅毒發病呈迅速上升趨勢[2]。應用疫苗是梅毒防控的根本措施[3-4],但至今未獲成功。
目前認為,梅毒疫苗分子主要存在于Tp的外膜蛋白(OMPs)中[3-4]。TprF是近年來屬于Tpr家族蛋白第Ⅰ亞家族成員,包含369個氨基酸,軟件分析預測為OMP[5]。研究發現,在不同Tp株感染兔的體內存在針對重組TprF的N端保守區域(TprF蛋白全長第21~284位氨基酸,簡稱TprFN)的強烈的抗體應答和T細胞應答,免疫兔后明顯減輕不同Tp株攻擊所致損傷和減緩疾病進展[5-6],表明TprFN是頗具希望的候選疫苗分子[3]。
聯合多個具有保護作用的抗原分子是研發梅毒高效疫苗的趨勢[3-4],但在同一載體上串聯多個抗原編碼基因技術上存在困難,并可能引起轉錄干擾[5]。多價表位疫苗能克服該困擾,同時去除了蛋白分子中可能的無關、干擾甚至抑制表位,且可避免免疫耐受,此外還具有保護范圍廣、針對性強、穩定、高效、制備簡單、經濟安全等優點,具有廣闊的應用前景[7]。本研究對TprF蛋白保守區的B細胞表位進行篩選和鑒定,為發展梅毒多價表位疫苗奠定基礎。
1 材料與方法
1.1 實驗材料 Tp(Nichols株)為上海皮膚性病醫院檢驗科顧偉鳴饋贈,本研究所傳代保存。表達載體pET30a(+)、限制性內切酶、Tp基因組提取試劑盒分別為美國Novagen、Thermo、德國Qiagen公司產品。羊抗兔HRP-IgG、羊抗人HRP-IgG購自北京康為世紀有限公司。96孔高結合力酶標板為美國Corning公司產品。確診梅毒陽性血清、健康人血清標本分別來自南華大學附屬第一醫院檢驗科和健康體檢中心。
1.2 方法
1.2.1 TprFN的B細胞表位預測 獲得TprF氨基酸序列(http://www.ncbi.nlm.nih.gov/protein/AGF43524.1),將去除信號肽后的保守區氨基酸序列(TprF全長第21~284位)輸入在線Mobyle軟件(http://mobyle.pasteur.fr/cgi-bin/portal.py?#forms::antigenic)和ABCpred軟件(http://www.imtech.res.in/raghava/abcpred/ABC_submission.html),結果按分值高低大小依次顯示。再應用在線預測軟件IEDB(http://www.immuneepitope.org/tools/bcell/iedb_input)綜合分析TprF氨基酸序列表面可及性、親水性、抗原性、β拐角區域以及易屈性等理化因素。綜合以上3種軟件預測結果預測TprFN的B細胞抗原表位。
1.2.2 預測表位多肽合成 委托北京澤溪源生物科技股份有限公司人工合成預測的表位多肽,經反相高效液相色譜純化,質譜分析鑒定。
1.2.3 TprFN的表達與鑒定 從GenBank查取TprF全基因序列,設計針對TprFN的一對特異性引物F:5′ CGCGGATCCTATGCAGGCGTACTCACTCC 3′ (下劃線為BamH I酶切位點)、R:5′ CCGCTCGAGGGCAGGGTCCTGGTGTTG 3′ (下劃線為Hind Ⅲ酶切位點),以試劑盒提取的Tp DNA為模板,PCR擴增保守區基因片段,構建pET30a(+)/TprFN的原核表達重組體,經測序和雙酶切鑒定,30 ℃、以1.0 mmol/L IPTG誘導表達蛋白4 h,以1∶100稀釋梅毒患者血清為一抗,1∶2 000稀釋標記的山羊抗人HRP-IgG,Western Blot(WB)鑒定純化蛋白。
1.2.4 TprFN多抗制備 以純化TprFN加弗氏佐劑免疫5只體重約2.5 kg的新西蘭兔(200 μg/只·次),間隔2 w免疫1次,共5次,末次免疫后第10 d收集免疫血清,ELISA測定重組蛋白抗體效價。
1.2.5 TprFN預測B細胞表位鑒定 分別將10 μg/mL合成的B表位多肽和重組TprFN(陽性對照)作為抗原包被在酶標板上(100 μL/孔),以1∶100稀釋血清(TprFN免疫兔血清、正常兔血清各5份、梅毒患者血清、正常人血清各10份)為一抗,同時設空白對照,每個樣品做3個平行孔,1∶15 000稀釋的羊抗兔或羊抗人HRP-IgG進行間接ELISA,酶標儀雙波長測定吸光值A630/450nm。實驗重復3 次。
1.3 統計學處理 結果以A均值表示,采用SPSS 19.0軟件進行統計分析,各組間進行t檢驗,P<0.05代表有統計學意義。
2 結 果
2.1 TprF蛋白B細胞表位在線軟件預測與合成 綜合ECBI、ABCpred和Mobyle在線軟件,預測出如下7段氨基酸序列有可能為TprFN蛋白的B細胞表位:P1 (43-62AA)、P2(57-71AA)、P3(81-88AA)、P4(89-103AA)、P5(125-138AA)、P6(231-251AA)、P7(268-279AA)。合成多肽純度均大于90%,質譜分析合成多肽分子量測定值與理論值一致。

表1 預測的TprFN蛋白的B細胞表位
2.2 重組TprFN的表達、鑒定及免疫原性分析 經PCR、雙酶切及測序鑒定的目的基因表達了一分子量大小約36 kDa的可溶性蛋白條帶(圖1-A);Western blot檢測顯示,重組蛋白僅與梅毒陽性病人血清發生特異性反應(圖1-B);ELISA檢測其免疫兔多抗效價均在1∶12 800以上,顯示良好的免疫原性。
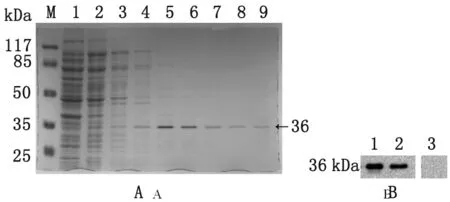
A. M:Protein molecular weight maker; 1:flow-through fluid; 2-4:W1-W3; 5-9:purified recombinant protein.
B. 1,2:syphilitic sera; 3:normal human sera.
圖1 純化重組蛋白TprFN的SDS-PAGE分析與Western Blot鑒定
Fig.1 Analysis and identification of purified recombinant protein TprFNby SDS-PAGE (A) and Western Blot (B)
2.3 TprF蛋白預測B細胞表位的鑒定 間接ELISA結果顯示,在P1和P3與TprFN免疫兔血清(圖2) 和梅毒血清(圖3)反應的吸光度A值與相應對照組差異均有統計學意義(P<0.05),表明P1和P3為TprFN蛋白的潛在優勢B細胞表位。
3 討 論
各種類型疫苗均基于表位(抗原決定簇)發揮作用。B細胞表位是抗原分子中能被BCR/抗體特異性識別的基本單位。在抗Tp感染中,B細胞分泌特異性抗體介導的體液免疫應答發揮著重要作用,主要方式包括阻斷Tp粘附、促進巨噬細胞的吞噬(調理作用)、激活補體溶菌作用等[3-4,8]。本研究預測鑒定TprF的B細胞表位,為構建多價表位疫苗奠定基礎。

The “*” symbol indicate a significant difference (P<0.05) compared with normal rabbit sera control groups.
圖2 ELISA檢測TprFN預測表位多肽與兔血清免疫反應性 (A450 /630)
Fig.2 Immunoreactivity of predicted TprFNepitopes with rabbit sera by ELISA
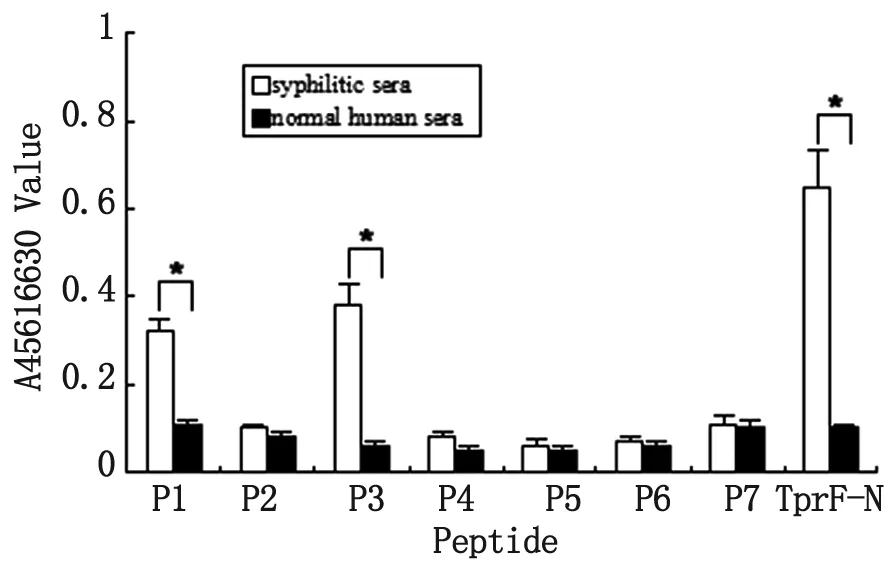
The “*” symbol indicate a significant difference (P<0.05) compared with normal human sera control groups.
圖3 ELISA檢測TprFN預測表位多肽與人血清免疫反應性 (A450 /630)
Fig.3 Immunoreactivity of predicted TprFNepitope with human sera by ELISA
傳統篩選鑒定表位的方法主要有合成重疊肽法[9]、噬菌體肽庫展示法[10]、水解法[11]、生物學或化學方法[12]等,其優點是能較全面篩選抗原蛋白可能表位,但工作難度大,耗時長。應用計算機軟件分析篩選抗原表位,可減少盲目篩選的龐大工作量,大大提高了發現新表位的效率[13]。目前幾乎所有B細胞表位預測軟件均以唯象理論為基礎,即通過計算蛋白亞序列二級結構或者理化性質,再利用B細胞表位與其相關性來預測。為提高預測的準確性,通常需綜合多種方案來分析[14-15],本文運用IEDB在線軟件中提供多種方案分析親水性、抗原性、易屈性、表面可及性與β-轉角,再結合ABCpred和Mobyle在線軟件總結得出7條TprF抗原最有可能的B細胞表位多肽。由于在線軟件均基于現有的表位數據模擬和推測待預測氨基酸序列,存在這一定風險和偏差,需鑒定所預測的表位。
鑒定TprF的B表位首先要確定其免疫原性和免疫反應性。由于至今Tp不能體外人工培養,且其外膜蛋白量少而脆弱,無法直接分離TprF,為此本研究首先通過E.coli表達了可溶性TprFN。可溶性表達無需對純化蛋白進行變性和復性處理,保持了抗原天然的空間構象,以其免疫能獲得針對TprFN的各種B細胞表位的混合抗體。以重組TprFN免疫兔,獲得了高滴度的抗體,表明該重組抗原具有良好的免疫原性;免疫印跡法(WB)鑒定重組抗原也表明其有良好的免疫反應性和特異性。WB和ELISA均可用于B細胞表位的鑒定,但人工合成的表位多肽分子量太小,不可采用WB來鑒定。本研究以各合成短肽為抗原,ELISA結果顯示,預測的P1和P3能與TprFN免疫兔血清發生特異性強免疫反應,為進一步確認,同時以梅毒感染者血清鑒定,結果與免疫兔血清的反應一致,進一步表明P1和P3為TprFN的優勢B細胞表位。
本研究篩選出TprFN的優勢B細胞表位,為進一步構建多表位梅毒疫苗奠定了基礎,但其是否具有免疫保護性及通過何種方式發揮抗Tp感染作用有待深入研究。
[1]Douglas JM Jr. Penicillin treatment of syphilis:clearing away the shadow on the land[J]. JAMA, 2009, 301(7):769-771. DOI:10.1001/jama.2009.143
[2]Tucker JD, Cohen MS. China’s syphilis epidemic:epidemiology, proximate determinants of spread, and control responses[J]. Curr Opin Infect Dis, 2011, 24(1):50-55. DOI:10.1097/QCO.0b013e32834204bf
[3]Cameron CE, Lukehart SA. Current status of syphilis vaccine development:need, challenges, prospects[J]. Vaccine, 2014, 32(14):1602-1609. DOI:10.1016/j.vaccine.2013.09.053
[4]Cullen PA, Cameron CE. Progress towards an effective syphilis vaccine:the past, present and future[J]. Expert Rev Vaccines, 2006, 5(1):67-80. DOI:10.1586/14760584.5.1.67
[5]Sun ES, Molini BJ, Barrett LK, et al. Subfamily ITreponemapallidumrepeat protein family:sequence variation and immunity[J]. Microbes Infect, 2004, 6:725-737. DOI:10.1016/j.micinf.2004.04.001
[6]Hevner GLK, Centurion-Lara A. Gene organization and transcriptional analysis of the tprJ, tprI, tprG, and tprF loci inTreponemapallidumstrains Nichols and Sea 81-4[J]. J Bacteriol, 2005, 187(17):6084-6093. DOI:10.1128/JB.187.17.6084-6093.2005
[7]Pishraft Sabet L, Taheri T, Memarnejadian A, et al. Immunogenicity of multi-epitope DNA and peptide vaccine candidates based on core, E2, NS3 and NS5B HCV epitopes in BALB/c mice[J]. Hepat Mon, 2014, 14(10):e22215. DOI:10.5812/hepatmon.22215
[8]Lafond RE, Lukeart SA. Biological basis for syphilis[J]. Clin Microbiol Rev, 2006, 19:29-49. DOI:10.1128/CMR.19.1.29-49.2006
[9]Williams KM, Bigley EC 3rd, Raybourne RB. Identification of murine B-cell and T-cell epitopes ofEscherichiacolioutermembrane protein F with synthetic polypeptides[J]. Infect Immun, 2000, 68(5):2535-2545. DOI:10.1128/IAI.68.5.2535-2545.2000
[10]Freund NT, Enshell-Seijffers D, Gershoni JM. Phage display selection, analysis, and prediction of B cell epitopes[M]. Curr Protoc Immunol, 2009, Chapter 9:Unit 9.8. DOI:10.1002/0471142735.im0908s86
[11]Reynolds SR, Dahl CE, Harn DA.T and B epitope determination and analysis of multiple antigenic peptides for theSchistosomamansoniexperimental vaccine triose-phosphate isomerase[J]. J Immuno, 1994, 152(1):193-200.
[12]Morris GE. Epitope mapping by chemical fragment[J]. Methods in Mol Biol, 1996, 66:121-127.
[13]Soria-Guerra RE, Nieto-Gomez R, Govea-Alonso DO, et al. An overview of bioinformatics tools for epitope prediction:implications on vaccine development[J]. J Biomed Inform, 2015, 53:405-414. DOI:10.1016/j.jbi.2014.11.003
[14]Lian Y, Ge M, Pan XM. EPMLR:sequence-based linear B-cell epitope prediction method using multiple linear regression[J]. BMC Bioinformatics, 2014, 15(1):414. DOI:10.1186/s12859-014-0414-y
[15]EL-Manzalawy Y, Honavar V. Building classifier ensembles for B-cell epitope prediction[J]. Methods Mol Biol, 2014, 1184:285-294. DOI:10.1007/978-1-4939-1115-8_15
Prediction and identification of B-cell epitopes ofTreponemapallidumrepeat protein F
CAO Long-gu1,2,LING Hui3,CAI Heng-ling1,ZHAO Fei-jun1,OUYANG Dan-ming2,CHEN Su-fang2,WU Yi-mou1,ZENG Tie-bing1
(1.InstitutionofPathogenicBiology,MedicalCollege,UniversityofSouthChina,Hengyang421001,China;2.DepartmentofPreventiveMedicineandMedicalExamination,XiangnanUniversity,Chenzhou423000,China;3.InstituteofCancerResearch,MedicalSchool,UniversityofSouthChina,Hengyang421001,China)
To predict and identify the dominant B-cell epitopes of conserved region ofTreponemapallidumrepeat protein F (TprFN) and provide the basis for development of polyvalent epitope-based syphilis vaccine, the amino acid sequence of TprFNwas obtained from GenBank and analyzed with comprehensive meta-analysis Mobyle, ABCpred and IEDB online software. The peptides containing predicted epitopes were artificially synthesized. To obtain and measure the titers of antibodies against TprFN, New Zealand rabbits were immunized with recombinant protein TprFNexpressed inE.coliand identified by Western blot (WB) . Sera from TprFN-immunized rabbits, syphilis patients, and normal human and normal rabbits were used to determine the immunoreactivity and specificity of 7 predicted peptides of TpFNby indirect ELISA. Comprehensive meta-analysis of online software showed that P1 (43-62AA), P2(57-71AA), P3(81-88AA), P4(89-103AA), P5(125-138AA), P6(231-251AA) and P7(268-279AA) might be the B-cell epitopes. A protein was expressed in a soluble form and identified as TpFNby WB. The ELISA indicated that P1 and P3 were active with TprFN-immunized rabbit sera and syphilis patient sera but not with negative control sera. These results indicate that P1 and P3 are the potential dominant B-cell epitopes.
Treponemapallidum; TprF; B-cell epitope
國家自然科學基金(No. 81273322,81373230);湖南省教育廳資助科研項目(No.13C876);郴州市科技局資助科研項目(No.2012CJ126);特殊病原體防控湖南省重點實驗室資助項目(湘科計字[2014]5號)
曾鐵兵,Email:nhdxztb@126.com
1.南華大學醫學院病原生物學研究所,衡陽 421001; 2.湘南學院預防醫學與醫學檢驗系,郴州 423000; 3.南華大學醫學院腫瘤研究所,衡陽 421001
10.3969/j.issn.1002-2694.2015.10.005
R377
A
1002-2694(2015)10-0919-04
2015-05-19;
2015-07-24

