胃癌中P53誘導相關微小RNA靶基因預測及生物信息學分析*
曹 輝,許 鐘,白班俊,張玲玲
(貴州省人民醫院:1.腫瘤科;2.消化內科,貴陽 550002)
微小RNA (miRNA)通過使其靶mRNA的降解或翻譯抑制,在轉錄后水平調控基因表達[1],參與調控細胞的增殖、分化及腫瘤發生、發展等生理和病理過程,因而受到廣泛關注。多項研究對胃癌臨床標本檢測發現,相對于癌旁組織,胃癌中出現多種miRNA表達的異常上調或下調;同時,miRNA的表達也受到多種轉錄因子的調控,如P53可誘導miR-34a、miR-26b、miR-92、miR-29a、miR-25、miR-103、miR-128a等一系列miRNA的表達上調[2]。對野生型P53胃癌來說,探討P53通過miRNA間接作用參與腫瘤的發生有助于深入理解該類腫瘤的發生機制。本研究擬通過生物信息學預測胃癌中P53誘導相關miRNA的靶基因,并進一步對其靶基因進行功能富集分析和信號轉導通路富集分析,為后續的表達調控實驗奠定理論基礎。
1 材料與方法
1.1目標miRNA的篩選 通過在線人類疾病相關microRNA數據庫mir2disease[3],檢索胃癌中高表達的miRNA;用檢索結果與P53誘導相關miRNA對比,取交集得到胃癌中P53誘導相關miRNA。
1.2靶基因的預測 確定目標miRNA后,利用在線預測軟件miRNA 靶基因數據庫miRGen(v3.0)進行靶基因預測(含PicTar、miRanda、DIANA-microT、TargetScanS 等miRNA 靶基因預測工具)[4],得到目標miRNA的候選靶基因數據集。
1.3確立胃癌中的預測靶基因 通過在線基因表達譜數據庫Gene Expression Atlas[5]檢索獲得胃癌中表達下調的基因,用預測的候選靶基因數據集與之比對,獲得胃癌中目標miRNA的靶基因數據集。
1.4功能富集分析 通過在線軟件DAVID[6]對預測的靶基因數據集進行功能富集分析;Pathway Miner進行信號轉導通路分析。
2 結 果
2.1目標miRNA的篩選情況 應用在線mir2disease數據庫檢索胃癌相關miRNA,根據其詳細注釋篩選出在胃癌中表達上調的基因,共18個;匯總胃癌中高表達的miRNA與P53誘導相關miRNA[2]兩者對比,初步獲得本研究候選miRNAs,見圖1。

圖1 目標miRNA篩選圖
2.2miR-21靶基因的預測 從候選目標miRNA中選擇文獻報道較多的miR-21,通過在線軟件預測其靶基因,用集成常用預測工具的miRGen(v3)分析,結果共獲得1 090個基因。
2.3確立胃癌中的預測靶基因 應用在線基因表達譜數據庫Gene Expression Atlas檢索,選擇 “(All genes) and down in gastric carcinoma”,限定為“Homo sapiens”,獲得胃癌中表達下調的基因共2 737個,用預測的1 090個候選靶基因數據集與之比對,獲得胃癌中目標miRNA的靶基因數據集共115個,見表1。
2.4功能富集分析結果 應用DAVID進行Gene_Ontology分析時,選擇系統默認的GOTERM_BP_FAT、GOTERM_CC_FAT、GOTERM_MF_FAT 3個庫,其中P<0.01部分結果見表2。通過Pathway Miner分析相關信號通路(表3),細胞調節通路包括黏附連接、脂肪細胞因子信號轉導通路、胰島素信號轉導通路、細胞凋亡、鈣信號轉導通路、JAK-STAT信號轉導途徑等,代謝途徑包括賴氨酸、纈氨酸、亮氨酸和異亮氨酸降解、丁酸代謝、脂肪酸代謝、丙酮酸代謝等,見圖2。
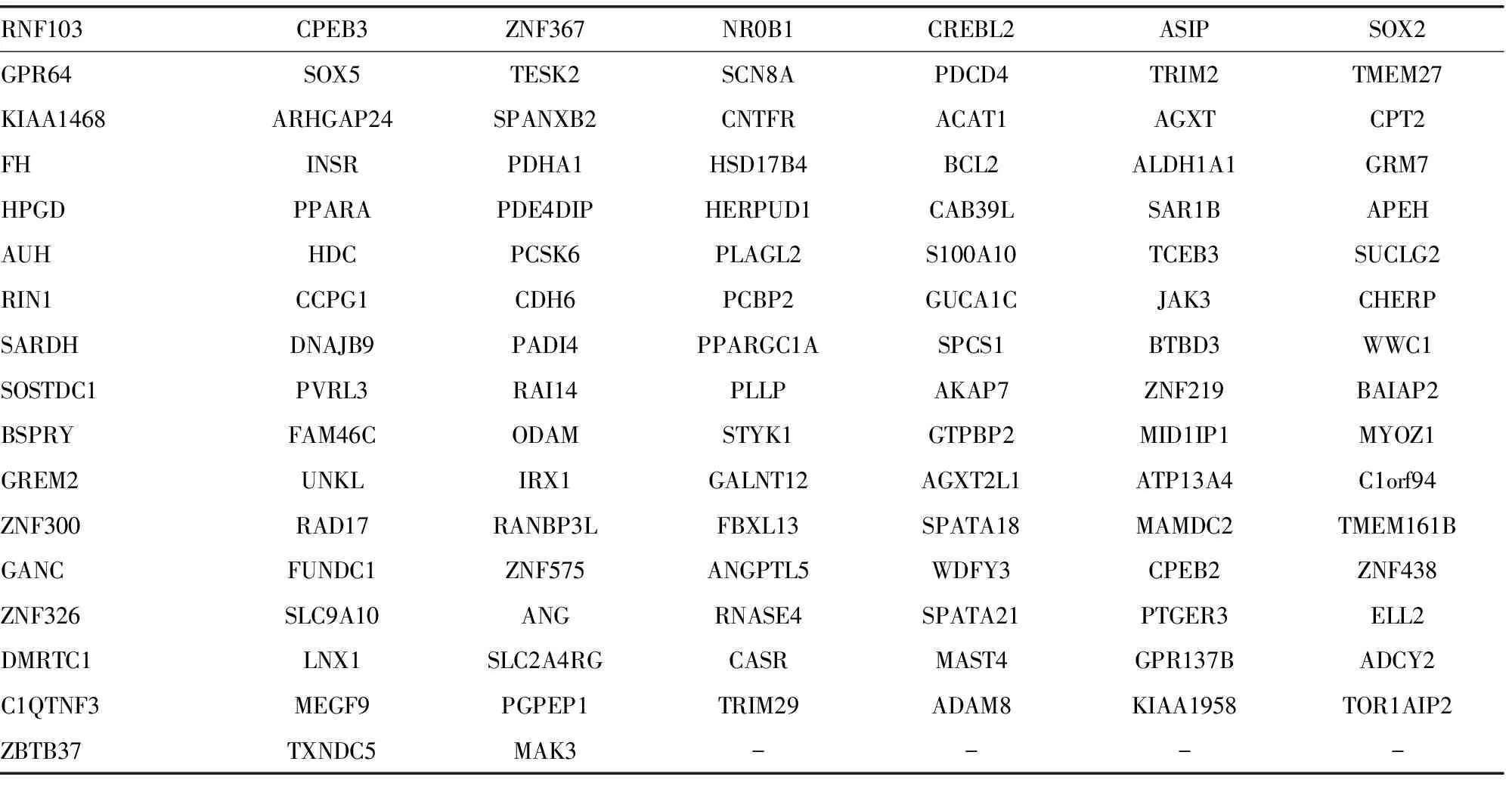
表1 胃癌中miR-21的靶基因預測與篩選結果
-:此項無數據。
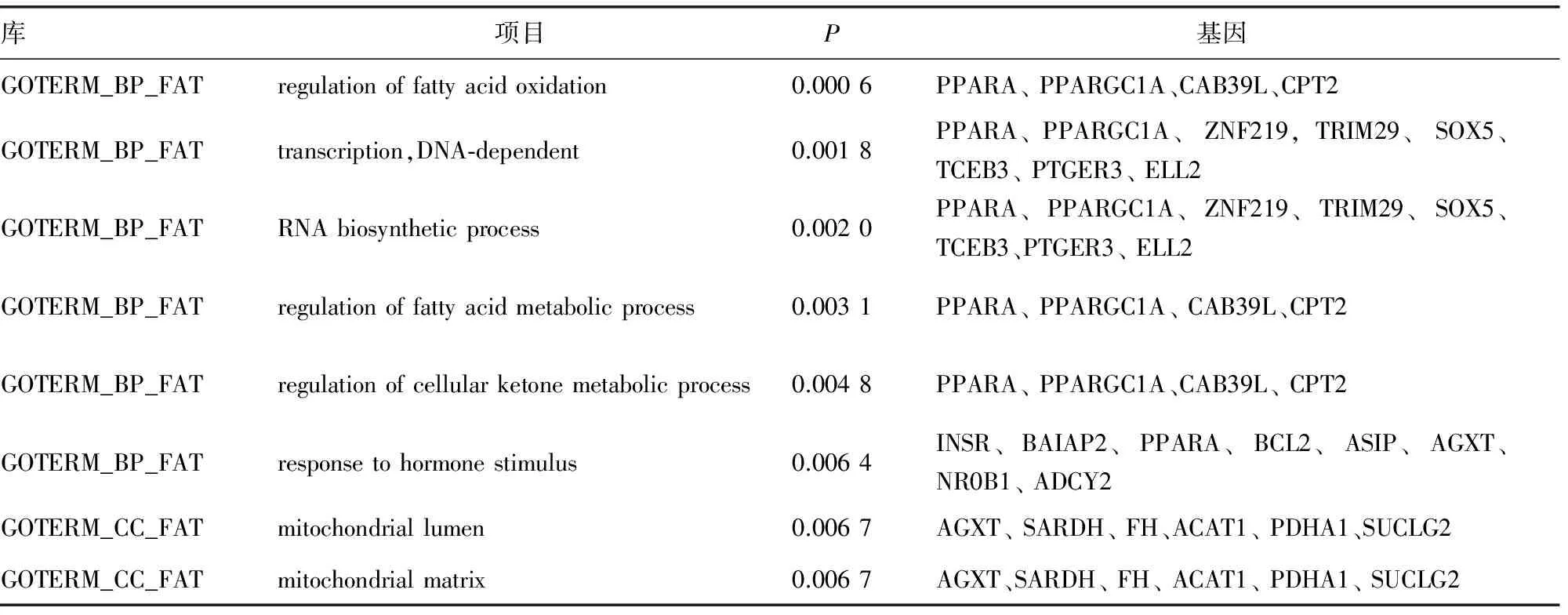
表2 應用DAVID進行Gene_Ontology功能富集分析(部分)
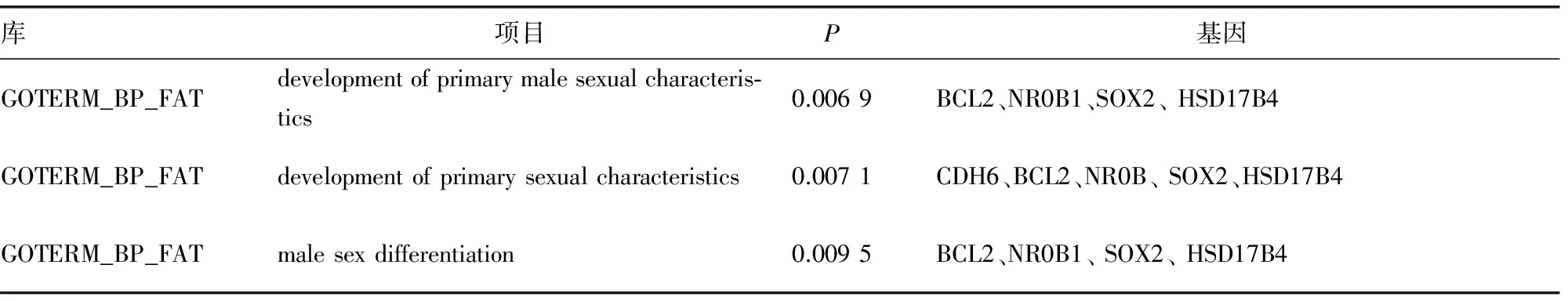
續表2 應用DAVID進行Gene_Ontology功能富集分析(部分)

表3 Pathway Miner分析中KEGG部分相關信號通路名稱
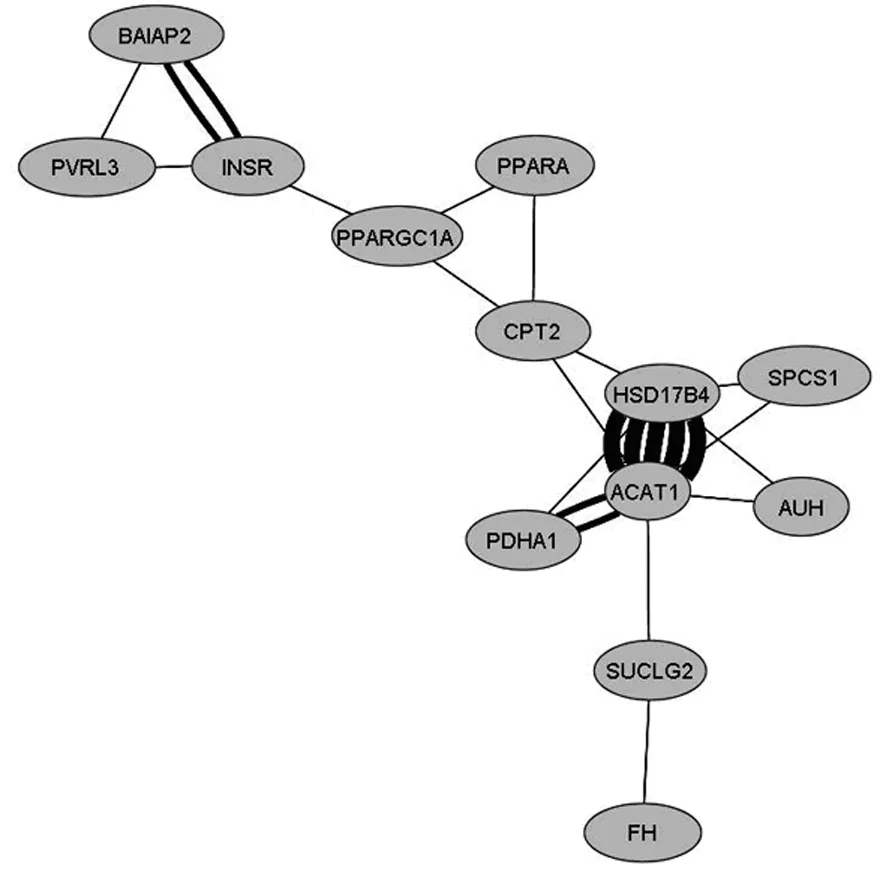
圖2 Pathway Miner分析中部分信號通路涉及基因(KEGG)
3 討 論
胃癌是我國最常見的惡性腫瘤之一,在胃黏膜癌變過程中,P53等多種抑癌基因的變化參與了胃癌形成。P53和多種轉錄因子之間存在著相互作用,但仍難以充分闡明胃癌發生機制。近年來發現P53可影響一組miRNA的表達[2],為本研究提供了新的契機。
miRNA作為一類具有調控基因表達的非編碼RNA(約18~22 nt),通過使其靶mRNA的降解或翻譯抑制,在轉錄后水平調控基因表達[1,7]。miRNA通過這種機制參與調控細胞的增殖、分化及腫瘤發生、發展等生理、病理過程。同時,miRNA的表達也受到多種轉錄因子的調控,P53可誘導一系列miRNA的表達上調[2]。通過對胃癌臨床標本的檢測,發現相對于癌旁組織,胃癌中出現多種miRNA表達的異常表達,如mir-34、mir-128a、miR-92、miR-25、miR-103等的上調,mir-128b、mir-129、mir-148等的下調[8-10]。正是由于miRNA與轉錄因子之間存在相互調控作用,推測在野生型P53胃癌中,P53可能在誘導miRNA的表達后,進而通過miRNA對下游基因的轉錄后水平調控,從而促進腫瘤的發生。因此,作者進行了相關生物信息學分析,為后續研究提供理論基礎。
通過在線數據的深度挖掘,成功縮小胃癌中目標miRNA分析范圍。miR-21與胃癌的發生和轉移有關,有望作為胃癌分子標志[11],選擇其進一步分析預測靶基因。經過整合常用miR靶基因預測軟件的靶基因數據庫miRGen(v3)進行靶基因預測后,應用基因表達譜數據庫有效縮小靶基因范圍,獲得在胃癌中相關的預測靶基因集。應用在線軟件DAVID和Pathway Miner分別進行GO基因注釋、功能富集分析和信號通路分析。結果顯示在胃癌中MiR-21的靶基因涉及系列信號通路中,包括黏附連接、胰島素信號轉導通路、細胞凋亡、鈣信號轉導通路、JAK-STAT信號轉導途徑等與腫瘤發生、發展及轉移密切相關的信號通路[12-13]。
經過充分數據發掘,利用生物信息學分析結果所提供的重點信號轉導通路為基礎,可以開展針對性的實驗研究,將有助于進一步理解P53、miRNA及下游靶基因組成的調控網絡在胃癌發生、發展中的作用機制。
[1]Wu L,Fan J,Belasco JG.MicroRNAs direct rapid deadenylation of mRNA [J].Proc Natl Acad Sci U S A,2006,103(11):4034-4039.
[2]Tarasov V,Jung P,Verdoodt B,et al.Differential regulation of microRNAs by p53 revealed by massively parallel sequencing:miR-34a is a p53 target that induces apoptosis and G1-arrest [J].Cell Cycle,2007,6(13):1586-1593.
[3]Jiang Q,Wang Y,Hao Y,et al.miR2Disease:a manually curated database for microRNA deregulation in human disease [J].Nucleic Acids Res,2009,37(Database issue):D98-104.
[4]Alexiou P,Vergoulis T,Gleditzsch M,et al.miRGen 2.0:a database of microRNA genomic information and regulation [J].Nucleic Acids Res,2010,38(Database issue):D137-141.
[5]Petryszak R,Burdett T,Fiorelli B,et al.Expression Atlas update——a database of gene and transcript expression from microarray- and sequencing-based functional genomics experiments [J].Nucleic Acids Res,2014,42(1):D926-932.
[6]Huang da W,Sherman BT,Lempicki RA.Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources [J].Nat Protoc,2009,4(1):44-57.
[7]Zeng Y,Cullen BR.Sequence requirements for micro RNA processing and function in human cells [J].RNA,2003,9(1):112-123.
[8]Volinia S,Calin GA,Liu CG,et al.A microRNA expression signature of human solid tumors defines cancer gene targets [J].Proc Natl Acad Sci U S A,2006,103(7):2257-2261.
[9]Tchernitsa O,Kasajima A,Schafer R,et al.Systematic evaluation of the miRNA-ome and its downstream effects on mRNA expression identifies gastric cancer progression [J].J Pathol,2010,222(3):310-319.
[10]Li X,Zhang Y,Zhang H,et al.miRNA-223 promotes gastric cancer invasion and metastasis by targeting tumor suppressor EPB41L3 [J].Mol Cancer Res,2011,9(7):824-833.
[11]Li BS,Zhao YL,Guo G,et al.Plasma microRNAs,miR-223,miR-21 and miR-218,as novel potential biomarkers for gastric cancer detection [J].PLoS One,2012,7(7):e41629.
[12]Souma Y,Nishida T,Serada S,et al.Antiproliferative effect of SOCS-1 through the suppression of STAT3 and p38 MAPK activation in gastric cancer cells [J].Int J Cancer,2012,131(6):1287-1296.
[13]Wei Q,Zhou W,Wang W,et al.Tumor-suppressive functions of leucine zipper transcription factor-like 1[J].Cancer Res,2010,70(7):2942-2950.

