苜蓿全基因組WRKY轉錄因子基因的分析
江騰,林勇祥,劉雪,江海洋,朱蘇文
(安徽農業大學生命科學學院 安徽省作物生物學重點實驗室,安徽 合肥230036)
轉錄因子在高等植物的生長發育及其對外界環境的反應中起著重要的調控作用,典型的高等植物轉錄因子含有DNA結合域、轉錄調控域、寡聚化位點和核定位信號,轉錄因子通過這些結構域與順式元件相互作用調控基因的表達。轉錄因子也稱反式作用因子,其主要功能是激活或抑制基因的轉錄效應[1,2]。WRKY蛋白是植物所特有的轉錄因子家族。因WRKY結構域是一個60個氨基酸左右的保守結構,所有結構域均含有高度保守的WRJKYGQK氨基酸序列而得名[3,4]。WRKY基因是首先從植物中分離得到的一類調節蛋白質。至今已從多種植物中分離,如甜土豆(Ipomoea batatas)、野燕麥(Avena sativa)、皺葉歐芹(Petroselinum crispum)、擬南芥(Arabidopsisthaliana)、煙草(Nicotiana tabacum)、水稻(Oryza sativa)等[5-9]。WRKY 基因家族的名字來源于這類蛋白質的一個主要特征即含有WRKY結構域。
在擬南芥、煙草和水稻中,已有大量的文獻報道WRKY轉錄因子參與了水楊酸和茉莉酸信號傳導相關基因的調控和抗病性[10-12]。水稻的一個WRKY45基因被證實與稻瘟病的抗性相關,過量表達WRKY45基因導致水稻的抗稻瘟病能力增強[13]。擬南芥中的WRKY18,WRKY40和WRKY60基因與灰霉菌(Streptomyces griseus)和假單孢菌(Pseudomonasaeruginosa)的抗性密切相關[14]。近年來,隨著多種植物全基因組數據的公布,利用全基因組數據分析某一基因家族基因的特征越來越多,最早是擬南芥中抗病基因的全基因組分析,利用生物信息學方法從擬南芥中共發現149個NBS(nucleotide binding site)基因,并對該基因家族進行染色體定位,分類,保守的基序(motif)分析[15],該研究對近幾年擬南芥NBS抗病基因的克隆和功能驗證起著重要的作用。隨后利用生物信息學方法對其他基因家族的全基因分析的報道也日益增多,而WRKY基因作為一類較為重要的基因家族,一直是分子生物學中研究的重點,在擬南芥全基因組中有72個WRKY基因,共含有85個WRKY結構域,其中有13個WRKY蛋白包含著2個WRKY結構域,對水稻全基因組WRKY基因研究表明,水稻中共有102個WRKY轉錄因子基因[16],對擬南芥和水稻WRKY基因的研究大大加速了對這類基因的了解。
蒺藜苜蓿(Medicago truncatula),豆科蝶形花亞科苜蓿屬一年生黃花苜蓿,耐旱,已經成為研究豆科植物的模式植物[17,18],基因組小(全基因組470 Mb),因其轉化和培養較為容易,目前,在豆科植物功能基因的研究中應用較多[19-21]。苜蓿全基因組的精細序列(Mt2.0)已經公布,目前,利用生物信息學方法分析基因家族的特征、進化關系成為熱點[22,23]。本研究利用生物信息學方法分析了苜蓿全基因組WRKY基因的種類、數目和進化特點,對促進苜蓿WRKY基因家族功能基因組學的進一步研究具有重要意義。
1 材料與方法
1.1 試驗材料
本研究以苜蓿最新的全基因組數據庫(Mt2.0)為對象,苜蓿全基因組數據和蛋白質數據下載于苜蓿基因組測序網站(http://www.medicago.org)。
1.2 試驗方法
1.2.1 WRKY基因的確定 因為苜蓿基因組的注釋并不完全,因此利用蛋白家族數據庫(Pfam)中選取的WRKY蛋白家族序列(PF03106),利用序列的同源性搜索工具(Blastp)篩選苜蓿全部基因,P值設為10-3[24],把候選WRKY基因進行Pfam分析,以證實其蛋白含有保守的WRKY結構域。而水稻的WRKY基因已經在基因組中有較好的注釋,直接從水稻基因組網站中下載全基因序列,獲取水稻的102條WRKY基因序列。
1.2.2 WRKY類型基因分類 對基因家族進行分組是功能分析的一項重要內容,轉錄因子基因家族常含有高度保守的功能域或有DNA結合功能的保守域。根據預測得到的WRKY域的氨基酸序列對苜蓿WRKY基因進行分組。參照Eulgem和Somssich[3]的分組原則及結合系統發育分析的結果進一步分組,含有2個WRKY保守結構域的定義為Ⅰ類,含有1個WRKY保守結構域且含有C2H2保守鋅指基序(motif)的定義為Ⅱ類,含有1個WRKY保守結構域且含有C2HC保守的鋅指motif的定義為Ⅲ類。這樣WRKY域可以分為3個大組(Ⅰ、Ⅱ、Ⅲ)。
1.2.3 WRKY類型基因的染色體定位 WRKY類型基因通過苜蓿基因組數據庫(http://www.medicago.org/genome/)提供的染色體BLAST工具進行苜蓿染色體物理定位,首先將每個WRKY基因與苜蓿全基因組進行BLAST搜索,尋找最匹配的染色體位置,從而確定每個WRKY類型基因在染色體上的具體位置。
1.2.4 WRKY類型基因系統發生學分析 選取苜蓿中所有的WRKY類型基因與水稻102個已鑒定的WRKY基因利用CLUSTALW軟件進行序列比對[25],然后利用MEGA 4.0軟件進行系統進化樹的構建,分析苜蓿與水稻WRKY基因間的進化關系[26]。此外,對所有WRKY基因中的所有WRKY結構域進行序列比對,然后根據結構域的比對結果繪制WRKY結構域的系統進化樹。
1.2.5 WRKY結構域(domain)的motif分析 苜蓿WRKY類型基因的預測motif分析通過MEME(multiple expectation maximization for motif elicitation)在線分析[27],MEME是美國圣地亞哥超級計算機中心(SDSC)開發的一套用來尋找一組相關的DNA序列或者蛋白質序列基序的程序。利用此軟件來預測苜蓿WRKY基因中的保守motif以及WRKY結構域上的保守motif。
2 結果與分析
2.1 WRKY類型候選基因的確定與染色體定位
利用Pfam數據庫的WRKY結構域的標準序列,在苜蓿全基因組中進行BLAST搜索,P值設為10-3,然后把候選序列進行序列比對。通過這種方法,從苜蓿基因組中共找到28個WRKY候選基因,分別命名為Mt-WRKY1-MtWRKY 28(表1)。水稻已經報道共有102個WRKY基因,即使是同為雙子葉植物的擬南芥中也發現了72個WRKY基因,苜蓿的基因組大小與水稻相當,這說明了WRKY基因家族并不與基因組大小有直接的對應關系,擬南芥的基因組是苜蓿的1/4左右,但是其WRKY基因總數是苜蓿的3倍左右。進一步把28個苜蓿的WRKY進行染色體定位,發現WRKY基因并沒有均勻的分布在染色體上,其中,5號染色體分布最多,共含有8個WRKY基因,1號染色體有4個基因,3和4號染色體都有3個基因,7號染色體有2個基因,8號染色體有6個基因,而2和6號染色體上不含有WRKY基因,水稻、擬南芥等植物的WRKY基因分布也有此特點。此外,26個WRKY基因都已經精確定位到染色體上,而MtWRKY14和MtWRKY15兩個基因并沒有定位在任何染色體上,主要是因為其所在的BAC(細菌人工染色體,bacterial artificial chromosome)克隆目前沒有被定位,導致基因不能夠與拼接的染色體序列匹配。
2.2 WRKY基因序列比對與分類
擬南芥和水稻WRKY基因的研究已經對這個基因家族有了一些理解,一般而言該基因可以分為3個大類和若干小類,采用了文獻的方法將苜蓿的28個WRKY基因進行了分類,也可以分為3類,其中MtWRKY1-MtWRKY 5因為其含有2個保守的WRKY結構域而劃分為Ⅰ類。MtWRKY27與Mt-WRKY28因為其除了含有1個WRKY結構域外,還含有C2HC的保守motif,而劃分為Ⅲ類,其他的WRKY基因含有1個WRKY結構域且在C端包含C2H2的保守motif而劃分為Ⅱ類。此外,把所有基因的WRKY保守結構域的氨基酸序列進行比對分析,其中含有2個WRKY結構域的Ⅱ類基因,把N端的結構域命名為NW,發現WRKY結構域長度為51~63個氨基酸(圖1),相對于 C端的WRKY結構域,其N端WRKY結構域之間具有相似的序列構成。對WRKY結構域進行系統發生學分析,可以明顯的分為3個分枝(圖2),且所有基因的NW結構域基本上在同一進化分枝上,說明它們與C端的WRKY結構域并不是簡單的復制現象造成,有其單獨的起源和進化關系。此外,WRKYGQK 7個氨基酸被視為WRKY基因最保守的結構,從結構域的序列比較可以發現,第1組和第3組基本上比較保守,WRKYGQK基本上沒有變異,而第2組中WRKYGQK中的Q殘基變異較大,說明了不同類型的WRKY基因的選擇壓力和進化模式是不同的。
2.3 苜蓿WRKY基因的系統發生學關系
利用苜蓿的28個WRKY基因氨基酸序列與水稻102個WRKY基因氨基酸序列進行序列比對,然后進行系統進化樹繪制(圖3)。可以明顯的看出苜蓿的WRKY基因集中分布在幾個分枝上,共有17個基因是單獨位于分枝上,說明了苜蓿與水稻WRKY基因的起源和進化關系不同。如MtWRKY8-11單獨在一分枝上,與水稻WRKY具有較遠的進化距離。此外,可以明顯的看出水稻的WRKY基因分布在外圍分枝的基因較多,而且一些基因與苜蓿的WRKY基因差異較多,證實水稻的WRKY基因變異較多,苜蓿的WRKY基因更加保守。
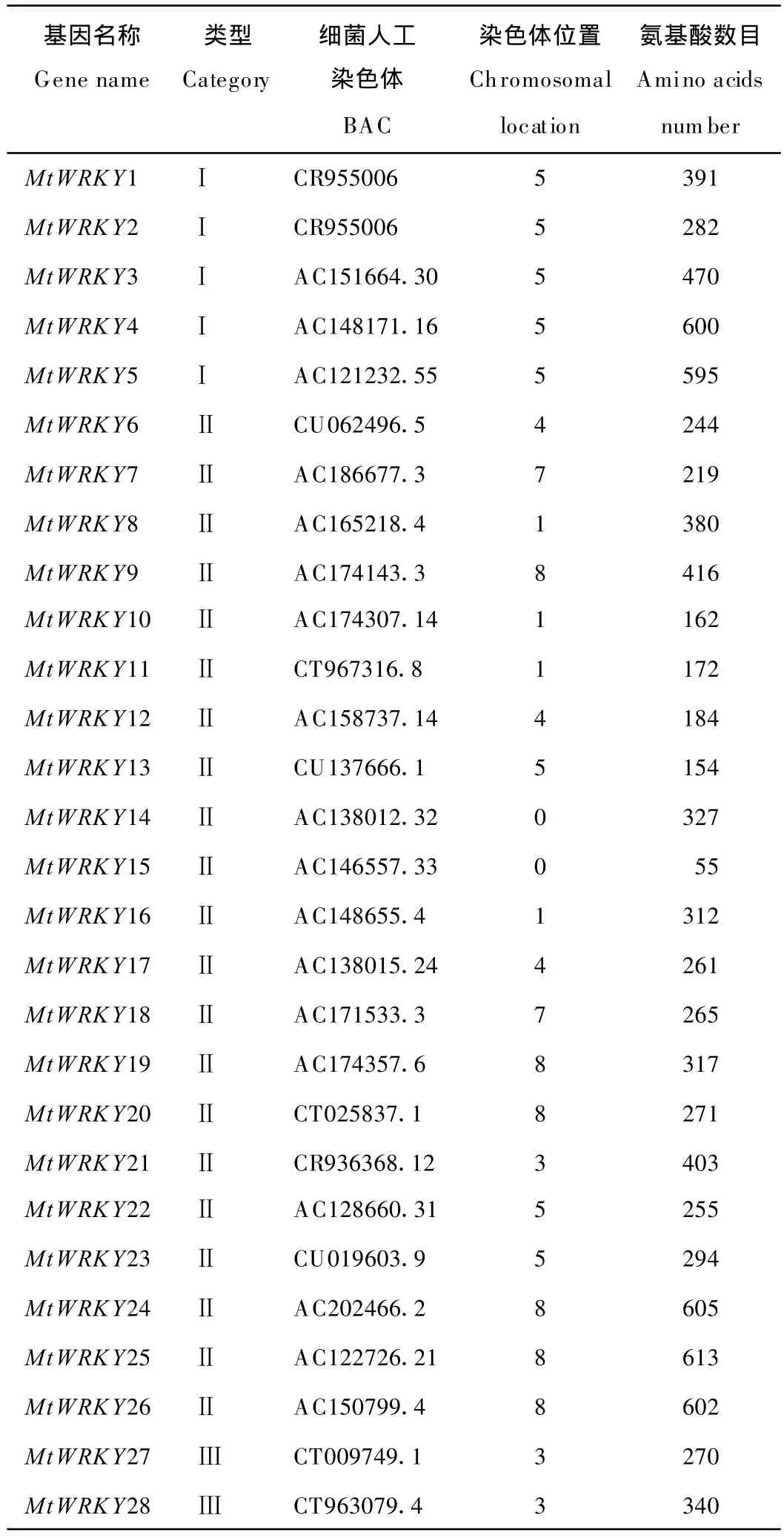
表1 苜蓿WRKY基因的分類和定位信息Table 1 Classification and location of WRKY genes in M.truncatula
2.4 苜蓿WRKY轉錄因子的motif分析
對28個WRKY基因的氨基酸進行MEME軟件在線分析,結果顯示,在苜蓿WRKY基因中共找到46個預測的motif,可以看出不同類型的WRKY基因含有的motif數目和種類有所差異,如Ⅰ組WRKY基本上都含有2個motif 1,這是WRKY結構域的特征,motif的多樣化也說明了WRKY基因廣泛參與細胞內的多樣代謝途徑和反應。此外選取28個基因的33個WRKY結構域序列進行motif分析,結果共發現5個motif(表2),其中最長的motif長度為26個氨基酸,最短的為10個氨基酸,其中motif 1最為保守,是WRKY基因最保守的motif(圖4)。

圖1 苜蓿WRKY結構域的氨基酸比對Fig.1 Amino acid alignment of M.truncatula WRKY domain
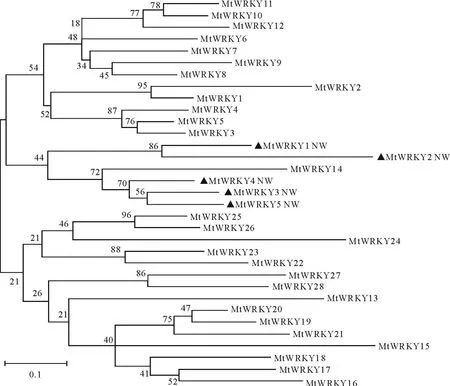
圖2 苜蓿WRKY結構域的系統發生樹Fig.2 Phylogenetic tree of M.truncatula WRKY domain
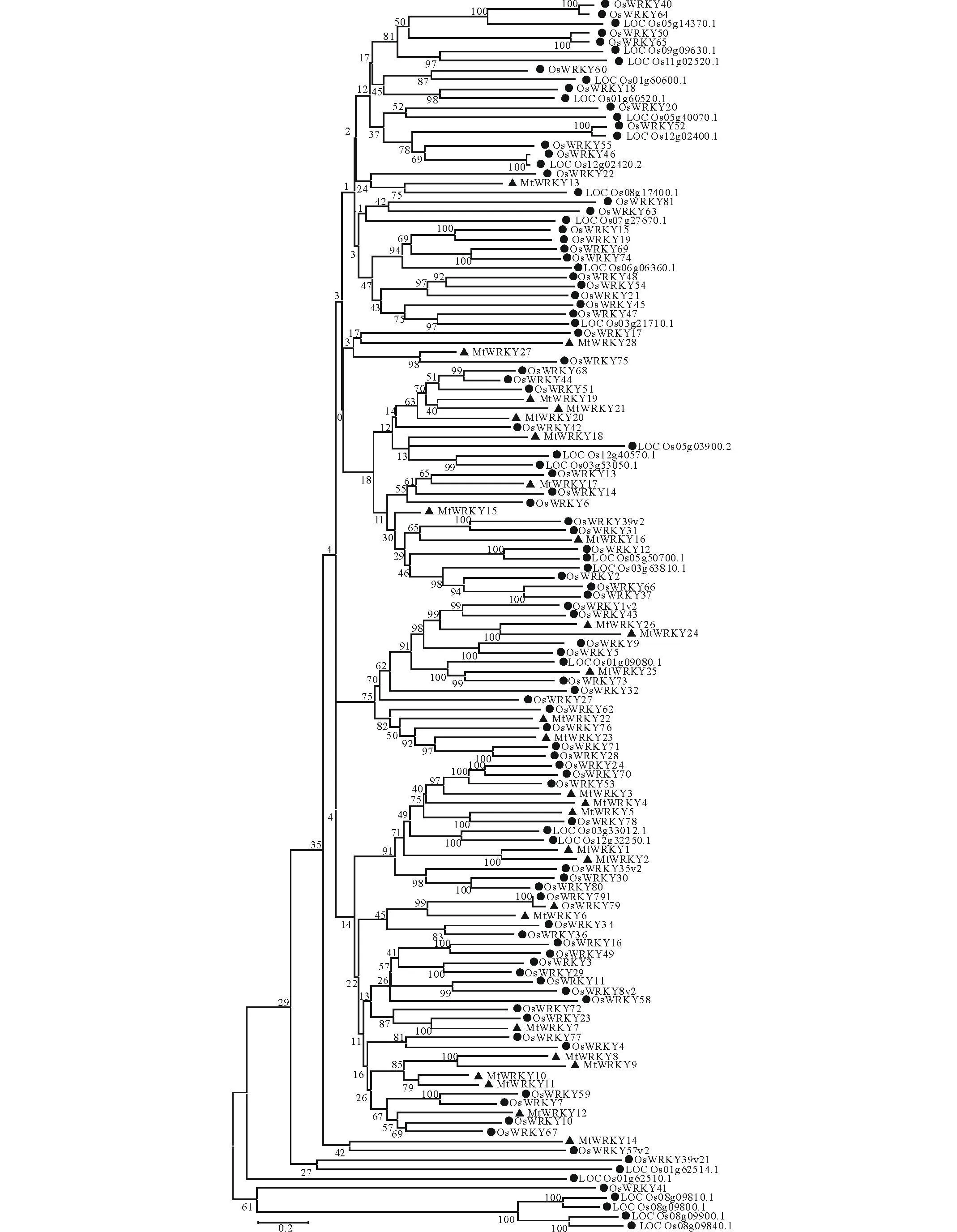
圖3 苜蓿和水稻WRKY基因的的系統發生樹Fig.3 Phylogenetic tree of rice and M.truncatula WRKY genes

圖4 苜蓿WRKY蛋白推測的保守motifFig.4 Forty six putative motifs identified in the WRKY family
3 討論
WRKY基因家族是植物特有的一類重要的轉錄因子基因,已經克隆的基因表明該基因廣泛參與植物的抗病、抗旱等逆境的調控,在細胞代謝和防衛反應中具有重要的作用。但是目前對WRKY基因的研究還處在一個初步認識的階段,大大地限制了人們對該類基因家族完全功能的認識和了解[28]。隨著眾多模式植物全基因組測序的完成,利用全基因組數據分析基因家族成為可能。本研究利用苜蓿最新的基因組數據(Mt2.0)進行WRKY基因家族的分析,詳細研究了該基因家族的特點和進化關系,并發現其與水稻、擬南芥WRKY基因具有一定的差異。

表2 苜蓿推測的WRKY結構域的motifTable 2 M.truncatula putative WRKY domain motif
在苜蓿全基因組中共有28個WRKY基因被確認,占整個基因總數的0.072%,而在擬南芥中這一比例為0.199%,水稻中這一比例為0.407%,無論是基因的總數還是所占比例,苜蓿都遠遠少于模式植物擬南芥和水稻。已經有研究表明,水稻和擬南芥WRKY基因家族存在較明顯的基因復制現象,而苜蓿中這一現象并不是很明顯,此外,苜蓿28個基因在7條染色體都有分布,相對于擬南芥和水稻而言分布不集中,說明了苜蓿基因組中WRKY基因復制事件較少,這可能是苜蓿WRKY基因較少的原因之一[29]。此外,苜蓿WRKY基因的同源性較高,在進化樹上分布相對集中,而水稻WRKY基因差異較大,不少基因分布在進化樹外圍,苜蓿相對水稻而言,WRKY基因較為保守。
對苜蓿和水稻WRKY基因的系統發生學分析證實了水稻和苜蓿的大部分WRKY基因并不在同一分枝上,說明了水稻和苜蓿的WRKY基因在進化上具有較大差異,進一步證實了這些差異可能早在單子葉與雙子葉植物分化前就已經產生,這也進一步說明了苜蓿的WRKY基因在進化上比較保守,而水稻的WRKY在進化上發生了更多的基因復制,導致WRKY基因的快速擴張。對苜蓿WRKY結構域的系統進化樹分析證實了位于N端的WRKY結構域與C端具有較大的差異,并不是結構域的簡單復制而來,可能起源于不同的結構域,特別是第1組5個基因的N端WRKY在一個分枝上,C端WRKY也在同一分枝上,說明了N端與C端WRKY結構域的起源不同。
[1]Arce A L,Cabello J V,Chan R L.Patents on plant transcription factors[J].Recent Patents on Biotechnology,2008,2(3):209-217.
[2]Jain M,Tyagi A K,Khurana J P.Genome-wide identification,classification,evolutionary expansion and expression analyses of homeobox genes in rice[J].Febs Journal,2008,275(11):2845-2861.
[3]Eulgem T,Somssich I E.Networks of WRKY transcription factors in defense signaling[J].Current Opinion in Plant Biology,2007,10:366-371.
[4]張秋平,楊宇紅,謝丙炎,等.植物轉錄因子的超表達及其在分子育種中的應用[J].分子植物育種,2005,3(3):401-408.
[5]Chujo T,Takai R,Akimoto-Tomiyama C,et al.Involvement of the elicitor-induced gene OsWRKY53 in the expression of defense-related genes in rice[J].Biochemica et Biophysica Acta,2007,1769:497-505.
[6]Li J,Brader G,Palva E T.The WRKY70 transcription factor:A node of convergence for jasmonate-mediated and salicylatemediated signals in plant defense[J].Plant Cell,2004,16:319-331.
[7]Liu X,Bai X,Wang X,et al.OsWRKY71,a rice transcription factor,is involved in rice defense response[J].Journal of Plant Physiology,2007,164:969-979.
[8]Peng Y,Laura E B,Chen X W,et al.OsWRKY62 is a negative regulator of basal and Xa21-mediated defense against Xanthomonas oryzae pv.oryzae in rice[J].Molecular Plant,2009,1:446-458.
[9]Qiu D,Xiao J,Ding X H,et al.OsWRKY13 mediates rice disease resistance by regulating defense-related genes in salicylateand jasmonate-dependent signaling[J].Molecular Plant-microbe Interactions,2007,20:492-499.
[10]Eulgem T,Rushton P J,Robatzek S,et al.The WRKY superfamily of plant transcription factors[J].T rends in Plant Science,2000,5:199-206.
[11]田云,盧向陽,彭麗莎,等.植物WRKY轉錄因子結構特點及其生物學功能[J].遺傳,2006,28(12):1607-1612.
[12]Shimono M,Sugano S,Nakayama A,et al.Rice WRKY45 plays a crucial role in benzothiadiazole-inducible blast resistance[J].Plant Cell,2007,19:2064-2076.
[13]Xu X,Chen C,Fan B,et al.Physical and functional interactions between pathogen-induced arabidopsis WRKY18,WRKY40,and WRKY60 transcription factors[J].Plant Cell,2006,18:1310-1326.
[14]Hara K,Yagi M,Kusano T,et al.Rapid systemic accumulation of transcripts encoding a tobacco WRKY transcription factor upon wounding[J].Molecular and General Genetics,2000,263:30-37.
[15]M eyersa B,Kozika A,Griego A,et al.Genome-wide analysis of NBS-LRR-encoding genes in arabidopsis[J].Plant Cell,2003,15:809-834.
[16]Wu K L,Guo Z J,Wang H H,et al.The WRKY Family of transcription factors in rice and arabidopsis and their origins[J].DNA Research,2005,12:9-26.
[17]張改娜,賈敬芬.豌豆清蛋白1(PA1)基因的克隆及對苜蓿的轉化[J].草業學報,2009,18(3):117-125.
[18]劉圈煒,王成章,嚴學兵,等.苜蓿青飼對波爾山羊屠宰性狀及肉品質的影響[J].草業學報,2010,19(1):158-165.
[19]何樹斌,劉國利,楊惠敏.不同水分處理下紫花苜蓿刈割后殘茬的光合變化及其機制[J].草業學報,2010,18(6):192-197.
[20]燕麗萍,夏陽,梁慧敏,等.轉BADH基因苜蓿T1代遺傳穩定性和抗鹽性研究[J].草業學報,2010,18(6):65-71.
[21]馮毓琴,曹致中.天藍苜蓿野生種質的品質分析研究[J].草業科學,2009,26(10):80-84.
[22]Boutrot F,Chantret N,Gautier M F.Genome-wide analysis of the rice and arabidopsis non-specific lipid transfer protein(nsLtp)gene families and identification of wheat nsLtp genes by EST data mining[J].BioMed Central Genomics,2008,9:86.
[23]Wang D,Guo Y,Wu C,et al.Genome-wide analysis of CCCH zinc finger family in arabidopsis and rice[J].BioMed Central Genomics,2008,9:44.
[24]Zhou T,Wang Y,Chen J Q.Genome-wide identification of NBS genes in japonica rice reveals significant expansion of divergent non-TIR NBS-LRR genes[J].Molecular Genetics and Genomics,2004,271(4):402-415.
[25]Higgins D G,Thompson J D,Gibson T J.CLUSTAL W:Improving the sensitivity of progressive multiple sequence alignment through sequence weighting,position-specific gap penalties and weight matrix choice[J].Nucleic Acids Research,1994,22:4673-4680.
[26]Tamura K,Dudley J,Nei M,et al.MEGA4:molecular evolutionary genetics analysis(M EGA)software version 4.0[J].M olecular Biology and Evolution,2007,24:1596-1599.
[27]Bailey T L,Elkan C.The value of prior knowledge in discovering motifs with MEME[J].Proceedings International Conference on Intelligent Systems for Molecular Biology,1995,3:21-29.
[28]Pandey S P,Somssich I E.The role of WRKY transcription factors in plant immunity[J].Plant Physiology,2009,150(4):1648-1655.
[29]Lai Z,Vinod K,Zheng Z.Roles of arabidopsis WRKY3 and WRKY4 transcription factors in plant responses to pathogens[J].BioMed Central Plant Biology,2008,8:68.

