批次和流加培養中國倉鼠卵巢工程細胞的細胞周期相關基因轉錄譜分析
劉興茂,葉玲玲,劉紅,李世崇,王啟偉,吳本傳,陳昭烈
軍事醫學科學院生物工程研究所,北京 100071
應用全基因表達譜芯片考察哺乳動物細胞在培養過程中的基因表達譜,不僅可以反映調控細胞生長、代謝、增殖、凋亡的相關信號通路的關鍵基因表達狀態,而且可以揭示細胞生長代謝動力學變化的內在原因,為細胞培養過程的優化控制提供理論依據[1-4]。近年來,隨著基因組學和蛋白組學研究的拓展和深入,DNA芯片技術及蛋白組芯片技術越來越廣泛地用于考察哺乳動物細胞自身及其在不同細胞培養工藝條件下的基因表達差異[5-8]。
批次培養和流加培養是哺乳動物細胞大規模培養技術體系中的兩種最為常用的操作模式[9-10]。批次培養和流加培養過程中營養成分耗竭和代謝產物積累等環境因素的變化,必將影響到細胞周期調控基因的表達,進而對培養細胞的生長密度和細胞表達產物的生產效率產生負面影響[11]。基于此,本研究以表達人重組尿激酶原CHO工程細胞系11G-S為研究對象,試圖運用基因芯片技術揭示CHO工程細胞在批次培養及流加培養不同生長階段參與細胞周期調控相關基因的表達譜及調控細胞增殖相關信號通路的關鍵基因的表達狀態,為探尋進一步提高細胞生長密度和細胞表達產物生產效率的可能途徑提供依據。
1 材料與方法
1.1 材料
1.1.1 細胞株
表達人重組尿激酶原 CHO工程細胞系 11G-S (由軍事醫學科學院生物工程研究所構建、保藏)。
1.1.2 培養基
CHO工程細胞批次培養和流加培養所用無血清培養基為添加了胰島素、腐胺、轉鐵蛋白、微量元素、Pluronic F-68及硫酸葡聚糖等成分的DMEM/F12 (1∶1,V/V)[12]。流加培養基為由葡萄糖、谷氨酰胺、氨基酸、微量元素、維生素、無血清添加成分、無機鹽及次黃嘌呤等組成的混合液。
1.1.3 基因芯片
CHO工程細胞基因表達水平分析采用安捷倫公司 (Aglient Technologies) 的小鼠全基因組表達譜芯片小鼠全基因組芯片 (Whole mouse genome microarray kit),其性能參數見表1。
1.1.4 基因芯片分析樣品
選取細胞接種時、無血清懸浮批次培養和流加培養的不同生長階段的 CHO工程細胞用作基因分析樣品,樣品的選取和說明見表2。
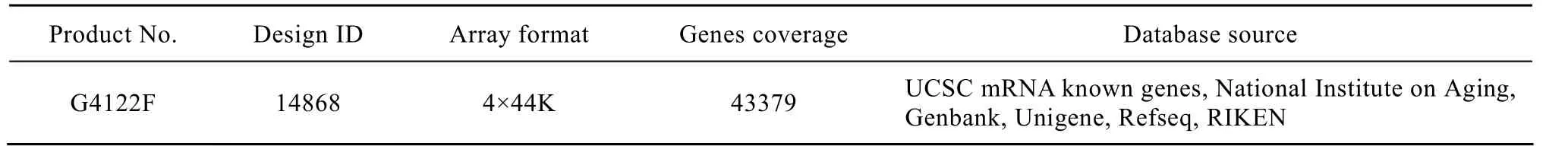
表1 Aglient小鼠全基因組表達譜芯片Table 1 Specifications of Aglient whole mouse genome microarray kit
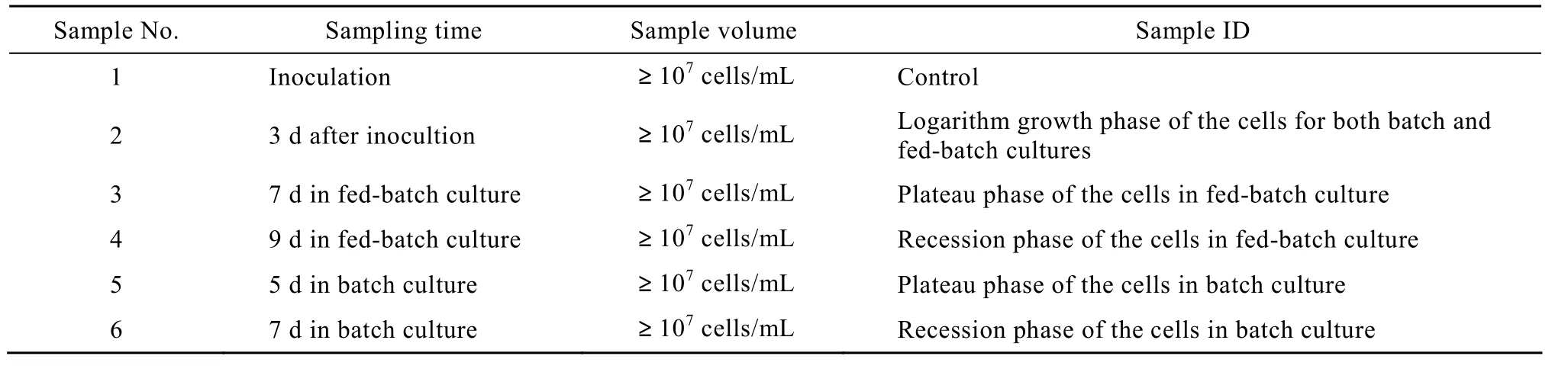
表2 用于基因芯片分析的樣品Table 2 Samples for comparative transcriptional analysis by gene chip
1.2 方法
1.2.1 細胞培養
CHO工程細胞以3×105cells/mL接種于100 mL三角瓶內,培養體積 35 mL,培養時加入含有5 mmol/L谷氨酰胺、0.1% (W/V) Pluronic F-68及25 μg/mL硫酸葡聚糖的無血清培養基,37 ℃、90 r/min懸浮培養。流加培養于接種后72 h,每24 h加入3 mL流加培養基。
1.2.2 細胞周期的檢測
分別于CHO工程細胞批次培養24、72、120、168 h和流加培養72、120、168、216 h各取細胞約1×106個,用75%的冰乙醇固定1 h,?20 ℃保存。流式細胞儀分析前先用PBS洗滌細胞,加入0.2 mL RNase A,37 ℃水浴消化30 min,再加入0.3 mL碘化丙啶 (Propidium iodide,PI) 置暗處染色20 min,用流式細胞儀 (BD FACSCalibur,USA) 檢測細胞周期。
1.2.3 樣品RNA抽提、基因芯片雜交及分析
取約1×107個細胞加入1.5 mL EP管中,采用Trizol 試劑盒提取RNA。采用苯酚/氯仿層相分離法純化總RNA,將提取的RNA保存于?70 ℃超低溫冷柜保存。用Nanodrop分光光度計測定RNA在260、280及230 nm的吸收值,以計算濃度并評估純度。同時,用甲醛電泳試劑進行變性瓊脂糖凝膠電泳,檢測RNA的純度及完整性。
用cy3熒光標記RNA樣品,然后利用等量的探針進行雜交,雜交條件為42 ℃、16 h,55 ℃洗片。采用Agilent掃描儀對芯片進行掃描,讀取數據,分辨率為5 μm、光電耦合裝置 (Photoelectric multiplication tube,PMT) 電壓100%,采用Gene spring軟件進行標定處理分析,并將樣品與對照比值取以2為底的對數,所得數值大于或等于1作為基因上調的篩選標準,所得數值小于或等于?1作為基因下調的篩選標準。
1.2.4 數據分析
根據測得的基因數據,首先將標準化的目標基因數據導入Genemapp軟件中,并設定標準以界定上調或下調基因差異表達的程度,運用已知有關小鼠細胞周期及信號通路圖,找出顯著差異表達的基因。
2 結果與分析
2.1 CHO 工程細胞批次和流加培養時的生長周期分布
圖1所示為CHO工程細胞無血清批次培養和流加培養過程中的活細胞密度變化。培養的前 3天,無血清批次培養和流加培養的 CHO工程細胞處于相同的培養條件,細胞處于對數生長期,活細胞密度呈現相同快速增加態勢。批次培養CHO工程細胞于第4天達到41.5×105cells/mL的最大活細胞密度,隨后活細胞密度呈現下降趨勢,培養10 d后的活細胞密度減低至18.4×105cells/mL;流加培養CHO工程細胞的快速生長期持續至第5天,于第6天達到78.1×105cells/mL的最大活細胞密度,隨后活細胞密度呈現下降趨勢,培養10 d后的活細胞密度減低至50.4×105cells/mL (圖1)。
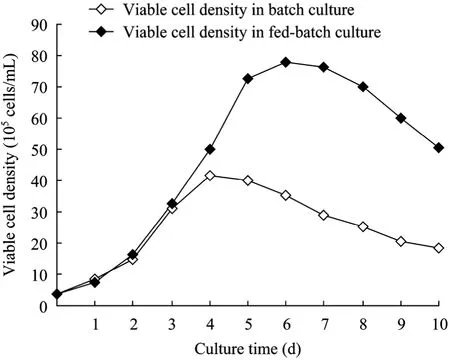
圖1 CHO工程細胞在批次和流加培養過程中的活細胞密度變化Fig. 1 Profile of viable cell density of the CHO cells in both batch and fed-batch cultures.
依照 CHO工程細胞無血清批次培養和流加培養過程中的活細胞密度變化,選取批次培養 24、72、120和168 h分別代表CHO工程細胞批次培養的對數生長早期、對數生長晚期、平臺期和衰退期;選取流加培養72、120、168和216 h分別代表CHO工程細胞流加培養的對數生長中期、對數生長晚期、平臺期和衰退期。批次培養的CHO工程細胞隨培養時間延長,G1期所占細胞比例明顯升高(圖2A);流加培養的 CHO工程細胞處于G2期的細胞比例隨培養時間延長明顯升高 (圖2B)。批次培養和流加培養的CHO工程細胞中,處于S期的細胞比例均隨培養進程呈現逐步下降的趨勢 (圖2A,B)。CHO工程細胞無血清批次培養和流加培養過程中的細胞周期分布變化與其各自的活細胞密度變化基本一致。
2.2 CHO工程細胞樣品的RNA的質量鑒定
圖3為各CHO工程細胞樣品經RNA抽提處理后的變性瓊脂糖凝膠電泳結果,各泳道均可見明亮的28S和18S核糖體RNA區帶以及稍顯暗淡和彌散tRNA和5S核糖體RNA區帶,表明各細胞樣品RNA抽提成功。表3為各提取RNA樣品的紫外分光光度計檢測結果,OD260/OD280比值介于1.8到2.0之間,OD260/OD280比值大于1.8,表明提取的總RNA樣品質量完好。
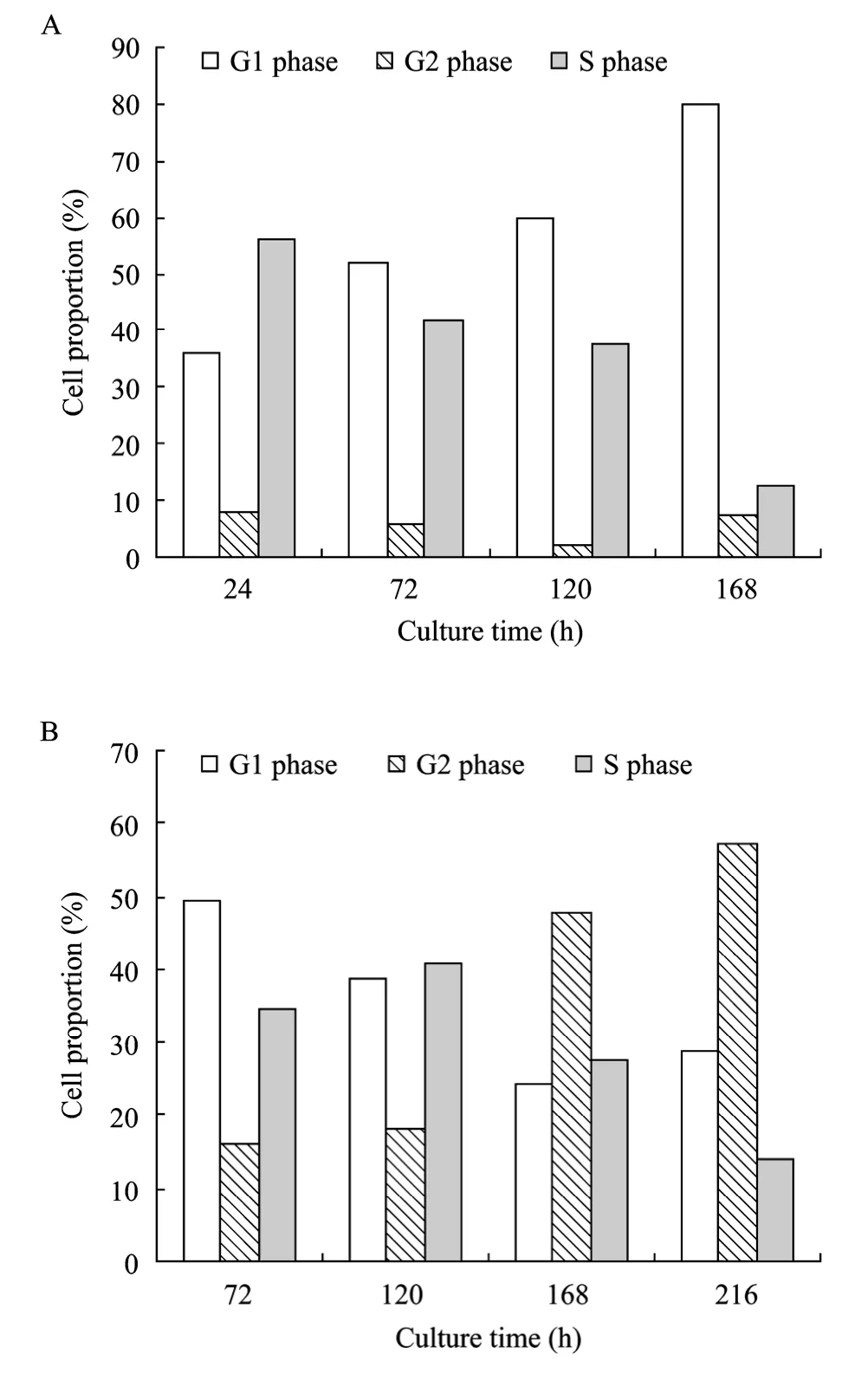
圖2 CHO工程細胞批次培養 (A) 和流加培養 (B) 過程中不同生長階段的細胞周期分析Fig. 2 Cell cycle analysis of the CHO cells in different growth phase in both batch (A) and fed-batch (B) cultures.
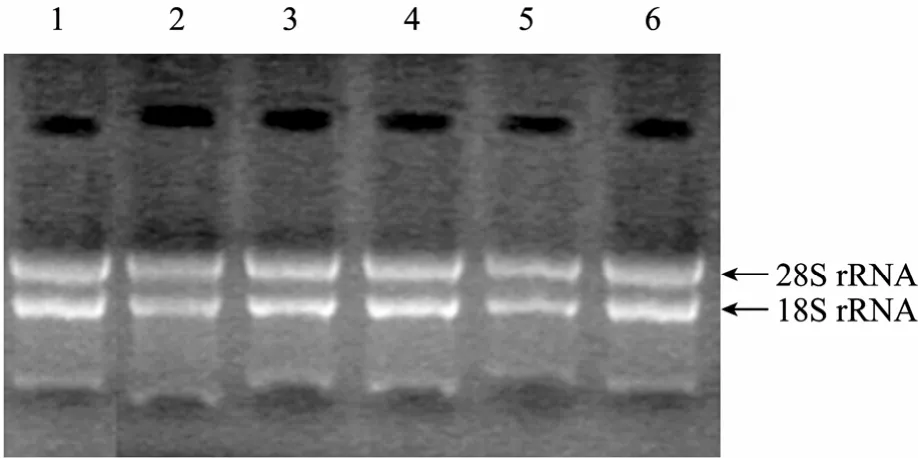
圖3 總RNA樣品的變性瓊脂糖凝膠電泳Fig. 3 Total RNA analysis of the CHO cells by denatured agarose electrophoresis. 1?6: total RNA of sample 1 to 6 respectively.
2.3 基因芯片實驗數據分析
基因芯片實驗數據來自于 CHO工程細胞批次培養及流加培養對數生長期、平臺期及衰退期 3個階段的樣品,以培養起始接種的細胞作為對照,其他樣品的每個基因標準化值與之相比,所得數值取以2為底的對數。圖4A、B分別為CHO工程細胞在兩種培養模式下比值大于1或小于?1及大于2或小于?2的基因上調及下調表達的差異狀況。在基因芯片分析涉及的19 191個目標基因中,有差異表達的基因較多。總體而言,下調表達的基因數量多于上調表達基因數目,且上調表達基因其比值大多介于1和2之間,下調表達基因其比值大多介于?2和?1之間。在細胞生長的對數期及平臺期,上調及下調表達的基因數目雖有差異,但總體上處于相對平衡的狀態,說明細胞在較好的培養環境中,其細胞正負調控基因的表達相對平衡;在細胞生長的衰退期,下調表達的基因數目明顯多于上調表達的數目,特別是顯著下調表達的基因所占的比例顯著升高,說明隨著培養環境的惡化,細胞更多是以下調表達基因的方式,來適應不斷變化的培養環境及維持自身的活力。對比兩種培養模式的差異表達基因的方式及數量也有著明顯的差異,特別是在流加培養的衰退期,下調表達的基因數量明顯多于批次培養的衰退期,提示在流加培養模式下,隨著培養體系中營養物、有毒代謝廢物累積及滲透壓的提高,細胞對環境的應激調控強度遠遠高于細胞在批次培養模式下由于少數營養物缺乏所產生的應激調控。
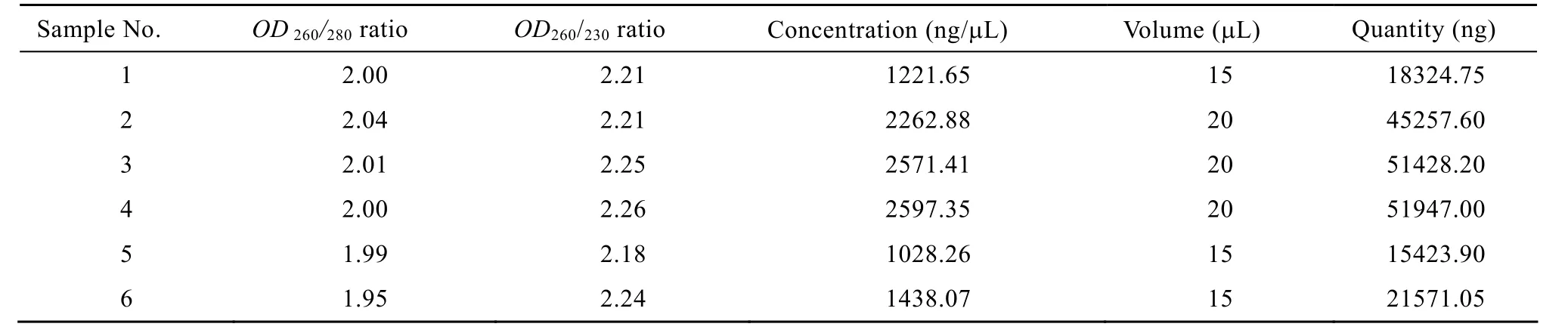
表3 用紫外吸光法測定RNA濃度和純度Table 3 Concentration and purity of the total RNA from the CHO cells determined by ultroviolet absorption

圖4 細胞無血清批培養及流加培養的基因表達差異Fig. 4 Number of differentially regulated genes of both batch and fed-batch cultures at different cell growth phases using two different significance criteria: fold change≥1 (A) or 2 (B) at P-value≤0.05. a: logarithm growth phase; b: plateau phase; c: recession phase of the cells.
2.4 兩種培養模式下與細胞周期相關基因表達的差異
CHO工程細胞在批次培養和流加培養過程中,細胞周期調控基因雖然呈現基本相同的以下調表達為主的轉錄譜,但流加培養過程中參與細胞周期調控的上調表達基因僅為 Smad4,明顯少于批次培養CHO工程細胞中的上調基因 (圖5)。
在涉及細胞周期的86個主要基因中,批次培養對數期生長期細胞的差異表達基因包括,G1期上調表達的 Hdac7a基因及下調表達的 Tfdp1、Ccne2、Hdac5、E2f5基因;S期上調表達的Orc1l及下調表達的 Orc5l基因。細胞生長進入平臺期時,有差異表達的基因明顯增多,主要有 G1期上調表達的Gsk3b、Ccnd2、Tfjp1、Hdac7a基因及下調表達的Ccne1、Ccne2、Cdk6、Hdac5、E2f5基因;S期上調表達Trp53、Orc1l、Orc2l、Mcm2基因及下調表達的 Orc5l、Ywhag、Cdc45l、Pcna,M 期的基因Espl1。當細胞生長進入衰退期后,上調表達的基因明顯減少,上調表達的基因主要有 G1期的 Smad4及Hdac7a基因;下調表達因與平臺期相比沒有太大的差異,下調表達的基因主要包括 G1期的基因Ccne1、Ccne2、Cdk2、Hdac5及E2f5;S期的Ywhag、Pcna、Cdk2、Cdc45l、Orc4l及Orc5l基因;G2期的Cdc2a基因和M期的Espl1基因。Hdac7a基因在CHO細胞批次培養的各階段均上調表達,Ccne2、Orc5l、Hdac5及E2f5在批次培養的各階段均下調表達,Ywhag、Pcna、Ccne1、Cdc45l及Espl1在批次培養的平臺期及衰退期均下調表達。
細胞在流加培養模式下的流加培養階段差異表達的基因以下調表達為主,且與批次培養模式下細胞的平臺期及衰退期差異表達的基因大體相近,上調表達的基因僅有Smad4基因。在流加培養的平臺期,下調表達的基因主要有G1期的Ccne1及E2f1,S期的Ywhag,G2期Cdc2a及Plk1,M期的Espl1;當細胞生長進入衰退期后,差異表達的基因明顯增加,并且基本上都為下調表達,僅有 G1期的基因Smad4上調表達。下調表達的基因包括G1期的基因Ccne1、Ccne2、Hdac5、Tfdp1、Hdac7a及E2f1,S期的基因Ywhag、Ccnh、Orc5l、Cdc45l及Cdc7,G2期的基因Mad2l1、Cdc2a及Plk1,M期的基因Espl1。
3 討論
細胞周期進程的實現有賴于各級調控因子對細胞周期精確而嚴密的調控。這些調控因子的核心是細胞周期蛋白依賴性蛋白激酶 (Cyclin dependent kinases,CDKs) 及其正、負性調控因子——細胞周期蛋白 (Cyclin) 和細胞周期蛋白依賴性蛋白激酶抑制劑 (CDI)[13-14]。CDKs屬于絲氨酸/蘇氨酸蛋白激酶家族,可在特定的細胞周期被激活,之后磷酸化相應的底物,從而引起后續事件的發生。Cyclin作為蛋白激酶復合體的調節亞基,對 CDKS起著正性調節作用。在高等真核細胞中,Cyclin 分為A、B、C、D、E和H六類。它們分別在細胞周期的不同時相中合成、積累,并與相應的CDKs結合,激活CDKs的蛋白激酶活,從而調節細胞周期進程。同時 CDKs的活性也可以被細胞周期抑制蛋白(Cell cycle inhibitory proteins,CKIs) 所抑制。
從依照細胞周期調控的信號通路圖分析,批次培養對數期生長期的CHO工程細胞,僅Cyclin E2發生了下調表達,其他基因表達均沒有明顯差異。隨著細胞生長進入平臺期及衰退期,CDKs、Cyclin家族中的相應基因的數目在細胞的G1、S及G2期下調表達明顯增加,主要有基因 Cdk6、Cdk2、Cdc2a、Ccne1及Ccne2,且細胞在衰退期下調表達基因多于平臺期。同時TGF2β信號轉導途徑的中心分子Smad4在批次培養CHO工程細胞的衰退期上調表達。研究表明,Smad4的失活是引起細胞生長抑制喪失的一個重要原因[15]。批次培養CHO工程細胞在衰退期通過上調表達Smad4進一步抑制了細胞的生長。反映在細胞周期分布上,CHO工程細胞在對數期 G1期細胞所占的比例較低,S期細胞所占的比例較高,細胞可以快速增殖;在進入培養的平臺期及衰退期后,G1期細胞所占的比例不斷升高,S期細胞所占的比例卻逐漸降低,大多數細胞停滯于G1期,細胞生長緩慢或停止。
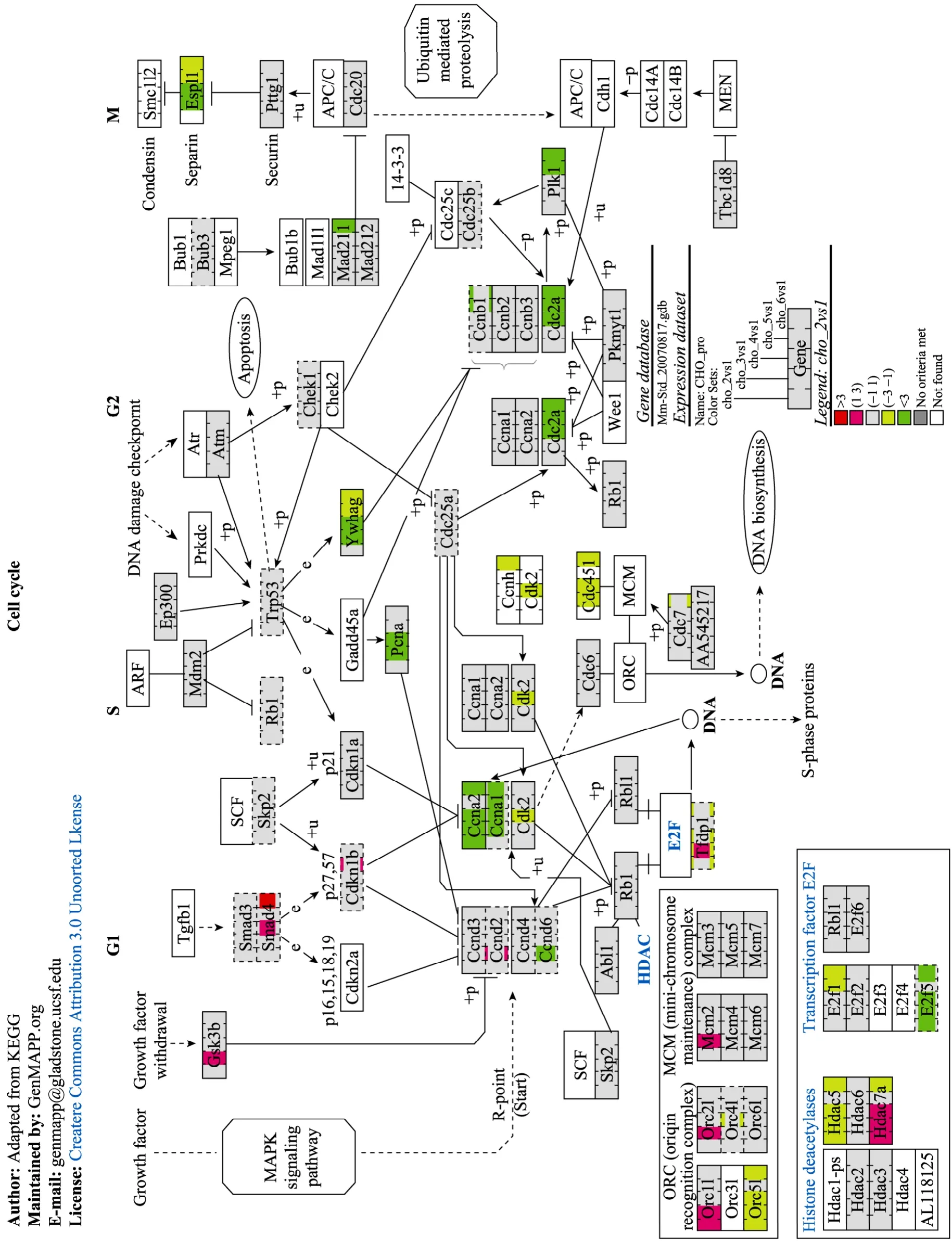
圖5 批次和流加培養中細胞的細胞周期相關基因轉錄譜Fig. 5 Transcriptional profiling of cell cycle regulatory networks of cells in batch and fed-batch cultures.
流加培養 CHO工程細胞在細胞生長的平臺期及衰退期,CDKs、Cyclin及CKI家族中的相應基因在G1、S及G2期下調表達基因的數量逐漸增加,且衰退期下調表達的基因多于平臺期。與批次培養平臺期及衰退期 CHO工程細胞下調表達的基因數目相比,流加培養相應階段CHO工程細胞下調表達的基因少于批次培養。同時Smad4在培養衰退期也發生了上調表達,且強度高于批次培養。表明由于導致 CHO工程細胞在批次培養及流加培養模式下增殖減緩的原因不同,因此在細胞周期調控基因的表達也有所差異。反映在細胞周期分布上,CHO工程細胞在流加培養過程中,G1期細胞所占的比例呈現出先降后升的變化趨勢,S期細胞所占的比例呈現出先升后降的變化趨勢。
本研究獲得的批次培養和流加培養 CHO工程細胞的細胞周期調控基因表達譜提示,CHO工程細胞在批次培養和流加培養過程中,主要是通過下調表達CDKs、Cyclin及CKI家族中的Cdk6、Cdk2、Cdc2a、Ccne1、Ccne2基因及上調表達Smad4基因,實現細胞增殖的調控。
REFERENCES
[1] Griffin TJ, Seth G, Xie H, et al. Advancing mammalian cell culture engineering using genome-scale technologies. Trends Biotechnol, 2007, 25(9): 401?408.
[2] Kantardjieff A, Nissom PM, Chuah SH, et al. Developing genomic platforms for Chinese hamster ovary cells. Biotechnol Adv, 2009, 27(6): 1028?1035.
[3] Charaniya S, Karypis G, Hu WS. Mining transcriptome data for function-trait relationship of hyper productivity of recombinant antibody. Biotechnol Bioeng, 2009, 102(6): 1654?1669.
[4] Yee JC, Gerdtzen ZP, Hu WS. Comparative transcriptome analysis to unveil genes affecting recombinant protein productivity in mammalian cells. Biotechnol Bioeng, 2009, 102(1): 246?263.
[5] Lee YY, Wong KTK, Nissom PM, et al. Transcriptional profiling of batch and fed-batch protein-free 293-HEK cultures. Metab Eng, 2007, 9(1): 52?67.
[6] Wong DC, Wong KTK, Lee YY, et al. Transcriptional profiling of apoptotic pathways in batch and fed-batch CHO cell cultures. Biotechnol Bioeng, 2006, 94(2): 373?382.
[7] Yee JC, de Leon Gatti M, Philp RJ, et al. Genomic and proteomic exploration of CHO and hybridoma cells under sodium butyrate treatment. Biotechnol Bioeng, 2008, 99(5): 1186?1204.
[8] djieff AK, Jacob NM, Yee JC, et al. Transcriptome and proteome analysis of Chinese hamster ovary cells under low temperature and butyrate treatment. J Biotechnol, 2010, 145(2): 143?159.
[9] Merten OW. Introduction to animal cell culture technology?past, present and future. Cytotechnology, 2006, 50(1/3): 1?7.
[10] Whitford WG. Fed-batch mammalian cell culture in bioproduction. BioProcess International, 2006, 4: 30?40.
[11] Wlaschin KF, Hu WS. Fedbatch culture and dynamic nutrient feeding. Adv Biochem Engin/Biotechnol, 2006, 101: 43?74.
[12] Liu XM, Liu H, Ye LL, et al. Serum-free medium for suspension culture of recombinant Chinese hamster ovary (11G-S) cells. Chin J Biotech, 2010, 26(8): 1116?1122.劉興茂, 劉紅, 葉玲玲, 等. CHO工程細胞 (11G-S) 懸浮培養的無血清培養基的設計. 生物工程學報, 2010, 26(8): 1116?1122.
[13] Loyer P, Trembley JH, Katona R, et al. Role of CDK/cyclin complexes in transcription and RNA splicing. Cell Signal, 2005, 17(9): 1033?1051.
[14] Morgan DO. Cyclin-dependent kinases: engines, clocks, and microprocessors. Annu Rev Cell Dev Biol, 1997, 13: 261?291.
[15] Attisano L, Wrana JL. Signal transduction by the TGF-beta superfamily. Science, 2002, 296(5573): 1646?1647.

