牛糞便微生物總DNA提取方法的優化
劉虎虎, 李亞丹, 薛高尚, 陳東明,3, 田 云*, 盧向陽*
(1.湖南省農業生物工程研究所,湖南長沙 410128; 2.湖南農業大學生物科學技術學院,湖南長沙 410128;3.懷化學院生命科學系,湖南懷化 418000)
21世紀能源危機日顯突出,開發可再生能源已引起世界各國的關注.發掘微生物資源對于發展生物質能源起到關鍵性作用,然而傳統常規方法篩選功能微生物存在一定的不足,即不能有效地開發未培養微生物資源 (約占99%).宏基因組技術的出現為開發未培養微生物提供了一條新的途徑.宏基因組是以生態環境中全部DNA作為研究對象[1],避開了微生物分離培養的問題,極大地擴展了微生物資源的利用空間.通過構建宏基因文庫研究微生物群落、挖掘新功能基因已日益成為熱點,樣品總DNA的質量是能否成功構建高質量的宏基因組文庫關鍵.
關于糞便微生物總DNA提取方法很多,如試劑盒法、化學—凍融研磨法與化學裂解法等.試劑盒法即采用生物公司生產的商業化試劑盒直接提取DNA;化學裂解法主要是利用溶菌酶、SDS與蛋白酶K等試劑使微生物細胞破裂進而提取DNA;化學—凍融研磨法主要是將物理方法與化學方法相結合提取DNA.但是,適合提取牛糞便微生物總DNA方法報道很少.據此本文選用試劑盒提取法、凍融-CTAB法、CTAB-SDSⅠ法與CTAB-SDSⅡ法四種不同的方法提取牛糞便微生物總DNA.通過分析DNA純度、顏色與提取量等指標,確定一種較為合適的牛糞微生物總DNA提取方法,以解決構建牛糞宏基因組文庫瓶頸問題.
1 材料與方法
1.1 實驗材料
飼養牛新鮮糞便,保存于-80℃超低溫冰箱.
1.2 主要儀器與試劑
1.2.1 儀器
CF-16RX型高速冷凍離心機、UVIpro型凝膠成像分析系統、Biophemeter型蛋白核酸定量檢測儀.
1.2.2 試劑
Biotech公司Lysozyme(10 mg/mL)、SIG MA公司proteinaseK、TIANGEN 公 司 RNaseA (10mg/mL)、INVITROGEN公司 PurelinkTMGenomic DNA MiNi KiT、DNA提取液 (100 mM磷酸鈉緩沖液pH 8.0、100 mM EDTA pH 8.0、100 mM Tris-HCl pH8.0、1.5 M NaCl、1%CTAB、2%SDS)、磷酸緩沖液 (PBS) (137 mM NaCl、2.7 mM KCl、10 mM Na2HPO4、2 mM KH2PO4,HCl調pH至7.4)
1.3 實驗方法
1.3.1 試劑盒提取法
樣品預處理:取-80℃保存牛糞樣品5 g,加20 mL PBS溶解,再加2%PVPP,37℃220 rpm搖30 min,500 rpm離心10 min取上清,重復2次.合并上清,8 000 rpm離心10 min,收集沉淀,加10 mL PBS,8 000 rpm離心10 min,棄上清,重復2次.所得沉淀中加入1 mL無菌水,按照PurelinkTM Genomic DNA MiNi Kit試劑盒說明提取DNA,加100μL含RNaseA(10 mg/mL)無菌水37℃消化1 h,-20℃保存.
1.3.2 凍融-CTAB法
參照Sangwon Lee等方法[2]稍作修改.取-80℃保存牛糞樣品5 g加入2 mL變性液研磨,加液氮冷凍再研磨,重復幾次,將樣品研磨成細粉狀即可.樣品移入50 mL離心管,加18 mL提取液,65℃水浴40 min,每15~20 min顛倒搖勻.1 800 g離心10 min,收集上清.沉淀中再加入10 mL提取緩沖液65℃水浴40 min,1 800 g離心10 min,收集上清.合并2次所得上清,加入等體積氯仿/異戊醇 (24∶1)并使其混合,1 800 g離心10 min,取上清.向上清液中加入0.6倍體積異丙醇,室溫靜置30 min.12 000 g離心20 min,棄上清.將所得沉淀無菌風吹干,加100μL含RNaseA(10 mg/mL)無菌水37℃消化1 h,-20℃保存.
1.3.3 CTAB-SDS法
參考ZHOU等方法[3],稍作修改.樣品預處理:取-80℃保存牛糞樣品5 g,加20 mL PBS溶解,再加2%PVPP,37℃220 rpm搖30 min,500 rpm離心10 min取上清,重復2次.合并上清,8 000 rpm離心10 min,棄上清,再加入10 mL PBS,8 000 rpm離心10 min,棄上清,重復2次.所得沉淀中加10 mL DNA提取緩沖液、10μL proteinase K(10 mg/mL)、10μL Lysozyme(10 mg/mL),37℃220 rpm搖30 min.加入5 mL 20%SDS,65℃水浴2 h,每15-20 min顛倒搖勻.12 000 rpm離心10 min,收集上清.沉淀中再加入4 mL DNA提取緩沖液,37℃220 rpm搖30 min,再加入1 mL 20%SDS,65℃水浴2 h,每15-20 min顛倒搖勻.12 000 rpm離心10 min,合并2次所得上清,加入等體積氯仿/異戊醇(2431),12 000 rpm離心10 min,取上清,如此重復2-3遍.Ⅰ:向上清液中加入預冷的2倍體積無水乙醇,1/10體積3 M醋酸鈉,-20℃靜置30 min;12 000 rpm離心10 min,棄上清,加入試管1/2體積70%乙醇,12 000 rpm離心10 min,重復2遍,將所得沉淀無菌風吹干,加100μL含RNaseA(10 mg/mL)無菌水37℃消化1 h,-20℃保存.Ⅱ:向上清液中加入0.6倍體積異丙醇,室溫靜置30 min.12 000 rpm離心10 min,棄上清,加入試管1/2體積70%乙醇,12 000 rpm離心10 min,重復2遍,將所得沉淀無菌風吹干,加入100 μL含RNaseA(10 mg/mL)無菌水37℃消化1 h,-20℃保存.
1.4 數據統計與分析


表1 四種提取方法獲得DNA相關比較
由表1可知,試劑盒法、凍融-CTAB法、CTABSDSⅠ法與CTAB-SDSⅡ法四種方法所得DNA A260/A280分別為 1.233±0.311、1.427±0.040、1.787±0.035、1.930±0.433;DNA提取量 (μg/g)分別為 2.453±0.311、15.727±0.427、17.867±0.162、19.870±0.433.DNA的純度可通過A260/A280比值表示,一般比值在1.8-2.0說明DNA純度較好,比值低于1.8說明樣品DNA存在蛋白質污染,比值高于2.0說明樣品存在RNA污染.樣品雜質的含量可以通過A260/A230比值來確定,即比值越高,雜質含量越低.試劑盒法提取DNA過程中需要過柱離心,由于雜質原因使得所得DNA純度與得率較低,提取效果較差;CTAB-SDSⅡ法使用異丙醇沉淀DNA,所得DNA純度較好,提取量較高;凍融-CTAB法使用液氮冷凍迅速研磨,由于物理作用可能影響DNA結構,提取量相對較少;CTAB-SDSⅠ法使用無水乙醇沉淀DNA出現白色晶體沉淀,可能導致提取量降低.白色晶體沉淀形成機理目前文獻尚未有具體說明,有待進一步研究.
2 結果
2.1 不同提取方法獲取總DNA樣品顏色差異
由圖1可知,四種方法在獲取DNA樣品顏色上存在差異:試劑盒提取法與CTAB-SDSⅠ法提取的DNA樣品顏色淺黃,凍融-CTAB法與CTAB-SDSⅡ法提取的DNA樣品顏色較透明.采用四種不同方法在獲取DNA樣品顏色上存在差異,可能是在沉淀DNA過程中由于使用不同有機溶劑造成的.試劑盒提取法與CTAB-SDSⅠ法使用無水乙醇沉淀DNA,凍融-CTAB法與CTAB-SDSⅡ法使用異丙醇沉淀DNA.為何使用無水乙醇與異丙醇沉淀DNA對樣品顏色造成不同的影響,有待進一步研究.
2.2 不同提取方法獲得DNA電泳結果
取5μL不同方法所提總DNA進行1%瓊脂糖凝膠電泳檢測,見圖2.由電泳圖可知四種方法所獲得DNA片段都在20 kb左右,其中方法3與4所得DNA片段在23 kb左右.四種方法所提取DNA電泳檢測不同程度出現彌散帶,而且電泳條帶亮度存在差異.電泳條帶亮度差異可反映不同提取方法在DNA提取效率方面存在的差異.CTAB-SDSⅡ法相對獲得DNA提取效率最高、電泳條帶亮;試劑盒法相對獲得DNA提取效率最低、電泳條帶較其他三種方法較暗.CTAB-SDSⅡ法所獲取DNA電泳條帶相對于凍融-CTAB法獲取DNA電泳條帶稍暗,這與CTAB-SDSⅠ法與凍融-CTAB法所相對應獲取DNA提取效率方面存在差異.出現這種現象一方面可能是由于CTAB-SDSⅠ法所獲取DNA中存在結晶物質引起;一方面可能CTAB-SDSⅠ法提取DNA降解程度相對較嚴重.樣品DNA電泳檢測出現彌散帶的原因:一方面DNA可能自行發生降解;另一方面可能是樣品背景DNA.本研究未設計實驗去除腸道脫落細胞與未消化食物等背景DNA,但是四種方法提取DNA片段大小在20 kb左右,滿足構建質粒文庫要求,可以進行后續分子實驗.
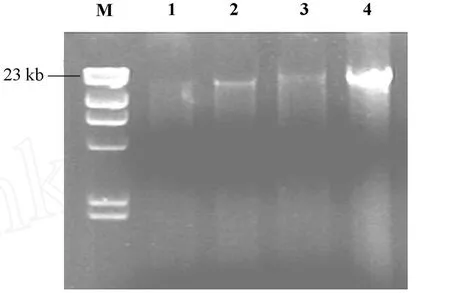
圖2 四種提取方法獲得DNA電泳檢測
3 討論
3.1 樣品取樣與保存
動物實驗樣品取樣一般包括傷害性取樣、非傷害性取樣與非損傷性取樣[7].糞便取樣則屬于非損傷性取樣,對動物體不造成生命危險.由于糞便中含有大量微生物,糞便一旦排出體外,須及時將樣品進行保存以防止目的DNA的降解.對于如何高效保存糞便樣品的方法的研究較少,但目前常用的保存方法一般包括低溫保存法、福爾馬林保存法、DETs(DMSO/EDTA/Tris鹽溶液)保存法、干燥保存法與乙醇保存法等[8].不同材料可以選擇不同的保存方法,本文將實驗材料直接-80℃保存,對于DNA提取無明顯影響.
3.2 糞便DNA提取與純化
食草反芻動物糞便中除含有參與食物代謝消化的腸道微生物之外,還存在大量腸道脫落細胞、未消化食物、膽堿、植物多糖與膽紅素等.這些雜質易導致目標DNA的降解,還對相關分子實驗 (如PCR反應、DNA連接與轉化等)有強烈的抑制作用.糞便DNA在質和量上的不足加大了抽提糞便DNA技術的難度,同時也增加了被污染的危險.因此,如何有效地去除糞便樣品內來自代謝 (消化酶、膽鹽等)或食物組分(植物多糖等)抑制劑,是實驗成功與否的關鍵[4].DNA的得率與純度受地理環境、操作條件、實驗方法等多方面因素的影響,但主要受細胞裂解方法和緩沖溶液這兩方面的影響[5].DNA提取方法大致分為物理法、化學法與酶促法.目前文獻中提取DNA的方法常是將三種方法搭配使用.添加PVPP與CTAB的作用主要是結合樣品中PCR抑制物,提高DNA純度.有文獻報道PVPP會吸附DNA,可導致DNA含量的損失[6].提取緩沖液中EDTA可作為金屬離子的螯合劑能抑制DNA酶的活性,從而防止DNA降解;Tris-HCl(pH8.0)為樣品提供一個緩沖液環境,防止核酸結構被破壞;NaCl可以提供一個高鹽環境;SDS的主要作用是裂解細胞以及使蛋白質與核酸分離.
DNA純化是獲取高純度DNA的關鍵步驟,目前純化DNA的方法一般包括電泳分離法、商品化的羥基磷灰石柱或硅膠柱法、葡聚糖凝膠柱法與玻璃粉吸附法等.純化過程應盡量避免DNA損失,保證構建基因文庫的有效性.
4 結論
經DNA純度檢測、樣品顏色以及電泳檢測分析,實驗認為CTAB-SDSⅡ法在獲取DNA純度以及含量上達到要求.與其他方法相比,該方法適合牛糞便微生物DNA提取.
[1]Rondon MR,August!PR,Bettermann AD,et al.Cloning the soil metagenome:a strategy for accessing the genetic and functional diversity of uncultured microorganisms[J].Appl Environ Microbiol,2000,66:2541-2547.
[2]Lee S,Hallam S J.Extraction of High Molecular Weight Genomic DNA from Soils and Sediments[J].J Vis Exp,2009,33(10):1569.
[3]Zhou J,Bruns MA,Tiedje JM.DNA recovery from soils of diverse composition[J].Appl Environ Microbiol,1996,62:316-322.
[4]趙建元,李進華.對影響哺乳動物糞便DNA提取相關因素的探討 [J].生物學雜志,2008,25(3):5-8.
[5]曹志明,鄭 維,權春善.土壤DNA提取方法的研究[J].大連名族學院學報,2005,7(3):94-94.
[6]Zhou J,Bruns MA,Tiedje JM.DNA recovery from soils of diverse composition[J].Appl Environ Microbiol,1996,62(2):316-322.
[7]Tabcrlet P,Waits L P,Luikart G.Noninvasive sampling:look before you leap[J].Trends Ecol Evolution,1999,14:323-327.
[8]李娜,李迪強,王秀磊,等.糞便不同保存方法對動物基因組DNA提取效果的影響 [J].國際遺傳學雜志,2006,29(5):341-346.

