應用16S rDNA克隆文庫篩查腹瀉病暴發病原體及特異毒力基因驗證*
黃新蓉,劉 劼,葉長蕓
2.湖北省黃岡市婦幼保健院,黃岡 436100;
3.傳染病預防控制國家重點實驗室,中國疾病預防控制中心傳染病預防控制所,北京 102206
感染性腹瀉病(Infectious diarrhea diseases)是目前世界性的公共衛生問題〔1〕,是一組由多種病原體引起的以腹瀉為主要癥狀的疾病,這些病原體包括細菌〔2〕、病毒〔3〕和寄生蟲〔4〕,成人主要以細菌性和寄生蟲性腹瀉為主,兒童主要以病毒性腹瀉為主〔5〕。目前,引起感染性腹瀉病的細菌主要有大腸桿菌屬(包括傳統的大腸埃希菌、志賀菌以及新近出現的O157 ∶H7 大腸桿菌〔6〕)、沙門氏菌屬 、霍亂弧菌 、金黃色葡萄球菌屬(產腸毒素型)等。長期以來,對于腹瀉患者的臨床細菌學檢測主要是分離糞便標本中的沙門氏菌、志賀菌、霍亂弧菌等腹瀉病原菌,然而,由于大多數患者發病后很快使用抗生素,而使細菌的分離培養結果不理想。同時,對一些新出現的腹瀉病原菌可能目前還沒有成熟的方法進行分離培養。因此,傳統的糞便細菌學檢驗方法已不能滿足診治的要求。近年來,16S rDNA克隆文庫作為一種不依賴于細菌培養、適合于在細菌種群多樣性體系中快速篩查疑似病原菌的新技術而引人關注。
傳染病預防控制國家重點實驗室經過反復試驗,建立了一套較完善的16S rDNA細菌學檢測技術平臺,該平臺適用于不同部位的標本,如糞便、痰液等〔7-8〕。本研究以該技術平臺為基礎,對某地腹瀉病暴發的病原體進行了篩查,并對篩查的結果進行了驗證。現將結果報告如下。
1 材料與方法
1.1研究對象 2006年7月,某地突然暴發100余例腹瀉病,當時實驗室沒有檢測到明確的病原體,因此,確定本次為不明原因腹瀉病暴發。本研究共收集到12例腹瀉患者的糞便標本,隨機抽取3例進行糞便總細菌16S rDNA檢測。3例腹瀉患者的基本資料為:1男,25歲,發病于2006年7月15日,2女,分別為46歲和 26歲,發病于2006年7月16日和18日。
1.2 方法〔7-8〕
1.2.1 標本采集 取腹瀉患者的水樣便或膿血便10g置于30ml無菌管中,加入磷酸鹽緩沖液(pH 7.2)15ml,振蕩混勻,200 r/min離心10min,棄掉沉渣,混懸液振蕩混勻,-20℃保存。
1.2.2 核酸提取 從-20℃凍存的試管中取200μ l水樣便或膿血便,按 QIAamp DNA Stool Mini Kit說明書提取糞便中的總細菌基因組。
1.2.3 引物設計 引用文獻上常用的27F-519R引物〔9〕,引物序列為上游引物(27F):5'-aga gtt tga tcy tgg yty ag-3',下游引物(519R):5'-gwa tta ccg cgg ckg ctg-3',簡并堿基的含義為:y代表t或c,w代表a或t,k代表 g或t。
1.2.4 PCR反應體系和反應條件:含Mg2+10×buffer 5μ L,10mmoL/l dNTPs 3μ L,正反向引物(濃度均為 10mmol/L)1μ L,混合酶(Taq:pfu=4 ∶1)5U,BSA(1mg/ml)5μ L,DNA 模板 100ng,加滅菌去離子水至總體系50μ L。反應條件:95℃預變性5min;95℃30s,58℃ 30s,72℃1 min,30個循環;72℃10 min。
1.2.5 連接、轉化和克隆子篩選 將PCR純化產物與T載體16℃水浴連接16 h,轉化JM109感受態細胞,并設空白對照和陽性對照。根據α-互補的原理進行克隆子藍白斑篩選,每份標本隨機挑選150個左右的白色克隆接送測序公司測序。
1.2.6 序列比對與分析 將得到的有效序列在網上進行Blast比較,根據比對結果判定每一個序列所對應的細菌,其判定標準為:1)取相似性(Identities)最高者;2)同一菌屬的相似性≥97%;3)當相似性最高者為 Uncultured bacteria,但在相似性 ≥97%中存在已知菌屬時,選擇已知菌屬作為比對結果。數據庫建立和統計圖繪制使用SPSS 14.0。
1.2.7 糞便標本中志賀菌的驗證 對用16S rDNA技術檢測到志賀菌屬的糞便標本,用志賀菌的特異性毒力基因ipaH引物進行兩次PCR擴增,取第一次PCR產物1μ L作為第二次PCR的模板。ipaH引物序列〔10〕為上游引物(ipaHF):5'-tgg aaa aac tca gtg cct ct-3',下游引物(ipaH R):5'-cca gtc cgt aaa ttc att ct-3'。預期PCR產物大小為420bp。
1.3 主要試劑與儀器 T-easy載體購自Promega,Taq酶、pfu酶、dNTPs、JM109感受態細胞購自Takara公司,糞便核酸提取試劑盒、質粒提取試劑盒、PCR產物膠純化試劑盒等購自QIAGEN公司,IPTG、X-Gal購自Sigma公司。引物合成和測序均在上海生物工程有限公司進行。PCR儀為型號為PE 9600。
2 結 果
2.1 細菌分離培養結果3例患者的糞便標本志賀菌、沙門氏菌、霍亂弧菌、副溶血弧菌的培養結果均為陰性。
2.2 16S rDNA檢測結果
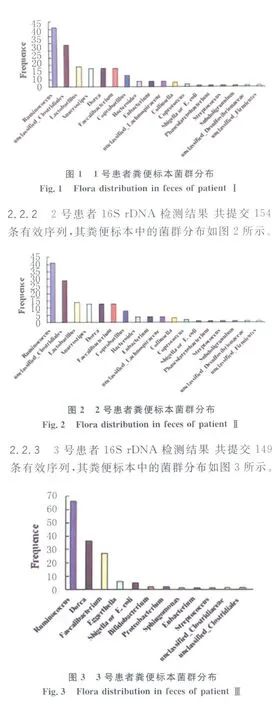
2.2.1 1號患者16S rDNA檢測結果共提交130條有效序列,其糞便標本中的菌群分布如圖1所示。
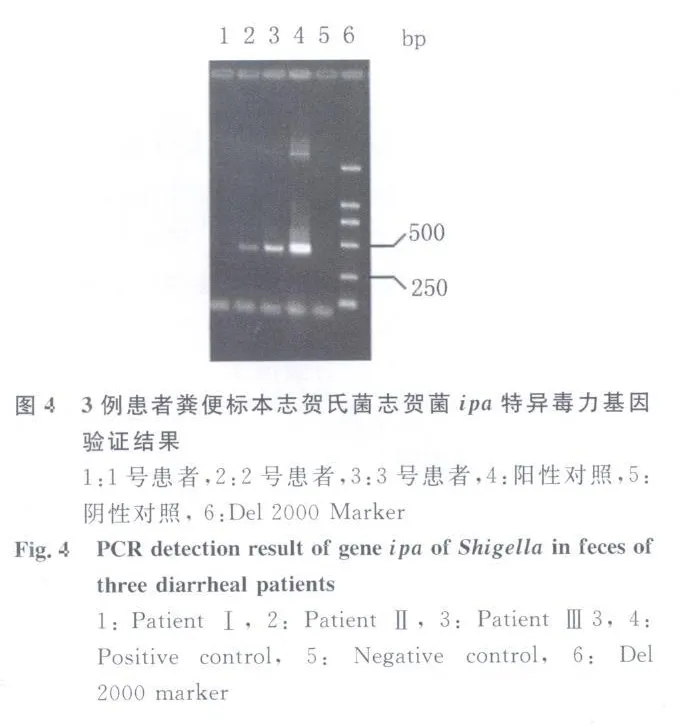
2.3 志賀菌特異性毒力基因驗證結果 對以上3例標本進行志賀菌的特異性毒力基因iapH檢測,結果見圖4。
2.4 12例腹瀉標本中志賀菌的檢出率 用針對志賀菌的特異基因iapH的引物對其它9份腹瀉糞便標本進行PCR檢測,共檢出6份陽性,總的檢出率為66.7%(8/12)。
3 討 論
目前,人類對腸道細菌的認識主要來自分離培養〔11〕,正常人群糞便標本的細菌數為 1012個菌細胞/g干便〔12-13〕,人的胃腸道寄居有400~500種細菌〔11〕,但約有60%~80%的胃腸道細菌是不可培養的〔12-13〕,因而影響了對腸道菌群的正確認識。雖然隨著培養基的不斷改進和新的細菌自動鑒定儀器的使用,以及對病毒性腹瀉和寄生蟲性腹瀉病原體的認識加深,腹瀉病原體的檢出率不斷提高,但仍有相當部分細菌性腹瀉難以查明病因,一方面是由于標本采集前可能已經使用抗生素,使致病菌達不到分離培養的數量,另一方面可能是由于新的病原菌引起。自 1996年 Wilson KH〔14〕用 16S rDNA 基因克隆文庫的方法分析一個正常人的結腸菌群以來,有不少關于16S rDNA菌群分布的研究,但研究對象大多是健康人或慢性疾病患者〔15-16〕,如 Antonia Suau等〔17〕分析了一個健康成年男子糞便標本的284個克隆子,發現了許多新的細菌核酸種類,提示了糞便標本中細菌的多樣性和16S rDNA基因克隆檢測未知細菌的可能性。
利用16S rDNA基因克隆文庫的方法可能從種類繁多的微生物群中得到病原菌的信息提示。本研究所涉及的某地腹瀉病暴發,糞便標本采集時大多數患者都經過2~3 d的抗生素治療,實驗室培養沒有檢測到明確的致病菌。在未明原因的情況下,隨機抽查3例患者進行16S rDNA基因檢測,發現3例患者的菌群分布基本一致,第一優勢菌均為Ruminococcus(瘤胃菌屬),該細菌屬于腸道正常菌群,其主要作用是分解纖維素,有利于食物的消化。瘤胃菌屬成為3位腹瀉患者的第一優勢菌群,其主要原因可能是患者使用抗生素以后,致病菌受到了抑制。在2號患者和3號患者的糞便標本中都篩到志賀菌或大腸桿菌,志賀菌是腹瀉的致病菌,因此,16S rDNA克隆文庫篩查的結果提示志賀菌可能是導致某地腹瀉暴發的原因。進一步用志賀菌的特異性毒力基因ipaH進行PCR檢測,在2號患者和3號患者中均檢測到了志賀菌核酸,驗證了16S rDNA克隆文庫的篩查結果。在所有收集的12份標本中,志賀菌檢出標本占66.7%(8/12),因此,志賀菌可能是本次腹瀉病暴發的主要病原體。16S rDNA克隆文庫篩查結果顯示,3號標本志賀菌在糞便細菌組成中占3.4%(5/149),2號標本只占0.6%(1/154);志賀菌iapH基因的檢測結果顯示,在相同的PCR反應體系和反應條件下,3號標本ipaH基因的PCR條帶明顯比2號標本的亮,說明3號標本的志賀菌基因組在糞便總細菌基因組中所占的比例比2號標本高,與16S rDNA克隆文庫篩查的結果一致。因此,16S rDNA基因克隆文庫的檢測結果基本能反映菌群組成的比例,經特異性毒力基因的驗證,其篩查的結果比較可靠。
在傳統分類學上,志賀菌和大腸桿菌分別屬于志賀菌屬和埃希菌屬,但二者的16S rDNA序列相似性大于99%,即用16S rDNA序列分析的方法不能鑒別志賀菌和大腸桿菌,但大腸桿菌多數情況下屬于正常菌群,且缺乏ipaH毒力基因,本實驗用16S rDNA序列結合ipaH基因的方法證明在12份糞便標本中8份存在志賀菌,排除了大腸桿菌是本次腹瀉暴發的原因。
傳染病預防控制工作多是應對群體性事件,應用16S rDNA克隆文庫研究暴發比散發更容易得到臨床和流行病學資料的支持,因人群中多個個體感染同一種病原體,其菌群改變具有相似性,分析起來更有規律,更易發現目標病原菌;同時,相同病例多,從身體其它部位(如血液、組織等)檢測到細菌的機會增多;多種支持證據是確證病原菌的重要條件。因糞便標本菌群組成復雜,細菌相互之間及與宿主之間關系復雜,腹瀉的原因又多種多樣,用16S rDNA基因文庫分析腸道菌群,在大海撈針般的病原菌搜索中,可起到定位和導航的作用,是一種較好的篩查方法。然而,該方法也受到很多因素的影響(如不同病程患者糞便中的致病菌數量不同,糞便細菌基因組的提取等),且工作量大,費用昂貴,因而實際中很難進行大樣本的操作。因此,要利用16S rDNA克隆文庫篩查及確證有菌部位標本中的病原菌,尚需大量的經驗和各種相關資料的支持。
〔1〕Cheng AC,McDonald JR,Thielman NM.Infectious diarrhea in developed and developing countries〔J〕.J Clin Gastroenterol,2005,39(9):757-773.
〔2〕Brooks JT,Ochieng JB,Kumar L,et al.Surveillance for bacterial diarrhea and antimicrobial resistance in rural western Kenya,1997-2003〔J〕.Clin Infect Dis,2006,43(4):393-401.
〔3〕Alain S,Denis F.Epidemiology of infectious acute diarrhoea in France and Europe〔J〕.Arch Pediatr,2007,Suppl 3 :S132-144.
〔4〕Stanley SL Jr.Amoebiasis〔J〕.Lancet,2003,361(9362):1025-1034.
〔5〕Vu Nguyen T,Le Van P,Le Huy C,et al.Etiology and epidemiology of diarrhea in children in Hanoi,Vietnam〔J〕.Int J Infect Dis,2006,10(4):298-308.
〔6〕徐建國,程伯鯤,馮麗,等.腸出血性大腸埃希菌O157∶H7引起的溶血性尿毒綜合征患者血清抗體調查〔J〕.中華流行病學雜志,2002,23(2):114-8.
〔7〕李振軍,戎建榮,張守印,等.16S rDNA序列分析在腹瀉糞便標本菌群分析應用〔J〕.中國人獸共患病學報,2008,24(2):100-103.
〔8〕李振軍,周永運,葉長蕓,等.咽部標本 16S rDNA序列分析方法的建立與應用〔J〕.中國人獸共患病學報,2008,24(3):229-232.
〔9〕Baker GC,Smith JJ,Cowan DA.Review and re-analysis of domain-specific 16S primers〔J〕.J Microbiol Methods,2003,55(3):541-555.
〔10〕Faruque S M,R Khan,M Kamruzzaman,et al.Isolation ofShigelladysenteriae type 1 andS.f lexneristrains from surface waters in Bangladesh:comparative molecular analysis of environmental Shigella isolates versus clinical strains〔J〕.Appl Environ Microbiol,2002,68(8):3908-3913.
〔11〕Moore WEC,Holdeman LV.Human fecal flora:the normal flora of 20 Japanese-Hawaiians〔J〕.Appl Environ Microbiol,1974,27(5):961-979.
〔12〕Langendijk PS,Schut F,Jansen GJ,et al.Quantitative fluorescence in situ hy bridization ofBif idobacteriumspp.with genus-specific 16S rRNA-targeted probes and its application in fecal samples〔J〕.A ppl Environ Microbiol,1995,61(8):3069-3075.
〔13〕Suau A,Bonnet R,Sutren M,et al.Direct analysis of genes encoding 16S rRNA from complex communities reveals many novel molecular species within the human gut〔J〕.Appl Environ Microbiol,1999,65(11):4799-4807.
〔14〕Wilson KH,Blitchington RB.Human colonic biota studied by ribosomal DNA sequence analysis〔J〕.Appl Environ Microbiol,1996,62(7):2273-2278.
〔15〕Ley RE,Backhed F,Turnbaugh P,et al.Obesity alters gut microbial ecology〔 J〕.Proc Natl Acad Sci U S A,2005,102(31):11070-11075.
〔16〕van Tongeren SP,Slaets JPJ,Harmsen HJM,et al.Fecal microbiota composition and frailty〔J〕.Appl Environ Microbiol,2005,71(10):6438-6442.

