大珠母貝微衛星DNA標記的分離與篩選
柳 明,喻達輝黃桂菊
(1.農業部海水養殖生態與質量控制重點開放實驗室,中國水產科學研究院 南海水產研究所,廣東 廣州510300; 2.上海海洋大學 水產與生命學院,上海 201306)
大珠母貝(Pinctada maxima),俗稱白蝶貝,屬于軟體動物門雙殼綱異柱目珍珠貝科珠母貝屬(Pinctada)。主要分布于熱帶海域,如澳大利亞、菲律賓、馬來西亞、印度尼西亞等地[1]。大珠母貝經濟價值很高,其肉(閉殼肌)質味道鮮美、營養豐富,堪稱為宴席佳品。它的殼形獨特,珍珠層厚而有美麗光澤,是名貴的工藝原料。用大珠母貝養殖出來的珍珠顆粒大,色澤好,價格高,既是貴重的裝飾品,又是名貴藥材。我國的大珠母貝資源本來就很少,屬于國家二級保護動物,過度利用使其更處于瀕危狀態,因此不能直接用野生大珠母貝進行育珠生產而必須使用人工繁殖個體。人工養殖貝育成的珍珠一致性和質量都較高。然而大珠母貝的海區養殖一直難以成功,幼貝出現大規模死亡現象,導致其珍珠養殖不能實現產業化開發。其原因除了養殖環境惡化外,種質資源的衰退也是重要原因之一。因此開展遺傳選育,改良種質是有效開發大珠母貝資源的重要途徑。在開發過程中,對選育群體進行遺傳多樣性分析和監測是十分必要的。微衛星DNA是遺傳多樣性分析和分子標記輔助育種最適遺傳標記之一。但大珠母貝的微衛星標記數量不多。Evans等[2]報道了6個多態標記和Smith等[3]報道了8個微衛星標記,遠不能滿足應用的需要,因此必須開發大量的微衛星標記。
本實驗采用生物素-磁珠富集法和探針雜交相結合的方法,構建了大珠母貝的微衛星富集文庫,篩選大珠母貝的微衛星標記,為大珠母貝的遺傳育種、保護生物學和種群遺傳多樣性等研究奠定基礎。
1 材料與方法
1.1 材料來源
用于微衛星分離的大珠母貝樣品于2008年8月采集于海南三亞,用于微衛星多態引物篩選的大珠母貝30個樣品個體于2007年7月采集于海南三亞養殖群體,取閉殼肌保存于95%酒精中。
1.2 實驗方法
1.2.1 基因組DNA提取
按照傳統的酚-氯仿法[4]提取基因組DNA,溶于100 μL滅菌雙蒸水中,-20℃保存待用。
1.2.2 基因組DNA酶切、接頭連接與PCR擴增
基因組DNA用內切酶MseI酶切3 h,20 μL反應體系包括:10×NEB Buffer2 2 μL,100×BSA 0.2 μL,MseI (10 U/μL) 0.5 μL,20 mg/LDNA 模板 3 μL,ddH2O 14.3 μL。將酶切產物與雙鏈接頭(接頭A序列為:5′-TAC TCA GGA CTC AT-3′,接頭 B 序列為:5′-GAC GAT GAG TCC TGA G-3′) 于 16 ℃連接過夜,反應體系如下:10 mmol/L T4 DNA ligase緩沖液2 μL,50 mmol/L 接頭 1.8 μL,酶切產物 11 μL,400 U/μL 的T4 DNA ligase 0.2 μL。連接產物用簡并引物MseI-N[5′-GAT GAG TCC TGA GTA A(N)-3′]進行 PCR 擴增,PCR 反應總體積 20 μL,包括 10×PCR buffer 2.0 μL,10 mmol/L dNTPs 0.4 μL,50 mmol/LMseI-N 0.2 μL,1UTaq酶,10×連接產物 2 μL。反應程序如下:95 ℃預變性5 min,然后進行PCR循環。循環參數為:95℃變性30 s,53 ℃退火1 min,72 ℃延伸1 min,循環數進行梯度篩選,分別做 14、17、20、23、26個循環,以確定最佳循環數,最后72 ℃延伸5 min。
1.2.3 探針雜交與磁珠富集
將雜交緩沖液70 μL、PCR產物25 μL和生物素標記的(CA)15探針(10 μmol/L)5 μL 混合,于 65 ℃雜交1 h。結束后,將磁珠加入到100 mL雜交液中,室溫溫浴 30 min,移去雜交液并保存。用 400 μL的TEN1000[5]于室溫下洗滌磁珠 3次,每次 5 min,保留第 3次的洗脫液。用 400 μL 的 0.2×SSC(含0.1%SDS)洗3次,每次5 min,保留第3次的洗脫液。用 400 μL TEN1000 再洗 5 min,保存洗脫液。加 100 μL TE重懸磁珠,100 ℃水浴10 min,將DNA從磁珠-探針-DNA混合物中分離,水浴結束后將離心管置于磁架上,吸出含有 DNA片段的洗脫液,然后立刻置于冰上。用100 μL TE再次重懸磁珠,保存洗脫液。以上述6管洗脫液為模板,用引物MseI-N進行PCR擴增,將 PCR擴增產物于 1.5%的瓊脂糖凝膠上電泳,檢測富集效果。富集效果好的樣品用 DNA Gel Extraction Kit(AXYGEN)回收純化PCR產物,純化產物按常規方法與pMD20-T載體16 ℃連接過夜,將連接產物轉化到大腸桿菌感受態細胞中進行培養,構建微衛星富集文庫。
1.2.4 富集文庫的篩選、測序與引物設計
挑選單克隆,接種到含有氨卞青霉素的LB培養基中,37 ℃振蕩培養2 h,菌液用載體通用引物和探針序列引物進行PCR篩選鑒定,經1.5%瓊脂糖凝膠中電泳檢測,選取符合條件的候選克隆送上海生工生物技術有限公司進行DNA測序。利用Clustal X[6]對序列特征進行分析,并用引物設計軟件primer 5.0對所得的微衛星序列設計引物。
1.2.5 微衛星多態引物的篩選
將引物稀釋成10 μmol/L,取海南三亞養殖群體共 30個個體,進行 PCR 擴增,體系如下:10×PCR buffer 2.0 μL,10 mmol/L dNTPs 0.4 μL,10 μmol/L正、反向引物各 0.5 μL,5 g/LTaq酶 0.2 μL,20 mg/L DNA模板2μL,ddH2O 14.4 μL。通過調整退火溫度,來獲得最佳PCR條件,將PCR產物用1.5%瓊脂糖凝膠進行電泳,EB染色,將有目的條帶出現的PCR產物再進行5%(w/v)聚丙烯酰胺凝膠電泳,篩選具有多態性的引物。
1.3 數據統計與分析
統計每個位點的等位基因數量(A),用 Pop-Gen1.32軟件[7]來計算群體的有效等位基因數(Ae)、觀測雜合度(Ho)和期望雜合度(He)、Hardy-Weinberg平衡偏離指數(D)以及卡方檢驗(PHW)。根據Botstein等[8]的公式計算每個微衛星位點的多態性信息含量(CPI),計算公式如下:
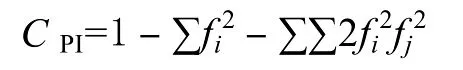
式中fi和fj分別表示某一位點第i個和第j個等位基因在群體中的頻率。
2 結果與分析
2.1 微衛星DNA的分離篩選
用內切酶MseI對基因組 DNA進行酶切后用1.5%瓊脂糖凝膠電泳檢測,發現酶切片段主要集中在200~1 000 bp,符合構建基因組文庫的要求。通過探針雜交,磁珠吸附,清洗和洗脫,PCR擴增,載體連接和轉化等步驟,構建了大珠母貝基因組微衛星富集文庫,共獲得近 2 000個陽性克隆,隨機挑選1 200個克隆進行PCR檢測。選取具有穩定擴增帶的陽性克隆298個進行測序,分析測序結果獲得了211個含有重復次數大于或等于6的微衛星序列。211個序列共含有 251個微衛星位點,一般每個克隆含有1~3個位點,其中1個克隆含有6個位點,微衛星序列長度大小范圍為 101~787 bp。對所得微衛星位點進行分類,發現(CA/GT)n微衛星重復序列204個,占了絕大多數,此外還發現了AG、AT、GC、TC、CAA、AGG、GATA、GACA、GTGC、GACG等重復類型。根據Weber[9]提出的微衛星序列分類標準,在所獲得的微衛星位點中,完美型159個,占63%; 非完美型80個,占32%; 復合型12個,占5%。
2.2 微衛星引物的篩選
根據包含微衛星位點的序列,用引物設計軟件primer 5.0設計引物,共設計引物90對,隨機挑選了其中30對送去上海生物工程有限公司合成。通過優化PCR反應條件,篩選出21對能夠穩定擴增的引物。將有目的條帶出現的PCR產物再進行5%(w/v)聚丙烯酰胺凝膠電泳,篩選出10對多態性引物(表1)。

表1 10對多態性微衛星引物序列Tab.1 Sequences of ten pairs of polymorphic microsatellite primers

表2 10個微衛星多態性位點的數據統計Tab.2 The statistics of ten microsatellite loci
2.3 微衛星位點多態性分析
使用PopGen1.32軟件對所得的微衛星多態性位點進行數據分析,由表2可知,10個位點共獲得59個等位基因,其中M412位點檢測出的等位基因最多(8個),M393位點的等位基因最少(2個),大部分位點為5~7個,大小在133~444 bp之間,表現出高度多態性。有效等位基因數(Ae)為1.342 3~6.000 0,平均為4.124 0; 觀測雜合度(Ho)的范圍為 0.300 0~0.800 0,平均為 0.533 3; 期望雜合度(He)范圍為 0.259 3~0.847 5,平均為 0.717 9; 多態性信息含量(CPI)在0.222 5~0.811 8之間,平均為0.671 4。根據雜合度計算的遺傳偏離指數(D),發現除了M393和M781位點為正值外,其余各位點均表現為不同程度的雜合子缺失。根據 Hardy-Weinberg平衡的卡方檢驗(PHW),只有 M393、M781和 M1758位點處于平衡狀態(PHW>0.05),其他位點都不同程度地偏離了平衡。
3 討論
磁珠富集法是一種簡單快速的篩選微衛星的方法,通過帶有鏈霉親和素的磁珠親和捕捉生物素標記的微衛星探針結合的DNA基因組片段,從而獲得高度富集微衛星小插入片段的基因組文庫,且獲得的微衛星重復序列的比例比小克隆的比例高[10],目前國內外較多采用這種方法[11,12]。本實驗在所測298個克隆中有 211個含有微衛星序列,陽性克隆率高達70.81%。姬長虹等[13]的報道為95.3%,李小寧等[14]為 88.5%,說明此法是一種高效快速的微衛星標記分離方法。但貝類多態位點的篩選效率相對較低,需要設計大量的引物才能獲得為數不多的多態標記[15],本實驗合成的 30對引物中僅有10對擴增出具有多態性的目的條帶,曲妮妮等[16]在篩選合浦珠母貝微衛星標記的研究中49對引物只有9對具有多態性。出現這類情況的原因可能與貝類個體之間的遺傳變異程度較大有關。Arias 等[17]對扇貝的 SNPs檢測,發現大約 100 個堿基就出現 1 次 SNP。Sauvage等[18]對太平洋牡蠣(Crassostrea gigas) 的研究,發現在編碼區平均每60 bp 出現1 次SNPs,在非編碼區每40 bp 就出現1 次SNPs。這些高頻率的點突變為微衛星引物的擴增篩選帶來了很大困難。
在動物基因組中微衛星大約 6~10 kb就出現一次[19],其中二堿基重復類型(CA/GT,AG/TC,AT/TA)的微衛星最為常見[20,21]。本實驗采用了其中的(CA)15探針,在所得的 251個微衛星位點中,含(CA/GT)n的有 204個,約占總數的 81%,其次是(AG/TC)n和(AT)n,各有24和6個,分別占總數的10%和2%。結果表明在大珠母貝中除了有大量的雙堿基序列外,還有其他三堿基、四堿基和四堿基以上的微衛星序列,因此可以考慮采用其他類型的探針研究開發大珠母貝基因組微衛星,以便獲得更多的微衛星分子標記。
微衛星核心序列突變率相對較高,造成了微衛星核心序列重復次數的變化,這是微衛星多態性的基礎[22]。Ellegren[23]認為真核生物中微衛星重復堿基重復序列長度大多在30次重復以下,本實驗所得序列重復次數在8~25的有58%,與上述分析微衛星的結論相似。關于微衛星重復數與多態性的關系,多數學者認為微衛星重復次數與多態性之間存在正相關,Valdes[24]認為重復次數低于 5的微衛星幾乎檢測不出多態性,一般微衛星的核心序列重復次數越高,其等位基因數也就越多,即多態性也就越高[25]。但較長的(AT)n容易形成二級結構而造成PCR效率下降,以及在電泳分離中造成較大誤差,并會給測序工作帶來一些麻煩[26]。本研究所得的大珠母貝微衛星序列中,除去(AT)n重復單元以外,重復次數在 5次以上的約占94%。因此理論上講,設計的引物可以進行群體的遺傳多樣性研究
雜合度是度量群體變異的一個重要參數[27],包括觀測雜合度和期望雜合度。本實驗所獲得的10個微衛星位點有 8個位點的觀測雜合度低于期望雜合度,其范圍相應地低于Smith等[3]報道的Ho(0.479~0.891)和He(0.872~0.972),與 Evans 等[2]報 道 的Ho(0.172~0.813)和He(0.163~0.878)比較接近,與谷龍春等[28]報道的Ho(0.167~0.833)相近,而略低于其He(0.658~0.915)。CPI值是等位基因頻率和數目變化的函數,是衡量片段多態性的較好指標,能反映出某個遺傳標記所含的遺傳信息容量。根據 Bostein等[8]提出的衡量基因變異程度高低的多態信息含量指標,當CPI>0.5 時,該基因座為高度多態基因座;當 0.25 <CPI< 0.5 時,為中度多態基因座; 當CPI<0.25 時,則為低度多態基因座。試驗中開發的10個微衛星基因座除M393為低度多態性外,其他9個均為高度多態性,表明這些微衛星位點適合大珠母貝的遺傳多樣性研究。結果為進一步開展大珠母貝的遺傳多樣性、遺傳連鎖圖譜構建和遺傳選育等研究工作奠定了一定基礎。
[1]姜因萍,何毛賢.大珠母貝的研究概況[J].海洋科學,2009,33(2):92-96.
[2]Evans B S,Knauer J,Taylor J U,et al.Development and characterization of six new microsatellite markers for the silver-or gold-lipped pear oyster,Pinctadamaxima(Pteriidae) [J].Molecular Ecology Notes,2006,6(3):835-837.
[3]Smith C,Benzie J A H,Wilson K J.Isolation and characterization of eight microsatellite loci from silver-lipped pearl oysterPinctada maxima[J].Molecular Ecology Notes,2003,3 (1):125-127.
[4]Sambrook J,Fritsch E F,Maniatis T.Molecular cloning:A Labortory Manual[M].NewYork:Cold Spring Harbour Press,1989.463-468.
[5]Zane L,Bargelloni L,Patarnello T.Strategies for microsatellite isolation:a review[J].Molecular Ecology,2002,11:1-16.
[6]Jeffs P.Multiple sequence alignment with Clustal X[J].Computer Corner,1998,23(1):78-80.
[7]Yeh F C,Boyle T J B.Population genetic analysis of Co-dominant and dominant markers and quantitative traits [J].Belgian Journal of Botany,1997,129:157.
[8]Botstein D,White R L,Skolnick M,et al.Constructio of a genetic linkage map in man using restriction fragment length polymorphisms[J].American Journal of Human Genetics,1980,32(3):314-331.
[9]Weber J L.Informativeness of human (dC-dA)n(dG-dT)npolymorphisms[J].Genomics,1990,7(3):524-530.
[10]孫效文,賈智英,魏東旺,等.磁珠富集法與小片段克隆法篩選鯉微衛星的比較研究[J].中國水產科學,2005,12(2):126-132.
[11]Li Q,Kijima A.Identification of novel microsatellite loci in the Pacific oyster (Crassastrea gigas) by magnetic bead hybridization selection[J].Tohoku Journal of Aqricultural Research,2002,53 (1-2):25-32
[12]孫效文,魯翠云,梁利群.磁珠富集法分離草魚微衛星分子標記[J].水產學報,2005,29(4):482-486.
[13]姬長虹,孫效文.用磁珠富集法快速制備銀鯽微衛星標記[J].大連水產學報,2007,22(6):460-464.
[14]李小寧,張殿昌,江世貴,等.合浦珠母貝微衛星DNA 標記分離與分析[J].福建水產,2009,13(5):48-54.
[15]Herbinger C M,Smith C A,Langy S.Development and characterization of novel tetra- and dinucleotide microsatellite markers for the French Polynesia black-lipped pearl oyster,Pinctada margaritifera[J].Molecular Ecology Notes,2006,6(1):107-109.
[16]曲妮妮,龔世圓,黃桂菊,等.基于 FIASCO 技術的合浦珠母貝微衛星標記分離與篩選研究[J].熱帶海洋學報,2010,29(3):1-8.
[17]Arias A,Freire R,Boudry P,et al.Single nucleotide polymorphism for population studies in the scallopsAequipecten opercularisandMimachlamys varia[J].Conserv Genet,2008,56:1000-1007.
[18]Sauvage C,Bieme N,Lapegue S,et al.Single Nucleotide polymorphisms and their relationship to codon usage bias in the Pacific oysterCrassostrea gigas[J].Gene,2007,406(1-2):13-22.
[19]Wang Y,Guo X.Chromosomal rearrangement in Pectinidae revealed by rRNA loci and implications for bivalve evolution[J].Biological Bulletin,2004,207(3):247-256.
[20]Chistiako D A,Helleman B,Volckaert F A M.Microsatellites and their genomic distribution,evolution,function and applications:A review with special reference to fish genetics[J].Aquaculture,2006,255(1-4):1-29.
[21]Brenner S,Elgar G,Sandford R,et al.Characterization of the pufferfish (Fugu) genome as a compact model vertebrate genome [J].Nature,1993,366:265-268.
[22]劉志毅,相建海.微衛星 DNA分子標記在海洋動物遺傳分子中的應用[J].海洋科學,2001,25(6):11-13.
[23]Ellegren H.Microsatellite evolution:a battle between replication slippage and point mutation[J].Trends in Genetics,2002,18:70.
[24]Valdes A M.Allele frequencies at microsatellite loci:the stepwise mutation model revisited[J].Genetics,1993,133(3):737-749.
[25]Ma Z Q,Roder M,Sorrells.Frequencies and sequence characteristics of di,tri-,and tetra-nucleotide microsatellite in wheat[J].Genome,1996,39(1):123-130.
[26]林能鋒,蘇永全,丁少雄,等.赤點石斑魚微衛星DNA篩選[J].福建農業學報,2007,22(2):115-119.
[27]陳微,張全啟,于海洋,等.牙鲆微衛星標記的篩選及群體多態性分析[J].中國水產科學,2005,12(6):682-687.
[28]谷龍春,黃桂菊,喻達輝,等.大珠母貝兩個野生群體遺傳多樣性的微衛星分析[J].漁業科學進展,2009,30(4):98-101.

