山茶花SEPALLATA(SEP)家族基因克隆及表達分析




摘要:花型特征是山茶花重要的觀賞性狀之一,為了揭示山茶花SEP基因結構特征及其與山茶花重瓣花型的關系,采用同源克隆結合RACE方法進行山茶花SEP基因克隆,對其進行生物信息學分析,并采用實時熒光定量PCR檢測SEP基因在不同花型山茶花品種中的表達特征。結果顯示:從山茶花胭脂牡丹中克隆得到3個SEP基因CjSEP1a(1 235 bp)、CjSEP3(967 bp)、CjSEP4a(1 725 bp),GenBank登錄號分別為OP172861、OP172863、OP172864,分別編碼249、185和243個氨基酸。山茶花3個SEP基因的編碼蛋白均呈不穩定、親水、細胞核定位特征,具有MADS-box和K-box保守結構域,與同屬物種茶樹的SEP蛋白具有高度同源性。qPCR分析表明,完全重瓣型品種胭脂牡丹3個SEP基因的表達量顯著高于其他品種,其次為半重瓣品種和單瓣型品種,3個特異重瓣花型品種的SEP表達量總體較低,因此,隨著花型規則重瓣程度的加深,SEP基因的表達量呈上調表達。CjSEP基因與山茶花規則重瓣性狀存在緊密關聯性,不同CjSEP成員具有協同表達作用。
關鍵詞:山茶花;重瓣;E類基因;SEPALLATA(SEP);RACE克隆
中圖分類號:S685.14文獻標志碼:A文章編號:0253?2301(2024)12?0028?09
DOI:10.13651/j.cnki.fjnykj.2024.12.006
Cloning and Expression Analysis of SEPALLATA(SEP)Gene Family in Camellia japonica LAI Gong-ti1,PAN Ruo1,HE Li-yuan1,LIN Jun-xuan1,GUO Ao-lin1,CHE Jian-mei2,LAI Cheng-chun1*
(1.Institute of Food Science and Techology,Fujian Academy of Agricultural Sciences,Fuzhou,Fujian 350003,China;2.Institute of Resources,Environment and Soil Fertilizer,Fujian Academy ofAgriculturalSciences,Fuzhou,Fujian 350003,China)
Abstract:The traits of flower types are one of the significant ornamental features of Camellia japonica.In order toreveal the structural characteristics of the SEP genes in Camellia japonica and its relationship with the double-petalflowertype,the homologous cloning combined with RACE method was used to clone the SEP genes of Camelliajaponica,and their bioinformatics analysis was performed.Then,the expression characteristics of SEP genes indifferent flower types of Camellia japonica were detected by the real-time fluorescence quantitative PCR.Theresultsshowed that three SEP genes were cloned from Camellia japonica Yanzhimudan,including CjSEP1a(1 235 bp),CjSEP3(967 bp),and CjSEP4a(1 725 bp).The GenBank accession numbers were OP172861,OP172863 andOP172864,respectively.These genes encoded the proteins consisting of 249,185 and 243 amino acids,respectively.The proteins encoded by the three SEP genes of Camellia japonica were unstable,hydrophilic and localized in thenucleus,featuring conserved domains of MADS-box and K-box,and had high homology with the SEP genes of teaplants of congeneric species.The qPCR analysis indicated that the expression levels of the three SEP genes in the fullydouble-petal cultivar Yanzhimudan were significantly higher than those in other cultivars,followed by the semi-double-petal cultivar and the single-petal cultivar.The expression levels of SEP genes in three other specific double-petal cultivars were generally lower.Therefore,as the prominence of double-petal phenotype increased,the expression of SEP genes was up-regulated.A strong relationship existed between CjSEP genes and the characteristic double-petal trait in Camellia japonica,with different CjSEP genes exhibiting synergistic effects in their expression.
Key words:Camellia japonica;Double-petal;E-type gene;SEPALLATA(SEP);RACE cloning
花型是花器官發育的形態,是觀賞植物的重要性狀之一,花器官發育調控模型由“ABC”[1?2]經“ABCD”[3?5]逐漸發展形成經典的“ABCDE”模型[6],該模型指出,花器官結構的萼片形成、花瓣發育、雄蕊發生、心皮發育和胚珠發育分別由A/E、A/B/E、B/C/E、C/E和C/D/E類基因參與調控。“ABCDE”模型顯示了植物花器官發育過程E類基因的不可或缺性,E類基因SEPALLATA(SEP)屬于MADS-box基因家族,能夠與其他類型基因形成蛋白四聚體,通過調控下游靶基因的表達進而參與花器官形成和花分生組織的發育[7?9],E類基因SEP參與了花器官發育的多個進程,是花器官發育的關鍵基因。除了E類基因,其他四類基因分別為:A類的APETALA1(AP1)和APETALA2(AP2);B類的APETALA3(AP3)和PISTILLATA(PI);C類的AGAMOUS(AG);D類的FLORAL BINDING PROTEIN(FBP7/11)、SEEDSTICK(STK)和SHATTERPROOF(SHP1/2)[7]。目前,已經從擬南芥、非洲菊、桃等植物中成功克隆獲得SEP基因[10?13],研究表明E類基因中SEP1/2參與心皮發育調控,SEP3參與調控花瓣和雄蕊的形成與發育,SEP4協同A型基因調控花萼生長發育[10]。SEP基因的沉默能夠導致“花內花”結構的產生,花分生組織確定性喪失和花器官的同源異型轉化[14],阻礙SEP3與AG蛋白二聚體的形成,也將導致雌雄蕊向花被轉化,形成重瓣花[15]。因此,重瓣特征結構等花型決定與SEP基因的調控具有緊密的聯系。
山茶花Camellia japonica L.為我國傳統十大名花之一,常用于室內觀賞、園林造景等,同時,山茶花多酚和黃酮類物質具有保健和藥理功能,有較高的經濟價值。山茶花具有多樣的花型和豐富的顏色,山茶花不同品種特異花型花瓣特征,為花器官形成調控研究奠定了良好的材料基礎。本研究通過同源克隆和cDNA末端快速擴增(rapid amplifi-cation of cDNA ends,RACE)技術,對山茶花的SEP家族基因進行克隆,并進行SEP基因編碼蛋白的基本理化特性、磷酸化修飾、跨膜結構、信號肽、亞細胞定位、保守結構域、二級、三級結構預測分析,構建SEP同源蛋白的系統進化樹,進一步分析SEP基因在不同花型山茶花品種花瓣中的表達特征。研究豐富了植物SEP家族基因成員的數量,填補了山茶花SEP全長基因無從獲取的空白,為山茶花SEP家族基因調控花器官發育的研究提供理論支撐。
1材料與方法
1.1試驗材料
山茶花不同花型品種美人茶(單瓣型)、芙蓉香波(半重瓣型)、胭脂牡丹(完全重瓣型)、緋爪芙蓉(牡丹型)、迷茫的春天(玫瑰型)和獅子笑(托桂型)保存于福建省農業科學院農產品加工研究所山茶花種質資源圃。采集盛花期無病蟲害、無損傷的山茶花花瓣用于后續試驗,SEP家族基因克隆采用胭脂牡丹為材料,上述6個不同花型代表品種均進行RNA提取和cDNA合成,用于熒光定量PCR試驗分析。
1.2試驗方法
1.2.1山茶花RNA提取采集6個不同花型的山茶花品種(美人茶、芙蓉香波、胭脂牡丹、緋爪芙蓉、迷茫的春天和獅子笑)盛花期花瓣,使用天根RNAprep Pure多糖多酚植物總RNA提取試劑盒,進行RNA提取,采用瓊脂糖凝膠電泳檢驗其完整性,并通過BMG CLARIOstar多功能酶標儀對RNA進行濃度和純度檢測。
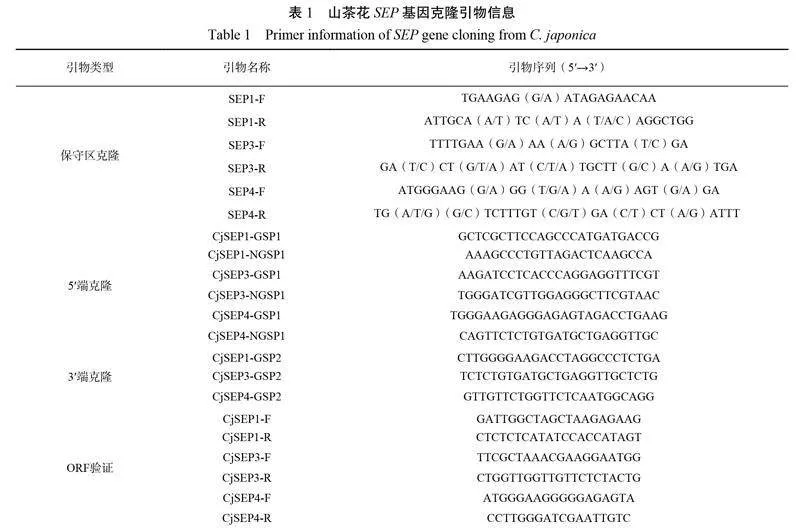
1.2.2山茶花SEP基因保守區克隆以山茶花胭脂牡丹RNA為模板,采用TRAN EasyScript One-Step cDNA Removal and cDNA Synthesis SuperMix試劑盒進行cDNA合成。從NCBI數據庫下載不同物種SEP同源基因序列,使用DNAMAN 6.0對比序列得到保守區序列,根據保守區設計簡并引物,引物序列見表1。將目的條帶進行純化回收,利用天根pLB零背景快速克隆試劑盒(VT205)進行連接轉化,挑選陽性大腸桿菌菌落,PCR鑒定后液體振蕩培養,菌液委托北京擎科生物進行測序。
1.2.3山茶花SEP基因5′端和3′端序列克隆根據SMARTer?RACE 5′/3′Kit操作說明書分別進行5′端和3′端cDNA第一鏈合成,以試劑盒提供的通用引物(Universal Primer Mix,UPM)為基礎,根據保守區序列設計配對引物GSP與NGSP,引物長度為23~28 nt為宜,GC含量為50%~70%,引物信息見表1,5′端序列克隆為巢式PCR進行兩輪反應,3′端序列克隆進行一輪PCR反應,具體步驟按照說明書進行。
1.2.4山茶花SEP基因ORF驗證與序列分析根據5′端、保守區與3′端得到的序列在DNAMAN 6.0中進行拼接,利用其他物種同源基因信息和NCBI-ORFfinder分析開放閱讀框,并設計ORF驗證引物,引物信息見表1。
1.2.5山茶花SEP基因編碼蛋白生物信息學分析利用ExPASy-ProtParam在線軟件進行蛋白質基本理化特性分析;ProtScale軟件分析SEP蛋白質的親水性;利用NetPhosv3.1對SEP蛋白進行磷酸化位點預測分析;通過TMHMM 2.0預測該蛋白跨膜區域;使用SignalP-5.0進行信號肽預測;通過CELLO2GO預測亞細胞定位;使用NCBI-Con-served Domains Search分析SEP蛋白序列的保守結構域;利用SOPMA數據庫進行SEP蛋白二級結構預測;利用SWISS-MODEL構建三個蛋白的三級結構模型。
1.2.6山茶花SEP蛋白系統進化樹構建采用NCBI-BLAST對SEP蛋白進行比對分析,下載30條同源蛋白序列,使用MEGA6.0軟件(Neighbor-joining)1 000次Boot-strap計算構建系統進化樹。
1.2.7山茶花SEP基因實時熒光定量PCR分析
根據驗證的山茶花SEP基因序列設計特異RT-qPCR引物,產物長度為80~200 nt,引物序列信息見表2。cDNA合成和熒光定量PCR采用寶生物工程有限公司提供的PrimeScriptTM RT reagent Kit和TB Green?Premix Ex TaqTM試驗于Roche LightCycler 480中進行,反應結束后確認qPCR的擴增曲線及溶解曲線,并獲取CT值。
1.2.8數據分析數據分析和作圖采用Excel和Origin8.5進行,熒光定量PCR結果采用2-ΔΔCT法[16]對數據進行表達量計算,GAPDH為內參基因。
2結果與分析
2.1山茶花RNA提取
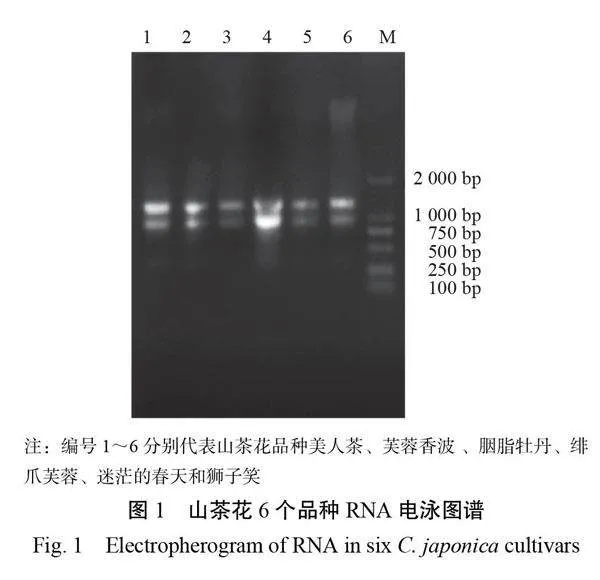
山茶花6個品種花瓣提取的總RNA電泳圖譜見圖1,結果顯示6個RNA樣品均具有明亮、清晰、完整的28S和18S的經典帶型,RNA濃度200~500 ng·μL?1,OD260/OD280約為2.0,具有較高的濃度和純度。因此,所提取的6個山茶花品種RNA質量好,具有較高的完整性、濃度和純度。
2.2山茶花SEP家族基因保守區克隆
以山茶花品種胭脂牡丹RNA合成獲得cDNA,使用簡并引物進行RT-PCR保守區序列擴增,電泳結果見圖2,經測序得到SEP1、SEP3、SEP4保守區序列,去除冗余序列得到的保守區長度分別為599、325和419 bp,并在NCBI網站做核酸序列同源性比對,結果顯示3個基因保守區片段與其他物種SEP基因高度同源。
2.3山茶花SEP基因5′端和3′端序列RACE克隆
以胭脂牡丹總RNA合成5′端和3′端cDNA第一鏈,以此為模板,通過5′/3′RACE擴增得到山茶花SEP1、SEP3、SEP4基因的5′端及3′端序列(圖3),克隆測序后去除冗余序列,5′端長度分別為207、114和716 bp,3′端長度分別為435、528和581 bp。利用DNAMAN6.0軟件進行SEPs的5′端、保守區和3′端序列拼接,并在NCBI數據庫進行同源基因比對驗證。
2.4山茶花SEP基因開放閱讀框(ORF)克隆驗證與序列分析
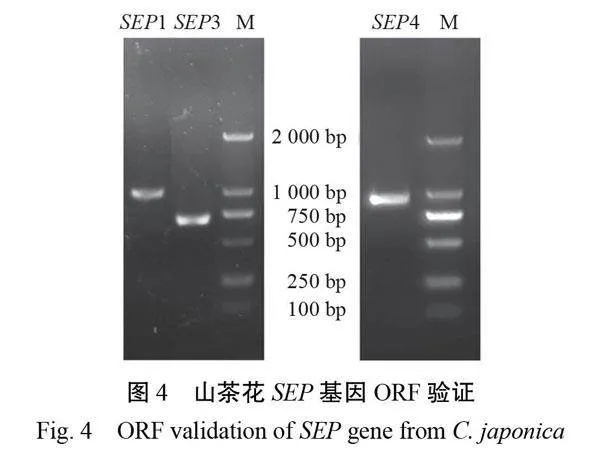
以拼接獲得的SEP基因序列為模板,進行ORF-PCR擴增驗證(圖4),結果與預期相符,因此,成功從山茶花中克隆獲得3個SEP基因CjSEP1a(1 235 bp)、CjSEP3(967 bp)和CjSEP4a(1 725 bp),NCBI登錄號分別為:OP172861、OP172863、OP172864。CjSEP1a全長1 235 bp,ORF長度為750 bp,編碼249個氨基酸;CjSEP3全長967 bp,ORF長度為558 bp,編碼185個氨基酸;CjSEP4a全長為1 725 bp,ORF長度為732 bp,共編碼243個氨基酸。
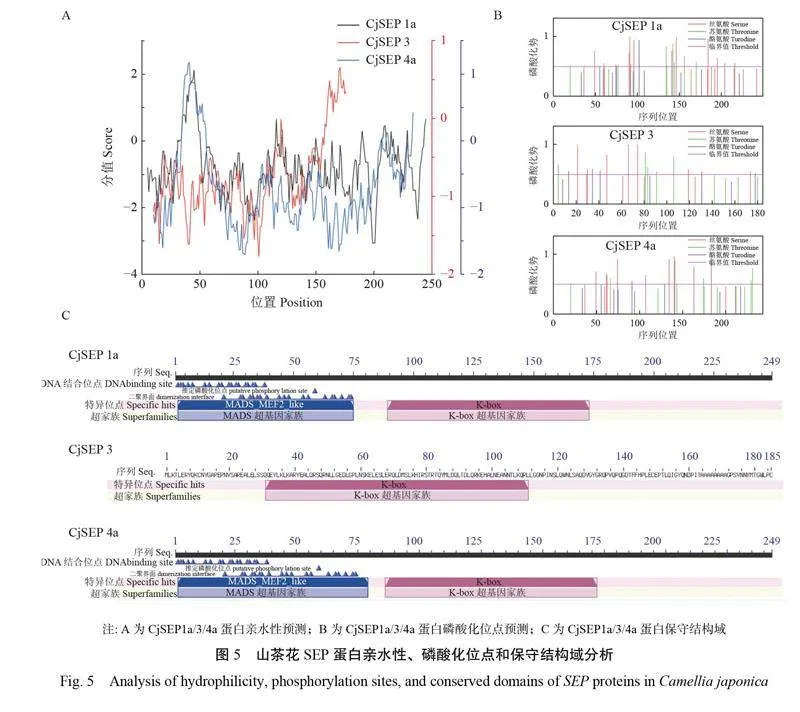
2.5山茶花SEP基因編碼蛋白基本理化特性及生物信息學特征
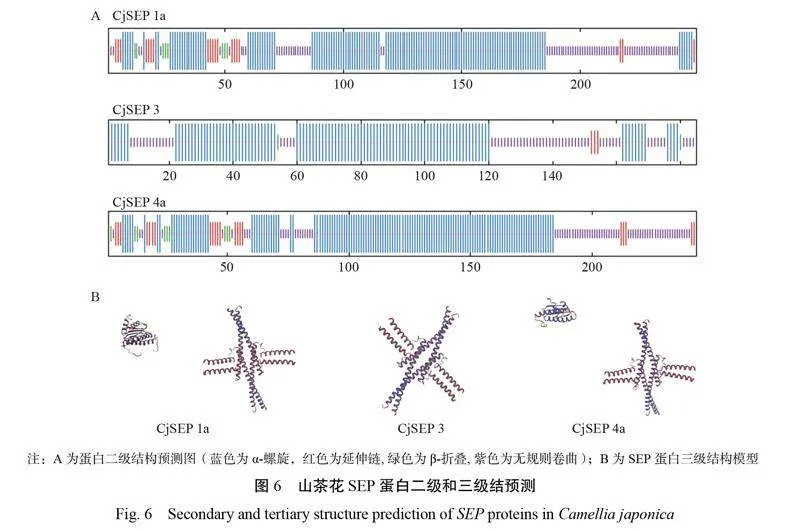
山茶花3個SEP蛋白相對分子量20~30 kD,其不穩定系數均大于40,平均親水性系數小于?0.6(圖5A),是不穩定的親水性蛋白。磷酸化預測顯示其修飾主要集中在絲氨酸、蘇氨酸和酪氨酸位點上(圖5B)。進一步預測分析顯示3個SEP基因編碼蛋白均不具有跨膜螺旋結構和信號肽,亞細胞定位預測顯示為細胞核定位蛋白。3個SEP蛋白均具有MADS-box中典型的MIKC結構域(圖5C),CjSEP1a和CjSEP4a含有特異的MADS-MEF2位點,3個蛋白均包含K-box特異位點。
根據SOPMA軟件對SEP蛋白二級結構預測結果如圖6A,二級結構主要由α-螺旋、延伸鏈、β-折疊和無規則卷曲4個結構組成,3個蛋白質的α-螺旋含量最高,CjSEP1a、CjSEP3和CjSEP4a的α-螺旋比例達到55.28%、61.08%和56.38%。SEP蛋白三級結構見圖6B,3個蛋白均呈現出十字形三維結構特征,此外,SEP1a和SEP4a具有額外的特異構型。
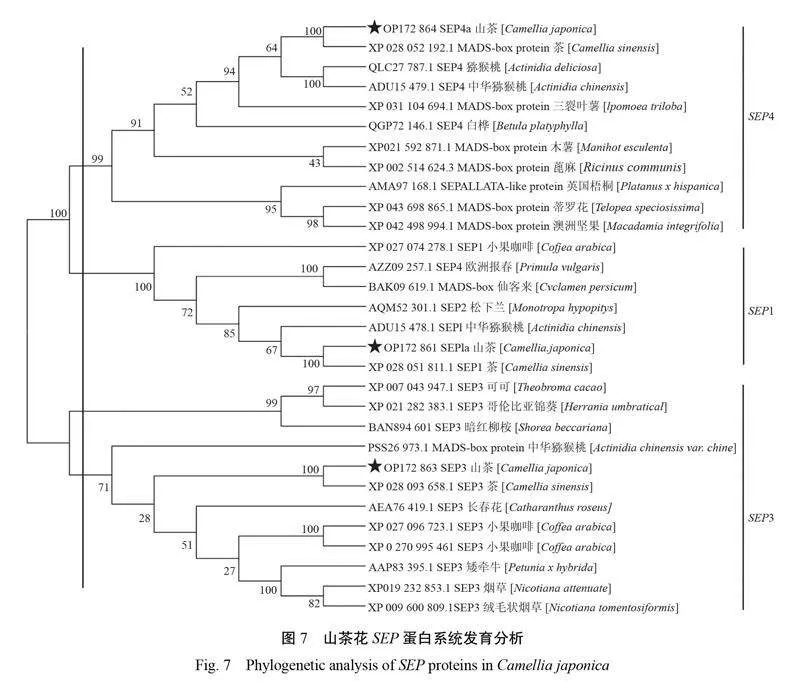
2.6山茶花SEP蛋白系統進化分析
由圖7可知,30條SEP蛋白聚類為4個分支,并劃分成3個組別,這3個分組總體分別對應SEP1、SEP3和SEP4蛋白成員類群。CjSEP1a、CjSEP3和CjSEP4a正確聚類到相應的蛋白成員類群中,山茶花SEP蛋白與其他木本植物蛋白或MADS-box特征蛋白具有較高同源性,尤其是與同屬的茶樹高度同源,而與草本植物的同源性較低,如煙草、矮牽牛等。
2.7 SEP基因在不同花型山茶花品種中的相對表達分析
利用實時熒光定量PCR對6個不同花型的山茶花品種SEP基因轉錄水平進行分析(圖8),除了緋爪芙蓉CjSEP4a的表達量顯著高于美人茶和芙蓉香波,山茶花3個SEP基因在不同花型中具有相似的表達模式,表達量由高到低的總體順序依次為胭脂牡丹、芙蓉香波、美人茶、緋爪芙蓉、迷茫的春天和獅子笑。完全重瓣型品種胭脂牡丹3個SEP基因的表達量顯著高于其他品種,其次為半重瓣品種芙蓉香波和單瓣型品種美人茶,3個特異花型(緋爪芙蓉、迷茫的春天和獅子笑)SEP表達量總體較低,因此,隨著花型規則重瓣程度的加深,SEP基因的表達量呈上調表達特征。同一花型的3個SEP基因表達量具有協同效應,不同SEP成員的表達模式總體呈現CjSEP4a>CjSEP3>CjSEP1a特征。綜上所述,CjSEP基因與山茶花規則重瓣性狀存在緊密關聯性,不同CjSEP成員具有協同表達作用。
3討論與結論
花器官發育并不是由單個或多個基因簡單控制,而是由“ABCDE”模型基因編碼的蛋白質通過特異性結合構成四聚體,進而控制下游靶基因的表達后最終達到調控花器官發育的目的[1,17?18]。在植物體花器官的花徑大小、花瓣形狀與重瓣等性狀的調控途徑中,早期的研究認為ABC類基因占主導地位,但僅憑ABC類基因無法單獨調控花器官性狀的形成,并且難以解釋營養器官和生殖器官的轉變機制,因此,E類基因在其中扮演不可或缺的角色[19]。E類基因參與了花萼、花瓣、雄蕊、心皮和胚珠全部5輪花器官發育調控,E類基因SEP包含4個成員,分別為SEP1、SEP2、SEP3和SEP4,它們參與調節植物的生殖生長尤其是花器官的發育,參與植物成花全過程[20]。花型相關基因在模式植物擬南芥中得到了深入開展[21],在山茶花中也已經被證實參與花器官形成和發育調控的“ABC”模型[22?23],但鮮有在山茶花中關于E類基因的研究報道。
本研究從山茶花中成功克隆獲得3個E類SEP基因CjSEP1a、CjSEP3和CjSEP4a,該基因編碼的蛋白呈不穩定、親水性、細胞核定位特征,具有MADS-box和K-box保守結構域,與同屬物種茶樹的SEP蛋白具有高度同源性。MADS-box結構基因與花器官形成調控和果實發育有關[24],不同物種研究也表明SEP基因參與多輪花器官發育調控[25?28]。山茶花CjSEPs在重瓣品種胭脂牡丹花瓣中表達量顯著高于其他花型,在半重瓣品種中的表達也高于單瓣型品種,完全重瓣存在花萼瓣化、雄蕊和雌蕊的退化,這也與E類基因通過蛋白四聚體調控萼片、花瓣及萼片瓣化的研究結果相符[29]。綜上,CjSEP基因與山茶花規則重瓣性狀存在緊密關聯性,不同CjSEP成員具有協同表達作用,CjSEP家族基因的克隆和表達分析為山茶花E類基因的花器官發育調控研究奠定了一定理論基礎。
參考文獻:
[1]BOWMAN J L,SMYTH D R,MEYEROWITZ E M.Genetic interactions among floral homeotic genes of Arabidopsis[J].Development,1991,112:1?20.
[2]COEN E S,MEYEROWITZ E M.The war of the whorls:geneticinteractions controlling flower development[J].Nature,1991,353:31?37.
[3]ANGENENT G C,FRANKEN J,BUSSCHER M,et al.A novel class of MADS box genes is involved in ovule development in petunia[J].The Plant Cell,1995,7(10):1569?1582.
[4]PINYOPICH A,DITTA G S,SAVIDGE B,et al.Assessing the redundancy of MADS-box genes during carpel and ovule development[J].Nature,2003,424(6944):85?8.
[5]YANOFSKY,M F.Floral meristems to floral organs:genes controlling early events in Arabidopsis flower development[J].Annual Review of Plant Physiology and Plant Molecular Biology,1995,46:167?188.
[6]THEI?ENG.Development of floral organ identity:stories from the MADS house[J].Current Opinion in Plant Biology,2001,4(1):75?85.
[7]相元萍,黃云彤,賀洪軍,等.羽衣甘藍SEPALLATA-like基因的系統發育與表達分析[J].生物工程學報,2020,36(11):2398?2412.
[8]PELAZ S,DITTA G S,BAUMANN E,et al.B and C floral organ identity functions require SEPALLATA MADS-box genes[J].Nature,2000,405(6783):200?203.
[9]DITTA G,PINYOPICH A,ROBLES P,et al.The SEP4 gene of Arabidopsis thaliana functions in floral organ and meristem identity[J].Current Biology,2004,14(21):1935?1940.
[10]王瑩,穆艷霞,王錦.MADS-box基因家族調控植物花器官發育研究進展[J].浙江農業學報,2021,33(6):1149?1158.
[11]SHI Q,ZHOU J,WANG P,et al.Protein expression and characterization of SEP3 from Arabidopsis thaliana[J].Genetics and Molecular Research,2015,14(4):12529?12536.
[12]ZHANG T,ZHAO Y,JUNTHEIKKI N,et al.Dissecting functions of SEPALLATA-like MADS box genes in patterning of the pseudanthial inflorescence of Gerbera hybrida[J].New Phytologist,2017,216(3):939?954.
[13]LI J,LI F,QIAN M,et al.Characteristics and regulatory pathway of the PrupeSEP1 SEPALLATA gene during ripening and softening in peach fruits[J].Plant Science,2017,257:63?73.
[14]WANG P,LIAO H,ZHANG W,et al.Flexibility in the structure of spiral flowers and its underlying mechanisms[J].Nature Plants,2015,2(1):1?10.
[15]GALIMBA K D,TOLKIN T R,SULLIVAN A M,et al.Loss of deeply conserved C-class floral homeotic gene function and C-and E-class protein interaction in a double-flowered ranunculid mutant[J].Proceedings of the National Academy of Sciences,2012,109(34):E2267?E2275.
[16]KENNETH J L,THOMAS D.Schmittgen analysis of relative gene expression data using real-time quantitative PCR and the 2?ΔΔCT method[J].Methods,2001,25(4):402?408.
[17]ZAHN L,KONG H,LEEBENS-MACK J,et al.The evolution of the SEPALLATA subfamily of MADS-Box genes:A preangiospermorigin with multiple duplications throughout angiosperm history[J].Genetics,2005,169(4):2209?2223.
[18]LESEBERG C H,EISSLER C L,WANG X,et al.Interaction study of MADS-domain proteins in tomato[J].Journal of Experimental Botany,2008,59(8):2253?2265.
[19]陳艷.我國觀賞植物花型基因研究進展[J].安徽農學通報,2018,24(6):43?45.
[20]姚曉琳,張曉芳,王錚,等.大豆MADS-box基因功能研究進展[J].植物遺傳資源學報,2024,25(6):909?918.
[21]GRANDI V,GREGIS V,KATER M M.Uncovering genetic and molecular interactions among floral meristem identity genes in Arabidopsis thaliana[J].The Plant Journal,2012,69(5):881?893.
[22]SUN Y,FAN Z,Li X,et al.Distinct double flower varieties in Camellia japonica exhibit both expansion and contraction of C-class gene expression[J].BMC Plant Biology,2014,14:288.
[23]SUN Y,FAN Z,LI X,et al.The APETALA1 and FRUITFUL homologs in Camellia japonica and their roles in double flower domestication[J].Molecular Breeding,2014,33:821?834.
[24]LYU J.Tomato development:MADS-box engineering[J].Nature Plants,2017,169:1142?1155.
[25]SIMON T M,ELIZABETH A K.SEPALLATA gene diversification:brave new whorls[J].Trends in Plant Science,2005,10(9):427?435.
[26]DAVIES B,EGEA-CORTINES M,DE ANDRADE SILVA E,et al.Multiple interactions amongst floral homeotic MADS box proteins[J].EMBO Journal,1996,15:4330?4343
[27]TZENG T Y,HSIAO C C,CHI P J,et al.Two lily SEPALLATA-Like genes cause different effects on floral formation and floral transition in Arabidopsis[J].Plant Physiology,2003,133(3):1091?1101.
[28]BONHOMME F,SOMMER H,BERNIER G,et al.Characterization of SaMADS D from Sinapis alba suggests a dual function of the gene:in inflorescence development and floral organogenesis[J].Plant Molecular Biology,1997,34:573?582.
[29]THEI?EN G,MELZER R,RIIMPLER F.MADS-domain transcription factors and the floral quartet model of flower development:linking plant development and evolution[J].Development,2016,143(18):3259?3271.
(責任編輯:柯文輝)

