瀕危植物黑牡丹KASP鑒定方法的建立
吳晶 劉曉宇 伏建國 郭靜 楊曉軍 李洋 梁超 李井干


摘要? 為了建立瀕危植物黑牡丹的KASP鑒定方法,選取6種13份巖牡丹屬植物樣品為材料,根據磷酸烯醇式丙酮酸羧化酶(PEPC)基因上的單核苷酸多態性(SNP)位點,設計黑牡丹特異性KASP引物,用于PCR擴增。結果表明,通過PEPC序列的SNP位點所設計的KASP引物可以在黑牡丹與其近似種之間形成明顯的基因分型,能夠將其與同屬近緣種及其他仙人掌科物種準確地區分開來,可用于黑牡丹的真假鑒定及進出境口岸瀕危物種的查驗鑒定。
關鍵詞? KASP;黑牡丹;鑒定方法
中圖分類號? Q943.2? 文獻標識碼? A? 文章編號? 0517-6611(2024)01-0098-03
doi:10.3969/j.issn.0517-6611.2024.01.020
開放科學(資源服務)標識碼(OSID):
Establishment of KASP Identification Method for Endangered Plant of Ariocarpus kotschoubeyanus
WU Jing,LIU Xiao-yu,FU Jian-guo et al
(Animal,Plant and Food Inspection and Quarantine Center of Nanjing Customs,Nanjing,Jiangsu 210019)
Abstract? In order to establish a KASP identification method for the endangered plant Ariocarpus kotschoubeyanus,13 samples of 6 species of Ariocarpus kotschoubeyanus were selected as materials.According to the single nucleotide polymorphism (SNP) site on the phosphoenolpyruvate carboxylase (PEPC) gene,a specific KASP primer was designed for PCR amplification of Ariocarpus kotschoubeyanus.The test results show that,KASP primer designed by SNP site of PEPC sequence can form obvious genotyping between Ariocarpus kotschoubeyanus and its similar species,can accurately distinguish it from its relatives and other cactus species,and can be used for identification of true and false Ariocarpus kotschoubeyanus and identification of endangered species at entry and exit ports.
Key words? KASP;Ariocarpus kotschoubeyanus;Identification method
基金項目? 南京海關科研項目(2022KJ19) 。
作者簡介? 吳晶(1981—),女,云南昆明人,高級農藝師,碩士,從事進境物種、雜草鑒定研究。*通信作者,農藝師,碩士,從事進境物種、雜草鑒定研究。
收稿日期? 2023-01-04
黑牡丹(Ariocarpus kotschoubeyanus)是仙人掌科巖牡丹屬一種多肉植物,形態獨特,自然古樸,形似巖石而又有生命,且花色鮮艷,是多肉植物中的經典種類,不少植物愛好者將其視為收藏級品種[1]。巖牡丹屬植物主要分布于墨西哥、美國,近年來,由于自然氣候的變化、非法采集和貿易、生長緩慢等原因,黑牡丹野生種群數量越來越少,在世界自然保護聯盟(IUCN)瀕危物種紅色名錄中被評為“瀕危”級別,被列入《瀕危野生動植物種國際貿易公約》(ITES)附錄I[2]。物種的瀕危及滅絕有多方面因素,非法過度采集及貿易就是其中之一,海關各口岸每年都會查獲大量非法入境的黑牡丹植株和種子。因此,加強口岸物種資源查驗工作可以在一定程度上打擊瀕危物種的非法貿易,對于黑牡丹的保護具有重要意義。物種的準確鑒定是口岸查驗工作的關鍵,由于黑牡丹與同屬其他種類形態相似,特別是幼株和種子在形態上相似度極高,難以區分,因此需要建立準確、快速的分子鑒定方法,為黑牡丹的鑒定提供技術支撐。
競爭性等位基因特異性PCR(kompetitive allele-specific PCR,KASP)技術是一種基于SNP(單核苷酸多態性single nucleotide polymorphism)的高通量基因分型技術。利用獨特的等位基因特異性PCR原理,采用雙色熒光檢測1個SNP位點的2種基因型,可對基因組DNA樣品中的目標SNP進行精準的雙等位基因分型檢測[3],目前該技術已經應用于輔助育種、品種檢測和物種鑒定等領域。鐘婷婷等[4]對甘藍型油菜抗根腫病和感病植株進行基因分型,基于SNP位點開發的KASP標記可以準確高效地應用于油菜抗根腫病材料的分子標記輔助選擇育種。王富強等[5]利用KASP標記,構建了不同葡萄種類的SNP指紋圖譜,驗證了KASP技術在我國葡萄品種鑒定的可行性。
KASP技術的關鍵是在目標物種及其近似種中找到穩定且特異的SNP位點[6]。磷酸烯醇式丙酮酸羧化酶(phosphoenolpyruvate carboxylase,PEPC)是一種細胞質酶,是C4代謝和景天酸(CAM)代謝植物光合作用的關鍵酶,其主要功能是在Mg2+或Mn2+的輔助下,催化磷酸烯醇式丙酮酸(PEP)和HCO3-生成草酰乙酸(OAA)和無機鹽酸鹽(Pi)的不可逆反應[7]。磷酸烯醇式丙酮酸羧化酶廣泛分布于古菌、細菌、藍藻、綠藻、原生動物和植物中,在動物和真菌中不存在[8]。每個家族可包含1至多個基因成員,它們的序列在不同植物之間存在著一定的保守性[9]。筆者基于巖牡丹屬PEPC序列,將KASP基因分型技術成功運用于巖牡丹屬物種鑒定中,建立了黑牡丹與同屬近似種類的分子鑒定方法。
1? 材料與方法
1.1? 試驗材料
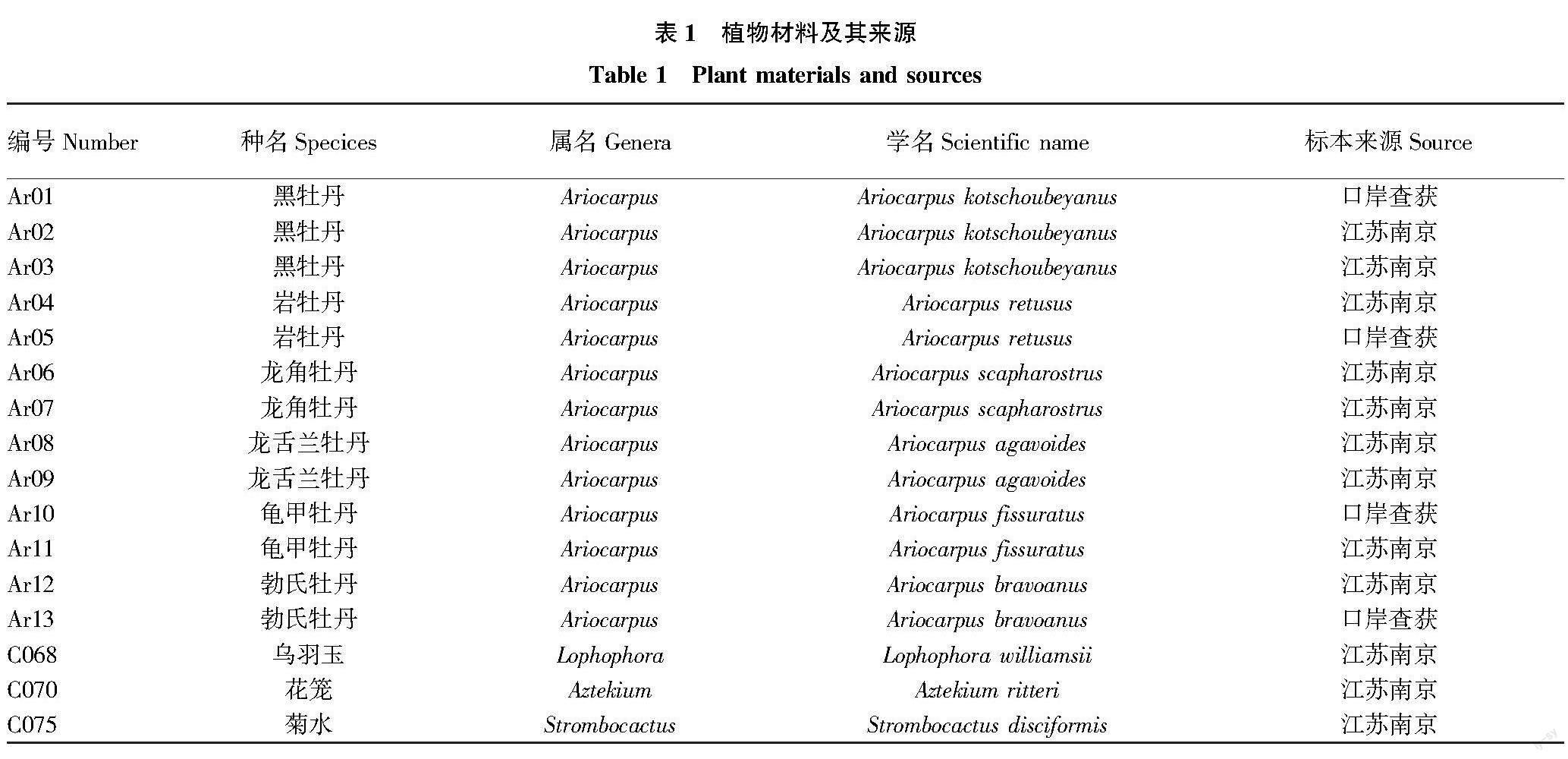
收集13份巖牡丹屬及3份仙人掌科共16份試驗材料。試驗材料來自江蘇南京(南京中山植物園)和海關口岸查獲,均由相關專家鑒定,采集新鮮植物組織進行試驗。樣品信息見表1。
1.2? 基因組DNA提取
先將供試樣品進行表面消毒,用液氮將植物葉片研磨成粉,參照DNeasy Plant Mini Kit試劑盒說明書上的步驟方法,提取試驗樣品基因組DNA。提取基因組DNA置于4 ℃保存備用。
1.3? 引物與探針設計
通過轉錄組測序數據分析篩選,克隆得到長度為679~1 079 bp的巖牡丹屬PEPC序列。使用BioEdit軟件比對6種巖牡丹屬植物PEPC序列,發現黑牡丹與其他巖牡丹屬植物相比較,存在穩定的SNP位點。根據黑牡丹特有SNP位點,按照LGC KASPgenotyping試劑盒的使用說明,結合引物設計軟件Primer 5設計了特異性的黑牡丹KASP反應引物。PCC-A-kot-f1-1、PCC-A-kot-f1-2、PCC-A-kot-r1的序列分別為GGATTTCTCTTGAGCTGTGAC、GGATTTCTCTTGAGCTGTGAT和ACCCTATTACCTGACACCAT,擴增產物為61 bp。在2條正向引物序列的5′端加上FAM(5′GAAGGTGACCAAGTTCATGCT 3′)或HEX(5′GAAGGTCGGAGTCAACGGATT 3′) 熒光基團的接頭序列。
1.4? 反應體系與反應條件
將引物PCC-A-fis-f1-1、PCC-A-fis-f1-2、PCC-A-fis-r1濃度分別調整為36、36、90 μmol/L,并按體積比1∶1∶1配制成引物混合液。反應體系:取DNA(調整濃度為5 ng/μL) 5.00 μL,KASP master mix 5.00 μL,Primer mix 0.14 μL,使用PCR反應板進行KASP,每次反應2次重復;將5.00 μL模板DNA替換成5.00 μL 滅菌雙蒸水作為陰性對照(NTC),操作步驟和反應條件與其他樣品一致。反應條件參照KASP試劑盒說明書進行設置,簡述如下:94 ℃預變性15 min;第1步擴增反應:94 ℃變性20 s,61~55 ℃梯度退火并延伸60 s(每個循環降低0.6 ℃),10個循環;第2步擴增反應:94 ℃變性20 s,55 ℃退火并延伸60 s,26個循環。
1.5? 特異性測試
以16份供試樣品的DNA為模板,利用引物混合液與所述試劑配置成反應液。按編號裝入 96 孔PCR 板中,用于擴增檢測在7500Fast上進行KASP反應,按照設定反應條件進行擴增,觀察反應體系的特異性。
2? 結果與分析
2.1? 序列擴增和SNP位點標記
通用BioEdit軟件比對分析巖牡丹屬PEPC序列發現,3份黑牡丹樣本在322 bp位點為胞嘧啶(C),其余巖牡丹屬樣本對應位點為胸腺嘧啶(T),具體信息見圖1。
2.2? PEPC序列? 黑牡丹PEPC序列長度為679 bp。序列中畫橫線的部分是引物區,紅色標記的是黑牡丹的SNP位點,該試驗以該SNP位點設計了黑牡丹KASP檢測方法的特異性引物。
1?? TGGATACTGA AACACTATCG ACTGCGACAA ATTTCATTTT GTTAATGGTC GAGAGAATTT
61? AAAGGAAGAT TTTCACATAT TGATAAGCTA AATCCCCTTT TTTCCATATC TGATGCGGTT
121 TTACATCAGA CCATAGATAG TATTTGACAG CTACGAAACT AATGGCTCAG TAATTTGTAT
181 TGGAACTGAA CAAGTGGGAC TCGTGTTTGG GGCGGGTCAG TTGCATTCAA ATTGAAGCCC
241 TCCACCAAGC TCATGAAGAA AATGAAGTGT GAGTCTTGTT GGTTGGGTTG GGTCAACTGG
301 ATTTCTCTTG AGCTGTGACA TTGGGTTTGG TTAAAGTATA TGGTGTCAGG TAATAGGGTA
361 CAATTTAGTC TTGCCAAACC AACCCACATT CTATTTCCTT CCAAGAATAA TTCAAATCAT
421 GAAATATCTC TACTACCTGC CCGAGTTCTA CAAAGTACAA ACCAAGTCCG ACTGACCTAA
481 ATTTCCATTG TATTTTTGCA GGTCGTCGGG CACAAGGAGC TGCTTGAAGG AGATCCTTAC
541 ATGAAACAGA GGCTCCATAT TCGAGATCCA TACCTTACAA TCCTTAATGT CTTCCAAGCA
601 AATACTCTCA AGCGTATTCG TGAGCCAAAC TTCCACGTAA CACAGAACCC ACGATCATCC
661 GAGGAAATAG CGGAATCCA
2.3? 黑牡丹KASP特異性檢測
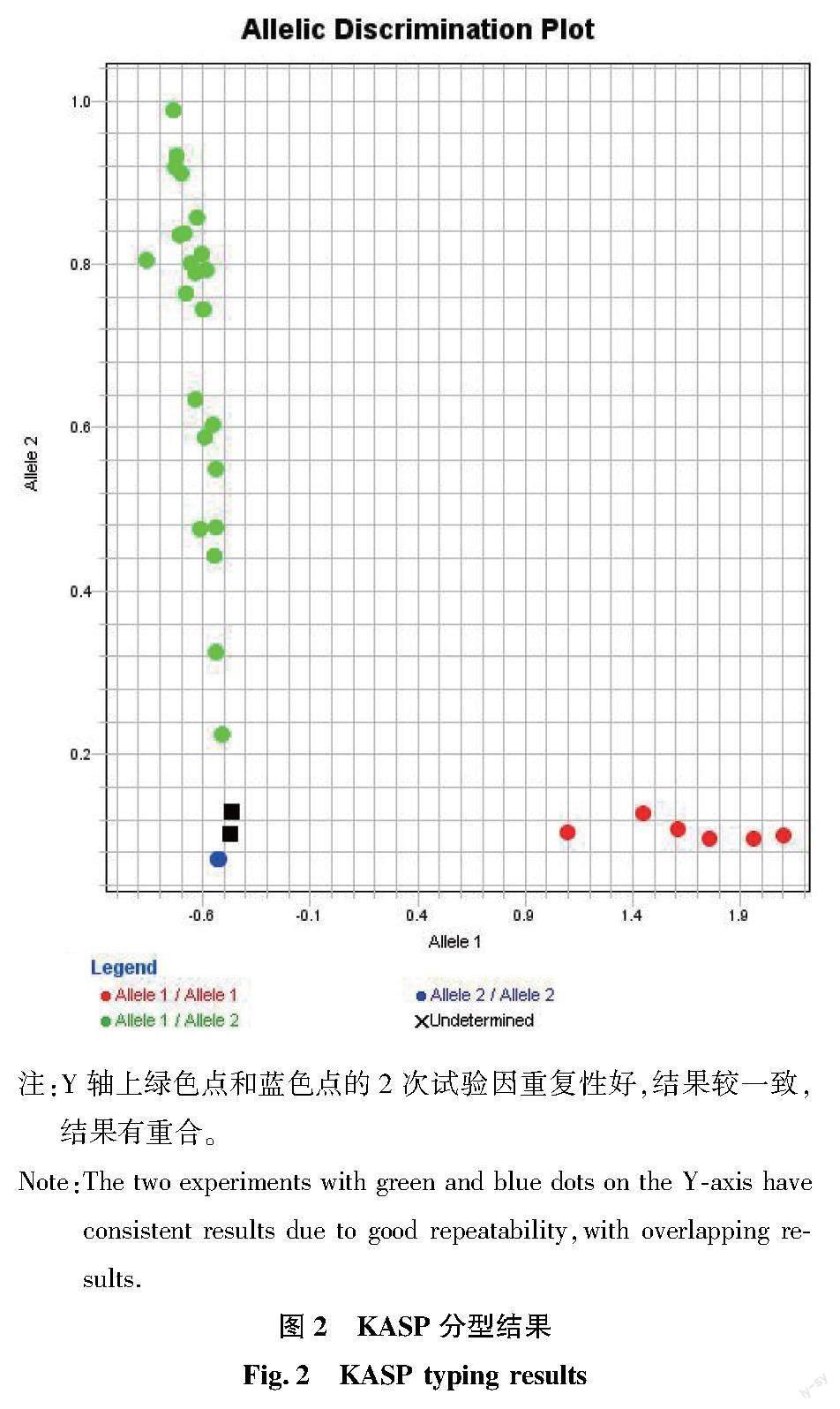
應用設計的特異性引物,對16份樣品DNA進行KASP反應,KASP分型結果見圖2。結果顯示,出現在X軸附近的紅色圓點是黑牡丹的基因型,Y軸附近的綠色圓點是同屬近緣種及花籠、菊水的基因型,坐標軸附近的藍色圓點是烏羽玉的基因型,黑色標記是陰性對照。通過PEPC序列的SNP位點所設計的KASP引物可以形成明顯的基因分型,準確鑒定黑牡丹,將其與同屬近緣種及其他仙人掌科物種進行區分。
3? 討論
當前,SNP分子標記方法作為國際植物新品種權保護聯盟(UPOV)BMT分子測試指南中構建DNA 指紋數據庫和國內《植物品種鑒定DNA 指紋方法總則》(NY/T 2594—2016)中推薦的標記方法[10]。競爭性等位基因特異性PCR(KASP)技術是由英國LGC公司開發的新一代高通量自動化SNP檢測技術,現已成為國際上SNP分型以及插入缺失變異檢測的主要方法之一[11]。KASP技術只需合成2個帶有不同顏色的熒光基團和雙鏈通用的探針,不需要針對每個SNP位點分別設計熒光探針,具有高通量、準確、省時、便捷的特點,實現了更加靈活的檢測,已成功應用于超過100余個物種的基因分型、遺傳多樣性分析、指紋圖譜構建等方面[12]。
目前巖牡丹屬的鑒定主要集中在形態學上,少有對巖牡丹屬分子鑒定的研究。該研究將PEPC基因應用于植物物種資源鑒定,結果表明,設計的PEPC引物在巖牡丹屬中具有100%的擴增效率和測序成功率,表現了良好的通用性。相比綠體序列matK[13]和psbA-trnH[14],PEPC序列具有更多的變異位點,更加適用于SNP分子標記方法和基因分型技術。該研究選擇巖牡丹屬PEPC序列上的SNP位點,基于KASP基因分型技術建立了黑牡丹的分子鑒定方法。該方法對瀕危物種巖牡丹屬黑牡丹的分型檢測既準確又可靠,對其他種類的鑒定具有極高的參考價值。同時,相對于DNA條形碼技術,KASP技術不需要DNA測序,大大縮短了檢測周期,在物種鑒定上有很好的應用潛力,為海關口岸查驗工作提供技術手段,也為瀕危野生動植物保護提供技術支撐。
參考文獻
[1]
VOVIDES A P,LUNA V,MEDINA G.Relación de algunas plantas y hongos mexicanos raros,amenazados o en peligro de extinción y sugerencias para su conservación[J].Acta botanica mexicana,1997(39):1-42.
[2]兌寶峰.黑牡丹與姬牡丹[J].園林,2004(9):36.
[3]BROCCANELLO C,CHIODI C,FUNK A,et al.Comparison of three PCR-based assays for SNP genotyping in plants[J].Plant methods,2018,14:1-8.
[4]鐘婷婷,郭詩芬,盧文斌,等.甘藍型油菜抗根腫病KASP標記開發和利用[J].華北農學報,2021,36(4):184-190.
[5]王富強,張建,溫常龍,等.基于KASP 標記的葡萄品種鑒定[J].中國農業科學,2021,54(13):2830-2846.
[6]HU W,ZHOU T H,WANG P F,et al.Development of whole-genome agarose-resolvable LInDel markers in rice[J].Rice,2020,13(1):1-11.
[7]IZUI K,MATSUMURA H,FURUMOTO T,et al.Phosphoenolpyruvate carboxylase:A new era of structural biology[J].Annual review of plant biology,2004,55:69-84.
[8]O′LEARY B,PARK J,PLAXTON W C.The remarkable diversity of plant PEPC(phosphoenolpyruvate carboxylase):Recent insights into the physiological functions and post-translational controls of non-photosynthetic PEPCs[J].The biochemical journal,2011,436(1):15-34.
[9]WANG X Y,GOWIK U,TANG H B,et al.Comparative genomic analysis of C4 photosynthetic pathway evolution in grasses[J].Genome biology,2009,10(6):1-18.
[10]WEI Z Y,LI H H,LI J,et al.Accurate identification of varieties by nucleotide polymorphisms and estab-lishment of scannable variety IDs for soybean germplasm[J].Acta agronomica sinica,2018,44(3):315-323.
[11]SEMAGN K,BABU R,HEARNE S,et al.Single nucleotide polymorphism genotyping using Kompetitive Allele Specific PCR(KASP):Overview of the technology and its application in crop improvement[J].Molecular breeding,2014,33(1):1-14.
[12]王富強,樊秀彩,張穎,等.SNP分子標記在作物品種鑒定中的應用和展望[J].植物遺傳資源學報,2020,21(5):1308-1320.
[13] CUNOUD P,SAVOLAINEN V,CHATROU L W,et al.Molecular phylogenetics of Caryophyllales based on nuclear 18S rDNA and plastid rbcL,atpB,and matK DNA sequences[J].American journal of botany,2002,89(1):132-144.
[14]HAMILTON M B.Four primer pairs for the amplification of chloroplast intergenic regions with intraspecific variation[J].Molecular ecology,1999,8(3):521-523.

