船舶壓載水中外來入侵魚類的快速檢測技術
劉璐 陳治 李揚 樊鑫

摘要:為消除壓載水中外來入侵魚類對生態環境的潛在威脅,解決傳統的入侵物種檢測方法存在的不足和局限性,采用傳統調查方法并結合環境DNA宏條形碼技術對壓載水中大量樣本的物種鑒定及目標物種進行信息獲取,構建外來魚種數據庫,并進行定性檢測及定量評估,探索建立適合我國國情的監測理論和方法,綜合分析評估魚類潛在入侵風險,為我國眾多港口進行壓載水處理和管控提供理論依據,并最終為降低魚類入侵風險甚至達到零風險提供技術支持。
關鍵詞:船舶壓載水;入侵魚類;快速檢測;環境DNA
中圖分類號:U691+.6;R185.3+2文獻標志碼:A文章編號:1672-0032(2023)01-0110-09
引用格式: 劉璐,陳治,李揚,等.船舶壓載水中外來入侵魚類的快速檢測技術[J].山東交通學院學報,2023,31(1):110-118.
LIU Lu, CHEN Zhi, LI Yang,et al.Fast detection technology for alien invasive fish in ship ballast water[J].Journal of Shandong Jiaotong University,2023,31(1):110-118.
0 引言
生物入侵、環境污染、資源過度開發和氣候變化帶來的環境問題已對全球生態系統構成巨大威脅[1]。近年來,以人類為傳播媒介的生物入侵事件在全球范圍愈演愈烈。密集的航運業、水產品和水生寵物貿易及水產養殖等導致外來水生生物快速傳播,進而引發生物入侵[2]。每年通過船舶貨運進入我國的壓載水超過3.5億t,且還在以年均5%左右的速度遞增[3]。
據統計,為保證航行的安全性和穩定性,一條普通的商業散貨船在單次航程中攜載的壓載水約3萬t,船舶壓載水每天攜帶的海洋生物多達7000種。大量的浮游植物、浮游動物、無脊椎動物幼蟲、魚卵和仔稚魚及成魚被壓載水從原有海域帶到新的海域。船舶壓載水已成為無意間引入外來水生生物的最主要途徑[4]。外來物種從被引入到成為入侵物種一般需要經過3個階段:到達→建群→擴散,最終達到某種相對穩定的狀態[5],而且一旦入侵成功,極易造成生態失衡或疾病流行(瘧疾、乙腦、霍亂、登革熱等)[6]。因此,對壓載水實施有效處理和管控已成為港口船舶管理的必要措施和關鍵步驟。很多國家都采用多種檢測方法和監測手段,研究和監測壓載水引入的魚類。
遠洋運輸在全球對外貿易貨物運輸中發揮著不可替代的主導作用。2020年我國港口完成貨物吞吐量145.5億t,包括沿海港口94.8億t和內河港口50.7億t,位居世界第一,同比增長4.3%。內河等級航道里程6.73萬km。港口規模不斷擴大,貨物吞吐量超過2億t的港口達24個。中國出入境船舶壓載水輸入和輸出總量巨大,基本呈逐年增長趨勢。壓載水的輸入量從2015年的約2.8億t增至2020年的約3.6億t,增幅近50%[7]。通過壓載水傳播到我國海域的海洋入侵生物的種類和數量越來越多[8-9],給我國海洋環境保護帶來了前所未有的挑戰,對我國近岸養殖業和人民生命財產安全構成嚴重威脅。
因此,需重點關注被壓載水引入到我國海域的外來生物有哪些?它們是否有成為入侵物種的潛在風險?通過完善壓載水潛在入侵生物檔案庫并構建早期監測預警技術體系和平臺,對壓載水引入中國海域的外來生物數量、分布及其對中國境內其他生物和環境造成的影響實行有效的跟蹤監測,成為我國港口遠洋船舶管理和海洋生態環境保護的當務之急,這也是防止壓載水生物入侵的最直接、最有效的方式。
1 船舶壓載水防治生物入侵現狀
壓載水置換是防控外來生物入侵的常規操作[10],是指將船舶在離港時在近岸海域所加的壓載水在航行過程中更換為遠洋海水,其中可能引入的遠洋海洋生物一般難以在近岸海洋環境生存,可避免將其他港口的近岸海水帶入目的港而導致生物入侵。我國明確要求船舶必須安裝壓載水處理系統進行壓載水置換,且要求在離岸超過321.9 m或水深大于2000 m的遠洋中進行[5]。但載質量超過4萬t的船舶在大洋中更換壓載水存在一定風險,影響航行安全[11]。壓載水置換對于距離較短的國際航運線路也較困難,置換水域的離岸距離可能無法滿足國際海事組織(International Maritime Organization,IMO)的要求[12-13]。
在航行過程中也可通過壓載水處理系統對壓載水進行生物殺滅處理。傳統的處理手段有機械處理法、物理處理法和化學處理法等[3]。機械處理法包括過濾法、旋流分離法,物理處理法包括加熱法、紫外線照射法,化學處理法包括電解法、臭氧法和羥基自由基法等,但實際應用時均存在一定局限性,且不能殺滅壓載水中的所有生物。目前還有部分國家沒有施行壓載水處理系統的裝載要求,壓載水不但沒有在遠洋海域中置換,而且沒有進行生物殺滅處理,給海洋生物入侵埋下極大隱患,對近海生態環境保護造成嚴重威脅。
船舶是否安裝了壓載水處理系統,是采用壓載水置換還是生物殺滅處理,均不能保證壓載水排放的絕對安全性。對壓載水引發的生物入侵應以防為主,防治結合。
2 船舶壓載水中魚類入侵的潛在風險
我國壓載水的入侵生物監測對象大多為微生物、浮游植物和小型無脊椎動物等[9,14-16],往往忽略魚類等較大體型生物,很多學者認為壓載水并不是外來魚種入侵的主要途徑,相關研究也沒有引起足夠重視。在壓載水置換或排放時通常采用防護柵欄等設施粗濾壓載水,但不能阻擋原生生物、硅藻、真核生物幼體及橈足類等出入壓載艙,在航運過程中經常出現防護柵欄和壓載艙的泵葉被腐蝕甚至缺失等情況,這就為海洋魚類(幼魚、成魚)等被泵入壓載艙創造了機會[17]。我國于2018年底正式加入《國際船舶壓載水及沉積物控制與管理公約》,并于2019年正式履約,對壓載水引入外來入侵生物的監測和管控仍處在經驗摸索期。國外在壓載水魚類入侵方面的調查研究相對較早,已經有蝦虎魚(Neogobius melanostomus)、鲇(Blenniidae)、刺魚(Gasterosteus aculeatus)、鯡(Clupeidae)、鰈(Pleuronectidae)、梭魚(Liza ramada)、獅子魚(Pterois volitans/miles complex)、褐菖鲉(Sebastiscus marmoratus)等被壓載水引入和入侵的魚類記錄,其中,蝦虎魚和鲇為最常見種類,大多數魚類曾被多次引入[17-20]。穴居、底棲、耐缺氧和低溫、卵小、耐鹽幅寬或具有發達側線系統的魚類能夠更好地適應壓載水黑暗、缺氧和不平穩的環境。當魚類隨著壓載水被排放至近岸港口時,礁石、洞穴等為這些魚類提供了過渡環境。大多數魚類因為不能適應當地的餌料、水文等條件而無法繼續生存,只有極少數魚類能夠存活,經過到達、建群和擴散3個階段成為外來入侵物種。
魚類一旦入侵成功,比其他入侵水生生物種類的活動能力更強,活動范圍更廣,生長更快,壽命更長,往往更難管控和治理,最終對當地海洋環境、海洋生物群落甚至漁業經濟造成破壞,代價可能是海洋生物多樣性減少或本土魚類滅絕,海洋生態系統的結構和功能失調,給漁業經濟造成巨大損失,本國需要支付高額的防治費用[21]。通過其他途徑,如觀賞魚引入、生物防控和人工養殖等入侵我國的非本土魚類已對我國水域造成嚴重影響,如食蚊魚、羅非魚等[22-25]。1985年,印度洋-太平洋海域的紅獅子魚首次被報道出現在美國加利福尼亞附近海域,隨后的十幾年逐漸發展成為美國南部大部分海域的優勢魚種,對當地魚類和無脊椎動物的多樣性產生負面影響。直到2016年,科學家根據連續8 a的跟蹤調查和研究推斷,最終發現了紅獅子魚是通過壓載水轉運至美國太平洋沿岸港口并最終成為入侵魚種[19]。
3 船舶壓載水入侵魚類的傳統檢測方法
目前,壓載水中入侵魚類的監測及鑒定方法包括采用鏡檢(或熒光染色后鏡檢)等傳統的形態學手段[17,20],或采用流式細胞技術、激光質譜及透射光譜技術等方法監測浮游植物和微生物等單細胞生物[26-28],這些方法能否用于壓載水中的魚類監測還有待驗證。調查時,壓載水滿倉時通常采用浮游生物網、抄網和抽水泵捕獲魚類生物,在空倉時通常采用肉眼觀察、先拍照后鑒定的方式。
采用傳統形態學鑒定方法進行壓載水中生物監測可直觀鑒定所檢測生物并對樣本生物量化,有效區分活體和死亡個體,增加監測結果的準確性。傳統形態學鑒定法的局限性是:1)效率低、費用高[26],早期發現和快速采取措施是應對生物入侵的關鍵環節,但在早期入侵階段,生物量較小,傳統的形態學檢測方法工作量大,形態鑒定試驗周期長,且容易低估潛在入侵風險,待檢測報告出來可能為時已晚,船舶或已靠港排放壓載水,使入侵防控變得被動;2)對形態鑒定人員的專業知識水平要求高[27];3)靈敏度低,許多類群有變態發育的特點,不同生活史階段的形態結構和習性發生較大變化,部分類群在同一生活史時期的形態學特點極為相似,增加了物種鑒定的難度,大多數情況也只能鑒定到科或科以上分類階元[29]。采樣方式和物種自身形態特征也對魚類物種鑒定工作產生較大影響。樣品保存不當導致形態特征缺失,給準確的分類鑒定造成困難[30]。目前亟需建立集快速、準確、高靈敏度、高分辨率于一體的監測壓載水外來魚種的試驗方法。
4 環境DNA 宏條形碼技術的應用
環境DNA(environment DNA,eDNA)是生物體釋放到環境中的DNA的總稱,主要來源于生物體的配子、皮膚碎屑、粘黏或排泄物等[31]。該技術從環境樣品(水體、土壤、空氣等)中直接提取DNA開展生物信息監測,不分離任何目標生物。
eDNA技術在20世紀80年代首先應用在微生物的群落研究中。Ficetola等[31]在2008年采用水中提取的DNA監測到美國牛蛙的分布情況,開啟了eDNA在水生生物實時監測中的應用研究。該技術已應用于生物多樣性評價、水生生物監測和珍稀瀕危物種的保護等研究領域[32-35]。DNA宏條形碼技術是采用基因組內一段標準化的DNA片段鑒定生物物種的分子鑒定技術,目前在物種的快速鑒定和起源、進化等研究領域得到了廣泛應用[36-38]。eDNA宏條形碼技術通過提取環境樣品中的DNA并使用條形碼基因通過引物擴增,利用聚合酶鏈式反應(polymerase chain reaction,PCR)和二代測序等技術,在短時間內獲得可操作分類單元(operational taxonomic units,OTUs),通過與具有可靠物種分類信息的DNA條形碼參照序列比對,可實現對大量樣本的物種鑒定及目標物種的信息獲取[39-41]。傳統的DNA條形碼技術僅從單一樣品中獲取DNA信息,eDNA 宏條形碼技術可直接從環境樣品或生物混合樣品中大批量識別物種[41-42],省時、高效、成本低,如圖1所示。
目前,eDNA宏條形碼技術應用到船舶壓載水入侵生物的監測研究處于發展階段[43-50],國內尚未見相關研究。2015年,Zaiko的研究團隊發表關于eDNA 宏條形碼可用于壓載水生物多樣性調查和入侵生物監測的研究結果,通過細胞色素氧化酶(cytochrome oxidase subunit I,COI)基因開展歐洲蝸牛(Peringia ulvae)這一入侵生物的監測和風險評估研究[47]。2019年,Rey等[49]采用eDNA 宏條形碼技術在壓載水中檢測到非本地物種的橈足類生物。Gerhard等[50]采用16S rRNA基因對美國20個港口41個壓載艙進行基于eDNA 宏條形碼技術的微生物調查,結果顯示5艘商船壓載水中的大腸桿菌含量超標,未達到壓載水排放標準。目前,基于eDNA 宏條形碼技術開展壓載水中入侵魚類的調查研究尚未見報道。
未來研究中可選擇我國航運發達的沿海港口為研究區域,以外來入港船舶為研究對象,通過明確壓載水eDNA樣品采集的位置和途徑,設計針對船舶壓載水的高質量的eDNA采樣方法,通過高通量測序和eDNA 宏條形碼分析,結合形態學分析方法,準確、快速鑒定我國海域船舶壓載水中的外來魚種。eDNA宏條形碼檢測比形態學檢測更靈敏,為提高物種的檢出率和降低假陽性率,可基于eDNA宏條形碼技術進行多方位采樣和設置陰性對照,對比港口附近海域本土魚種的調查研究,驗證壓載水中外來魚種的鑒定結果。初步構建通過船舶壓載水引至我國海域的有潛在入侵風險的外來魚種信息數據庫和物種DNA條形碼的標準數據庫。完善基于eDNA 宏條形碼技術的壓載水外來魚種的鑒定方法,揭示外來魚種在中國海域入侵的潛在風險,向我國外來入侵物種防控工作發出預警,為我國壓載水處理和管控提供理論依據和資源信息共享平臺,為其他水生生物通過壓載水入侵我國海域的調查研究提供參考依據和技術支撐。
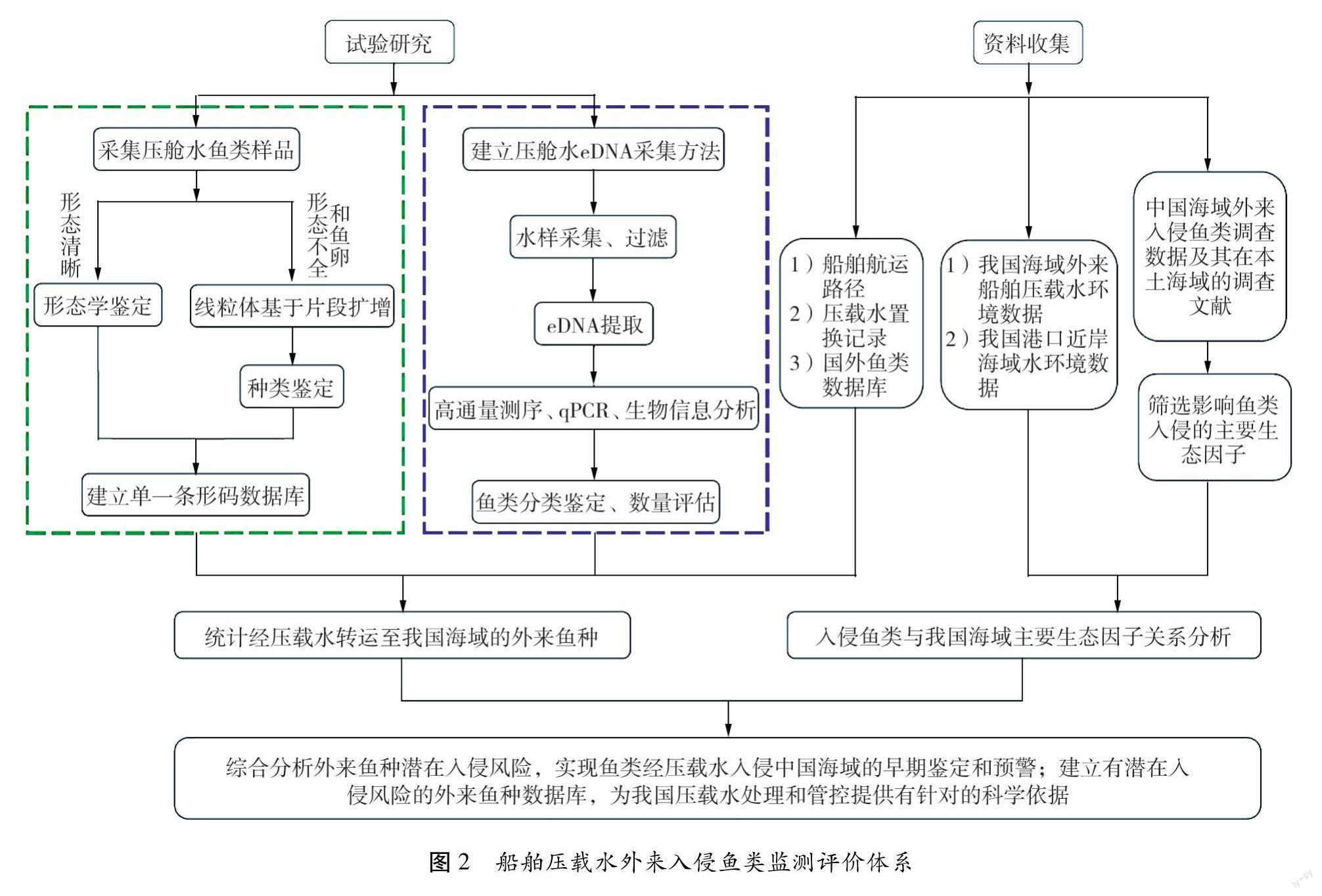
船舶壓載水外來入侵魚類監測評價體系如圖2所示。分別采集、調查壓載艙中壓載水、船舶所在港口附近海域水樣,基于eDNA宏條形碼測序的技術手段,對壓載水和港口水域、近岸海域中的魚類物種信息進行對比、排除,將測序片段與NCBI、EMBL、BOLD等數據庫比對,參考船舶航行路徑和壓載水更換地點等數據信息,初步確定外來魚種信息。與形態學鑒定結果進行相互印證,揭示壓載水引入外來魚類的種類組成,通過定量PCR開展外來魚種的定量評估。結合歷史調查數據和公開文獻報道,收集魚種自然分布海域的經緯度、溫度、鹽度、深度、pH值、溶解氧等基礎環境參數,通過與我國近岸海域的相應參數的對比分析,尋找不同海域間的異同,對比有記錄的成功入侵海洋魚類的共有特征及其與環境因子間的關系,初步探討外來魚種在我國海域成為入侵物種的潛在風險。構建魚類通過壓載水引至我國海域的有潛在入侵風險的外來魚種信息庫。
5 新方法應用的影響因素和局限性
科學合理的取樣方法是壓載水檢測的基礎,對生物入侵風險管理非常重要,但目前公約中未規定壓載水檢測采樣的標準化方法。船舶沒有專用的壓載水采樣口,可利用的采樣途徑有測深管、消防管道開口、人孔、通氣孔和壓載水排放口,根據這些管、孔的大小可歸類為大孔采樣和小孔采樣,但每種采樣途徑都有一定的局限性。在對進港船舶開展壓載水生物監測調查時,為保證監測結果的準確性,根據不同的船型和裝載情況,選擇的取樣具體位置和方法也不同[51-52]。需摸索出一套合理、可行的采集eDNA樣品的取樣方法,保證所采集到的水樣具有代表性,是保證研究結果準確性的前提。采集壓載水水樣,通過采水器法、潛水泵法、隔膜泵法和管道采樣法及不同的采樣途徑,采取控制變量等方法,結合核酸定量分析和定量PCR檢測,探索eDNA的最佳采樣途徑,確定適用于壓載水的高質量eDNA采樣方法。優化樣品收集保存技術,有效地規避樣品間交叉污染,設置陰性對照,提升鑒定結果的準確性和可靠性。
不同的數據分析方式可能給快速檢測外來魚種帶來不同的檢測結果,比如分項目閱讀值的不同設置方法可能引起結果的假陰性,影響外來物種的有效鑒別和檢出,適當選擇和設置生物信息學工具及參數尤為重要[53],注意開發準確性更高的軟件包,增強不同研究結果的可對比性及準確性。
參考數據庫的不完整性和不準確性是限制采用eDNA宏條形碼技術進行船舶壓載水中外來物種檢測的主要障礙[54],數據庫不完整將大大降低入侵魚種的檢出率,應盡快構建完善的物種DNA條形碼參考數據庫、建立全球范圍的資源信息共享和應用平臺,推動該技術的應用與推廣。檢測外來魚種應建立健全外來入侵魚種和各港口附近海域本土魚類資料數據庫,以便準確判定檢測到的生物是否為外來入侵魚種。
最優目標基因的選擇是影響采用eDNA宏條形碼技術進行船舶壓載水中外來物種檢測的另一重要因素。目前已有不少針對魚類設計的eDNA宏條形碼通用引物[55-57],其目標基因彼此存在差異,導致魚類鑒定方面的通用性差異較大。需根據實際調查的海域狀況和有記錄的魚類出現記錄及具體的預試驗等設定目標基因。魚類種間差異的閾值認定也不固定。標準的動物DNA 條形碼主要為線粒體細胞色素 c 氧化酶亞基 I 基因,但魚類eDNA 宏條形碼主要選用線粒體 12S 核糖體或 16S 核糖體基因,變異速度不穩定[58],且擴增片段多小于 200 bp(堿基對base-pair)。魚類物種系統發育分析過程的種間差異閾值目前也尚無定論。
在船舶壓載水采用eDNA宏條形碼技術鑒定入侵魚類的種類相對簡單,但如何實現入侵魚類的定量評估仍存在爭議,未來可開發準確鑒別特定外來物種和相關近緣物種的特異性條形碼,探索影響eDNA與生物量線性關系的生物和非生物因素,校正優化eDNA定量方法[59]等方面的研究手段,促進eDNA技術在壓載水入侵生物防治中的應用。
參考文獻:
[1]鞠瑞亭,李慧,石正人,等.近十年中國生物入侵研究進展[J].生物多樣性,2012,20(5):581-611.
JU Ruiting, LI Hui, SHI Zhengren, et al. Progress of biological invasions research in China over the last decade[J].Biodiversity Science, 2012,20(5):581-611.
[2]LOCKWOOD J L, HOOPES M F, MARCHETTI M P.Invasion ecology[M].Chichester, West Sussex:Wiley-Blackwell Scientific Publications, 2013.
[3]笪靖.船舶壓載水中有害水生物的處理技術研究[J].船舶工程,2016,38(增刊2):120-123.
DA Jing. Research on treatment technology of harmful aquatic organisms in ship ballast water[J].Ship Engineering, 2016,38(Suppl.2):120-123.
[4]ARDURA A, ZAIKO A, MARTINEZ J, et al.Environmental DNA evidence of transfer of North Sea molluscs across tropical waters through ballast water[J].Journal of Molluscan Studies, 2015, 81:495-501.
[5]劉艷,吳惠仙,薛俊增.海洋外來物種入侵生態學研究[J].生物安全學報,2013,22(1):8-16.
LIU Yan, WU Huixian, XUE Junzeng.The ecology of invasions by marine exotic species[J].Journal of Biosafety, 2013,22(1):8-16.
[6]吳昊,丁建清.入侵生態學最新研究動態[J].科學通報,2014,59(6):438-448.
WU Hao, DING Jianqing.Recent progress in invasion ecology[J].Chinese Science Bulletin, 2014,59(6):438-448.
[7]交通運輸部.2020年交通運輸行業發展統計公報[R].北京:交通運輸部,2021.
[8]白敏冬,張芝濤,薛曉紅.海洋外來有害生物和病原體防治新技術[M].北京:海洋出版社,2010.
[9]吳惠仙,邊佳胤,王飛飛,等.中國大陸到港船舶壓載水生物研究[J].上海海洋大學學報,2018,27(3):455-459.
WU Huixian, BIAN Jiayin, WANG Feifei, et al.Research of organisms in ballast water of ships arriving in the mainland of China[J].Journal of Shanghai Ocean University, 2018,27(3):455-459.
[10]International Maritime Organization.IMO (2004) International convention for the control and management of ships′ ballast water and sediments[J/OL].(2015-12-15)[2022-03-20].https://www.westpandi.com/publications/notice-to-members/2016-2017/notice-to-members-no-20-20162017/.
[11]王朝暉,陳菊芳,楊宇峰.船舶壓載水引起的有害赤潮藻類生態入侵及其控制管理[J].海洋環境科學, 2010,29(6):922-934.
WANG Zhaohui, CHEN Jufang, YANG Yufeng.Control and management of harmful algal bloom species introduced by ballast water[J].Marine Environmental Science, 2010,29(6):922-934.
[12]WOODWARD J B, PARSONS M G, TROESCH A W.Ship operational and safety aspects of ballast water exchange at sea[J].Canadian Journal of Chemistry,1994,31(4):315-326.
[13]郝林華,石紅旗,王能飛,等.外來海洋生物的入侵現狀及其生態危害[J].海洋科學進展,2005,23(增刊):121-126.
HAO Linhua, SHI Hongqi, WANG Nengfei, et al.Present status and ecological harming of alien marine organism invasion[J].Advances in Marine Science, 2005,23(Suppl.):121-126.
[14]王雷,薛俊增,吳惠仙.中國到港船舶壓載水中致病菌分析及檢測[J].船舶工程, 2020,42(2):92-98.
WANG Lei, XUE Junzeng, WU Huixian.Analysis and detection of pathogenic bacteria in ballast water of oceanic vessels moored in Chinese coastal ports[J].Ship Engineering, 2020,42(2):92-98.
[15]GHABOOLI S, ZHAN A, PAOLUCCI E, et al.Population attenuation in zooplankton communities during transoceanic transfer in ballast water[J].Ecology and Evolution, 2016, 17:6170-6177.
[16]楊帆,李捷,于淑婷,等.船舶壓載水浮游生物檢測方法研究進展[J].環境科學與技術,2017,40(4):45-49.
YANG Fan, LI Jie, YU Shuting, et al.Advances in detection of plankton in ballast water[J].Environmental Science & Technology, 2017,40(4):45-49.
[17]WONHAM M J, CARLTON J T, RUIZ G M, et al.Fish and ships:relating dispersal frequency to success in biological invasions[J].Marine Biology,2000,136:1111-1121.
[18]SEEBENS H, GASTNER M T, BLASIUS B, et al.The risk of marine bioinvasion caused by global shipping[J].Ecology Letters, 2013, 16(6):782-790.
[19]MACISAAC H J, DE ROY E M, LEUNG B, et al.Possible ballast water transfer of Lionfish to the eastern Pacific Ocean[J].Plos One, 2016, 11(11):e0165584.
[20]HANSEN H, KARLSBAKK E.Pacific false kelpfish, Sebastiscus marmoratus (Cuvier, 1829) (Scorpaeniformes, Sebastidae) found in Norwegian waters[J].Bioinvasions Records, 2018, 7(1):73-78.
[21]潘勇,曹文宣,徐立蒲,等.魚類入侵的過程、機制和研究方法[J].應用生態學報,2007,18(3):687-692.
PAN Yong, CAO Wenxuan, XU Lipu, et al.Process, mechanism, and research method of fish invasion[J].Chinese Journal Applied Ecology, 2007,18(3):687-692.
[22]李振宇,解炎.中國外來入侵種[M].北京:中國林業出版社,2002.
[23]何舜平,陳宜瑜.中國淡水魚類瀕危現狀及致危原因分析:保護中國的生物多樣性[M].北京:中國環境科學出版社,2001.
[24]解炎,李振羽,汪松.中國入侵物種綜述:保護中國的生物多樣性(二) [M].北京:中國環境科學出版社,1996.
[25]汪松,樂佩琦,陳宜瑜.中國瀕危動物紅皮書:魚類卷[M].北京:科學出版社,1998.
[26]HOELL I, OLSEN R, HESS-ERGA O, et al.Application of flow cytometry in ballast water analysis-biological aspects[J].Management of Biological Invasions,2017,8(4):575-588.
[27]EMAMI K, ASKARI V, ULLRICH M, et al.Characterization of bacteria in ballast water using MALDI-TOF mass spectrometry[J/OL].Plos One, 2012, 7:e38515[2022-03-21].https://doi.org/10.1371/journal.pone.0038515.
[28]MAHON A R, BARNES M A, LI F, et al.DNA-based species detection capabilities using laser transmission spectroscopy[J/OL].Journal of the Royal Society Interface, 2013, 10:20120637 [2022-05-21].http://dx.doi.org/10.1098/rsif.2012.0637.
[29]劉璐,孫典榮,李純厚,等.DNA條形碼技術在鯔科魚類鑒定中的應用[J].中國海洋大學學報,2016,46(11):178-186.
LIU Lu, SUN Dianrong, LI Chunhou, et al.Application of DNA barcoding in classification of mugiIidae fishes[J].Periodical of Ocean University of China, 2016, 46(11):178-186.
[30]DRAKE J M, LODGE D M.Rates of species introductions in the Great Lakes via ships′ ballast water and sediments[J].Canadian Journal of Fisheries and Aquatic Sciences, 2007, 64:530-538.
[31]FICETOLA G F, MIAUD C, POMPANON F, et al.Species detection using environmental DNA from water samples[J].Biology Letters, 2008, 4:423-425.
[32]李飛龍,楊江華,楊雅楠,等.環境DNA宏條形碼監測水生態系統變化與健康狀態[J].中國環境監測,2018,34(6):37-46.
LI Feilong, YANG Jianghua, YANG Ya′nan, et al.Using environmental DNA metabarcoding to monitor the changes and health status of aquatic ecosystems[J].Environmental Monitoring in China, 2018,34(6):37-46.
[33]單秀娟,李苗,王偉繼.環境DNA (eDNA)技術在水生生態系統中的應用研究進展[J].漁業科學進展,2018,39(3):23-29.
SHAN Xiujuan, LI Miao, WANG Weiji.Application of environmental DNA technology in aquatic ecosystem[J].Progress in Fishery Sciences, 2018, 39(3):23-29.
[34]李萌,尉婷婷,史博洋,等.環境DNA 技術在淡水底棲大型無脊椎動物多樣性監測中的應用[J].生物多樣性,2019,27:480-490.
LI Meng, WEI Tingting, SHI Boyang, et al.Biodiversity monitoring of freshwater benthic macroinvertebrates using environmental DNA[J].Biodiversity Science, 2019,27:480-490.
[35]RUPPERT K M, KLINE R J, RAHMAN M S.Past, present, and future perspectives of environmental DNA (eDNA) metabarcoding:a systematic review in methods, monitoring, and applications of global Edna[J].Global Ecology and Conservation, 2019,17:e00547.
[36]劉璐,高天翔,韓志強,等.中國近海棱梭拉丁名的更正[J].中國水產科學,2016,23:1108-1116.
LIU Lu, GAO Tianxiang, HAN Zhiqiang, et al.Latin name correction of Liza affinis from China[J].Journal of Fishery Sciences of China, 2016,23:1108-1116.
[37]LIU L, PANHWAR S, GAO T, et al.New genetic evidence from three keel-backed Liza species based on DNA barcoding confirms morphology-based identification[J].Pakistan Journal of Zoology,2017, 49:1901-1907.
[38]VALENTINI A, TABERLET P, MIAUD C, et al.Next-generation monitoring of aquatic biodiversity using environmental DNA metabarcoding[J].Molecular Ecology, 2016, 25(4):929-942.
[39]KUMAR S, CARLSEN T, MEVIK B H, et al.CLOTU:an online pipeline for processing and clustering of 454 amplicon reads into OTUs followed by taxonomic annotation[J].BMC Bioinformatics, 2011, 12:182-187.
[40]JI Y, ASHTON L, PEDLEY S M, et al.Reliable, verifiable and efficient monitoring of biodiversity via metabarcoding[J].Ecology Letters, 2013, 16(10):1245-1257.
[41]SHAWA J, CLARKEA L J, WEDDERBURNB S D, et al.Comparison of environmental DNA metabarcoding and conventional fish survey methods in a river system[J].Biological Conservation, 2016, 197:131-138.
[42]AYLAGAS E, BORJA A, IRIGOIEN X, et al.Benchmarking DNA metabarcoding for biodiversity-based monitoring and assessment[J].Frontiers in Marine Science, 2016, 1(3):96.
[43]JERDE C L, CHADDERTON W L, MAHON A R, et al.Detection of Asian carp DNA as part of a Great Lakes basin-wide surveillance program[J].Canadian Journal of Fisheries and Aquatic Sciences, 2013, 70(4):522-526.
[44]LIN M X, ZHANG S, YAO M.Effective detection of environmental DNA from the invasive American bullfrog[J].Biological Invasions, 2019, 21(7):2255-2268.
[45]AMBERG J, MERKES C, STOTT W, et al.Environmental DNA as a tool to help inform zebra mussel, Dreissena polymorpha, management in inland lakes[J].Management of Biological Invasions, 2019, 10(1):96-110.
[46]ARDURA A, ZAIKO A, MARTINEZ J, et al.Environmental DNA evidence of transfer of North Sea molluscs across tropical waters through ballast water[J].Journal of Olluscan Studies, 2015, 81:495-501.
[47]ZAIKO A, MARTINEZ J, SCHMIDT-PETERSEN J, et al.Metabarcoding approach for the ballast water surveillance:an advantageous solution or an awkward challenge[J].Marine Pollution Bulletin, 2015, 92(1/2):25-34.
[48]EGAN S P, GREY E, OLDS B, et al.Rapid molecular detection of invasive species in ballast and harbor water by integrating environmental DNA and light transmission spectroscopy[J].Environmental Science and Technology, 2015, 49(7):4113-4121.
[49]REY A, CARNEY K, QUINONES L, et al.Environmental DNA metabarcoding:a promising tool for ballast water monitoring[J].Environmental Science and Technology, 2019, 53(20):11849-11859.
[50]GERHARD W A, GUNSCH C K.Metabarcoding and machine learning analysis of environmental DNA in ballast water arriving to hub ports[J].Environment International, 2019,124:312-319.
[51]徐加偉,付立東,吳桂濤.船舶壓載水取樣探究[J].中國水運,2016,16(6):12-14.
[52]張弛,薛俊增,邵于豪,等.船舶壓載水浮游植物采樣技術的比較[J].上海海洋大學學報,2018,27:421-425.
ZHANG Chi, XUE Junzeng, SHAO Yuhao, et al.Effects of different sampling methods on the assessment of biomass quantity in ballast water[J].Journal of Shanghai Ocean University, 2018,27:421-425.
[53]SCOTT R, ZHAN A B, BROWN E A, et al.Optimization and performance testing of a sequence processing pipeline applied to detection of nonindigenous species[J].Evolutionary Applications,2018, 11(6):891-905.
[54]WEIGAND H, BEERMANN A J, C^IAMPOR F, et al.DNA barcode reference libraries for the monitoring of aquatic biota in Europe:gap-analysis and recommendations for future work[J].Science of the Total Environment, 2019, 678:499-524.
[55]SCHENEKAR T, SCHLETTERER M, LECAUDEY L A, et al.Reference databases, primer choice, and assay sensitivity for environmental metabarcoding:lessons learnt from a re-evaluation of an eDNA fish assessment in the Volga headwaters[J].River Research and Applications, 2020, 36(7):1004-1013.
[56]SHU L, LUDWIG A, PENG Z.Environmental DNA metabarcoding primers for freshwater fish detection and quantification:in silico and in tanks[J].Ecology and Evolution, 2021, 11(3):8281-8294.
[57]ZHANG S, ZHAO J, YAO M.A comprehensive and comparative evaluation of primers for metabarcoding eDNA from fish[J].Methods in Ecology and Evolution, 2020, 11(12):1609-1625.
[58]MIYA M, SATO Y, FUKUNAGA T, et al.MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes:detection of more than 230 subtropical marine species[J].Royal Society open science, 2015, 2(7):150088.
[59]林淵源, 趙崢.環境 DNA 技術在水生入侵生物監測中的應用[J].生態毒理學報, 2021, 16(6):1-12.
LIN Yuanyuan, ZHAO Zheng.Application of environmental DNA technologies in monitoring aquatic invasive species[J].Asian Journal of Ecotoxicology, 2021, 16(6):1-12.
Fast detection technology for alien invasive fish in ship ballast water
LIU Lu1, CHEN Zhi2,3, LI Yang4, FAN Xin1*
1.Naval Architecture and Port Engineering College, Shandong Jiaotong University, Weihai 264209,China;
2.Key Laboratory of Utilization and Conservation for Tropical Marine Bioresources, Hainan Tropical Ocean University, Ministry of Education,
Sanya 572022,China;3.Hainan Key Laboratory for Conservation and Utilization of Tropical Marine Fishery Resources,
Hainan Tropical Ocean University, Sanya 572022,China; 4.Weihai Ecological Environment Monitoring Center, Weihai 264200 China
Abstract:In order to eliminate the potential threat of alien invasive fish in ballast water to the ecological environment, and solve the shortcomings and limitations of traditional invasive species detection methods, traditional investigation methods combined with environmental DNA macro barcode technology are used to identify species and obtain information about target species in a large number of samples in ballast water. A database of alien fish species is built, and qualitative detection and quantitative evaluation are carried out. Monitoring theories and methods suitable for China′s national conditions are explored and established to comprehensively analyze and evaluate the potential invasion risk of fish. Theoretical basis for ballast water treatment and control in many ports in China is provided, and finally technical support for reducing the invasion risk of fish and even reaching zero risk is offered.
Keywords:ship ballast water; invasive fish; rapid detection; environmental DNA
(責任編輯:王惠)

