半蒴苣苔屬植物染色體制片優化及染色體數目和倍性研究
高丹 向小果 張強 張艷杰 金偉濤

摘 要: 染色體數目和倍性是系統與進化生物學和遺傳學研究中十分重要的基礎信息。為探索半蒴苣苔屬染色體制片的適宜條件以及染色體數目的進化模式及其與物種的進化關系,該研究基于半蒴苣苔屬染色體數目的進化歷史,并根據該屬植物具有葉片扦插繁殖的特性,采用葉片水培生根法獲取半蒴苣苔(Hemiboea subcapitata)、弄崗半蒴苣苔(H. longgangensis)、龍州半蒴苣苔(H. longzhouensis)、江西半蒴苣苔(H. subacaulis var. jiangxiensis)、華南半蒴苣苔(H. follicularis)和永福半蒴苣苔(H. yongfuensis)6種植物的根尖材料,分析不同實驗條件對染色體制片效果的影響,對染色體制片實驗的條件進行優化及染色體計數,結果表明:(1)9:30—10:00取材,解離10 min以及染色15 min為半蒴苣苔屬染色體制片的適宜條件。(2)上述6種半蒴苣苔屬植物均為二倍體,染色體數目均為32(2n=2x=32)。(3)除個別物種染色體數目有變化以外,該屬大部分物種染色體數目可能為2n=2x=32且染色體數目變化可能是非整倍化的作用,與物種進化沒有明顯關系。該研究結果為半蒴苣苔屬以及具有類似葉片再生植株特性類群的染色體制片提供了參考,并為該類群的分類、系統進化等方面的研究提供了啟示。
關鍵詞: 苦苣苔科, 半蒴苣苔屬, 葉片水培生根, 染色體數目, 基因組倍性
中圖分類號: Q943? 文獻標識碼: A? 文章編號: 1000-3142(2023)05-0833-13
Abstract: Chromosome number and genome ploidy are crucial basic information for systematic evolutionary biology and genetics. Hemiboea, a genus of Gesneriaceae, with about 44 species, is mainly distributed in southern China, which have important medicinal and ornamental values. It is difficult to obtain suitable root tips for the chromosome preparation because of their small twining roots and small chromosomes. So far, only the chromosome numbers from three species of Hemiboea have been reported, while the chromosome numbers and ploidy of most species are unknown. The relationship between the pattern of chromosome number evolution and species evolution is also unclear. In this study, in order to explore the optimal conditions for chromosome preparation and the relationship between chromosome numbers and species evolution of Hemiboea, firstly, the root tips of six species (including H. subcapitata, H. longgangensis, H. longzhouensis, H. subacaulis var. jiangxiensis, H. follicularis and H. yongfuensis), which were generated by the method of hydroponic rooting for cutting leaves based on the characteristics of leaf cuttage propagation of plants in Hemboea, were used for the chromosome preparation experiments. Then, the effects of different experimental conditions on chromosome preparation were assessed. Next, the conditions were optimized and the chromosome counting was performed. Finally, the evolutionary history of chromosome numbers in Hemiboea and the related genera including Anna and Loxostigma were traced based on the molecular phylogenetic relationships, and the evolutionary pattern of chromosome numbers and relationships with species evolution were studied. The results were as follows: (1) The optimal conditions for karyotype preparation of the plants in Hemiboea were sampling during 9:30-10:00, dissociating for 10 min, and dyeing for 15 min. (2) All the six species of Hemiboea were diploid and with 32 chromosome numbers (2n=2x=32). (3) Except for a few species in the genus, the chromosome numbers of most species might be 2n=2x=32, and the variation of chromosome numbers might be caused by aneuploidy, which had no obvious relationship with the species evolution. This study sheds light on chromosome preparation of Hemiboea and other groups with similar leaf regeneration characteristics, and provides implications for the classification and phylogeny of this group.
Key words: Gesneriaceae, Hemiboea, hydroponic rooting for cutting leaves, chromosome number, genome ploidy
染色體是生物遺傳信息的載體,在植物的屬間、種間甚至種內常有不同程度的分化。這為探討屬間和種間的進化關系以及種內的變異格局提供了重要依據且被廣泛用作植物分類學的依據(洪德元,1990)。染色體的數目和倍性是遺傳學和基因組學研究的重要基礎信息(Soltis & Soltis, 1999)。目前,關于苦苣苔科植物細胞學的研究已有很多。最早的報道來自Oehlkers(1923)對Monophyllaea horsfieldii的細胞學研究(李振宇和王印政,2005),自20世紀60年代起,苦苣苔科植物的細胞學研究工作開始大范圍地開展起來(Ratter, 1963;Ratter & Ppentice, 1964,1967,1970)。中國苦苣苔科植物的細胞學研究起步較晚,最早的是中國臺灣學者對臺灣半蒴苣苔(Hemiboea bicornuta)的研究(Hsu, 1968),在此之后苦苣苔科植物細胞學研究取得了很多進展(魯元學等,2002;曹麗敏等,2003;季慧等,2008;覃信梅等,2020)。例如,廣義報春苣苔屬(Primulina s. l. )植物的細胞學研究,共約100種植物的染色體數目被報道(劉瑞瑞,2013)。綜合這些研究發現,苦苣苔科植物染色體的體積通常比較小(Mller & Kiiehn, 2004)且該科植物染色體數目的變異范圍比較大(李振宇和王印政,2005)。但是,大部分實驗是選用植物的根尖作為材料(王印政和顧志建,1999;魯元學等,2002;季慧等,2008),少數采用花芽(Hsu, 1968)。選擇根尖為材料時,獲取根尖的方式主要有種子萌發和組織培養,而這些方式都存在不足。例如,種子萌發時容易發霉,盡管萌發前已將種子進行了消毒,但有些植物的種子仍會發霉,已萌發的幼苗也會出現腐爛現象(趙大克等,2010);植物組織培養的操作比較復雜,對實驗環境和操作能力的要求比較高。因此,尋找一個更簡便有效的獲取根尖材料的方式對于苦苣苔科植物的細胞學研究十分必要。
半蒴苣苔屬(Hemiboea)為苦苣苔科(Gesneriaceae)植物,該屬目前包括約44種6變種,其中不少種類都具有重要的藥用價值和觀賞價值。該屬廣泛分布于中國南部,少數種類分布于越南北部和日本南部,中國南部喀斯特地區是其分布和分化中心(李振宇和王印政,2005)。目前,關于半蒴苣苔屬植物的細胞學研究很少,僅涉及臺灣半蒴苣苔、貴州半蒴苣苔(H. cavaleriei)和單座苣苔(H. ovalifolia)3種且這些研究僅限于染色體數目和倍性的報道(Hsu,1968;魯元學等,2002;曹麗敏等,2003)。然而,染色體數目和倍性有何進化模式,與半蒴苣苔屬物種進化之間有何關系,該屬植物核型研究困難阻礙了這些問題的進一步研究。
Hsu(1968)在研究臺灣半蒴苣苔時選用了植物的花芽和根尖作為實驗材料且均不作預處理,僅報道了染色體二價體數目n=18;魯元學等(2002)在研究貴州半蒴苣苔時選用了植物的根尖作為實驗材料,用0.1%的秋水仙素進行預處理2 h,用1∶1的1 mol·L-1鹽酸和45%冰醋酸混合液在60 ℃水浴鍋內進行解離30 s,結果觀察到染色體不夠分散,有些染色體存在相互粘連的現象,這可能與材料本身染色體為小型染色體以及實驗過程中預處理時間不夠、解離時間過短有關;曹麗敏等(2003)在研究單座苣苔時選用植物實生苗的根尖作為實驗材料,用2∶1的1 mol·L -1鹽酸和45%冰醋酸混合液在60 ℃水浴鍋內進行解離30 s,結果觀察到染色體分散效果很差,染色體粘連現象十分明顯,這可能與材料本身染色體為小型染色體以及解離時間過短有關。由于半蒴苣苔屬于小型染色體,本身就難以分散且不易觀察(李振宇和王印政,2005),解離時間過短又會使細胞壁難以破開,染色體很難分散開、容易粘連在一起,因此半蒴苣苔屬植物的染色體制片仍不容易。此外,我們通過觀察還發現半蒴苣苔屬植物的根系十分細小且相互纏繞,很難直接得到良好的具有分生組織的根尖,通過植株直接獲取根尖材料研究染色體數目的方法比較困難。因此,該屬的染色體數目研究有待于新的取材策略和制片方法的優化。
李振宇和王印政(2005)的研究發現,苦苣苔科植物可以進行無性繁殖,只要很小的插穗就可以繁殖成新的植株,而在扦插繁殖方式中又屬葉插最為常見,多數苦苣苔科植物都可以用葉片進行葉插繁殖且葉片還可以水插,只要將葉柄浸入清水中就可逐漸生根出芽。覃信梅等(2020)在此前石山苣苔屬的細胞學研究中已經成功地通過葉片水培生根,我們通過實驗發現半蒴苣苔屬植物也易通過葉片水培生根。這為我們開展半蒴苣苔屬的細胞學研究提供了新的思路和方法。
本研究以半蒴苣苔屬的半蒴苣苔(H. subcapitata)、弄崗半蒴苣苔(H. longgangensis)、龍州半蒴苣苔(H. longzhouensis)、江西半蒴苣苔(H. subacaulis var. jiangxiensis)、華南半蒴苣苔(H. follicularis)和永福半蒴苣苔(H. yongfuensis)這6種植物為對象,采用葉片水培生根的方法對這6種植物進行細胞學研究,并在多種不同條件下對這6種半蒴苣苔屬植物進行實驗:(1)探索該屬染色體制片的適宜條件;(2)基于6種半蒴苣苔的染色體計數結果,結合已報道物種數據探討該屬染色體的變異情況;(3)結合分子系統學重建該屬染色體數目的進化歷史,探討其進化模式與物種進化的關系。本研究將對半蒴苣苔屬甚至具有類似葉片繁殖生物學特性類群的細胞學研究提供借鑒意義,為進一步研究該類群的分類、系統演化和物種形成等提供一些啟示。
1 材料與方法
1.1 材料
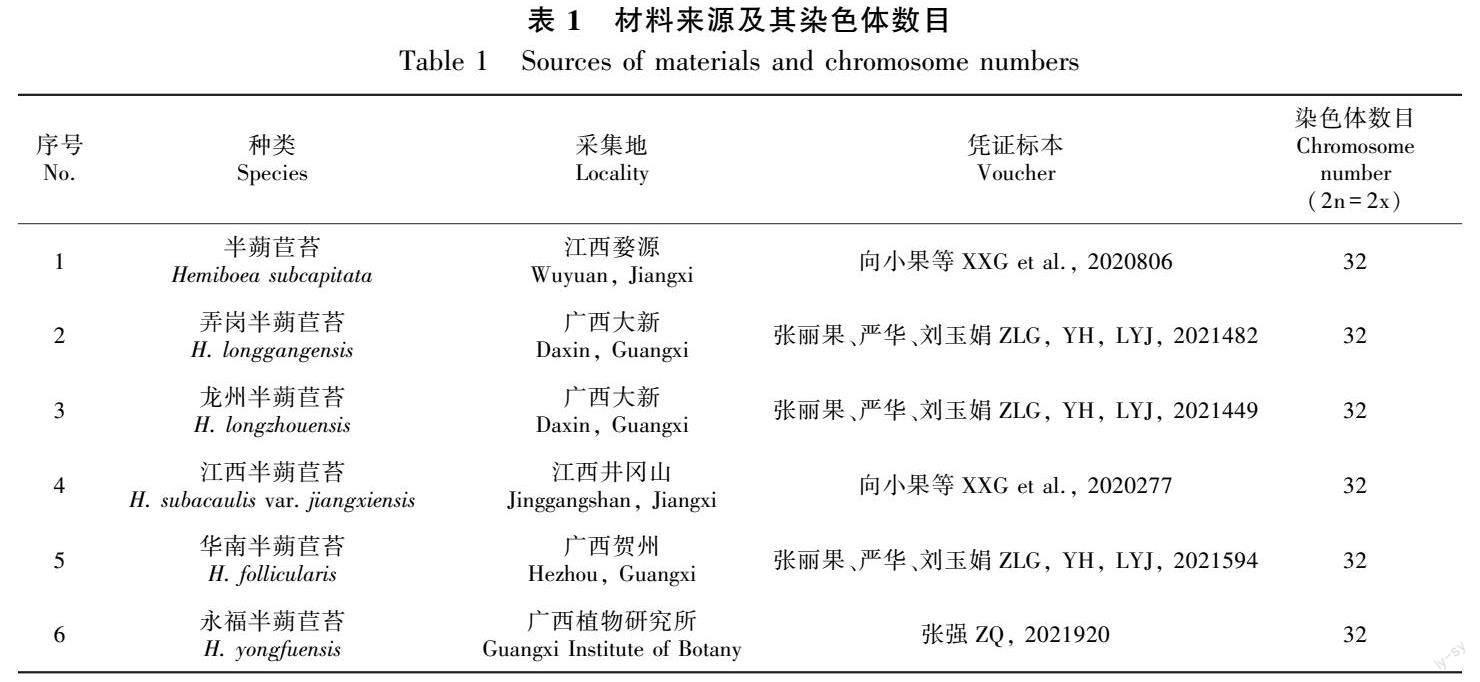
所涉及的研究材料來源如表1所示。現栽培于南昌大學流域生態研究所系統與進化研究室溫室內。本研究憑證標本存放于南昌大學標本館(JXU)中。
1.2 方法
1.2.1 半蒴苣苔屬植物染色體制片優化
1.2.1.1 取材 從6種半蒴苣苔屬植物上選取生長狀態良好的葉片,在室溫下用清水培養10~20 d,2~3 d換1次水,待葉片生根,選取生長良好的根尖進行取材,在9:30—10:00、10:00—10:30、10:30—11:00這3個時間段進行取樣。
1.2.1.2 預處理 將根尖浸入0.002 mol·L -1的8-羥基喹啉溶液中,以溶液浸沒根尖為度,在室溫下預處理4~5 h。
1.2.1.3 固定 先將處理后的根尖材料用純水沖洗2次,再轉入卡諾固定液[V(無水乙醇)∶V(冰醋酸)=3∶1]中,在4 ℃下固定30 min。
1.2.1.4 解離 先將固定后的根尖用無水乙醇洗2次,再用純水沖洗,之后轉入1 mol·L -1 HCl中,在60 ℃恒溫金屬浴鍋中分別解離8、10、12 min。
1.2.1.5 染色 將解離后的根尖用純水洗2次,每次5 min。接著,將根尖置于載玻片中間,用刀片從前端乳白色的分生區組織中切取盡可能薄的1片,滴加少量改良苯酚品紅染液,分別染色10、15 min。
1.2.1.6 壓片 對染色過后的材料進行常規壓片,壓片時用帶橡皮頭的鉛筆垂直敲打,注意不要滑動蓋玻片,這樣染色體分散效果更好。
1.2.1.7 鏡檢及染色體計數 將制作好的玻片在Leica DM2500顯微鏡下進行檢測,挑選染色體分散效果好的有絲分裂中期分裂相細胞,在100倍油鏡下進行觀察和拍照。染色體計數方法參照李懋學和陳瑞陽(1985)的植物核型分析標準。
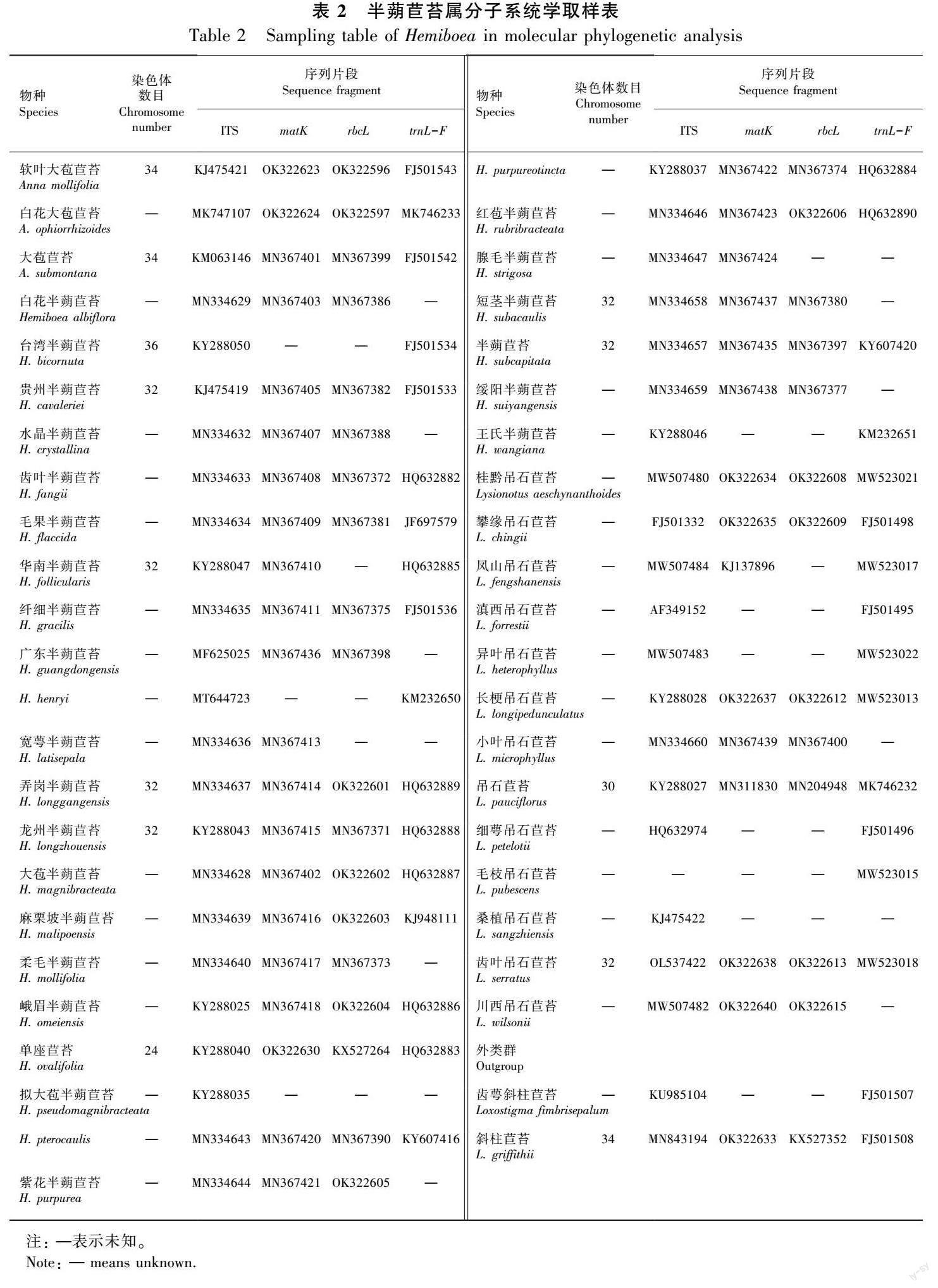
1.2.2 半蒴苣苔屬染色體數目祖先狀態重建 為探討半蒴苣苔屬植染色體數目的進化歷史,本研究以半蒴苣苔屬28種及其近緣的吊石苣苔屬(Lysionotus)13種和大苞苣苔屬(Anna)3種植物為內類群,以紫花苣苔屬 (Loxostigma)2種植物為外類群,基于下載自NCBI的核糖體ITS和3個葉綠體基因包括rbcL、matK和trnL-F(表2),使用MAFFT v7. 407(Katoh & Standley, 2013)分別進行矩陣排列,利用FASconCAT-G v1.04(Kück & Longo, 2014)將所有基因矩陣進行串聯,通過RAxML v. 8. 2. 12(Stamatakis, 2014)構建最大似然(maximum likelihood,ML)樹,堿基替代模型為GTRGAMMA,經過1 000次bootstraps重復抽樣估算支持率。
本研究通過WebCyte數據庫(http://elmer.rbge.org.uk/webcyte/webcyteintro.php)和CCDB 數據庫(Chromosome Counts Database,http://ccdb.tau.ac.il/browse/)對半蒴苣苔屬及其近緣的吊石苣苔屬、大苞苣苔屬物種的染色體數目數據進行收集。其中臺灣半蒴苣苔僅報道染色體二價體數目n=18,其二倍體染色體數目尚不清楚,此處暫以2n=36作為其染色體數目進行分析。對于尚未報道染色體數目的物種在性狀編碼時以缺失處理。在此基礎上,結合本研究的6種半蒴苣苔屬植物的染色體數據,基于上述系統發育樹,通過RASP v. 4. 2(Yu et al., 2020)軟件,采用Bayesian Binary MCMC (BBM)的方法進行染色體數目的祖先狀態重建,探究其可能的進化歷史。
2 結果與分析
2.1 半蒴苣苔屬植物染色體制片優化
2.1.1 取樣方法優化 6種半蒴苣苔屬植物如圖1所示。由圖2和圖3可知,半蒴苣苔、弄崗半蒴苣苔、龍州半蒴苣苔、江西半蒴苣苔、華南半蒴苣苔和永福半蒴苣苔6種植物通過葉片水培生根的方法得到了生長狀態良好的根,通過此方法得到的根比土生的根更粗壯、更易取材且染色體更易被壓散。
2.1.2 取樣時間優化 由表3和圖4可知,9:30—10: 00取材,根尖細胞分裂旺盛且染色體分散效果良好;10:00—10:30取材,染色體分散效果不如9:30—10:00;10:30—11:00取材,觀察到的分裂相不多且分裂效果不好。
2.1.3 解離時間優化 由表3和圖4可知,在解離時間為8 min時,觀察到的染色體不夠分散,出現粘連現象,計數時困難較大;在解離時間為10 min時, 染色體分散效果良好, 便于計數; 在解離時間為12 min時,有些染色體出現斷裂,影響計數的準確性。
2.1.4 染色時間優化 由表3和圖4可知,在染色時間為10 min時,染色體著色效果較差,不利于觀察;在染色時間為15 min時,染色體著色效果良好,觀察時很清晰;在染色時間為20 min時,染色體著色效果與15 min時差別不大,但由于時間過長,觀察時發現染液變干、有顆粒物的現象,影響了觀察視野和染色體計數。
2.2 半蒴苣苔屬植物染色體數目
由圖5可知,該6種半蒴苣苔屬植物的染色體數目均為2n=2x=32。
2.2.1 半蒴苣苔 本種分布于中國的華中、華南以及越南北部地區,生長在山坡溝邊、巖石山或林蔭下,海拔500~1 500 m。體細胞中期染色體數目為2n=32(圖5:A,B)。
2.2.2 弄崗半蒴苣苔 本種分布于廣西的龍州、大新、桂林、隆安等地,生長在石灰巖石山谷密林下陰處,海拔130~400 m。體細胞中期染色體數目為2n=32(圖5:C,D)。
2.2.3龍州半蒴苣苔 本種分布于廣西的龍州、桂林、寧明、隆安等地,生長于石灰巖石山山坡密林處,海拔170~800 m。體細胞中期染色體數目為2n=32(圖5:E,F)。A. 半蒴苣苔; B. 弄崗半蒴苣苔; C. 龍州半蒴苣苔; D. 江西半蒴苣苔; E. 華南半蒴苣苔; F. 永福半蒴苣苔。
2.2.4 江西半蒴苣苔 本種分布于江西的遂川、南康、井岡山、上猶、贛州等地,生長在山谷陰濕石面,海拔750~900 m。體細胞中期染色體數目為2n=32(圖5:G,H)。
2.2.5 華南半蒴苣苔 本種分布于廣東北部、廣西和貴州,生長在石灰巖山地林下陰濕石上或溝邊石縫中,海拔240~1 500 m。體細胞中期染色體數目為2n=32(圖5:I,J)。
2.2.6永福半蒴苣苔 本種分布于廣西桂林永福縣金鐘山,生長在喀斯特山石灰巖基質上。體細胞中期染色體數目為2n=32(圖5:K,L)。
2.3 半蒴苣苔屬染色體數目和倍性的進化
根據半蒴苣苔屬及其近緣的吊石苣苔屬(Lysionotus)、大苞苣苔屬(Anna)祖先狀態重建結果,3個近緣屬共有祖先的染色體數目可能是2n=34。其中大苞苣苔屬為2n=34,吊石苣苔屬可能從2n=34向2n=32,再向2n=30減少方向進化,而半蒴苣苔屬的染色體數目可能從祖先的2n=34到2n=24,32,36進化,其中2n=32出現多次。半蒴苣苔屬及其近緣屬均為二倍體,尚未見多倍化的現象。
3 討論與結論
3.1 半蒴苣苔屬植物染色體制片優化
半蒴苣苔屬植物由于根系十分細小且相互纏繞,因此難于從植株中直接獲取根尖。此外,該屬植物染色體小型,其根尖分生組織壓片后染色體不易分散且難以觀察到清晰的染色體形態,這也使得該屬細胞學研究較少。因此,本研究對該屬染色體制片的取材方法、取材時間、解離時間及染色時間等條件進行了優化。
實驗材料是實驗最重要的部分,取材越方便、根尖材料越健康,后續實驗的開展,成功的概率就越高。目前,已有一些植物使用過水培方法進行生根,如萱草、大蒜、洋蔥等,而在苦苣苔科里,僅在石山苣苔屬的研究中見有報道(田秋元和楊約田,2009;李國泰,2017;李永平等,2020;覃信梅等,2020)。本研究通過葉片水培生根的方法獲取了生長狀態良好的根尖材料,并且我們對水培生根方法進行了優化,只需要在室溫下,將半蒴苣苔屬植物健康葉片的葉柄插入清水中即可成功生根。這可能與苦苣苔科植物易于通過葉片扦插繁殖的生物學特性有關(李振宇和王印政,2005),本實驗的方法與以往的相比操作更加簡單方便。通過實驗我們還發現,在水培方式下長出的根比土生的根要更粗壯、染色體更易被壓散,良好的根尖材料為實驗奠定了堅實的基礎。
為獲得較多的有絲分裂中期分裂相細胞,就需要在植物有絲分裂旺盛的時期進行取樣。有研究表明,大多數植物在8:00—11:00取材,較容易獲得中期分裂相,但不同類群取樣的最佳時間段也會有所不同(韓毅科等,2003;楊寧等,2012;趙雁等,2019;覃信梅等,2020)。本研究中,6種半蒴苣苔屬植物最佳的取樣時間為9:30—10:00,這與大多數植物常規取材時間一致。
解離時間對最終染色體的制片效果影響非常大,解離時間過短,細胞壁很難破開,而時間過長則容易造成染色體斷裂或缺失。以往苦苣苔科植物染色體制片的解離時間大部分為30 s(王印政和顧志建,1999;魯元學等,2002;曹麗敏等,2003;季慧等,2008),此外,如報春苣苔為3 min、石山苣苔為3~4 min(劉瑞瑞等,2014;覃信梅等,2020)。在本研究中,半蒴苣苔屬植物解離時間為10 min時染色體分散效果最佳,這與具有體細胞小、染色體數目多的金盞菊最佳解離時間(9~10 min)一致(龍海海等,2020),而與本屬的貴州半蒴苣苔(魯元學等,2002)和單座苣苔(曹麗敏等,2003)2種解離時間(30 s)不同,但本研究的分散效果更佳。
以往的半蒴苣苔屬植物染色體制片對染色時間的描述較少(魯元學等,2002;曹麗敏等,2003),但染色時間對于染色體制片效果也產生較大影響。染色時間過短,染色體的著色淺,難以觀察;染色時間過長,染色劑容易出現顆粒物沉淀粘在玻片上,難以計數。根據本研究結果發現,該6種半蒴苣苔屬植物染色時間為15 min時,染色體著色效果最好,這與金盞菊染色8~12 min的效果最好比較接近(龍海海等,2020)。
綜上所述,不同類群染色體制片的方法可能不同,需要結合類群本身特性并多次嘗試才可能得到最佳的染色體制片方案。本研究主要通過葉片水培生根法對半蒴苣苔屬6種植物進行取材,此法有效地解決了半蒴苣苔屬植物染色體制片中取材困難的問題。在此基礎上,通過優化取樣、解離、染色時間等條件進行染色體制片,確定了該屬6種植物的染色體數目,這為半蒴苣苔屬、苦苣苔科甚至具有類似生物學特性類群的染色體制片提供了借鑒。
3.2 半蒴苣苔屬染色體數目和倍性
在半蒴苣苔屬中,臺灣半蒴苣苔與本研究中的弄崗半蒴苣苔和龍州半蒴苣苔所在支系聚為一支,該物種最早基于花芽進行染色體研究,表明其染色體二價體數目為n=18(Hsu, 1968)。該物種染色體數目可能為2n=2x=36,確切數目尚不清楚,需要進一步研究,后2種均為2n=2x=32。Weber 等(2011)根據分子系統學的研究,將單座苣苔屬(Metabriggsia)包括單座苣苔(H. ovalifolia)和紫葉單座苣苔(H. purpureotincta)合并到半蒴苣苔屬,這與本研究中的分子系統學結果一致,根據我們的研究,其中位于半蒴苣苔屬基部的單座苣苔是該屬已知染色體數目最少的物種(2n=2x=24)(曹麗敏等,2003),另外的紫葉單座苣苔與單座苣苔關系較遠且與毛果半蒴苣苔(H. flaccida)互為姐妹,其染色體數目尚未見報道。貴州半蒴苣苔(H. cavaleriei)(魯元學等,2002)與本研究中分布于各個支系的半蒴苣苔(H. subcapitata)、弄崗半蒴苣苔(H. longgangensis)、龍州半蒴苣苔(H. longzhouensis)、江西半蒴苣苔(H. subacaulis var. jiangxiensis)、華南半蒴苣苔(H. follicularis)和永福半蒴苣苔(H. yongfuensis)6種植物的染色體數目和倍性一致,均為二倍體(2n=2x=32)。
綜上所述,目前半蒴苣苔屬染色體數目和倍性的變異范圍與楊麗華等(2019)的研究結果一致,為2n=2x=24, 32,其中n=18的臺灣半蒴苣苔染色體數目可能需要進一步確認。此外,基于系統發育和染色體數目結合的研究表明,該屬染色體數目變化與物種進化并沒有明顯的變化趨勢,除個別物種染色體數目有變異外,該屬物種染色體數目相對穩定且大部分可能是2n=2x=32。由于該屬已有物種的染色體研究較少,確切的染色體變異范圍還有待于對其余物種進行進一步的細胞學研究。
3.3 半蒴苣苔屬染色體數目的進化模式
在植物進化過程中,染色體數目非整倍化非常普遍且對植物進化和物種形成具有重要意義(De Storme & Mason, 2014)。這種非整倍化往往由染色體裂變或融合導致染色體數目非整倍性的增加或減少,但這種染色體數目的非整倍性變化不涉及遺傳信息的增加或丟失(De Storme & Mason, 2014)。本研究結果表明,半蒴苣苔屬及其近緣的吊石苣苔屬和大苞苣苔屬的共同祖先可能是由2n=34進化而來,其中大苞苣苔屬保持祖先染色體數目狀態(2n=34),吊石苣苔屬可能是由2n=34向2n=32、2n=30逐漸減少。半蒴苣苔屬可能由2n=34向2n=24、2n=32、2n=36變化,因為該屬部分支系支持率不高,染色體變化趨勢并不明顯,其中2n=32多次平行出現。半蒴苣苔屬及其近緣的吊石苣苔屬染色體數目的變化可能與非整倍化進化有關,這與苦苣苔科許多類群如漢克苣苔屬和長蒴苣苔屬等染色體非整倍化進化模式類似(楊麗華等,2019)。目前,由于半蒴苣苔屬及其近緣屬染色體的研究較少且尚未見高分辨率的種間系統發育關系,需要進一步開展更多物種的細胞學與分子系統學結合的研究才能進一步揭示其染色體的進化模式。
參考文獻:
CAO LM, CAO M, TANG XL, et al., 2003. Chromosome numbers of 4 species in the Gesneriaceae from Guangxi [J]. Guihaia, 23(4): 331-333. [曹麗敏, 曹明, 唐咸來, 等, 2003. 廣西苦苣苔科四種植物的染色體數目報道 [J]. 廣西植物, 23(4): 331-333.]
DE STORME N, MASON A, 2014. Plant speciation through chromosome instability and ploidy change: Cellular mechanisms, molecular factors and evolutionary relevance [J]. Curr Plant Biol, 1: 10-33.
HAN YK, DU SL, WANG M, 2003. Study on chromosome preparing and ploidy in cucumber [J]. Acta Agric Boreal-Sin, 18(1): 72-74. [韓毅科, 杜勝利, 王鳴, 2003. 黃瓜染色體制片及倍性研究 [J]. 華北農學報, 18(1): 72-74.]
HONG DY, 1990. Plant cell taxonomy [M]. Beijing: Science Publishing House: ⅰ-ⅱ. [洪德元, 1990. 植物細胞分類學 [M]. 北京: 科學出版社: ⅰ-ⅱ.]
HSU CC, 1968. Preliminary chromosome studies on the vascular plants of Taiwan (Ⅱ) [J]. Taiwania, 14(1): 11-27.
JI H, GUAN KY, LU YX, 2008. Chromosome numbers of eight species in the genus Petrocosmea (Gesneriaceae) [J]. Acta Bot Yunnan, 30(3): 321-324. [季慧, 管開云, 魯元學, 2008. 石蝴蝶屬八種植物的染色體數目報道 [J]. 云南植物研究, 30(3): 321-324.]
KATOH K, STANDLEY DM, 2013. MAFFT multiple sequence alignment software version 7: improvements in performance and usability [J]. Mol Biol Evol, 30(4): 772-780.
KCK P, LONGO GC, 2014. FASconCAT-G: extensive functions for multiple sequence alignment preparations concerning phylogenetic studies [J]. Front Zool, 11(1): 81.
LI GT, 2017. The Liliaceae four plant chromosome karyotype analysis [J]. For By-Prod Spec Chin, 6(5): 21-25. [李國泰, 2017. 百合科4種植物染色體的核型比較 [J]. 中國林副特產, 6(5): 21-25.]
LI JH, HUANG ZP, LU YB, et al., 2020. Diversity, geographical distribution and species boundary of the Hemiboea subcapitata complex [J]. Guihaia, 40(10): 1477-1490. [李佳慧, 黃章平, 盧永彬, 等, 2020. 半蒴苣苔復合群的多樣性、地理分布和物種界限 [J]. 廣西植物, 40(10): 1477-1490.]
LI MX, CHEN RY, 1985. A suggestion on the standardization of karyotype analysis in plants [J]. J Wuhan Bot Res, 3(4): 297-302. [李懋學, 陳瑞陽, 1985. 關于植物核型分析的標準化問題 [J]. 武漢植物學研究, 3(4): 297-302.]
LIU RR, 2013. Cytological characteristics and systematic significance in Primulina sensu lato (Gesneriaceae) [D]. Beijing: University of Chinese Academy of Sciences. [劉瑞瑞, 2013. 廣義報春苣苔屬(苦苣苔科)的細胞學特征及其系統學意義 [D]. 北京: 中國科學院大學.]
LIU RR, SKOG LE, LIAO JP, et al., 2014. New chromosome counts and their taxonomic implications in Primulina sensu lato (Gesneriaceae) [J]. Plant Divers Resour, 36(1): 13-21. [劉瑞瑞, SKOG LE, 廖景平, 等, 2014. 廣義報春苣苔屬(苦苣苔科)的染色體新計數及其分類學意義 [J]. 植物分類與資源學報, 36(1): 13-21.]
LI YP, JIA ML, LIANG Z, et al., 2020. Karyotype analysis of three species of Hemerocallis [J]. J Shanxi Agric Sci, 48(1): 32-34. [李永平, 賈民隆, 梁崢, 等, 2020. 3種萱草屬植物染色體核型分析 [J]. 山西農業科學, 48(1): 32-34.]
LI ZY, WANG YZ, 2005. Plants of Gesneriaceae in China [M]. Zhengzhou: Henan Science and Technology Publishing House: 123, 576, 653-654. [李振宇, 王印政, 2005. 中國苦苣苔科植物 [M]. 鄭州: 河南科學技術出版社: 123, 576, 653-654.]
LONG HM, ZHANG NQ, LI ZY, et al., 2020. Study on chromosome technique of Calendula officinalis and its karyotype [J]. Acta Agric Zhejiang, 32(1): 86-92. [龍海梅, 張楠卿, 李宗艷, 等, 2020. 金盞菊根尖細胞染色體制片與核型分析 [J]. 浙江農業學報, 32(1): 86-92.]
LU YX, SUN XF, ZHOU QX, et al., 2002. Chromosome numbers in ten species in the Gesneriaceae from Yunnan [J]. Acta Bot Yunnan, 24(3): 377-382. [魯元學, 孫先鳳, 周其興, 等, 2002. 云南十種苦苣苔科植物的染色體數目報道 [J]. 云南植物研究, 24(3): 377-382.]
MLLER M, KIEHN M, 2004. A synopsis of cytological studies in Gesneriaceae [J]. Edin J Bot, 60(3): 425-447.
QIN XM, PAN B, LU YB, et al., 2020. Chromosome numbers and ploidy of four species (including one variety) in Petrocodon Hance [J]. Guihaia, 40(10): 1466-1476. [覃信梅, 盤波, 盧永彬, 等, 2020. 石山苣苔屬四種(含一變種)植物的染色體數目和倍性研究 [J]. 廣西植物, 40(10): 1466-1476.]
RATTER JA, 1963. Some chromosome numbers in the Gesneriaceae [J]. Notes Roy Bot Gard Edin, 24(1): 221-229.
RATTER JA, PPENTICE HT, 1964. Chromosome numbers in the Gesneriaceae: Ⅱ [J]. Notes Roy Bot Gard Edin, 25(3): 303-307.
RATTER JA, PPENTICE HT, 1967. Chromosome numbers in the Gesneriaceae: Ⅲ [J]. Notes Roy Bot Gard Edin, 27(2): 205-209.
RATTER JA, PPENTICE HT, 1970. Chromosome numbers in the Gesneriaceae: Ⅳ [J]. Notes Roy Bot Gard Edin, 30(1): 183-187.
SOLTIS DE, SOLTIS PS, 1999. Polyploidy: recurrent formation and genome evolution [J]. Trends Ecol Evol, 14(9): 348-352.
STAMATAKIS A, 2014. RAxML version 8: a tool for phylogenetic analysis and post-analysis of large phylogenies [J]. Bioinformatics, 30(9): 1312-1313.
TIAN QY, YANG YT, 2009. Karyotype analysis on onion and discussion on its related dialysis methods [J]. J Anhui Agric Sci, 37(25): 12341-12343. [田秋元, 楊約田, 2009. 洋蔥核型分析及有關制片方法的探討 [J]. 安徽農業科學, 37(25): 12341-12343.]
WANG YZ, GU ZJ, 1999. Karyomorphology of four species in Ancylostemon, Briggsiopsis and Lysionotus (Gesneriaceae) [J]. J Syst Evol, 37(2): 137-142. [王印政, 顧志建, 1999. 直瓣苣苔屬、筒花苣苔屬和吊石苣苔屬4個種的核形態學研究 [J]. 植物分類學報, 37(2): 137-142.]
WEBER A, WEI YG, SONTAG S, et al., 2011. Inclusion of Metabriggsia into Hemiboea (Gesneriaceae) [J]. Phytotaxa, 23(1): 37-48.
YANG LH, FENG C, XU MZ, et al., 2019. Synopsis of cytological studies on Didymocarpoideae (Gesneriaceae) under new classification system [J]. J Trop Subtrop Bot, 27(5): 548-557. [楊麗華, 馮晨, 徐梅珍, 等, 2019. 新分類系統下長蒴苣苔亞科 (苦苣苔科) 細胞學研究概述 [J]. 熱帶亞熱帶植物學報, 27(5): 548-557.]
YANG N, TAN YX, LI QX, et al., 2012. Optimization of a chromosome mounting technique and karyotype analysis of Thymus mongolicus [J]. Acta Pratac Sin, 21(1): 184-189. [楊寧, 談永霞, 李巧峽, 等, 2012. 百里香染色體制片優化及核型分析 [J]. 草業學報, 21(1): 184-189.]
YU Y, BLAIR C, HE XJ, 2020. RASP 4: ancestral state reconstruction tool for multiple genes and characters [J]. Mol Biol Evol, 37(2): 604-606.
ZHAO DK, LU YX, SHI JF, et al., 2010. Seed germination and chromosome numbers of Lysionotus pacuciforus and Briggsia longgipes [J]. J Yunnan Agric Univ (Nat Sci Ed), 25(2): 173-177. [趙大克, 魯元學, 石景峰, 等, 2010. 吊石苣苔和盾葉粗筒苣苔的種子萌發及染色體數目觀察 [J]. 云南農業大學學報(自然科學版), 25(2): 173-177.]
ZHAO Y, DU KH, LI WX, et al., 2019. Chromosome preparation optimization and karyotype analysis of two landscape plants in Araliaceae [J]. N Hortic, 22(1): 83-89. [趙雁, 杜康華, 李宛宣, 等, 2019. 兩種五加科園林植物染色體制片優化與核型分析 [J]. 北方園藝, 22(1): 83-89.]
(責任編輯 鄧斯麗 李 莉)

