基于單倍型肉牛屠宰性狀全基因組關聯分析研究
李宏偉,徐凌洋,王澤昭,蔡文濤,朱 波,陳 燕,高 雪,張路培,高會江,李俊雅
(中國農業科學院北京畜牧獸醫研究所 牛遺傳育種創新團隊,北京 100193)
隨著高通量測序技術的發展和大量SNP標記的出現,全基因組關聯分析逐漸成為定位復雜性狀遺傳變異或基因的常用方法。該方法于1996年由Risch和Merikangas[1]首次提出。近二十年來,科學家們在人類、牲畜和作物中開展了大量基于連鎖不平衡的GWAS研究工作[2-9]。GWAS研究有兩個具體目標,一是要確定與目標性狀關聯的QTLs或者基因,其次是探究復雜性狀的遺傳結構[10]。全基因組關聯分析開始廣泛應用更高密度的芯片及全基因組測序數據開展基因定位研究,大大提高了GWAS的檢測效率。伴隨GWAS研究的深入,GWAS新的統計模型和算法不斷發展,用以提高位點定位的精準性。2006年,Yu等[11]首次提出基于固定效應和隨機效應的混合模型。隨后,針對GWAS研究中的計算速度和假陽性等問題陸續提出了優化模型和方法。這些方法主要分為兩類,一類是精確算法,另一類是近似算法。精確算法如GEMMA和fast-LMM等針對每個標記重新擬合模型,并準確計算每個標記的效應值,然而,此類方法需要較長的計算時間[12-13]。相比之下,近似算法如EMMAX和GRAMMAR的計算速度非常快。單倍型是指在同一染色體上進行共同遺傳的多個基因座上等位基因的組合。多個等位基因之間存在一定的連鎖不平衡關系。單倍型標記與數量性狀基因座之間具有較強的LD關系,在基因定位、因果突變鑒定及QTL連鎖分析方面具有較高的統計效力。然而,目前大部分畜禽基因組研究工作都是基于SNP標記,利用單倍型標記的研究較少。有研究表明,基于單倍型的全基因組關聯分析能夠檢測到基于SNP檢測不到的位點[14-15]。單倍型可以捕獲SNP之間的局部上位性效應,因此可以提高解析復雜性狀的能力和準確性[16-17]。在人類的相關研究中已經揭示了利用單倍型開展GWAS研究的諸多優點:首先,與SNP標記相比,單倍型標記提供了更多的多態性信息[18];其次,單倍型標記可以用來判斷兩個等位基因在血統上的一致性[19];另外,利用單倍型標記可以捕獲更多的物種進化信息[20]。目前,單倍型已日益成為全基因組關聯分析中重要的遺傳標記[21-25]。
本研究基于中國農業科學院北京畜牧獸醫研究所構建的華西牛資源群體,分別利用SNP和單倍型標記對宰前活重(LW)和屠宰率(DP)開展全基因組關聯分析,定位目標性狀的分子標記及候選基因。
1 材料與方法
1.1 試驗群體
本試驗的研究群體為中國農業科學院北京畜牧獸醫研究所牛遺傳育種團隊構建的華西牛資源群體,選取該群體于2008—2021年間屠宰的共計1 478個個體進行研究,其中公牛1 333頭,母牛145頭。所有個體的表型數據從出生時開始收集整理,包括各個生長發育的階段的表型數據,犢牛斷奶后,公牛犢轉移至指定的肉牛育肥場進行育肥,按照相同的飼養方式進行統一管理,并于24月齡左右進行屠宰,按照國家標準GB/T 27643-2011進行屠宰、胴體、分割等性狀的測定和收集。
1.2 試驗方法
1.2.1 表型性狀測定及數據預處理 在1 478頭華西牛群體中,公牛1 333頭,母牛145頭,選擇宰前活重(LW)和屠宰率(DP)性狀進行研究。具體的測定計算方法:宰前活重為肉牛屠宰前禁水禁食12 h以上的體重;屠宰率是肉牛胴體重與宰前活重的比值。
首先對原始數據進行預處理,剔除各個性狀平均值加減3倍標準差之外的異常數據,然后對各性狀進行描述性統計分析,并進行表型校正。本研究根據資源群體數據的實際情況,考慮的固定效應為性別、出生年、育肥場、屠宰場和所在群體等,協變量包含育肥天數、進場重和屠宰日齡等。利用R語言中的GLM函數對影響原始表型的各個因素進行顯著性檢驗和校正,其模型如下:
y=u+Sex+Year+F1+F2+Pop+day1+enter_wt+day2+e
其中,y為表型值,u為總體均值,Sex為性別效應,Year為出生年效應,F1為育肥場效應,F2為屠宰場效應,Pop為所在群體效應,day1為育肥天數效應,enter_wt為進場重效應,day2為屠宰日齡效應,e為隨機殘差。
1.2.2 遺傳參數估計 本研究利用ASReml (v4.1)通過個體動物模型對目標性狀的遺傳力進行了估計[26]。具體模型如下:
y=1nμ+Za+e
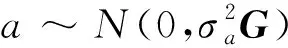
1.2.3 基因型數據處理 利用PLINK軟件(v1.07)[27]對1 478個個體的770K高密度芯片數據進行質量控制,質控后利用BEAGLE(v5.1)[28]對芯片數據中缺失位點進行填充,具體質控方法:首先,去除性染色體和質粒上的SNPs位點,僅保留常染色體上的位點。剔除染色體位置信息缺失的SNPs位點;剔除最小等位基因頻率小于0.01的SNPs位點;剔除缺失率大于0.05的SNPs位點;剔除哈代-溫伯格平衡檢驗P<10-6的SNPs位點;剔除樣本SNP缺失率大于0.05的個體。
1.2.4 基因型定相及單倍型構建 本研究利用BEAGLE (v5.1)軟件的默認參數對質控后的SNP數據進行定相(phase)。分別采用基于LD水平和基于連續固定的SNP個數的方式進行單倍型的構建,具體過程如下。
1.2.4.1 基于LD單倍型構建:利用相鄰兩個SNPs之間的連鎖不平衡程度(LD)進行單倍型的劃分。若某一區域彼此相鄰兩個SNPs之間的LD大于某一個閾值(r2),則將這部分連續SNPs劃分為一個單倍型塊。這種方法確保在物理距離盡可能近的情況下,具有一定LD關系的SNPs可以劃分到一個單倍型區域中。r2的計算公式如下:
其中,D=pA1B1pA2B2-pA1B2pA2B1,D是兩個雙等位基因之間的連鎖不平衡程度,pA1、pA2、pB1和pB2是4個等位基因(A1、A2、B1和B2)的頻率,pA1B1、pA2B2、pA1B2和pA2B1是4個基因型頻率(A1B1、A2B2、A1B2和A2B1)[29-30]。本研究選擇LD閾值水平為r2>0.3進行單倍型(0.3LD)的構建。
1.2.4.2 基于固定SNP個數進行單倍型構建:本研究將整個基因組按常染色體進行拆分(共29條),在每條染色體上,依據SNP的排列順序,設置每5個連續的SNPs劃分為一個單倍型塊(5_SNP_HAP)。若染色體末端SNP個數不足5個,將染色體末端最后剩余的所有SNP作為一個單倍型塊。在單倍型塊內部按照劑量可加效應,依據單倍型等位基因的重復個數進行編碼(0、1、2)。
為了排除低頻單倍型等位基因對GWAS結果的影響,對單倍型等位基因進行質量控制,去除單倍型等位基因頻率小于0.01的單倍型等位基因。
1.2.5 單倍型效應編碼 在劃分單倍型塊的基礎上構建單倍型矩陣。具體編碼規則是利用劑量可加效應,依據單倍型等位基因重復個數的方法進行編碼[31],兩個連續SNPs構建一個單倍型塊的劑量可加編碼規則如表1所示。
1.2.6 群體遺傳結構分析 在全基因組關聯分析時,為了減少假陽性位點的出現,在分析之前需要對群體分層進行校正。本研究利用高密度SNP芯片數據對華西牛群體做PCA分析,判斷群體中是否存在群體分層現象。
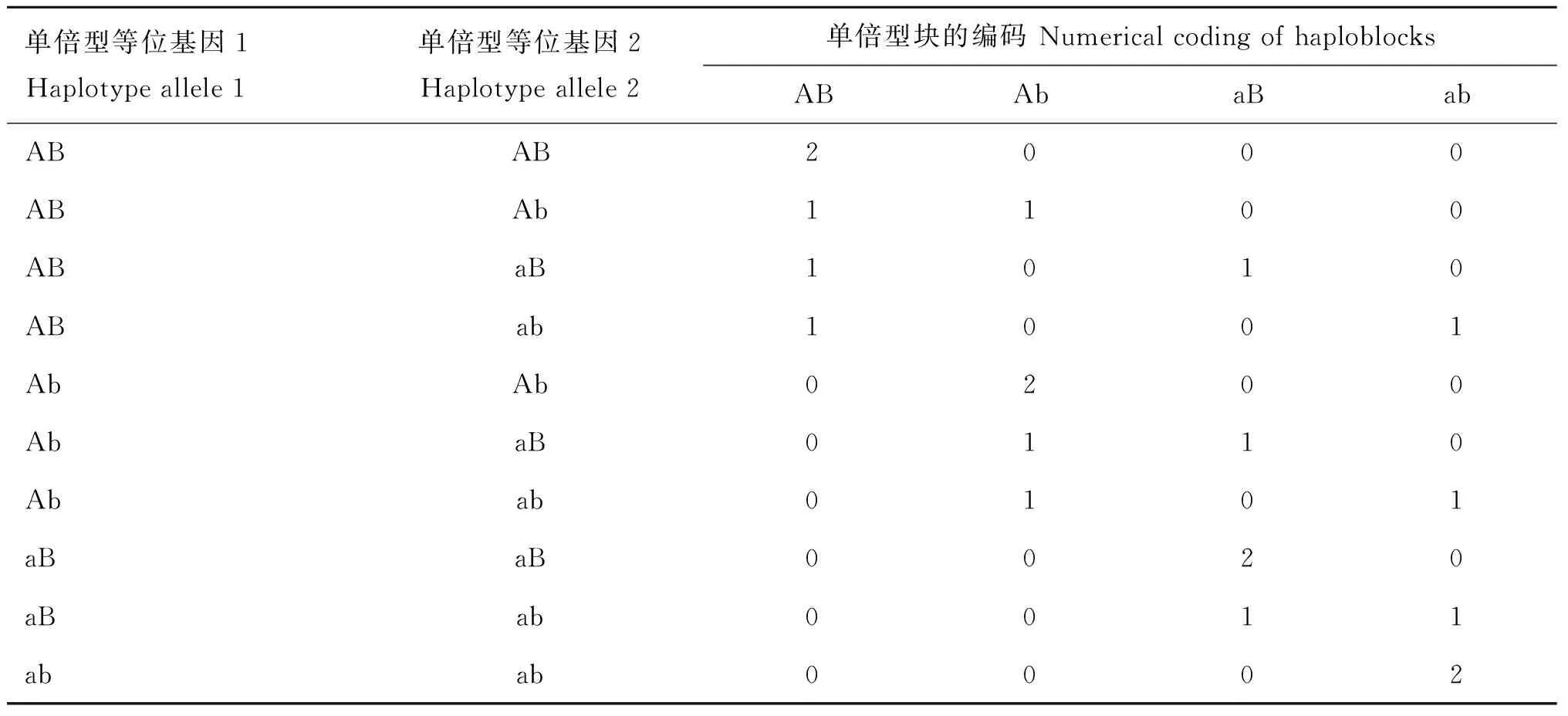
表1 兩個連續SNPs構建一個單倍型塊的劑量可加編碼
1.2.7 全基因組關聯分析
1.2.7.1 基于SNP的全基因組關聯分析:利用GCTA軟件(v1.93)[32]中的混合線性模型(mixed linear model,MLM)對目標性狀進行GWAS研究。計算模型如下:
y=Xβ+Ws+Zμ+e
其中,y是表型值向量;X是固定效應向量的關聯矩陣,β是固定效應向量;W是SNP基因型指示變量,aa、Aa和AA三種不同基因型分別用0、1、2數字編碼,s為SNP效應向量;Z為多基因效應向量的設計矩陣,μ~(0,Kσ2)是微效多基因效應向量,K為基因組親緣關系矩陣;e~N(0,Iσ2)表示隨機殘差。
1.2.7.2 基于單倍型的全基因組關聯分析:利用連續5個SNPs構建的單倍型的全基因組關聯分析模型如下:
y=Xβ+W′s′+Z′μ′+e
其中y是表型值向量;X為固定效應向量的關聯矩陣,β為固定效應向量;W′是單倍型的指示變量,依據單倍型等位的重復個數(0、1、2)進行編碼,s′為單倍型等位基因效應向量;Z′為多基因效應向量的設計矩陣,μ′~(0,K′σ2)為微效多基因效應向量,K′為單倍型等位基因計算的基因組親緣關系矩陣;e~N(0,Iσ2)表示隨機殘差。
由于基于LD進行單倍型構建時將整個基因組劃分為兩部分,一部分為具有LD的區域,利用單倍型進行重編碼,剩余的位于單倍型塊外部的SNP依舊利用原來的方式進行編碼。因此,利用LD單倍型全基因組關聯分析的模型如下:
y=Xβ+W″s″+Z″μ″+e
其中,y是表型值向量;X為固定效應向量的關聯矩陣,β為固定效應向量;W″是單倍型和SNP合集的指示變量,其中單倍型部分依據單倍型等位的重復個數(0、1、2)進行編碼,s″為SNP與單倍型等位基因效應向量;Z″為多基因效應向量的關聯矩陣,μ″~(0,K″σ2)為微效多基因效應向量,K″為單倍型等位基因和單倍型外部SNP計算的基因組親緣關系矩陣;e~N(0,Iσ2)表示隨機殘差。
采用wald檢驗法對得到的SNP或單倍型等位基因進行顯著性檢驗,利用bonferroni多重檢驗將SNP標記的顯著水平閾值設置為1/SNP數[33],單倍型等位基因顯著性水平閾值設置為1/單倍型等位基因數。
1.2.8 候選基因篩選 根據設定的顯著性閾值篩選與目標性狀顯著關聯的SNPs及單倍型塊,并通過生物數據庫ensembl中的BioMart模塊將篩選到的顯著SNPs與牛的參考基因組(Bos_taurus.UMD3.1)進行比對。然后利用SNPs或單倍型塊的物理位置尋找距離SNP最近的基因。且認為該基因是與目標性狀顯著相關的候選基因,利用NCBI網站的Gene數據庫檢索相關候選基因的生物學功能。
2 結 果
2.1 芯片數據質控與表型基本統計量
經過基因型數據質控及表型預處理后宰前活重和屠宰率性狀分別剩余1 387及1 366個個體,共672 060個SNPs位點的基因型數據用于下一步分析。各性狀表型的基本統計量,包括表型數據的記錄數、平均值及標準差、最大值、最小值和變異系數如表2所示。圖1展示了性狀的表型頻率分布情況。研究結果表明表型數據基本服從正態分布,可用于下一步研究。

表2 性狀基本統計量
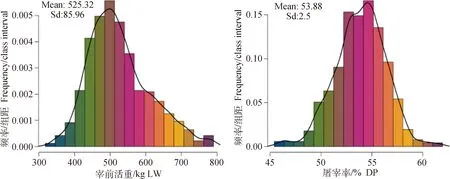
圖1 宰前活重和屠宰率性狀的表型頻率分布圖Fig.1 Phenotypic frequency distribution of LW and DP
2.2 固定效應及協變量顯著性
對性別、出生年、育肥天數、進場重、育肥場、屠宰場、所在群體和屠宰日齡等固定效應和協變量進行顯著性檢驗,使用GLM函數對各性狀進行表型校正,檢驗結果如表3所示。
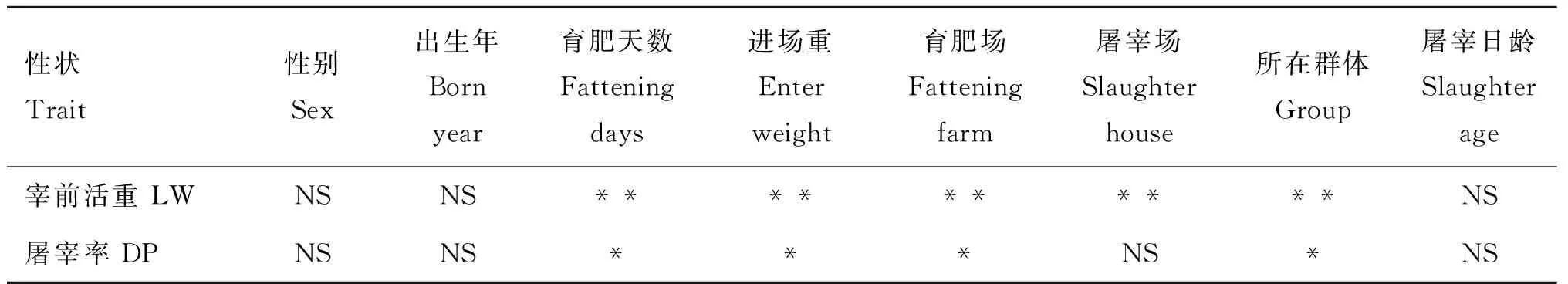
表3 各固定效應及協變量顯著性結果
結果顯示,各個性狀中,性別、出生年和屠宰日齡對宰前活重和屠宰率的影響不顯著(P>0.05),育肥天數、進場重、育肥場、屠宰場和所在群體對宰前活重的影響極顯著(P<0.01),育肥天數、進場重、育肥場和所在群體對屠宰率影響顯著(P<0.05)。
2.3 表型遺傳參數估計
利用ASReml(v3.0)軟件[34]中的個體動物模型對各性狀的遺傳參數進行估計。各性狀加性遺傳方差、殘差方差和遺傳力如表4所示。宰前活重和屠宰率的遺傳力分別為0.40和0.22。其中,宰前活重為高遺傳力性狀,屠宰率為中等遺傳力性狀。
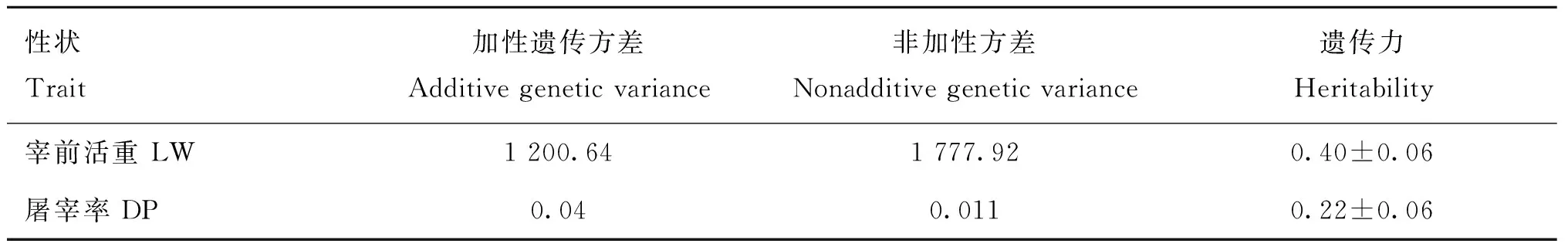
表4 性狀遺傳參數估計
2.4 群體遺傳結構
以第一、二主成分聚類的群體結構如圖2所示。群體中的個體大致聚成5類,表明群體中存在一定程度的群體分層現象。本研究根據前期研究的經驗,選取前5個主成分作為協變量放入GWAS分析模型以校正群體分層效應對關聯分析的影響。
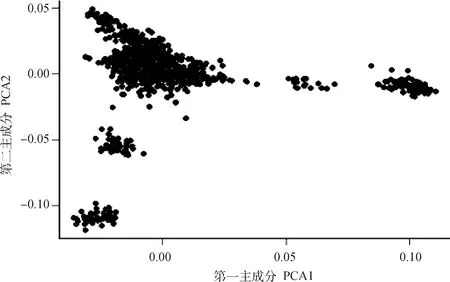
圖2 主成分分析繪制種群結構圖Fig.2 Population structure identified by principal components analysis
2.5 全基因組關聯分析結果
圖3和圖4分別展示了基于SNP、0.3LD單倍型和5_SNP_HAP單倍型標記對宰前活重和屠宰率等性狀的GWAS分析的Manhattan和QQ圖。QQ圖結果顯示,不論SNP標記還是單倍型標記,SNP顯著性分布的理論值與觀察值的基本吻合,表明本研究選取的固定效應和協變量較好的校正了群體結構和其他混雜因素,模型選擇合理。以bonferroni法矯正后的顯著閾值為標準,兩個性狀在全基因組范圍內共找到16個顯著SNPs及單倍型區域,利用ensembl網站BioMart模塊成功鑒定到與這些SNPs或單倍型區域密切相關的10個候選基因。
對于宰前活重,利用SNP和單倍型方法共檢測到15個顯著SNPs及單倍型區域(表5),分別位于第1、5、6、14、16和17號染色體上,分別鄰近或坐落于PPM1K、FAM184B、LCORL、PMP2、R1MS2、PEX14、EPHB3、MPST、RIMS2、SCOC、S100A10、ANGPTL4、FER1L6、FABP4和AARSD1等,其中,3種標記共同鑒定到FAM184B基因,SNP標記和0.3LD單倍型標記共同鑒定到PPM1K。5_SNP_HAP和0.3LD標記共同鑒定到R1MS2基因,0.3LD單倍型標記除了鑒定到SNP標記中的所有候選基因外,還鑒定到PEX14、EPHB3和MPST等基因。本研究表明單倍型方法,尤其是0.3LD單倍型標記鑒定到的基因遠遠多于SNP方法,且單倍型方法鑒定到的顯著性位點或區域大多位于基因內部。
對于屠宰率,共檢測到位于第1、14和28號染色體上4個顯著SNPs位點(表6),它們鄰近或坐落于EPHB3、RIMS2和ANTXRL等基因,其中,SNP和0.3LD標記共同鑒定到EPHB3基因。且所有被鑒定到的顯著位點均位于基因的內部。
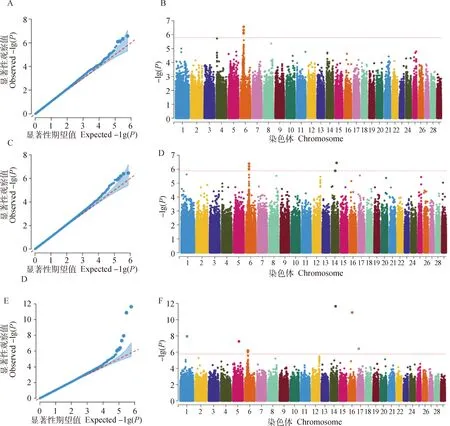
A和B表示利用SNP標記,C和D表示利用5_SNP_HAP單倍型標記,E和F表示利用0.3LD單倍型標記A and B represent SNP marker, C and D represent 5_SNP_HAP haplotype marker, E and F represent 0.3LD haplotype marker 圖3 基于SNP、0.3LD單倍型和5_SNP_HAP單倍型標記對宰前活重性狀關聯分析結果的Manhattan和QQ圖Fig.3 Manhattan and QQ plots for the GWAS results of LW by SNP, 0.3LD and 5_SNP_HAP marker
3 討 論
在肉牛生產中,屠宰性狀是備受關注的經濟性狀,在肉牛產業中發揮著著重要的作用。本研究通過GWAS策略鑒定影響華西牛屠宰性狀的分子標記或候選基因,有助于從遺傳水平上探索肉牛生長發育的遺傳機制,對該品種遺傳改良具有重要意義,為后續全基因組選擇、分子標記輔助選擇等提供可靠的理論基礎。本研究基于本團隊組建的華西牛資源群體的基因型及表型數據,通過SNP和單倍型標記分別對宰前活重(LW)和屠宰率(DP)等重要經濟性狀進行了GWAS研究。
通過對群體分層效應進行校正,可有效減少由于遺傳背景不同導致的假陽性位點的出現。在做GWAS分析時,選取放入到模型中PCA個數的依據主要有兩種,一種是根據經驗,選取一定數量的PCA放入模型中[35-38];另外還有根據EIGENSTRAT軟件計算各個主成分是否有顯著的統計學意義,將P值小于0.05的主成分納入群體分層校正中[39]。本研究的GWAS結果如圖2和圖3所示,整體上看,利用單倍型方法進行GWAS鑒定到的基因遠遠多于SNP方法,且所有SNP方法能夠鑒定到的顯著性位點,利用單倍型方法也可以鑒定到。在一定程度上說明,利用單倍型進行GWAS可以增加顯著性位點的檢測能力。這與前人的研究結果一致[14-15]。
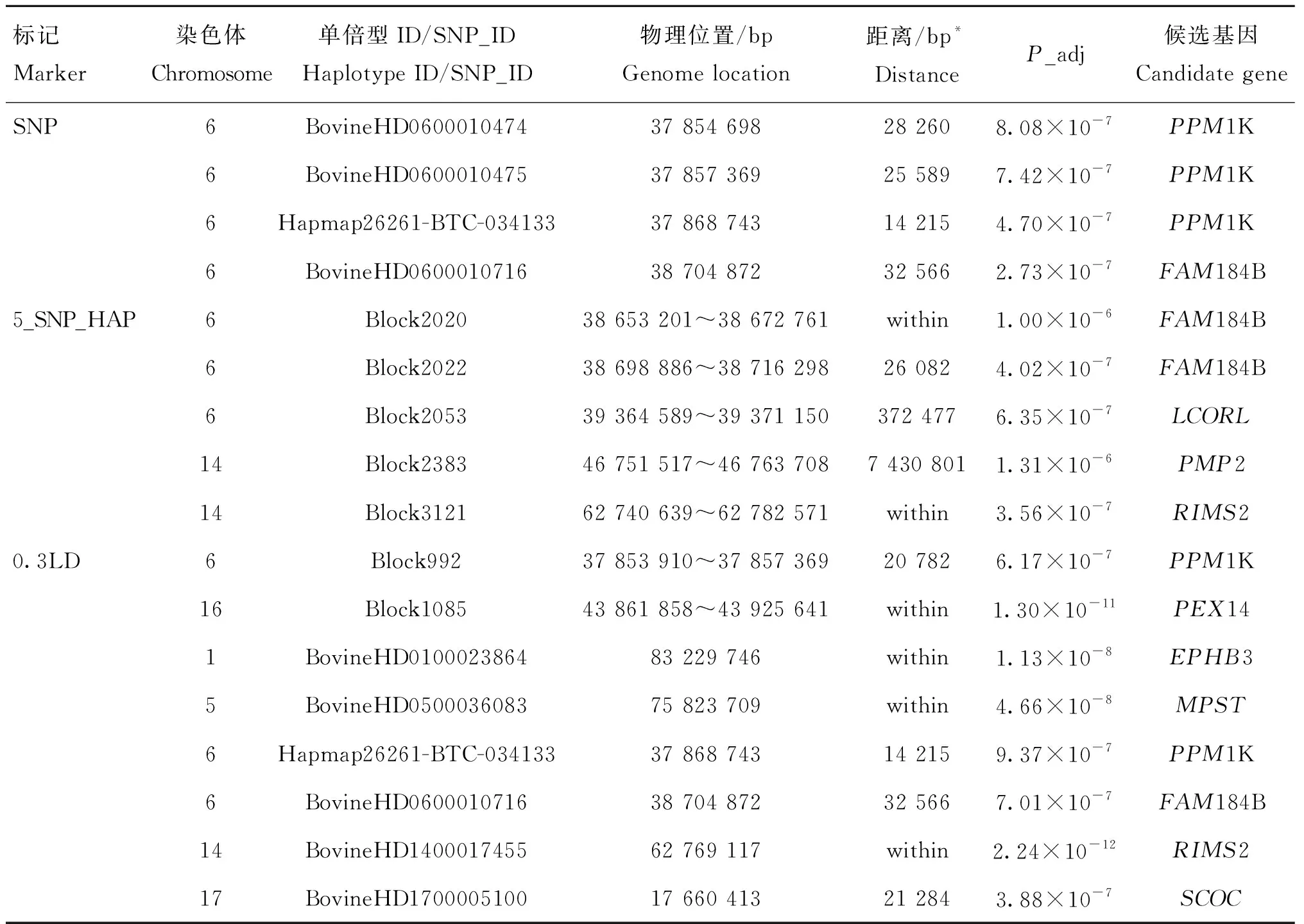
表5 基于SNP、0.3LD單倍型和5_SNP_HAP單倍型標記對宰前活重性狀的關聯分析結果
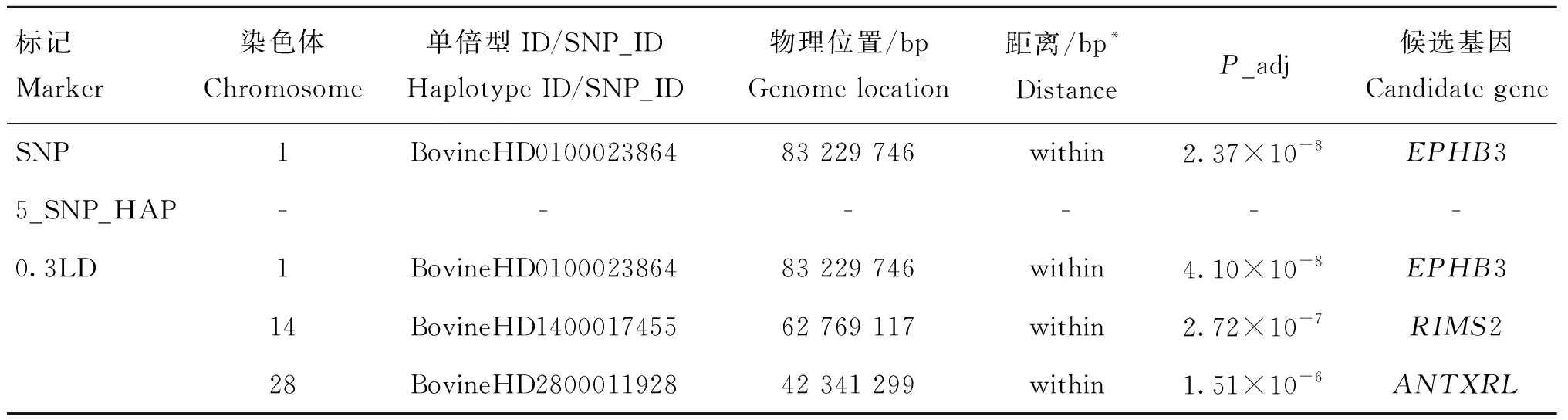
表6 基于SNP、0.3LD單倍型和5_SNP_HAP單倍型標記對屠宰率性狀的關聯分析結果
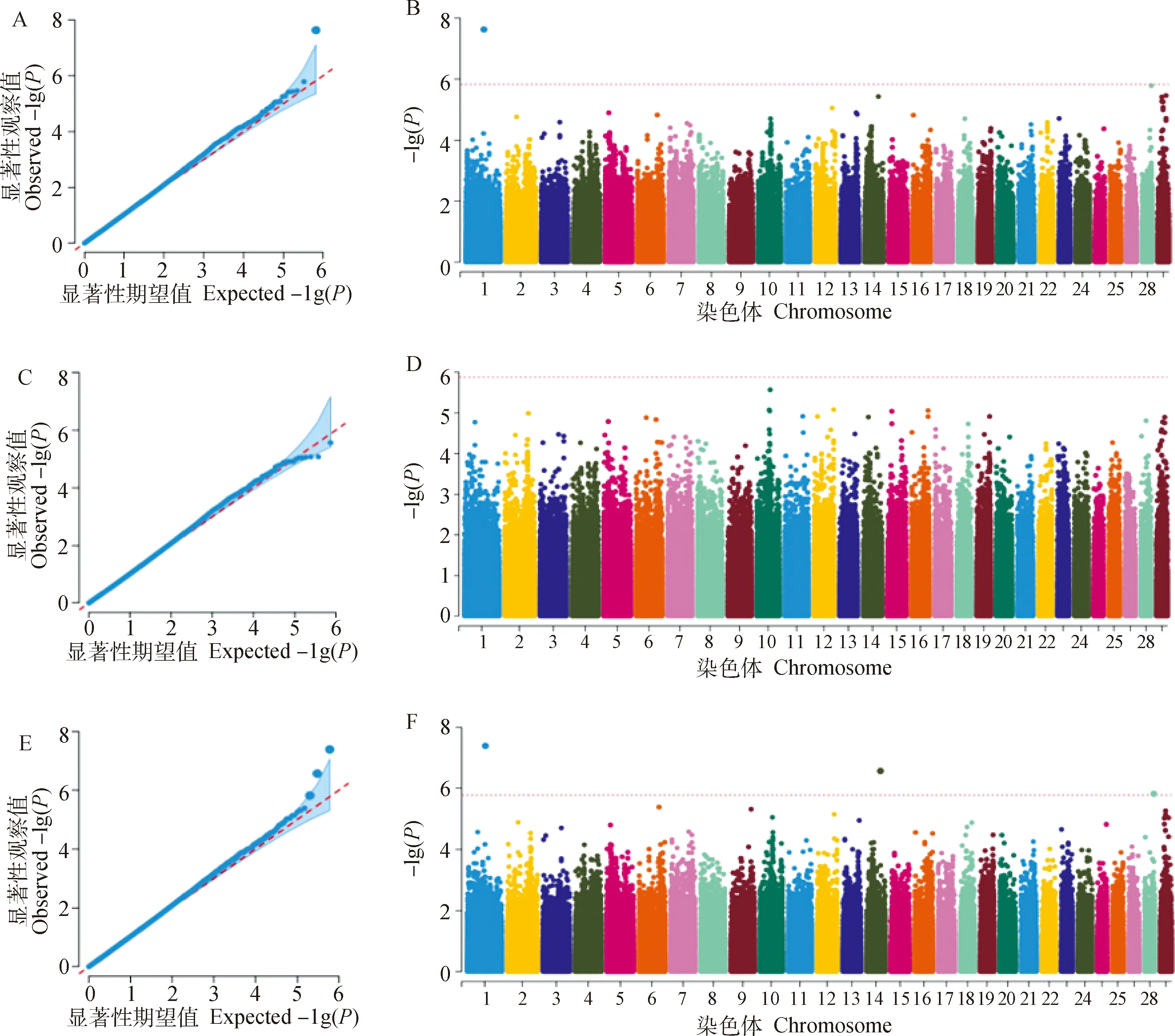
A和B表示利用SNP標記,C和D表示利用5_SNP_HAP單倍型標記,E和F表示利用0.3LD單倍型標記A and B represent SNP marker, C and D represent 5_SNP_HAP haplotype marker, E and F represent 0.3LD haplotype marker圖4 基于SNP、0.3LD單倍型和5_SNP_HAP單倍型標記對屠宰率性狀關聯分析結果的Manhattan和QQ圖Fig.4 Manhattan and QQ plots for the results of DP by SNP, 0.3LD and 5_SNP_HAP marker
本研究中的3種標記共鑒定到10個與宰前活重和屠宰率等屠宰性狀顯著相關的候選基因(表5和表6)。其中,SNP標記鑒定到的3個候選基因利用單倍型方法也可鑒定到。許多候選基因已經在其他牛品種或物種上被相繼報道。對于宰前活重性狀,FAM184B在3種標記中被共同鑒定到。FAM184B是一種蛋白質編碼基因,位于6號染色體的38 614 370~38 672 306 bp區域。已有研究表明,FAM184B是一種與生長發育和胴體性狀顯著相關的候選基因[40]。Xia等[41]在肉牛群體中進行全基因組關聯分析的研究中發現,FAM184B是與骨重顯著相關的候選基因。表5中的結果顯示,PPM1K在SNP標記和0.3LD單倍型標記中被鑒定到,據報道PPM1K是編碼Mn2+/Mg2+依賴蛋白磷酸酶的PPM家族成員。Abo-Ismail等[42]在加拿大安格斯、海福特及其雜交群體中進行全基因組關聯分析發現,PPM1K是與日增重、代謝體重及皮下脂肪沉積性狀顯著關聯的基因。RIMS2在SNP標記中沒有被鑒定到,但在兩種單倍型方法同時被鑒定到了。已有報道指出,該基因與胴體重和凈肉率性狀顯著相關[43-46],也有研究認為該基因與骨病顯著相關[47]。在5_SNP_HAP單倍型標記中成功鑒定到了LCORL,鑒定到的單倍型區域位于該基因的下游,距離該基因372 477 bp,該基因是與生長發育相關的候選基因,很多的研究報道均已指出LCORL是胴體重、日增重、骨重和眼肌面積等性狀重要的候選基因[41,48],盡管該單倍型區域距離LCORL較遠,但相比于SNP標記,單倍型方法鑒定到了該基因,在一定程度上證明了單倍型方法具有更強的檢測能力。同樣在0.3LD單倍型標記中鑒定到的SCOC是與體脂率和體重指數等性狀顯著關聯的候選基因。國內外幾乎未見對于屠宰率性狀的GWAS 研究,但在本研究中找到EPHB3、RIMS2和ANTXRL等候選基因,據報道EPHB3是重要的蛋白質編碼基因,主要在長骨的增殖軟骨細胞中持續表達[49]。RIMS2在宰前活重和屠宰率性狀中同時被鑒定到,證明其可能是調控生長發育的重要基因。
4 結 論
本研究基于SNP、0.3LD單倍型和5_SNP_HAP單倍型標記分別對宰前活重和屠宰率等屠宰性狀進行了GWAS研究。結果發現,3種標記共鑒定到10個與屠宰性狀顯著相關的候選基因,利用單倍型方法的GWAS鑒定到的基因多于基于SNP標記的GWAS,表明以單倍型開展GWAS可以綜合考慮SNP位點間連鎖關系,并能較好的揭示復雜性狀的遺傳結構。

