谷子PAL基因家族全基因組的鑒定和表達分析
孟亞軒, 孫穎琦, 趙心月, 王鳳霞, 甕巧云, 劉穎慧
( 河北北方學院 農林科技學院, 河北 張家口 075000 )
苯丙氨酸解氫酶(phenylalanine ammonia-lyase,PAL)是苯丙烷代謝途徑中的關鍵酶,參與木質素及酚類化合物的合成過程,廣泛存在于各種植物中(Fraser & Chapple, 2011;蓋江濤等,2016)。Koukol和Comm(1961)在大麥()中首次提取出PAL蛋白,后續馬鈴薯()(Joos & Hahl, 1992)、煙草()(Reichert et al., 2009)等高等植物也相繼分離純化得到PAL蛋白。楊郁文等(2017)研究發現PAL多以同源四聚體形式存在于細胞質和葉綠體中,通過轉錄水平等多層次調控方式,在植物防御系統中發揮重要作用。啟動子缺失實驗表明,MYB轉錄因子可通過結合啟動子區段,進行轉錄水平調控(楊郁文等,2017)。可通過調控HCA類酚類、類黃酮等產物的合成影響果實品質(張麗之等,2018)。此外,廣泛參與植物響應病原菌侵染過程。楊會曉等(2019)研究表明與香蕉抗病性密切相關,在枯萎病菌侵染過程中高豐度表達。活性增加與苯丙烷類產物的產量提高密切相關,其活性水平隨發育階段、細胞和組織的分化、不同應激刺激而改變。Lister等(1996)首次發現活性與蘋果()果實類黃酮含量存在顯著正相關。楊會曉等(2019)研究發現在香蕉()果實發育成熟過程高豐度表達,次級代謝物質在這一階段具有較高的表達效率。Olsen等(2008)研究發現擬南芥()1、2基因在氮脅迫和溫度變化過程中高豐度表達,并伴隨類黃酮化合物的積累。
隨著基因組學的發展,多種植物基因組測序相繼完成,植物中基因的功能陸續得到研究。大豆()1-1、2-1、2-3在木質素合成過程中發揮重要作用(候鵬等,2016);10強烈響應紋枯病菌侵染玉米()過程(鄧路長等,2019);參與山葡萄()花色苷的積累(陳蒙等,2018)。基因組學的迅速發展,標志著在分子水平上進行系統分析已成為當下生物學研究的主流趨勢(王燦等,2020)。谷子()為一年生禾本科作物,基因組小且為二倍體,具有抗旱性強、生育期短、產量高等特點,是挖掘作物抗旱基因和解讀抗逆分子機制的重要作物(宋健等,2020)。谷子生育期內需水量較少,屬環境友好型作物(宋健等,2019),抗逆育種靶向基因資源的研究已成為谷子增收工作的重要環節(Nadeem et al., 2020)。谷子基因組測序的完成為谷子研究搭建了數據基礎,標志著谷子遺傳研究已進入后基因組學時代(Bennetzen et al., 2012;Zhang et al., 2012)。基因家族廣泛存在于植物中,但關于谷子基因家族的研究卻鮮有報道。為明確谷子基因家族在逆境脅迫過程中的作用機理,本研究利用生物信息學方法對谷子基因家族進行鑒定,分析其結構特點及進化方式,并構建家族基因在非生物脅迫下的表達模式,以期為谷子基因家族的生物學功能研究提供參考。
1 材料與方法
1.1 材料
供試谷子()品種為一年生‘張雜谷8號’,盆栽于河北北方學院農場,分別置于自然光、黑暗、紅光、藍光、遠紅光條件下照射24 h,取幼嫩葉片,液氮速凍后,-80 ℃保存備用。
1.2 谷子PAL基因的鑒定及蛋白序列分析
利用Pfam數據庫查找并下載PAL蛋白結構域的隱馬爾可夫模型文件(Pfam號碼:00221)(Finn et al., 2008),利用plant Genes 基因組獲取Gene stable ID及Transcript stable ID,通過CDD和InterProScan對Gramene網站獲取的序列進行篩選鑒定,去除冗余,得到PAL蛋白序列、染色體定位等基因信息(Marchler et al., 2009; Hunter et al., 2009)。利用ProtParam數據庫通過序列信息得到氨基酸數量、等電點等用于推測蛋白質的相關因子(宋健等,2020)。
1.3 基因的染色體定位
通過MG2C在線工具,利用谷子基因位置信息以及從Ensemble Plants查詢到的谷子染色體長度,繪制基因的染色體定位。
1.4 谷子PAL蛋白分析及進化樹繪制
使用ProSite和Clustal X在線軟件對谷子PAL蛋白結構域位置進行序列對齊。通過MEGA 6.0軟件中的銜接法(neighbor-joining, NJ),采用泊松模型繪制蛋白進化樹(bootstrap為1 000)(Larkin et al., 2007;Sigrist et al., 2010)。通過上述方法對二穗短柄草()、高粱()、狗尾草()等54個PAL家族成員繪制蛋白進化樹(bootstrap為默認值)。
1.5 Motif獲取及蛋白預測
在MEME網站得到谷子PAL的motif模式(最小寬度設置為60,最大寬度設置為200)(Bailey et al., 2009);利用Weblogo分析得出PAL結構域motif(Stacks per Line設置為100);在SWISS-MODEL網站中利用其各基因位點出現頻率最高基因組成的保守序列預測其三維結構并得到螺旋模式(Kiefer et al., 2009),并根據上述方法對二穗短柄草、水稻等物種的PAL蛋白進行預測,分析對比各物種的蛋白結構差異。
1.6 PAL同源共線性分析
在PAL系統發育的基礎上,谷子和水稻()的直系同源片段已得到鑒定。利用Ensembl Plants網站的基因組比較功能得到谷子和水稻同源物的同源共線圖(Wang et al., 2012)。通過其染色體定位及基因位置,在Adobe Illustrator CS4軟件繪制谷子和水稻同系物的同源共線圖,分析其共線性。
1.7 PAL基因結構分析
通過Phytozome數據庫獲得谷子基因的序列信息,使用GSDS 2.0在線數據庫分析其編碼序列,得到內含子-外顯子結構模型(Hu et al., 2015)。在谷子基因組數據庫得到起始密碼子上游1 500 bp區域序列,利用plantCARE分析其順式作用元件,通過GSDS 2.0將其可視化(Lescot et al., 2002)。
1.8 谷子PAL基因表達模式分析
在Phytozome數據庫獲得11個基因在不同誘導處理下的表達模式,包括強光誘導2周的葉片、強光誘導1周的芽、黑暗誘導的地上組織、紅光誘導的地上組織、正常光誘導的根、干旱誘導的根、尿素誘導的根、強光誘導的穗,利用TBtools繪制熱圖(Chen et al., 2020)。
1.9 谷子PAL基因在不同光質照射下的RT-qPCR分析
利用DNAMAN設計引物,選擇為內參基因。使用植物總RNA試劑盒提取RNA(天根生化科技有限公司),FastQuant RT Kit合成cDNA,使用Agilent 3000P熒光定量PCR儀進行PCR擴增。反應條件:95 ℃預變性2 min,95 ℃變性15 s;60 ℃退火30 s,40個擴增循環,每個樣品進行3次重復;采用2進行數據處理。
2 結果與分析
2.1 谷子PAL基因家族鑒定
谷子中共鑒定11個家族基因,分別命名為1~11(表1)。11個PAL蛋白序列差異較小:氨基酸長度為698 aa(SiPAL1、SiPAL8)~891 aa(SiPAL7),開放閱讀框長度2 142 bp(3)~4 610 bp(2),分子質量為74.99 kD(SiPAL8)~95.01 kD(SiPAL7),等電點為5.82(5)~6.52(6),含有1個(3、4、5)~6個(7)外顯子。從表1可以看出,位于1號染色體的5個基因(1~5)具有相似的編碼蛋白特征,且基因位置緊密排列在一起,位于7號染色體的3個基因(9~11)雖然緊密排列,但其編碼蛋白的特征卻相差較大。所有PAL蛋白的等電點均在5.82~6.52之間,說明苯丙氨酸解氫酶具有酸性特征,PAL蛋白可能在弱酸性的環境中發揮作用。家族基因除7(6)外,外顯子數量較小,推測10個家族基因具有相似的功能。
從基因的染色體分布發現,1號染色體有5個緊密排列的基因,分布的基因數量最多;7號染色體具有3個基因且緊密排列,其他染色體上沒有成簇基因分布(表1,圖1);此外,基因不均勻分布在染色體各個部位,其中1號染色體和7號染色體基因形成簇狀分布。利用Phytozome數據庫的Gbrowse功能比對成簇分布的基因家族成員與側翼編碼蛋白質基因位置關系,參考Holub(2001)對基因簇的界定,暗示谷子家族基因可通過串聯復制實現家族擴增。
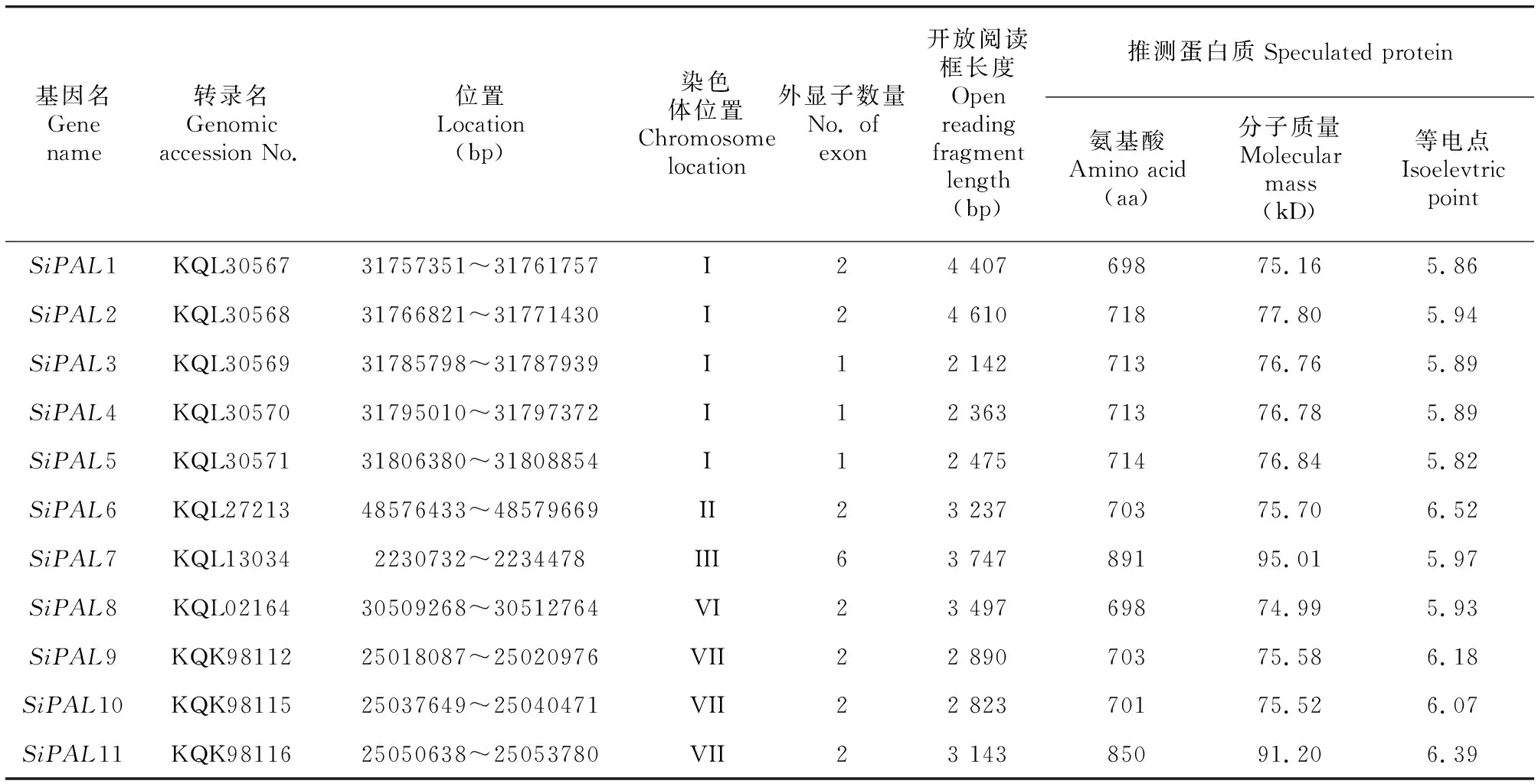
表 1 谷子PAL基因家族的信息Table 1 Information of millet PAL gene family
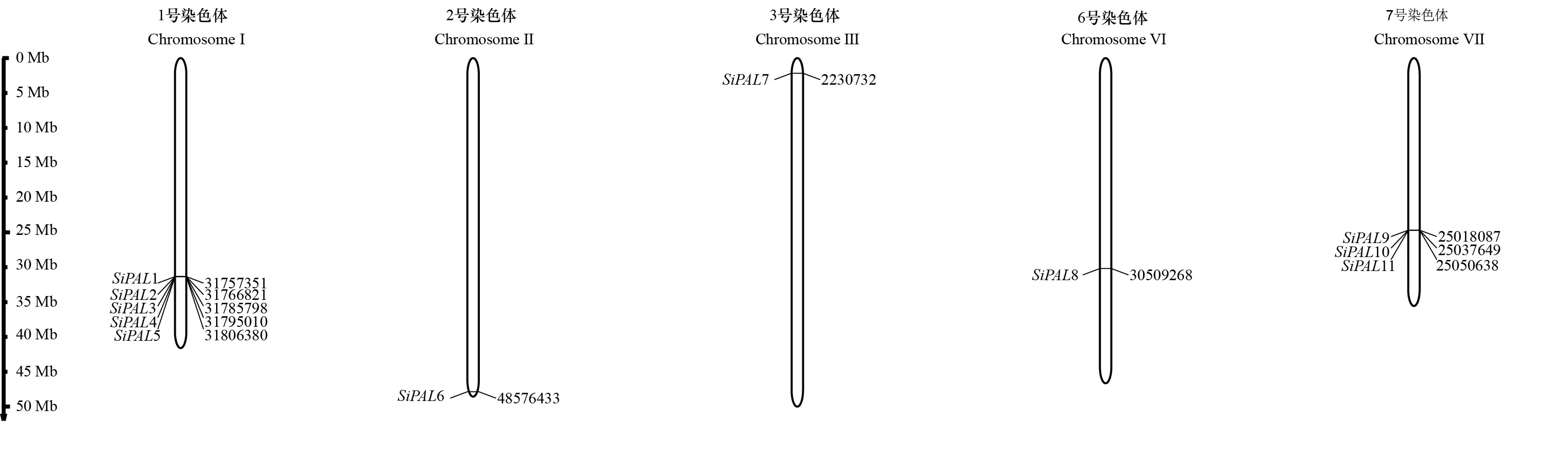
圖 1 谷子PAL基因家族的染色體分布Fig. 1 Chromosome distribution of millet PAL gene family
2.2 谷子PAL蛋白進化樹的構建
利用蛋白序列對齊文件,通過銜接法繪制出谷子PAL的進化樹(圖2)。由圖2可知,根據進化樹拓撲結構可分為3組蛋白,第1組和第2組蛋白數量相同且分支分布情況相似,此外小部分PAL蛋白的臨界值達到100。進化樹的拓撲情況表明,這兩組基因可能經過復制的方式得到或者執行相似基因功能。由進化樹聚類分析得出,相同PAL蛋白結構域與同一基因擴增或復制的蛋白聚合在一起,如SiPAL6和SiPAL9分散復制聚在一起,相同結構域聚合為一組。另外,SiPAL7獨自分支為一組,說明SiPAL7可能具有不同的進化軌跡。
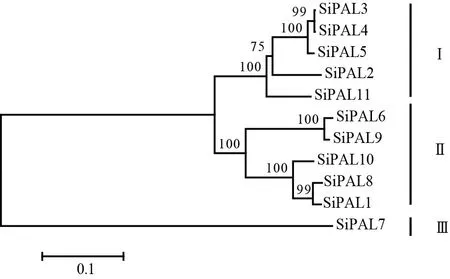
圖 2 PAL蛋白系統發育進化樹Fig. 2 Phylogenetic tree of PAL protein
2.3 不同物種PAL蛋白進化關系
利用MEGA 6.0軟件將單、雙子葉共6個物種的54個PAL蛋白利用銜接法構建系統發育進化樹(圖3)。由圖3可知,基因家族演化分析中選用二穗短柄草9個,高粱8個、狗尾草11個、水稻10個、擬南芥5個PAL蛋白發現,7個親緣關系組中部分物種PAL蛋白呈家族性聚集,不同綱目物種之間的PAL蛋白也具有較高的同源性。
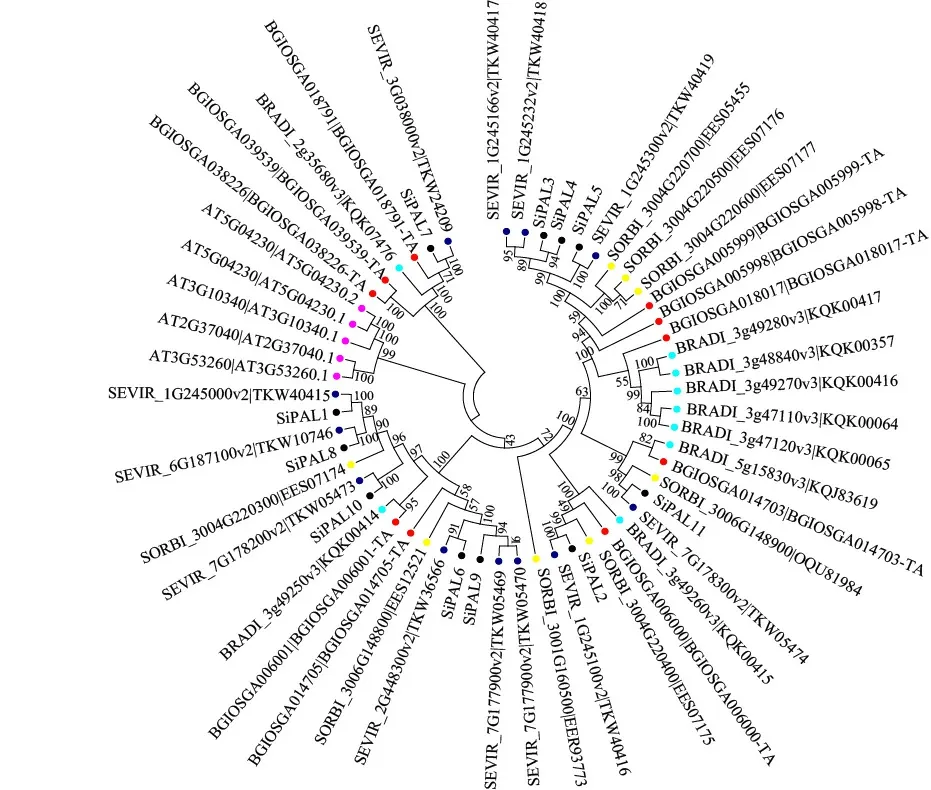
黑色為谷子,青色為二穗短柄草,黃色為高粱,藍色為狗尾草,紫色為擬南芥,紅色為水稻。Black is Setaria italica, cyan is Brachypodium distachyon, yellow is Sorghum bicolor, blue is Setaria viridis, purple is Arabidopsis thaliana, red is Oryza sativa.圖 3 不同物種PAL蛋白進化樹Fig. 3 Phylogenetic tree of PAL protein in different species
2.4 谷子PAL蛋白的結構域分析
通過ProSite軟件對谷子PAL蛋白的結構域分析結果發現,所有PAL蛋白均具有保守的PAL 結構域。由圖4可知,SiPAL7蛋白除含有PAL結構域外還具有HtRna 結構域、HGTP 結構域,其中HtRNA 結構域可能調控氨酰腺苷酸的合成,HGTP 結構域可能與相關蛋白質的合成有關,SiPAL7的氨基酸長度最長,擁有蛋白結構域最多,可能與其具有最多的外顯子(6個)有關,意味著擁有更多的遺傳信息;SiPAL11雖同樣具有較長的氨基酸序列,但只含有PAL蛋白結構域,暗示SiPAL11可能擁有一些低復雜度的蛋白結構域。通過MEME軟件對谷子PAL蛋白結構域分析發現,PAL結合結構域的氨基酸組成較為穩定。
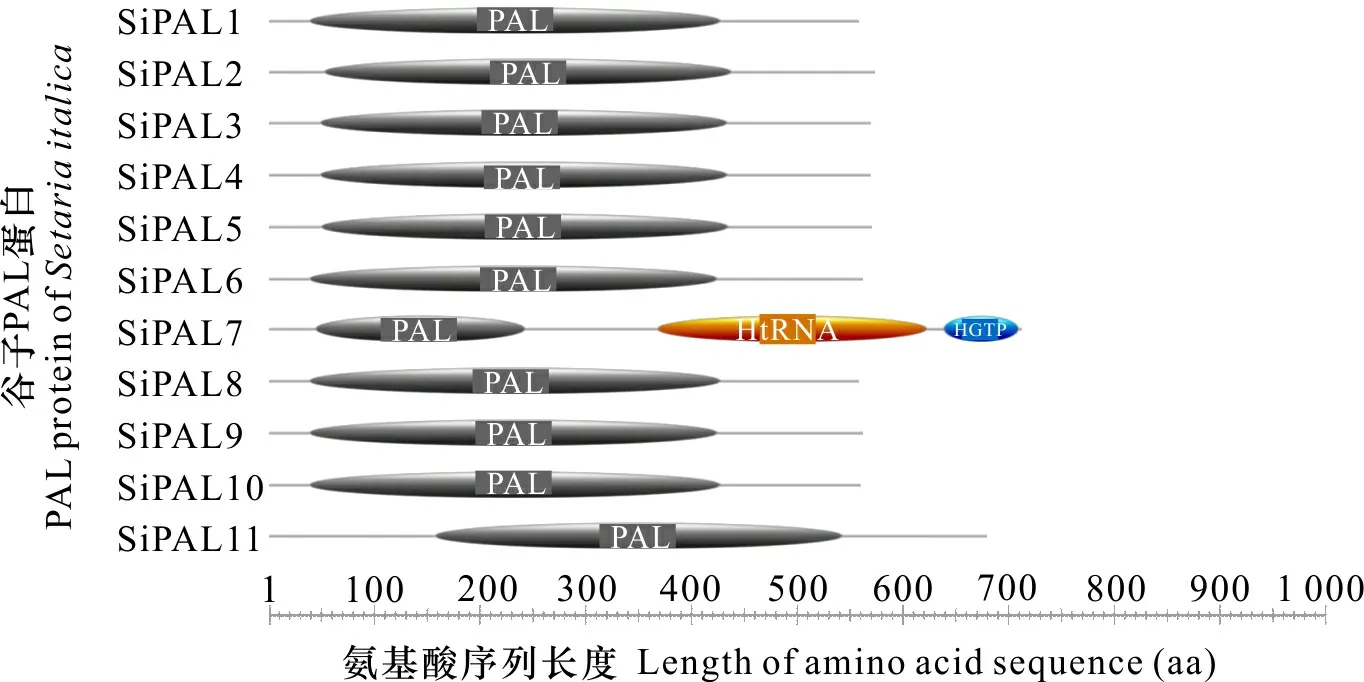
圖 4 谷子PAL蛋白結構域示意圖Fig. 4 Schematic diagram of PAL protein domain of millet
2.5 谷子PAL蛋白3D結構與序列分析
利用SWISS-MODEL軟件分析各基因位點出現頻率最高的保守基序,預測谷子PAL蛋白的3D結構發現,谷子PAL蛋白的PAL 3D 結構在對稱基礎上具有較多的螺旋、折疊方式(圖5:A)。通過Superpose Version 1.0將各物種間的PAL蛋白3D結構對比,比值越小證明物種間PAL蛋白結構越相似。由表2可知,狗尾草與谷子3D結構相差最小(0.38),與進化樹分支聚類情況一致,證明兩者具有最近的親緣關系,相差最大的是谷子與二穗短柄草(3.93),這與谷子和二穗短柄草為不同綱目的物種相對應。通過蛋白3D結構發現PAL蛋白的三級結構大多具有對稱性,說明其蛋白質的結構單元具有一定的相似性,從基因進化來看,這種對稱性代表著基因存在復制融合。
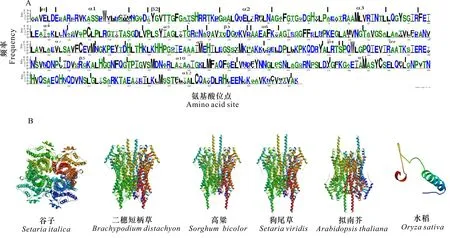
A. PAL氨基酸序列; B. PAL蛋白3D結構示意圖。A. PAL amino acid sequence; B. PAL protein 3D structure schematic diagram.圖 5 PAL蛋白的3D結構及序列Fig. 5 3D structure and sequence of PAL protein
2.6 谷子、水稻PAL同源共線性分析
利用Ensembl Plants網站synteny功能得到谷子和水稻同源物的同源共線圖。通過染色體定位及基因位置,繪制谷子和水稻同源物的同源共線圖(圖6僅展示代表成員)。圖6結果表明,谷子11個家族成員均可在水稻中找到共線基因,如含有020626100、020626400等的水稻2號染色體基因組區域與谷子1號染色體的基因組塊具有共線性,含有050150900的水稻5號染色體基因組區域與谷子3號染色體的基因組塊具有共線性;同樣含有040518100、040518400的水稻4號染色體基因組區域與谷子7號染色體組塊具有共線性。

圖 6 谷子和水稻PAL基因共線性分析Fig. 6 Collinearity analysis of PAL gene in millet and rice
2.7 PAL基因結構分析
利用GSDS 2.0對谷子序列信息分析并可視化,得到基因內含子-外顯子結構模型圖(圖7:A)發現,基因大多數只具有1個內含子(1、2、6、8、9、10、11),且這些基因的外顯子結構及大小具有一定的相似性,這種結構間的相似性說明他們可能在植物的生化過程中執行相同的作用。3、4、5同樣具有相同的外顯子結構,但均不含有內含子。7擁有最多的內含子數量(5)。
將plantCARE分析得到的順式作用元件區域可視化,得到順式作用元件分布圖,圖7:B結果顯示,家族中含有多種激素信號響應元件和環境信號響應元件。其中,分布最廣的為茉莉酸響應元件,存在于所有基因中,暗示基因通過響應植物內源激素參與植物抗病過程;分布最少的元件為胚乳表達元件,只存在于9中;其他不同類型順式作用元件如脅迫元件、光響應等不均勻分布在各個位置。可見,家族基因在谷子發育、抗病及脅迫過程中發揮重要作用。
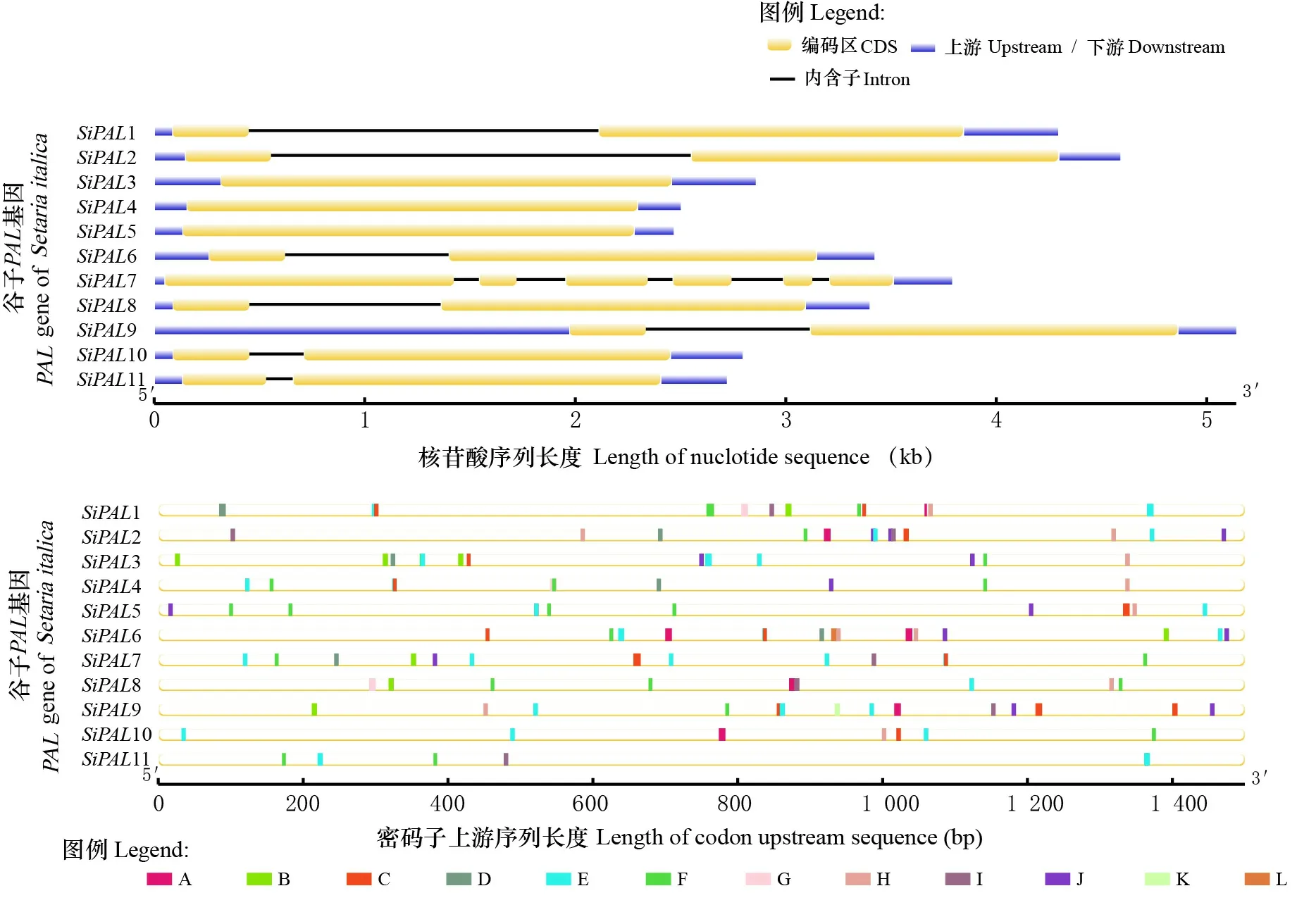
A. 防御與應激響應; B. 赤霉素響應; C. 脫落酸響應; D. 生長素響應; E. 光響應; F. 茉莉酸響應; G. 玉米醇溶蛋白調控元件; H. 分生組織誘導; I. MYB結合位點; J. 缺氧誘導; K. 胚乳表達; L. 低溫脅迫。A. Defense and stress responsiveness; B. Gibberellin responsiveness; C. Abscisic acid responsiveness; D. Auxin responsiveness; E. Light responsiveness; F. MeJA responsiveness; G. Zein metabolism regulatory elements; H. Meristem induction; I. MYB binding site; J. Hypoxia induction; K. Endosperm expression; L. Low temperature stress.圖 7 PAL基因內含子-外顯子結構及順式作用元件Fig. 7 PAL gene intron-exon structure and cis-acting elements
2.8 谷子PAL基因表達分析
從Phytozome數據庫獲得的11個基因在谷子不同組織中面對不同非生物脅迫的表達量數據,利用TBtools將結果可視化,繪制熱圖,如圖8所示,所有基因均檢測到表達量,其中1、2、8、10在強光誘導1周的芽、黑暗誘導的地上組織、紅光誘導的地上組織、正常光誘導的根、干旱誘導的根、尿素誘導的根、強光誘導的穗中均具有明顯高于其他基因的表達量; 在強光誘導生長2周葉片的表達量中只有2具有微弱表達,其他基因幾乎不表達。3、6、9在谷子不同組織中的表達量均明顯低于其他基因;家族基因為誘導型表達,即在干旱或強光等誘導條件下,表達量為增強或抑制,存在著不同基因的差異性。
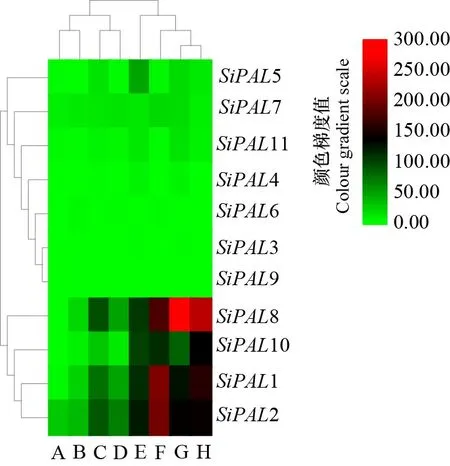
A. 強光誘導葉片2周; B. 強光誘導芽1周; C. 黑暗誘導的地上組織; D. 紅光誘導的地上組織; E. 尿素誘導的根; F. 強光誘導的穗; G. 正常光誘導的根; H.干旱誘導的根。A. Strong light induced leaves for two weeks; B. Strong light for one week; C. Dark induced above-ground tissue; D. Red light induced above-ground tissue; E. Urea induced roots; F. Bright light-induced spikes; G. Normal light-induced roots; H. Drought-induced roots.圖 8 谷子PAL基因在不同誘導下各組織的表達量Fig. 8 Expression of millet PAL gene in various tissues under different induction
2.9 谷子PAL基因在不同光質照射下的RT-qPCR分析
由圖9可知,紅光照射下,1、8、5上調表達,其余基因與對照組相比,表現出不同程度的下降。藍光照射下1、8、10等上調表達,7與對照組相比無明顯變化。黑暗處理下,1、8、2等上調表達,其他基因表現為下調,其中6表達量極低。遠紅光照射下,1、8、2、10、5、11等上調表達。家族基因在不同光質照射下表達量變化明顯,說明基因在谷子光形態建成中發揮重要作用,1、8為主要成員。
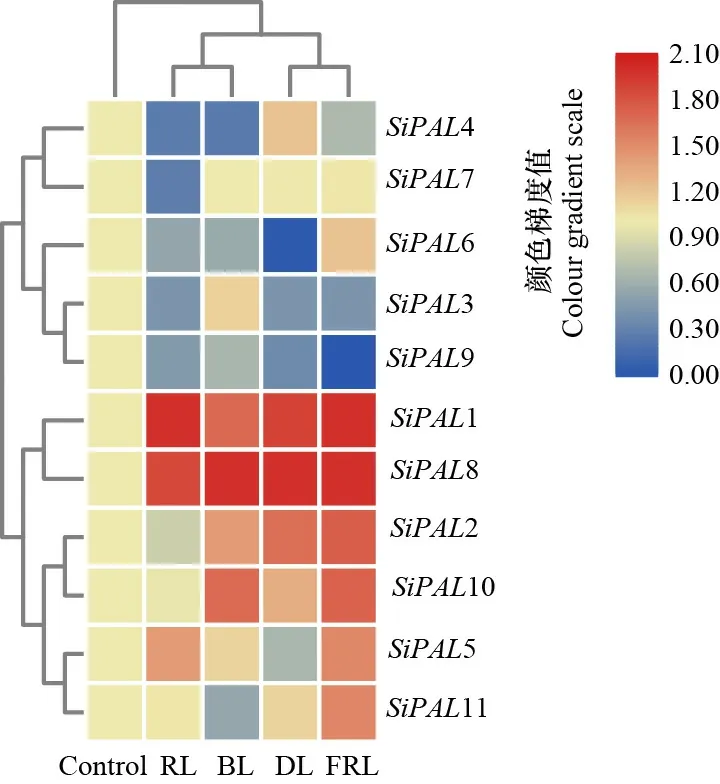
Control. 正常光誘導; RL. 紅光誘導; BL. 藍光誘導; DL. 黑暗誘導; FRL. 遠紅光誘導。Control. Normal light induction; RL. Red light induction; BL. Blue light inducation; DL. Dark induction; FEL. Far-red light induction.圖 9 谷子PAL基因在不同光質下的表達量Fig. 9 Expression of PAL gene in millet under different light qualities

表 2 PAL蛋白疊加比對Table 2 PAL protein stacking ratio
3 討論與結論
PAL是與植物抗性相關的關鍵酶(孫宇蛟等,2018),對其研究備受關注,前人已對多物種的基因家族進行了分析,如玉米(鄧路長等,2019)、蘋果(張麗之等,2018)和陸地棉()(楊郁文等,2017)。其中,玉米和陸地棉均含有13個基因,蘋果含有8個家族成員,與本研究中谷子鑒定出11個基因結果相差不大,說明基因以小基因家族形式存在,在物種分化過程中沒有出現大幅度擴增現象。谷子基因編碼的蛋白中有部分蛋白具有相近的分子量、等電點等,與香蕉(楊會曉等,2019)和青天葵()(黃瓊林等,2016)的研究較一致,說明其編碼的蛋白具有相似的功能。
基因復制事件是基因家族擴張的主要動力。本研究發現,谷子基因呈簇狀分布,在蘋果(張麗之等,2018)、大豆(候鵬等,2016)、西瓜()(Dong & Shang, 2013)中PAL染色體定位同樣存在簇狀分布現象,其中西瓜12個基因中有7個串聯排列在4號染色體,2個串聯排列在7號染色體上,其余單獨排列在染色體2號、3號、8號上,與谷子染色體分布有著高度的相似性,說明在基因家族的擴增中存在串聯復制、分散復制、片段復制(郭棟等,2019)。共線性分析發現,11個谷子基因家族成員在水稻染色體組中均存在共線性關系,說明基因在進化過程中保守性較高。
根據進化樹的拓撲結構,谷子PAL蛋白進化樹分為3組,SiPAL7獨自進化為一支,說明SiPAL7與其他成員同源性較低,可能具有不同起源或進化軌跡,本研究結果與陸地棉(楊郁文等,2017)具有一致性。系譜樹內同一進化枝成員基因結構較為一致,具有較高的保守性。但是,Ⅲ組成員7與其他家族成員相比基因結構差異較大,擁有更多的內含子數量,這暗示基因家族具有多樣化的轉錄調控過程。基因家族成員結構的差異性可能會影響其功能活性。
啟動子分析發現,基因家族不僅含有大量非生物脅迫元件,而且存在光響應、激素響應等多種類型元件,不同家族成員所含元件數量和種類不同,說明基因家族廣泛參與不同生物學調控過程,不同基因具有其特異調控模式。
植物在遭受干旱、高溫等脅迫時,會迅速產生大量活性氧(ROS)造成細胞結構損傷。苯丙烷代謝途徑中產生的類黃酮等次生代謝產物,具有清除ROS的抗氧化活性(楊會曉等,2019)。本研究發現,基因家族多為誘導型表達,在受到不同脅迫刺激時,表達量迅速提升,說明基因家族廣泛響應不同非生物脅迫,類黃酮等次級代謝產物在非生物脅迫過程中可能具有較高的合成活性。本研究還發現,部分基因具有相似的響應模式,說明可能存在功能冗余。光質在植株建成、生長發育過程中具有重要作用。熒光定量分析結果表明,在不同光照條件下,基因家族差異表達。5在紅光、紅遠光高表達,11在紅遠光高表達等,這些變化說明基因家族在谷子光調節途徑中存在復雜的調控機制,不同基因存在功能分化。

