銀杏類黃酮合成關鍵基因的鑒定和GbANR2的基因的克隆
季雅菲
(中國農業大學,北京 100089)
1 引言
1.1 背景介紹
銀杏(Ginkgo bilobaL.)俗稱“白果”,為我國特有珍貴樹種,在植物界享有“活化石”之稱,集生態防護、觀賞、經濟等價值為一體,具有良好開發前景和研究價值[1]。
類黃酮是大多數植物中含量較多的一類次生代謝物,原花青素是類黃酮中的主要成分之一,主要由花青素還原酶(ANR)調控合成[2]。由于原花青素等黃酮類化合物能為農作物提供良好性狀,提高食品的保健作用,還可以為轉基因材料著色[3],故而該類化合物的合成途徑的相關研究逐漸受到人們的關注。
1.2 研究目的及意義
近年來,研究者已經相繼從一些植物中,如杉木、擬南芥中克隆了ANR 基因,并進行了相關的遺傳和生化研究,但是銀杏GbANR2基因的克隆與表達分析的分子機制鮮有報道[4-5]。本研究以類黃酮合成途徑為切入點,通過基因共表達分析鑒定出合成途徑中的關鍵結構基因,對關鍵基因GbANR2進行了基因克隆,并對其進行生物學分析。本研究將為明確GbANR2對銀杏類黃酮的合成的作用提供理論基礎,為定向培育高類黃酮含量的優質銀杏植株提供理論基礎。
2 材料方法
2.1 銀杏黃酮類化合物合成相關結構基因的共表達分析
從NCBI Sequence Read Archive(SRA)數據庫(https://github.com/ncbi/sra-tools)中下載126 個銀杏樣本的高質量轉錄組(RNA-seq)數據,對其進行數據處理獲得126 個樣品中所有基因的表達數據[7-8]。將通過KEGG 鑒定出的銀杏類黃酮合成途徑中的48個結構基因進行基因共表達分析,計算所有基因之間的皮爾森相關系數(PCC),篩選PCC>0.75 的基因,并利用Cytoscape軟件實現基因的共表達網絡的可視化。
2.2 GbANR2基因克隆
2.2.1 試驗試劑
本試驗所用的主要試劑有:RNA 試劑盒(Omega,R6827-01,廣州)、反轉錄MonScript?RTIII All-in-One Mix with dsDNase 試劑盒、Phanta?Max Super-Fidelity DNA 高保真酶試劑盒、大腸桿菌感受態細胞(Tsingke,TSC01,北京)、DL5000DNA Marker(Tsingke,TSJ012-500,北京)、pClone007 Blunt Simple Vector Kit載體試劑盒、TAE溶液、ddH2O無菌水。
2.2.2 基因克隆
(1)RNA 提取及純度測定。試驗過程中的RNA 提取步驟按照試劑盒(Omega,R6827-01,廣州)說明進行。摘取銀杏幼葉,磨樣、渦旋、孵育、離心、渦旋,直至所有樣品轉移到色譜柱中。加入RWF洗滌緩沖液,丟棄濾液和收集管。
(2)引物設計。提取DNA 甲基化相關基因CDS 序列,使用Primer Primier 程序設計引物,采用直接擴增的方式進行克隆。正向引物(5’-3’)為ATGGCACCGCAGGCTTATCC,反向引物(5’-3’)為TTAGCAGGTAAGCAAGCCCTTGG。
(3)目的片段凝膠回收。切下含目的DNA 片段凝膠,加入等倍體積的Buffer GDP,水浴,短暫離心,將FastPure DNA Mini Columns-G 吸附柱置于Colletion Tubes 2 mL 收集管中,將≤700 μL的溶膠液轉移至吸附柱中,將濾液棄除,吸附柱置于收集管中。靜置,濾液棄除,重復前一步驟,將吸附柱置于在離心管中,加入Elution Buffer 至吸附柱中央,靜置,棄去吸附柱,離心管存于-20°C。
(4)目的片段連接與轉化。取100 μL大腸桿菌感受態細胞,加入連接產物,靜置。將其放入水浴中激活45 s,靜置。加入無抗性SOC/LB 培養液,搖1 h。將復蘇液涂抹至含氨芐青霉素(Amp)的培養基上,倒置過夜12~16 h。
2.3 GbANR2生物信息學分析
2.3.1GbANR2基因結構分析及染色體定位
基于銀杏基因組數據庫中基因的位置信息,運用TBtools軟件實現目的基因的基因結構的可視化。
2.3.2GbANR2蛋白結構預測
利用SWISS-MODEL 在線軟件分析銀杏ANR 蛋白三維結構模型,并與蛋白質二級結構預測結果進行比較。
2.4 GbANR2在銀杏不同組織中的表達
選取銀杏成年根(R)、成年莖(S)、未成熟葉片(IL)、成熟葉片(ML)、雌球果(OS)、大孢子葉球(M)、未成熟果實(IF)與成熟果實(MF),利用GraphPad 進行作圖,分析GbANR2 在銀杏不同組織中的表達量。
3 結果分析
3.1 基因共表達分析鑒定銀杏類黃酮合成途徑的核心結構基因
利用處理后的126 個銀杏公共轉錄組數據[6],對48 個類黃酮合成結構基因進行共表達分析(PCC>0.75),篩選出12個高度相關的核心基因[7],這些基因在類黃酮合成中發揮重要作用,現將其定義為參與類黃酮合成的核心基因。分別為CHS5、4CL2、DFR2、F3’H1、ANR3、PAL4、CHS1、ANS1、ANR2、CHI、CHS4、F3H1。
3.2 GbANR2基因克隆
3.2.1 目的基因的擴增與純化
以cDNA 為模板,利用Phanta? Max Super-Fidelity DNA Polymerase 進行GbANR2(Gb_10028)基因克隆,如圖1,將所有的PCR 產物進行電泳檢測并且進行切膠回收、轉化和陽性克隆鑒定。
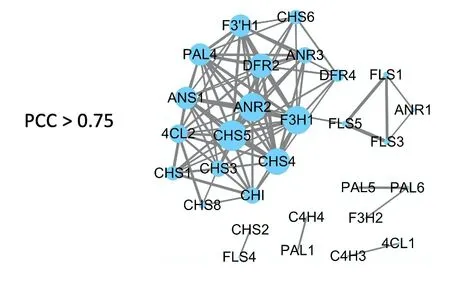
圖1 類黃酮通路相關核心結構基因的共表達分析Fig.1 The analysis on coexpression of core structure genes relatedto flavonoid pathway
3.2.2 克隆序列比對
將擎科公司測序結果使用NCBI 網站進行分析,將測序結果與原DNA 序列輸入進行比對,根據多序列比對結果可知,克隆序列與目的基因的序列完全相同,大小為1029bp,編碼341個氨基酸。
3.3 GbANR2的生物信息學分析
3.3.1GbANR2的基因結構和染色體定位
運用TBtools 進行基因結構可視化分析,得出GbANR2基因分布在3號染色體上,具有4個內含子、5個外顯子,長度為1029bp。通過DANMAN 軟件對GbANR2堿基序列進行翻譯,得到由341個氨基酸組成的蛋白質。
3.3.2GbANR2同源性分析
使用Blast 將GbANR 序列與其它植物的ANR 基因序列進行比對,結果顯示GbANR 基因的核苷酸序列與其它植物的ANR基因的核苷酸序列同源性非常高,尤其是裸子植物的白云杉和北美云杉,相似性分別為74.44%和74.24%,說明GbANR 與來自裸子植物的ANR 具有較近的親緣性。此外,GbANR 與日本柳杉、野茶樹、蘋果的相似性依次為72.59%,65.32% 和64.99%。因此,從蛋白質序列比較結果推測GbANR正是銀杏ANR基因家族成員之一。
3.3.3GbANR2蛋白結構預測
通過SOPMA 預測蛋白質的二級結構,結果顯示,GbANR2蛋白結構中α 螺旋有141 個氨基酸,所占比例為41.23%;β 轉角有27 個氨基酸,所占比例為7.89%,延伸鏈(Extended strand)有50 個氨基酸,所占比例為14.62%,無規則卷曲(Random coil)有124 個氨基酸,所占比例為36.26%。使用Swiss-model 對GbANR2蛋白的三級結構進行預測,結果顯示,GbANR2以花青素還原酶為模板構建,兩者蛋白的三級結構相似性為59.16%,模型主要包括12 個α 螺旋與10個β 轉角。
3.4 GbANR2在銀杏不同組織中的表達
銀杏發育過程中,GbANR2基因在根、莖中表達量較低,而生長初期相反,表達量較高,而后又迅速下調,使花青素得以積累。在不同的銀杏組織中,ANR 基因的表達量均有所不同,依次是IF>OS>IL>MF>R>S>ML>M ,表達量結果差異顯著。
4 結語
本研究從銀杏公共數據庫中下載了銀杏轉錄組數據126個,對原始數據進行處理和均一化,以皮爾森相關系數PCC>0.75 為標準,鑒定出12 個關鍵核心基因。其中GbANR2基因與其它基因的相互調控關系較為緊密,對該基因進行了克隆,并對其在染色上位置、基因結構、蛋白結構,以及在不同組織中的表達量進行了分析。研究表明ANR基因在在根、莖中的表達量較低,在葉、果實中的表達量較高,ANR基因在植物特定器官表達中起重要作用。
類黃酮在銀杏葉中具有重要作用,因此研究銀杏類黃酮代謝途徑,并利用相關基因進行有效的調控是獲得高產類黃酮的基礎。本研究鑒定了銀杏類黃酮合成關鍵基因,克隆了銀杏GbANR2基因。為了更好得進行后續研究,可以從銀杏幼葉中選取表達量較高的其它核心基因進行克隆,為研究銀杏類黃酮合成途徑提供豐富的理論依據。

