一株雞傳染性貧血病毒的分離鑒定及全基因組序列分析
胡峰 朱彤 高月花 黃兵 宋敏訓 劉丹丹 汪建華 李玉峰

摘 要:利用MDCC-MSB1細胞系從某養雞場疑似雞傳染性貧血病毒(chicken infectious anemia virus,CIAV)感染的發病雞組織病料中進行病毒分離,并通過PCR、病毒基因序列測序分析,確定分離到一株雞源CIAV,并命名為CZ16。將分離株全基因組進行PCR分段擴增、測序,結果顯示分離株CZ16基因組序列全長為2 298 bp,VP1、VP2、VP3基因大小分別為1 350、651、366 bp,兩端5'和3'非編碼區長度分別為358 bp和117 bp。全基因組序列同源性分析結果顯示CZ16與國內外已發表的36個CIAV毒株全基因組核苷酸同源性在95.6%~99. 6%之間,與國內毒株GD-102關系最近,與澳大利亞毒株(U65414.1)關系最遠。研究結果為雞傳染性貧血病病原學研究提供材料。
關鍵詞:傳染性貧血病毒;病毒分離;全基因組;進化分析
雞傳染性貧血病是由雞傳染性貧血病毒(CIAV)感染引起的雞主要免疫抑制病,其中雛雞最易感[1]。CIAV感染后主要病理特征是胸腺萎縮和骨髓黃化,從而造成貧血和免疫抑制,容易繼發其它病毒、細菌、寄生蟲感染[2-3]。CIAV不僅可以感染雞,還可感染野生鳥類[4]。CIAV屬于圓環病毒科,環形病毒屬,病毒粒子呈球形,無囊膜。病毒基因組為單股環狀DNA,編碼3個蛋白質(VP1、VP2、VP3)[5]。目前發現的CIAV均屬于同一個血清型,但不同地區不同毒株之間毒力不同,基因組序列也存在一定的差異[6]。本實驗室接診了某養殖公司送檢的疑似雞傳染性貧血病毒感染病例,通過病毒分離、PCR檢測、病毒基因序列測序分析,確定分離到1株CIAV,為進一步了解CIAV的流行規律提供材料。
1 材料與方法
1.1 試驗材料及試劑
病料:來自河北某養雞場。
試劑:高保真聚合酶,GenStar產品;DNA提取試劑盒,AxyGen公司產品;克隆載體pMD18T,購自寶生物(大連)生物技術有限公司;MDCC-MSB1細胞,哈爾濱獸醫研究所惠贈。
1.2 引物設計與合成
根據GenBank中已發表的CIAV毒株基因序列設計并合成3對引物用于病毒分離鑒定和全長基因組擴增。
病毒鑒定引物對:CAV-F:5'-GGACCATCAA
CGGTGTTCAG-3',CAV-R:5'-CCTCAAGTCCGG
CACATTCT-3',預計擴增產物長度為200 bp。
擴增VP1和3'UTR基因引物對:3VP1-F:5'-ATGGCAAGACGAGCTCGCAGAC-3',3VP1-R:5'-GATTGTGCGATAAAGCCATTTG-3',預計擴增產物長度為1 467 bp。
擴增VP2(含VP3)和5'UTR基因引物對:5VP2-F:5'-GCATTCCGAGTGGTTACTATTCC-3',
5VP2-R:5'-TTACACTATACGTACCGG-3',預計擴增產物長度為1 009 bp,引物由青島擎科生物技術有限公司合成。
1.3 病毒分離
無菌采集的病死雞肝臟、脾臟等臟器,經實驗室檢測初步確定為CIAV陽性。病料混合后以1∶5比例加入含有青霉素、鏈霉素的滅菌生理鹽水,研磨,離心取上清,過濾除菌后接種于MDCC細胞,定期觀察細胞生長狀態,待細胞出現明顯病變后收集細胞懸液,-80 ℃保存。
1.4 PCR鑒定
按照試劑盒(Axygen)說明書提取細胞懸液病毒核酸,利用設計的引物進行PCR鑒定,PCR反應體系25 μL:2 ×Taq PCR StarMix 12.5 μL,
上下游引物(20 μmol/L)各0.5 μL,DNA模板2 μL,超純水9.5 μL。擴增條件:94 ℃ 預變性2 min;94 ℃ 20 s,50 ℃ 20 s,72 ℃ 90 s,35個循環;72 ℃延伸5 min。PCR產物經瓊脂糖電泳鑒定并測序。
1.5 病毒全基因組克隆
取1.4中提取的病毒核酸,利用設計的引物(3VP1-F/3VP1-R, 5VP2-F/5VP2-R)PCR擴增病毒全基因組序列,PCR產物純化回收后與pMD18T載體連接,轉化DH5α感受態細胞,篩選陽性克隆,送青島擎科生物技術有限公司測序。
1.6 病毒全基因組序列分析
對全基因組序列進行拼接,并利用Mega6.0軟件將分離株與其它在GenBank已發表的36個毒株進行序列比對和遺傳進化分析。
2 結果
2.1 病毒分離
組織病料經無菌處理后接種MDCC細胞,48 h后開始出現細胞病變,細胞死亡,碎片增多,而對照組MDCC未出現細胞病變(圖1)。將病毒擴大培養,并將該分離株命名為CZ16。
2.2 特異性PCR檢測
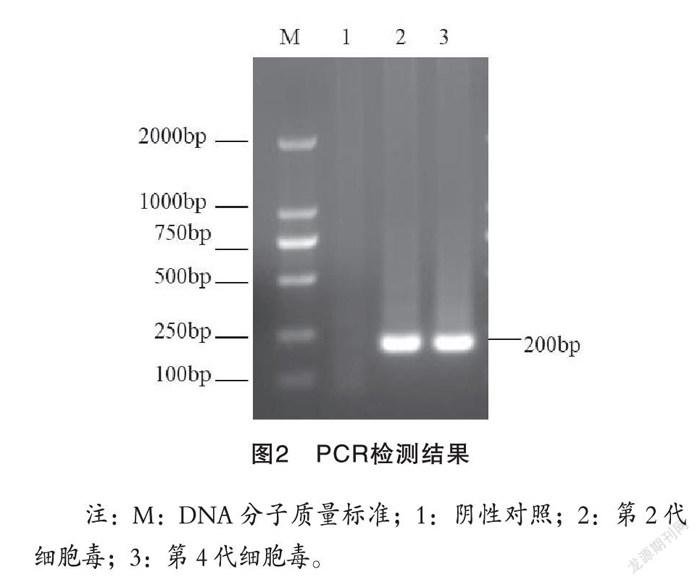
以提取的第2代和第4代細胞培養病毒DNA為模板,利用特異性引物進行PCR鑒定,瓊脂糖凝膠電泳結果顯示在約200 bp處出現目的條帶(圖2)。
注:M:DNA分子質量標準;1:陰性對照;2:第2代細胞毒;3:第4代細胞毒。
2.3 全基因組分段擴增和序列分析
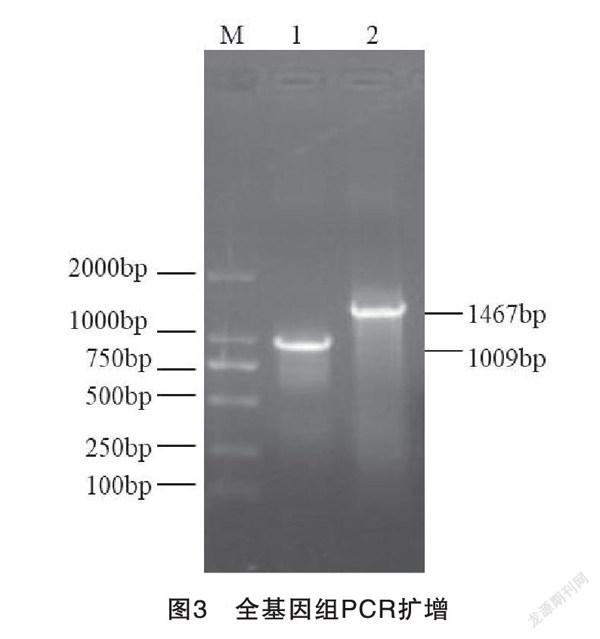
病毒全基因組PCR擴增結果顯示,擴增獲得兩條目的片段,且大小與預期結果相符(圖3)。將PCR產物連接到pMD18T載體中,測序結果顯示分離株CZ16基因組全長為2 298 bp, 其中VP1基因大小為1 350 bp,VP2基因大小為651 bp,VP3基因大小為366 bp,VP3基因完全包括在VP2基因中,基因組的兩端為5'和3'非編碼區,長度分別為358 bp和117 bp,與已知的CIAV基因組結構一致,均無缺失或插入。同源性分析表明CZ16與GenBank中收錄的CIAV流行株全基因組核苷酸同源性在98.61%~99. 56%之間。CZ16 VP1氨基酸序列與標準毒株Cux-1有12個氨基酸差異,而與中國毒株SC-MZ只有1個氨基酸差異。氨基酸序列分析還發現了CZ16與其它36個毒株VP1氨基酸序列的一些變異位點,這些變異位點主要分布于VP1基因的aa 136~157、aa 357~376及aa 436~448區域(圖4)。A6AD6A04-CE0E-4BF6-8BBB-8B1CF407BF53
2.4 分離株遺傳進化分析
分離株CZ16與其它GenBank已發表的CIAV毒株全基因組序列遺傳進化分析結果顯示, CZ16與亞洲流行毒株親源關系較近,其中與中國分離株GD-102(KU050677.1)關系最近,與澳大利亞的U65414.1關系最遠(圖5)。
3 討論
雞傳染性貧血病對雞群的主要危害是產生免疫抑制,繼而造成雞群易感其它病毒和細菌,使得疫病發生更加復雜[7]。雞傳染性貧血病是家禽重要的垂直傳播性疫病,嚴重影響家禽養殖,特別是種業生產。目前,種業已成為國家戰略性和基礎性核心產業,種源質量直接關系到家禽業健康發展的未來,對垂直傳播性疫病進行研究是保障家禽種源質量的核心和關鍵。由于CIAV容易與其它病毒和細菌混合感染,所以病原分離較困難。本研究基于CIAV易感細胞MDCC,成功分離獲得一株CIAV,并對分離株基因組序列進行分析。
基因序列分析結果表明,分離株CZ16與GenBank中收錄的CIAV流行株全基因組核苷酸同源性在98.61%以上,說明該分離株變異很小。VP1氨基酸序列分析結果表明變異位點散在分布于基因中,但主要還是集中在關鍵的氨基酸區域,特別是aa 136~157高變區域。VP1基因編碼CIAV的衣殼蛋白,也是病毒的主要抗原基因,它的變異可能與病毒和宿主相互作用有關,但具體變異機制還需要進一步研究。已有研究表明CIAV VP1蛋白第394位氨基酸是決定病毒致病性強弱的關鍵位點,谷氨酰胺(Q)代表強感染性,組氨酸(H)代表弱感染性[8]。本研究中,分離株CZ16 VP1蛋白第394位氨基酸是Q,說明該分離株是強感染性毒株,但其對雞的致病性還需要動物實驗進一步證實。系統發育進化樹分析結果顯示,所比對的37個CIAV毒株既有來自中國、韓國、日本、馬來西亞等亞洲毒株,也有美國、德國等歐美來源毒株,它們分布在各個進化分支中,沒有表現出明顯的地域性。本研究中,分離株CZ16與國內分離株GD-102關系最近,與澳大利亞分離株U65414.1關系最遠。
本研究成功分離到一株CIAV,并對其全基因組序列進行系統分析,為更全面了解CIAV流行規律、致病性提供材料,同時為雞傳染性貧血病的有效防控奠定一定的基礎。
參考文獻:
[1] Fatoba AJ, Adeleke MA. Chicken anemia virus: A deadly pathogen of poultry [J]. Acta Virol, 2019, 63 (1):19-25.
[2] Li Y, Yan N, Wang Y, et al. Molecular evolution and pathogenicity of chicken anemia virus isolates in China[J]. Arch Virol, 2021, 166(2): 439-449.
[3] Luan Q, Jiang Z, Wang D, et al. A sensitive triple nanoparticle-assisted PCR assay for detection of fowl adenovirus, infectious bursal disease virus and chicken anemia virus [J]. J Virol Methods, 2022, 303:114499.
[4] 劉婉思,高宏雷,秦立廷,等.1株野鴨源雞貧血病病毒的分離鑒定及其編碼區基因序列分析[J].中國獸醫學報,2013,33(05): 654-658.
[5] Rosario K,Breitbart M, Harrach B, et al. Revisiting the taxonomy of the family Circoviridae: establishment of the genus Cyclovirus and removal of the genus Gyrovirus [J]. Arch Virol, 2017, 162(5):1447-1463.
[6] Li Y, Fang L, Cui S, et al. Genomic Characterization of Recent Chicken Anemia Virus Isolates in China [J]. Front Microbiol, 2017, 8:401.
[7] Gimeno IM, Schat KA. Virus-Induced Immunosuppression in Chickens [J]. Avian Dis, 2018, 62(3):272-285.
[8] Eltahir YM, Qian K, Jin W, et al. Molecular epidemiology of chicken anemia virus in commercial farms in China [J]. Virol J, 2011,8:145.
Isolation and Complete Genomic Sequence Analysis of
a Chicken Infectious Anemia Virus Strain
HU Feng1, ZHU Tong1, GAO Yuehua1, HUANG Bing1, SONG Minxun1,
LIU Dandan1, WANG Jianhua2, LI Yufeng2
(1.Institute of Poultry Science, Shandong Academy of Agricultural Sciences / Shandong Provincial Key Laboratory of Immunity and Diagnosis of Poultry Diseases, Jinan ?250100,China;A6AD6A04-CE0E-4BF6-8BBB-8B1CF407BF53
2. Shandong Hekangyuan Biological Breeding Co., Ltd, Jinan ?250000,China)
Abstract: Chicken infectious anemia virus (CIAV) strain CZ16 was successfully isolated on MDCC-MSB1 cells from chicken flock suspicious of infected CIAV, and identified by PCR and gene sequence analysis. The complete genomic sequence of the virus was amplified by PCR, cloned, and sequenced. The results showed that the whole genome of CZ16 was comprised of 2298 nt, and the length of VP1, VP2, and VP3 gene was 1350,651 and 366 bp, respectively. The length of 5' and 3' untranslated regions was 358 bp and 117 bp, respectively. The nucleotide homology of complete genomic sequences between CZ16 strain and other CIAV isolates ranged from 95.6%~99.6%. Based on phylogenetic analysis of the complete genomic nucleotide sequence of CZ16 strain and other CIAV isolates, the phylogenetic tree showed a relatively close genetic distance from the internal strain GD-102, and a far genetic distance from the external strain (U65414.1) of Australia. A CIAV stain was isolated successfully and analyzed, which provided material for further study.
Keywords: Chicken infectious anemia virus;Isolation;complete genome;Phylogenetic analysisA6AD6A04-CE0E-4BF6-8BBB-8B1CF407BF53

