在中國大豆品種上創建ALSV誘導的基因沉默體系
董永鑫,衛其巍,2,洪浩,黃瑩,趙延曉,馮明峰,竇道龍,徐毅,陶小榮
在中國大豆品種上創建ALSV誘導的基因沉默體系
董永鑫1,衛其巍1,2,洪浩1,黃瑩1,趙延曉1,馮明峰1,竇道龍1,徐毅1,陶小榮1
1南京農業大學植物保護學院/農業農村部大豆病蟲害防控重點實驗室,南京 210095;2四川省達州市農業科學研究院,四川達州 635000
【】在中國大豆品種上建立以蘋果潛隱球形病毒(apple latent spherical virus,ALSV)為載體的基因沉默體系,為中國大豆品種的基因功能和遺傳育種提供一種簡便、省時、易操作的技術體系。構建以農桿菌介導接種的ALSV病毒侵染性克隆載體。從大豆品種威廉姆斯82(Williams 82)中特異擴增327 bp的八氫番茄紅素脫氫酶()基因cDNA片段,插入病毒載體pALSV2。通過農桿菌浸潤法將病毒載體導入模式植物本氏煙(),17 d后富集病毒粒子,摩擦接種大豆第一輪真葉,以接種病毒空載作為對照組,持續觀察測試大豆植株系統葉表型,并結合逆轉錄-聚合酶鏈式反應(RT-PCR)或熒光定量PCR(qRT-PCR)檢測ALSV衣殼蛋白基因()和的表達水平。ALSV﹕GmPDS接種大豆品種威廉姆斯82和南農1138-2,20 d后,前者系統葉未見白化表型,而后者系統葉出現明顯的白化表型;qRT-PCR檢測結果表明,發生白化的南農1138-2植株中的表達水平顯著降低,未出現白化表型的威廉姆斯82中的表達水平沒有出現顯著變化。在此基礎上,采用相同的方法,測試了ALSV在其他9種大豆品種中誘導的沉默效率,發現在南農47、安豆203、祥斗4號、中黃13、山寧29、齊黃34等大豆品種上接種ALSV﹕GmPDS后植株系統葉均產生白化表型,而菏豆12、中黃311和山寧16等3個品種的均不能誘導有效沉默。構建了農桿菌介導的ALSV病毒載體,利用本氏煙擴繁富集ALSV病毒,將提純的病毒粒體摩擦接種大豆真葉,在多個中國大豆品種上成功建立了基因沉默體系。
病毒誘導的基因沉默;大豆;蘋果潛隱球形病毒;大豆八氫番茄紅素脫氫酶基因
0 引言
【研究意義】大豆((L.) Merr.)起源于中國,已有五千年栽培歷史,是中國重要糧食作物之一,也是世界上最重要的豆類作物。大豆含有豐富的植物蛋白質,是植物油和蛋白的重要來源,在世界各地廣泛種植。中國既是大豆原產地,又曾是全球大豆貿易最大出口國,然而由于國內對于大豆的需求與日俱增,中國的大豆產量難以滿足國內日益增多的消費,超過八成的大豆消費依賴進口,目前,全球超過一半的大豆進口來自中國[1-2]。大豆遺傳轉化體系難度大,周期長,是阻礙大豆基因功能研究的主要瓶頸之一[3];此外,中國大豆品種多,在不同品種大豆上建立快速鑒定基因功能的手段和工具,對于進一步挖掘中國大豆重要農藝性狀基因,研究大豆品種中與產量和抗病相關的基因功能,提高中國大豆產量和質量,促進大豆產業發展具有重要的理論科學意義[4-5]。【前人研究進展】病毒誘導的基因沉默(virus-induced gene silencing,VIGS)是一種研究基因功能的重要方法,通過將插入植物cDNA片段的重組病毒載體接種植物,利用病毒產生的雙鏈RNA被植物中的RNA沉默元件識別,切割產生大量針對植物目的基因的小干擾RNA(small interference RNA),這些小干擾siRNA靶向植物內源目的基因mRNA,通過目的基因表達量降低后的植物表型或者生理指標的變化可以鑒定該基因的功能[6-7]。VIGS具有操作簡單、周期短、同時沉默多個靶標基因及不依賴植物遺傳轉化體系等優點,被廣泛用于不同植物基因功能的研究[8-10]。在大豆上的VIGS載體有菜豆莢斑駁病毒(bean pod mottle virus,BPMV)為基礎構建的沉默載體,但該病毒需要用粒子轟擊的方法將BPMV載體導入大豆的子葉中,或用體外轉錄的方法進行接種,方法復雜且BPMV侵染會在大豆上產生如褪綠、壞死等癥狀,影響表型觀察[11-12]。蘋果潛隱球形病毒(apple latent spherical virus,ALSV)載體也被報道用于沉默大豆基因[13-14]。ALSV屬于櫻桃銼葉病毒屬病毒(),具有直徑約25 nm的等軸形病毒顆粒,病毒粒體內包含2條單鏈基因組RNA(RNA1和RNA2)[15]。與其他報道的病毒載體相比,ALSV作為VIGS載體有幾個優點:第一,ALSV作為一種潛隱性病毒,在大部分寄主上不會產生嚴重的癥狀[13];第二,ALSV載體具有較高的穩定性,可以在被接種植物的整個生長周期持續存在[16];第三,ALSV的寄主范圍廣,除了模式植物擬南芥()[17]、本氏煙()[18]外,還可應用于辣椒()[19]、菎諾藜()[20]、豇豆()、黃瓜()、葡萄()和柑橘()等多個不同科植物的基因沉默[21-24]。【本研究切入點】目前,ALSV介導的VIGS體系只在國外多個大豆品種中進行了VIGS的測試[14],發現在國外不同大豆品種中ALSV沉默靶標基因的效率存在顯著差異,只有部分品種可以誘導高效的基因沉默。中國是大豆的起源地,大豆品種種類多,但目前還沒有利用ALSV誘導的基因沉默體系對中國大豆品種進行沉默效率的評估,到底哪些品種能夠利用該病毒載體進行高效的靶標基因沉默仍不清楚。【擬解決的關鍵問題】本研究基于ALSV病毒載體能夠侵染大豆誘導基因沉默的能力,擬利用ALSV病毒載體在中國大豆品種中建立有效的基因沉默體系,通過構建農桿菌介導法接種的ALSV載體,在本氏煙上擴繁病毒,快速分離高濃度的病毒粒子,建立穩定的病毒接種大豆方法,在中國多個大豆品種上建立高效的基因沉默體系,為進一步研究中國大豆品種的基因功能和重要農藝性狀基因挖掘提供重要的研究工具。
1 材料與方法
1.1 植物材料和生長環境
大豆種子:南農1138-2由南京農業大學大豆工程改良中心李凱教授提供,齊黃34、安豆203、南農47、祥斗4號、中黃13、山寧29、威廉姆斯82(Williams 82)、山寧16、菏豆12和中黃311等10種大豆品種由南京農業大學植物保護學院葉文武副教授惠贈。
本氏煙種子:由南京農業大學分子植物病毒學實驗室保存。
本氏煙和大豆的生長環境均為溫度25℃,日長16 h/黑暗8 h的隔離溫室。本氏煙基因沉默生長環境為溫度22℃,日長8 h/黑暗16 h的隔離溫室。
1.2 序列和引物設計
運用primer premier 5.0軟件設計引物(表1)。

表1 本研究所使用的引物
劃線部分序列為載體pALSV2上的序列The underlined sequence from pALSV2
1.3 ALSV-VIGS載體的構建
1.3.1 農桿菌介導的ALSV載體
參考Huang等[17]方法構建ALSV RNA1和RNA2的侵染性克隆載體pALSV1、pALSV2(圖1)和pALSV2-CH42(Chlorata42),pALSV2-CH42用于指示ALSV病毒在本氏煙中的侵染情況。
1.3.2 pALSV2-GmPDS載體構建
1.3.2.1 GmPDS沉默片段克隆 用NCBI(http://www. ncbi.nlm.nih.gov)已公布的擬南芥PDS基因(GenBank登錄號為NM_001340908.1)序列進行大豆序列blast,選取大豆PDS基因的3條同源基因,序列號分別為M64704.1、XM_003538489.4和NM_001249840.2。3個同源大豆PDS基因用DNAMAN軟件進行多序列比對(圖1),選取編碼區保守區域1 520—1 846 nt為目的片段。選用(GenBank登錄號AW350943.1)作為內參基因。以Williams 82大豆品種的葉片為植物材料,利用天根生化科技(北京)有限公司的總RNA抽提試劑盒提取RNA,具體操作方法依據產品說明書進行。采用Promega反轉錄試劑盒合成大豆cDNA。以大豆cDNA為模板,按照Phanta Max Super-Fidelity DNA Polymerase反應體系,用特異性引物(表1)進行片段擴增(圖1)。擴增反應條件為95℃ 3 min;95℃ 15 s,55℃ 15 s,72℃ 30 s,35個循環;72℃ 5 min。
1.3.2.2 pALSV2-GmPDS重組載體的構建 pALSV2載體經Ⅰ酶切后,用瓊脂糖電泳檢測(圖1),參考生工生物工程(上海)股份有限公司的DnaSpin柱式DNA膠回收試劑盒步驟進行純化回收,將GmPDS RT-PCR產物按照同樣的純化方法純化后,通過南京諾唯贊生物科技有限公司的同源重組試劑盒與Ⅰ酶切的pALSV2載體進行同源重組,重組產物轉化大腸桿菌DH,挑取陽性克隆,經菌落PCR檢測后,進行測序鑒定,驗證正確的克隆提取質粒保存。
1.4 農桿菌浸潤
將pALSV1、pALSV2、pALSV2-CH42、pALSV2- GmPDS以及病毒基因沉默抑制子P19(tomato bushy stunt virus P19)等載體通過電擊法分別轉入農桿菌GV3101中,在含有25 mg·L-1利福平和50 mg·L-1卡那霉素的YEP固體培養基上培養,挑取單菌落,在YEP液體培養基中28℃過夜培養,5 000 r/min離心8 min,棄上清液,加入適量的農桿菌浸潤緩沖液(10 mmol·L-1MES、10 mmol·L-1MgCl2和150 μmol·L-1乙酰丁香酮)重新懸浮菌體,在28℃恒溫培養箱避光放置3 h后,調菌液濃度至OD600=1,將攜帶pALSV1與pALSV2、pALSV2- GmPDS、pALSV2-CH42的農桿菌等比例混合,同時每個組合中加入等比例的含有P19的農桿菌,注射6—8葉期的本氏煙。由pALSV1和pALSV2構成的病毒命名為ALSV,由pALSV1、pALSV2-AtCH42構成的病毒命名為ALSV﹕CH42,由pALSV1、pALSV2-GmPDS構成的病毒命名為ALSV﹕GmPDS。
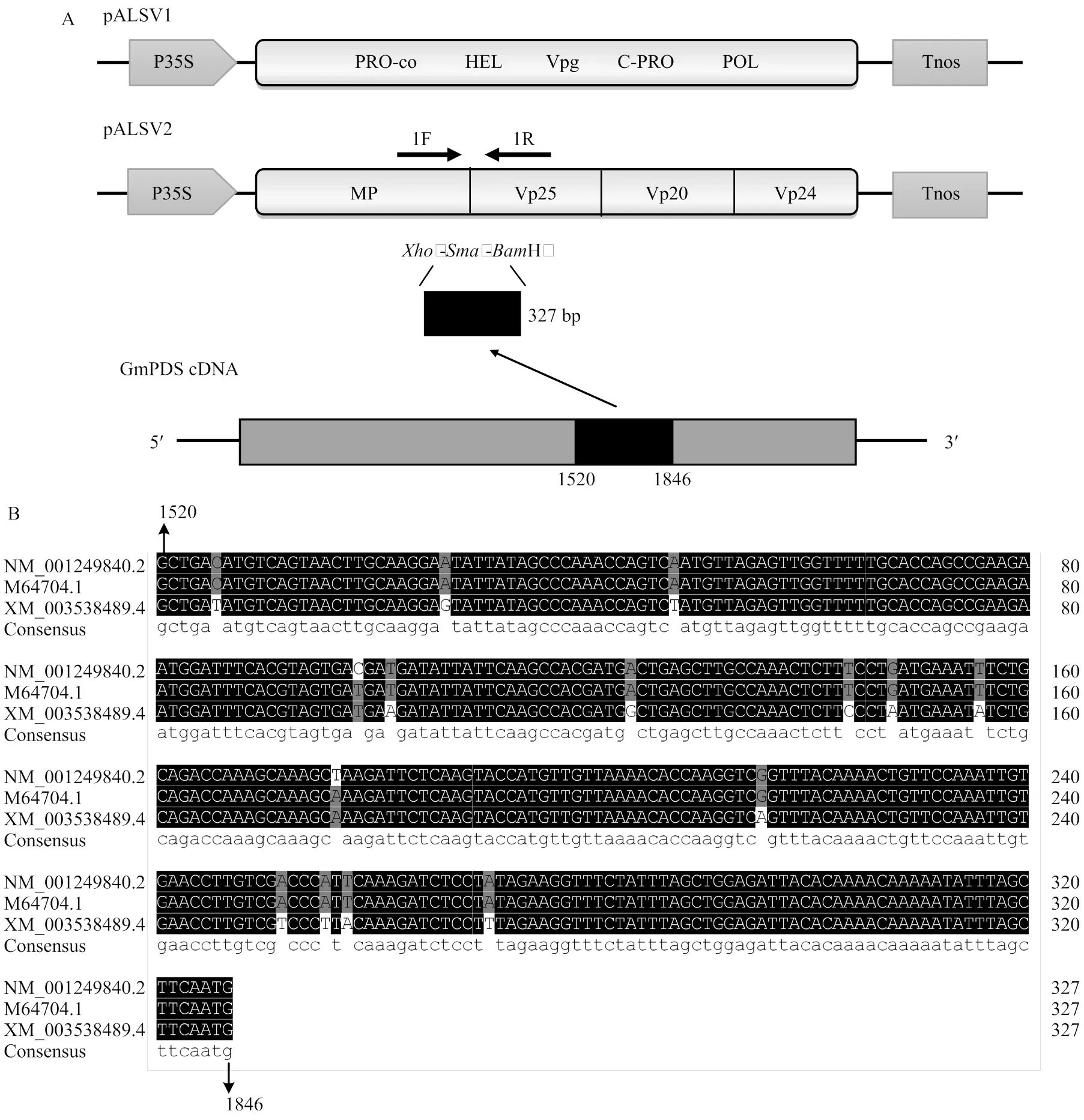
A:pALSV2-GmPDS載體構建示意圖。P35S:35S啟動子;Tnos:終止子;PRO-co:蛋白酶輔因子;HEL:NTP結合解旋酶;C-PRO:半胱氨酸蛋白酶;POL:RNA聚合酶;MP:42K運動蛋白;Vp25、Vp20和Vp24:衣殼蛋白。B:GmPDS cDNA擴增區域序列比對
1.5 RT-PCR驗證ALSV、ALSV﹕CH42、ALSV﹕GmPDS侵染本氏煙
分別對表達pALSV1、pALSV2和P19,pALSV1、pALSV2-CH42和P19,pALSV1、pALSV2-GmPDS和P19的本氏煙提取植物系統葉總RNA,反轉錄為cDNA,用表1中的相應引物進行PCR擴增,PCR產物經1%瓊脂糖電泳檢測。
1.6 病毒粒子富集
利用侵染ALSV空載和ALSV﹕GmPDS的本氏煙系統侵染葉進行病毒粒子富集。具體步驟如下(圖2):
1)農桿菌浸潤17 d后,稱取0.4 g系統侵染葉片,在研缽中加入液氮充分研磨成粉末,加入800 μL 0.01 mol·L-1pH=7.5的PB緩沖液,混勻后置冰上孵育30 min;
2)4℃,8 500 r/min離心10 min后取上清,加入相應體積的40% PEG/NaCl溶液(每100 μL上清液加入23 μL 40% PEG/NaCl),混勻后置冰上孵育1 h;
3)4℃,13 000 r/min離心15 min后棄上清,用100 μL 0.01 mol·L-1pH=7.0的PB緩沖液懸浮沉淀,即得到富集的病毒粒子。通過OD260測得病毒粒子濃度,將病毒終濃度調至2 μg·μL-1,4℃保存備用。
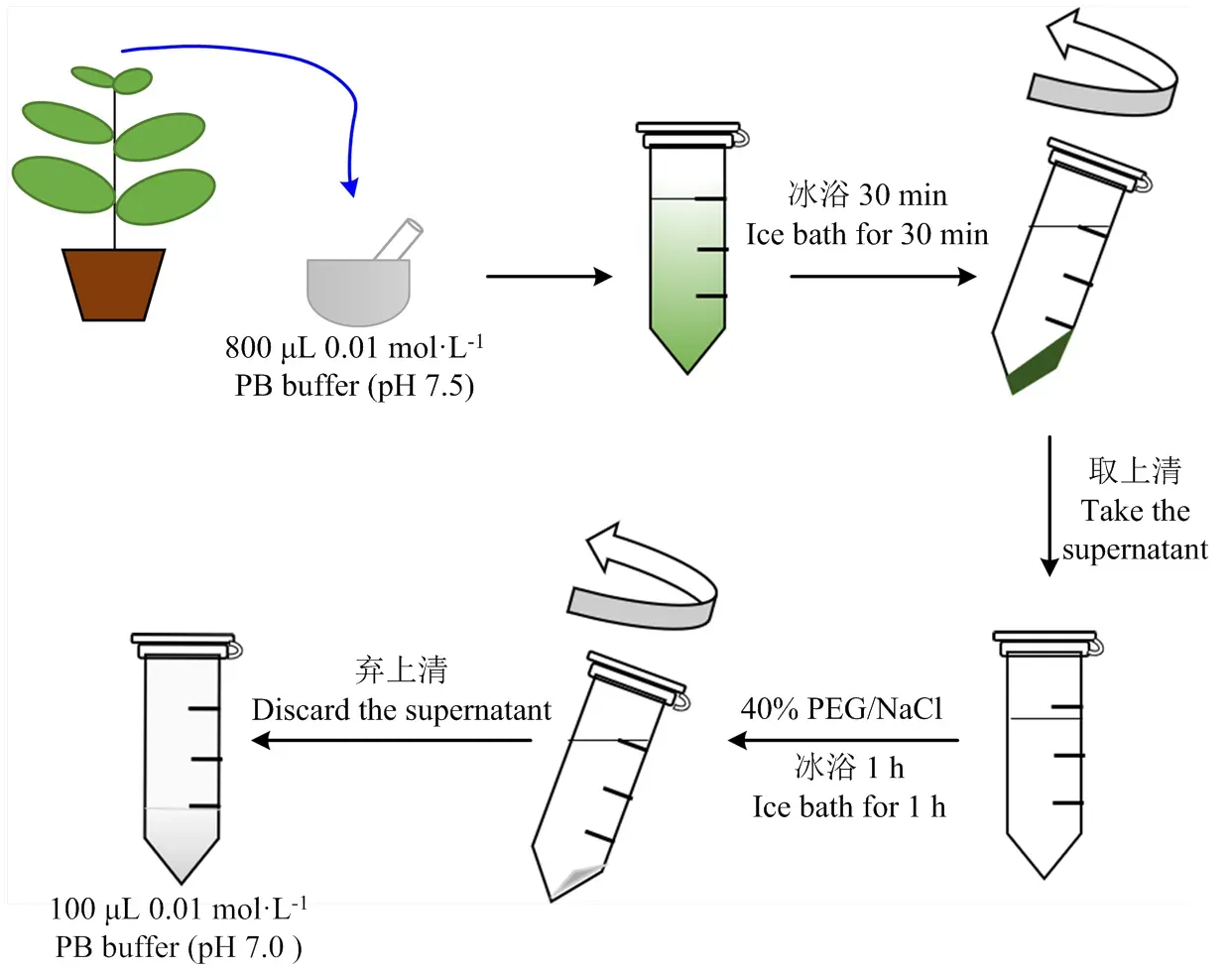
圖2 本氏煙中ALSV病毒粒子的提取和富集流程
1.7 ALSV病毒粒子摩擦接種大豆
選擇8—10日齡大豆幼苗,將金剛砂均勻撒在大豆葉片上,用移液槍吸取20 μL提取的病毒粒子,用一次性手套均勻涂抹在大豆真葉葉片上,每片真葉摩擦40 μg病毒。
1.8 qRT-PCR檢測大豆GmPDS的沉默效率
當接種ALSV﹕GmPDS的大豆系統葉出現白化表型時,采集大豆頂部白化葉提取總RNA,同時用摩擦接種ALSV空載體的大豆系統葉作為對照,按照南京諾唯贊生物科技有限公司的快速反轉錄試劑盒合成cDNA,再用ChamQ Universal SYBR qPCR Master Mix定量試劑盒進行定量PCR,用作為內參基因,按照表1中的相應引物檢測的沉默效率。
2 結果
2.1 農桿菌介導的ALSV侵染性克隆構建及通過侵染本氏煙擴繁病毒
ALSV載體在初始感染階段病毒RNA積累量少,需要接種到菎諾藜上進行擴增繁殖[18, 25]。為了能夠在實驗室常用病毒寄主本氏煙中進行ALSV的擴增繁殖,人工合成了ALSV基因組RNA1和RNA2的全長cDNA,將病毒2條基因組分別克隆到pCB301-2x35S- RZ-NOS,構建了農桿菌介導的pALSV1和pALSV2的侵染性克隆載體(圖1)[17]。將含有pALSV1、pALSV2和P19載體的農桿菌懸浮液等比例混合后浸潤本氏煙,同時用含空載體的農桿菌浸潤本氏煙作為陰性對照。在農桿菌接種后17 d,受感染的植株沒有出現明顯的病毒癥狀(圖3-A);RT-PCR檢測結果顯示,pALSV1、pALSV2和P19共處理的本氏煙的系統葉可檢測到ALSV病毒的表達,表明通過農桿菌介導的ALSV載體不誘導任何病癥但可成功侵染本氏煙(圖3-B)。
為了測試農桿菌介導的ALSV載體是否能夠在本氏煙上誘導基因沉默,首先選取了(),該基因編碼葉綠素產生所必需的鎂螯合酶復合物的組分之一,作為指示基因被廣泛應用于病毒誘導的基因沉默體系研究[26]。在pALSV2載體上插入了的cDNA片段,構建成pALSV2-CH42載體[17]。將攜帶pALSV1、pALSV2-CH42和P19的農桿菌等比例混合后注射本氏煙,浸潤17 d后,pALSV1、pALSV2-CH42和P19共表達的植株系統葉片出現黃化表型,結果表明,構建的ALSV載體能夠在本氏煙上誘導有效的基因沉默(圖3-C)。進一步測試不加沉默抑制子P19時,將攜帶pALSV1和pALSV2-CH42農桿菌等比例混合后注射本氏煙能否實現系統侵染。結果顯示,在不加入P19時,系統葉并沒有出現黃化的表型(圖3-C),表明不加基因沉默抑制子P19,ALSV載體不能有效侵染本氏煙。以上研究結果表明構建的ALSV載體可通過農桿菌浸潤的方式侵染本氏煙,并能夠誘導高效的基因沉默,基因沉默抑制子P19對于農桿菌介導的ALSV侵染本氏煙是必需的。
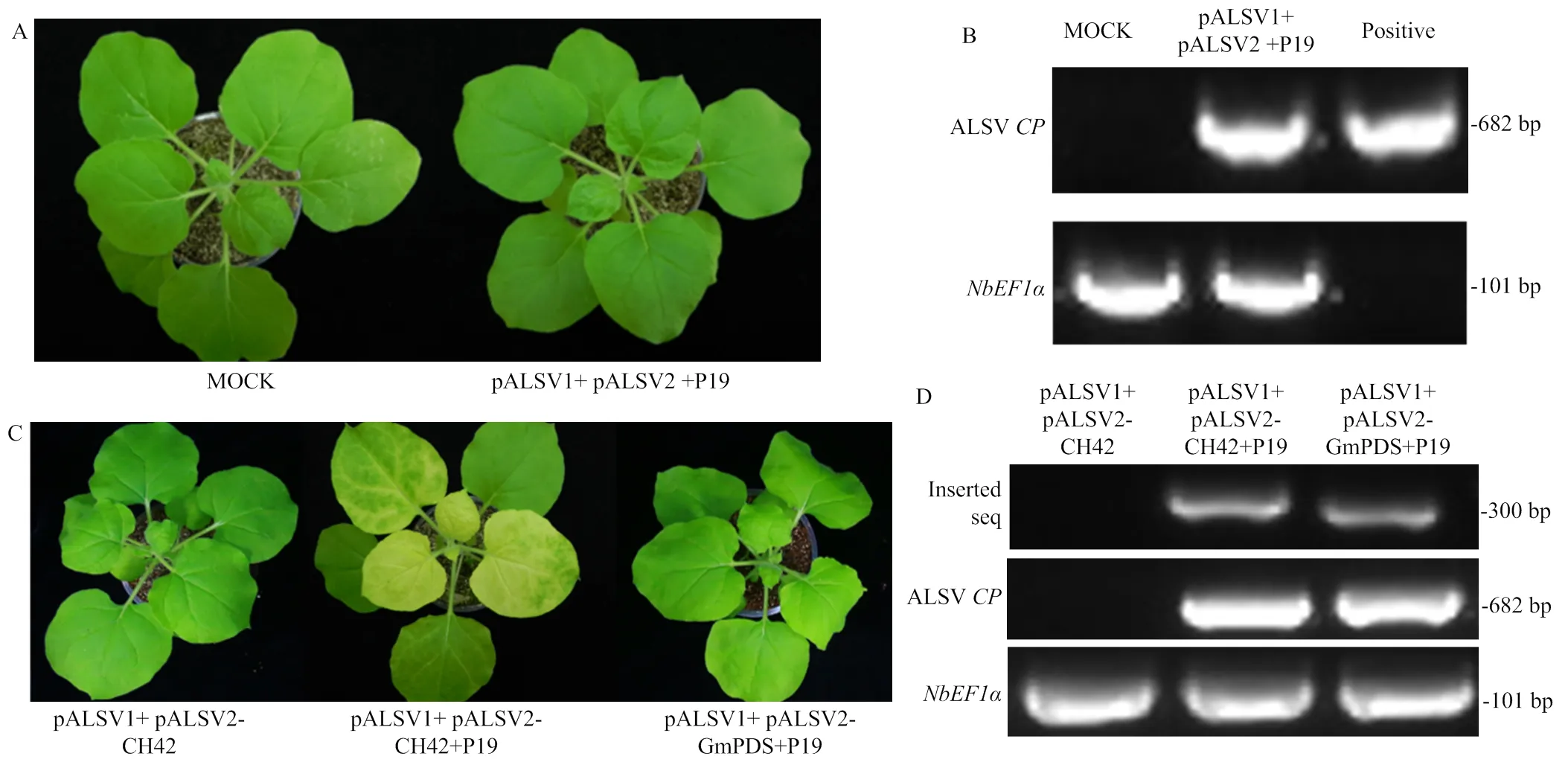
A:農桿菌介導ALSV侵染本氏煙。將含有pALSV1和pALSV2空載體、pALSV2-CH42、pALSV2-GmPDS和P19的農桿菌1﹕1﹕1混合后注射本氏煙,在侵染17 d后,對侵染植株的表型進行拍照。MOCK:空載體對照;P19:基因沉默抑制子;B:RT-PCR檢測本氏煙系統葉ALSV CP積累量;C:農桿菌介導的ALSV﹕GmPDS侵染本氏煙以及P19對ALSV侵染本氏煙的作用測定,CH42:葉綠素合成相關基因;D:RT-PCR檢測C圖ALSV插入片段CH42、GmPDS和CP在本氏煙系統葉中的表達
隨后,將327 bp大豆的cDNA片段插入pALSV2,構建pALSV2-GmPDS(圖1)。通過pALSV2-GmPDS、pALSV1和P19農桿菌懸浮液等比例混合后注射本氏煙,以pALSV1、pALSV2-CH42和P19農桿菌懸浮液浸潤的植株作為陽性對照。待浸潤ALSV﹕CH42的植株系統葉黃化后,采集ALSV﹕GmPDS侵染本氏煙植株的系統葉,經RT-PCR驗證,ALSV﹕GmPDS本氏煙成功實現系統侵染(圖3-D)。
2.2 ALSV載體在南農1138-2大豆品種上的基因沉默體系的建立
為了在大豆上建立基因沉默的體系,通過一種快速提純病毒的方法,對ALSV空載體和ALSV2﹕GmPDS的病毒粒子進行富集(圖2)。將富集后的病毒粒子,摩擦接種到南農1138-2和威廉姆斯82大豆品種的第一輪真葉上,每片真葉摩擦40 μg提純的病毒粒體。
以提純的ALSV空載體病毒粒子摩擦接種的大豆為對照組,以ALSV﹕GmPDS病毒粒子摩擦接種的大豆為試驗組。結果顯示,在南農1138-2大豆品種上接種ALSV﹕GmPDS病毒粒子20 d后,在接種植株第二輪和第三輪以上的真葉上均出現了完全白化的表型,而接種ALSV空載病毒粒子的對照組大豆則沒有出現白化(圖4-A—圖4-C),接種的6棵植物中全部出現白化表型(表2)。為明確ALSV﹕GmPDS在1138-2大豆品種中誘導的沉默效率,采集ALSV空載體大豆系統葉片和ALSV﹕GmPDS誘導白化的葉片,提取總RNA,qRT-PCR檢測的表達水平。結果顯示,在ALSV﹕GmPDS處理的南農1138-2大豆品種中,與對照組相比,出現白化表型的表達水平顯著降低(<0.05)(圖4-D)。在威廉姆斯82大豆品種,接種的8棵威廉姆斯82中都沒有出現白化表型,qRT-PCR顯示并沒有被沉默(圖4-E和圖4-F)。對未出現沉默的植株進行RT-PCR檢測,發現ALSV病毒在威廉姆斯82中的病毒積累量非常低或不能侵染該大豆品種(圖5)。結果表明,利用ALSV在南農1138-2大豆品種上成功建立了基因沉默的體系(圖4)。
2.3 ALSV介導的基因沉默在中國其他9種大豆品種的沉默效率測定
利用ALSV在南農1138-2品種建立基因沉默體系的基礎上,進一步選擇了國內其他9種大豆品種包括齊黃34、安豆203、南農47、祥斗4號、中黃13、山寧29等進行了沉默效率的測定。通過前面在南農1138-2誘導基因沉默相同的策略對供試大豆進行了ALSV﹕GmPDS病毒接種。結果顯示,ALSV﹕GmPDS侵染2周后,在齊黃34、安豆203、南農47、祥斗4號、中黃13、山寧29等大豆品種中均誘導產生了白化的表型(圖6),齊黃34中8棵接種植株有3棵出現了白化,安豆203中8棵接種的植株有3棵出現白化,南農47中接種8棵植株有3棵出現白化,祥斗4號的8棵接種植株有4棵出現白化,中黃13中6棵接種植株有4棵出現白化,山寧29中6棵接種植株有4棵出現白化(表2)。經RT-PCR檢測,在接種的8棵齊黃34中,3棵出現白化表型的植株中都能檢測到病毒,而在其余未出現PDS沉默的植株中,并沒有檢測到病毒侵染,表明未出現PDS沉默的植株是ALSV病毒未能夠成功侵染引起的(圖7-A)。此外,在ALSV﹕GmPDS接種的國內大豆品種菏豆12、中黃311、山寧16上,病毒接種后均沒有產生白化表型(圖6)。經RT-PCR檢測后發現ALSV可以在這些大豆品種產生系統侵染,表明ALSV在這些大豆品種中不足以誘導PDS的沉默(圖7-B—圖7-D)。這些研究結果表明,ALSV介導的基因沉默體系在中國不同大豆品種的上誘導的基因沉默表型有差異,ALSV在一些大豆品種可以實現系統侵染,但是沉默效率存在差異。
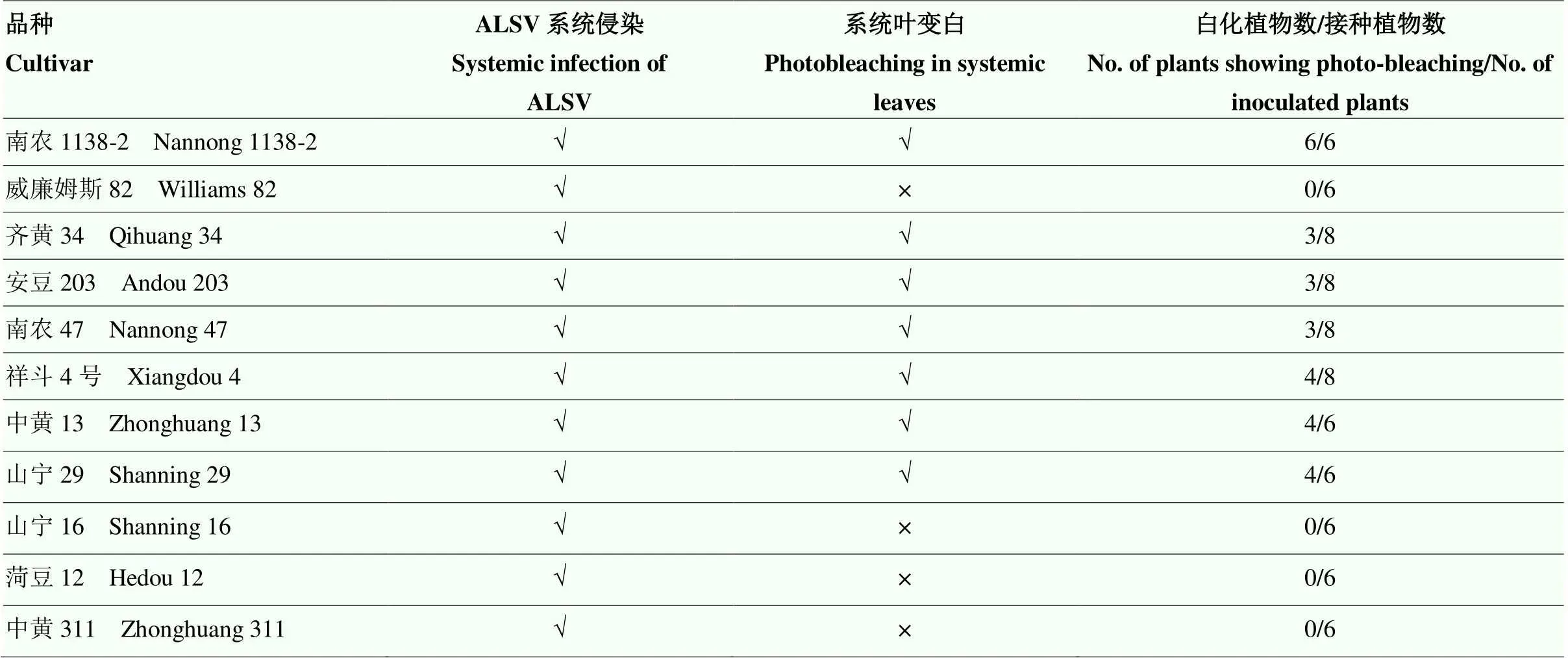
表2 ALSV誘導的基因沉默在11個大豆品種上的沉默效率
√:ALSV能夠系統侵染/ALSV﹕GmPDS侵染后能夠誘導系統葉變白;×:ALSV不能系統侵染/ALSV﹕GmPDS侵染后不能夠導致系統葉變白
√: ALSV can infect systematically/ALSV:GmPDS can cause photo-bleaching; ×: ALSV can’t infect systematically/ALSV:GmPDS can’t cause photo-bleaching

A:ALSV空載體病毒粒體接種南農1138-2 20 d后的表型;B:ALSV﹕GmPDS病毒粒體接種南農1138-2 20 d后的表型;C:A、B圖中對應南農1138-2系統葉GmPDS沉默表型,左邊為接種ALSV空載對照,右邊為接種ALSV﹕GmPDS上部白化葉片;D:qRT-PCR檢測ALSV空載體和ALSV﹕GmPDS病毒粒體處理的南農1138-2大豆品種系統葉中PDS基因的沉默效率(n=3,**:p<0.01);E:ALSV﹕GmPDS接種威廉姆斯82大豆品種20 d后的表型;F:qRT-PCR檢測ALSV空載體和ALSV﹕GmPDS病毒粒體處理的威廉姆斯82大豆品種系統葉中PDS基因的沉默效率(n=3,ns:p>0.05)
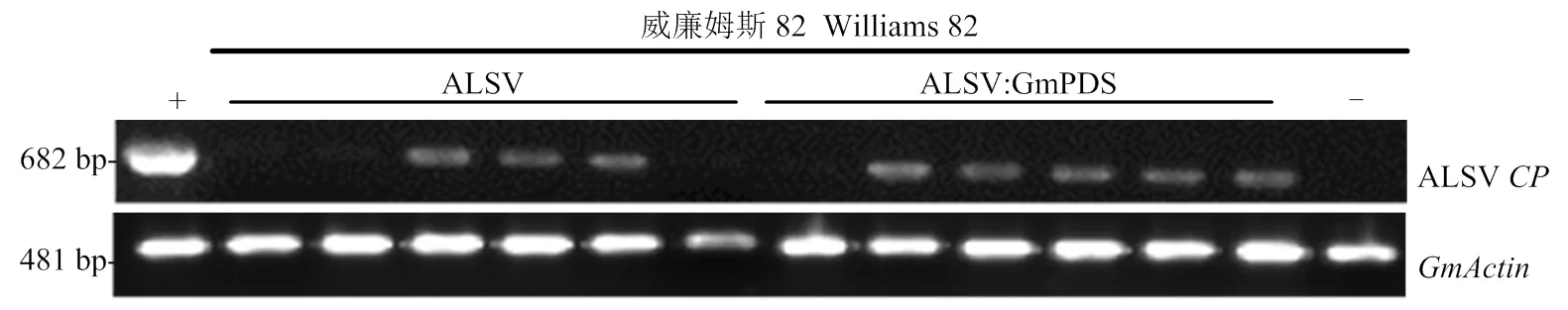
+:用已白化的南農1138-2系統葉cDNA為陽性對照;—:健康大豆cDNA為陰性對照

ALSV﹕GmPDS病毒純化粒體接種國內其他九種不同大豆品種20 d后的表型。ALSV﹕GmPDS在齊黃34、南農47、祥斗4號、中黃13、山寧29以及安豆203系統葉誘導白化表型;山寧16、菏豆12以及中黃311系統葉未能誘導出現白化表型
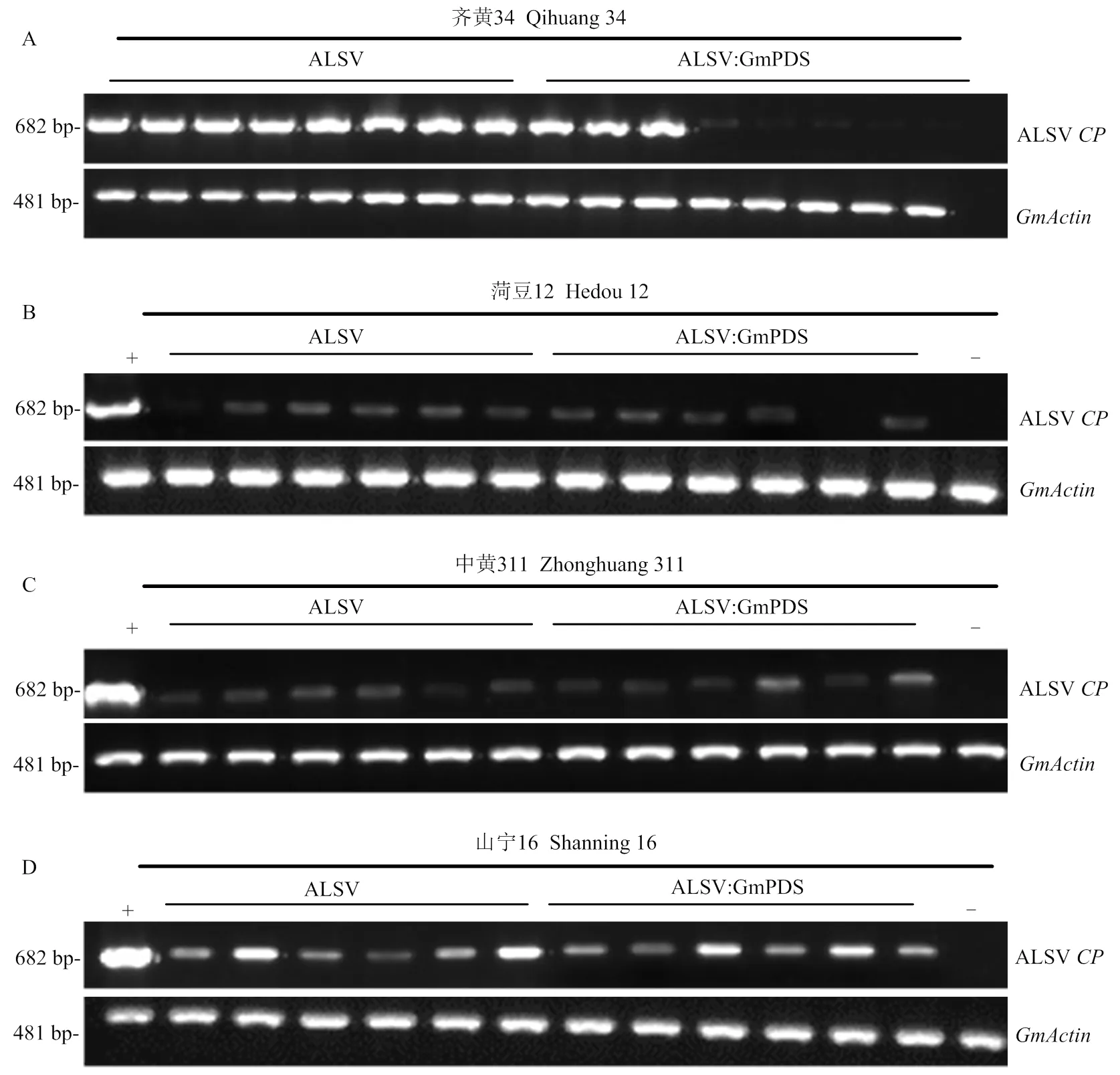
A:接種ALSV空載體的齊黃34在8棵植物系統葉都能夠檢測到病毒CP RNA的積累,接種ALSV﹕GmPDS的齊黃34在8棵植物有3棵發生沉默的植物系統葉能檢測到CP;B:ALSV空載體和ALSV﹕GmPDS接種的菏豆12檢測不到或只能檢測到很低的CP RNA積累量;C:ALSV空載體和ALSV﹕GmPDS接種的中黃311只能檢測到很低的CP積累量;D:ALSV空載體和ALSV﹕GmPDS接種的山寧16只能檢測到很低的CP積累量。+:用已白化的南農1138-2系統葉cDNA為陽性對照,—:健康大豆cDNA為陰性對照
3 討論
3.1 利用ALSV載體在中國多種大豆品種中成功建立有效的基因沉默體系
目前廣泛應用于大豆基因沉默的載體主要是BPMV,該載體在大豆上應用方法較為復雜且會導致植物產生癥狀[27-29]。原始的ALSV需摩擦到菎諾藜上,并且要進行基因槍轟擊到大豆上進行基因沉默,這一操作對一般的實驗室較難熟練掌握。本研究將ALSV改造為農桿菌介導的病毒載體,極大地方便了病毒誘導基因沉默的操作。基于ALSV病毒在植物中積累量少的特點,創立了富集本氏煙中擴繁的ALSV病毒粒子的方法,用富集的病毒粒子接種大豆。改進的方法在大豆上誘導的基因沉默效果穩定,可重復性好。這些改進顯著簡化了ALSV用于大豆基因沉默和基因功能的過程。本研究發現使用基因沉默抑制子P19對于農桿菌介導的ALSV在本氏煙上的侵染是必需的,ALSV在植物中的積累量較低,基因沉默抑制子P19的加入顯著促進了ALSV在本氏煙的侵染和積累,這為大量富集病毒粒子用于后續有效接種大豆創造了有利條件。基于此,本研究利用ALSV載體在南農1138-2、安豆203、齊黃34等中國多種大豆品種中建立了有效的基因沉默體系。
3.2 ALSV在不同大豆品種中誘導基因的沉默效率存在差異
發現ALSV-VIGS載體在不同大豆品種中沉默基因的效率存在顯著差異,這種差異可能是由于大豆的基因型決定的。對于Williams 82、菏豆12、中黃311、山寧16這4個品種,ALSV雖然可以系統侵染,但是沒有出現白化表型,RT-PCR結果表明,這些大豆品種系統葉中,病毒的積累量與對照組(1138-2品種)相比顯著較低,這暗示ALSV并不能很好地侵染一些大豆品種,或ALSV在這些品種中較難誘導基因沉默。之前報道中,ALSV在不同的國外大豆品種上誘導基因沉默效率也有較大的差異[14]。此外,為了排除PDS在不同大豆品種中沉默效率存在差異是由沉默片段的保守性差異造成的,本研究擴增了11個大豆品種的沉默區域進行測序,測序結果顯示不同大豆品種之間并無明顯差異。對于能夠產生白化表型的大豆品種,沉默效率也有差異,其中比較敏感的南農1138-2品種,沉默效率可以達到100%,而對于其他沉默效率稍低的大豆品種,未來可以對這個體系進行進一步優化,例如通過提高病毒接種濃度和接種量、嘗試不同的沉默片段長度或調整植物生長溫度[30],以提高ALSV在這些大豆品種的基因沉默效率。因此,利用該病毒在新的大豆品種中進行基因沉默和功能研究時,應先對供試大豆品種進行基因沉默效率的測定。
4 結論
將ALSV載體改造為農桿菌浸潤的病毒載體,以常用的本氏煙作為繁殖寄主,根據ALSV病毒在植物中積累量少的特點,創造性地采用了富集本氏煙中擴繁的ALSV病毒粒子的方法,創新了基于ALSV載體誘導的大豆基因沉默體系,并應用該體系在中國10種大豆品種上進行了沉默測試,明確了該病毒載體在國內不同大豆品種上的沉默效率。
[1] 吳汶珊. 中國大豆對外貿易的發展現狀及對策. 大陸橋視野, 2021(6): 88-89.
WU W S. Development status and countermeasures of Chinese soybean foreign trade. New Silk Road Horizon, 2021(6): 88-89. (in Chinese)
[2] 吳曰程, 王玉斌. 中國轉基因大豆進口及其影響分析. 大豆科學, 2019, 38(4): 635-643.
WU Y C, WANG Y B. Import of transgenic soybean in China and its impact analysis. Soybean Science, 2019, 38(4): 635-643. (in Chinese)
[3] 劉曉彬, 劉娜, 李福寬, 吳立柱, 張潔, 王冬梅. TRV介導的大豆基因瞬時沉默體系的建立. 中國農業科學, 2015, 48(12): 2479-2486.
LIU X B, LIU N, LI F K, WU L Z, ZHANG J, WANG D M. Establishment of TRV-mediated transient gene-silencing system in soybean.Scientia Agricultura Sinica, 2015, 48(12): 2479-2486. (in Chinese)
[4] 王爽, 郭兵福, 郭勇, 張麗娟, 金龍國, 楊慧, 邱麗娟. 病毒誘導的基因沉默(VIGS)技術及其在大豆基因功能研究和育種中的應用潛力. 大豆科學, 2016, 35(4): 536-540.
WANG S, GUO B F, GUO Y, ZHANG L J, JIN L G, YANG H, QIU L J. Virus-induced gene silencing and its usage in soybean functional genomics. Soybean Science, 2016, 35(4): 536-540. (in Chinese)
[5] 李敏, 關博文, 楊學, 陳慶山, 張睿, 武小霞, 金慧, 吳玉娥. 大豆種質資源遺傳多樣性分析. 農業科技通訊, 2021(11): 4-8.
LI M, GUAN B W, YANG X, CHEN Q S, ZHANG R, WU X X, JIN H, WU Y E. Genetic diversity analysis of soybean germplasm resources. Bulletin of Agricultural Science and Technology, 2021(11):4-8. (in Chinese)
[6] LANGE M, YELLINA A L, ORASHAKOVA S, BECKER A. Virus-induced gene silencing (VIGS) in plants: an overview of target species and the virus-derived vector systems. Methods in Molecular Biology, 2013, 975: 1-14.
[7] RAMEGOWDA V, MYSORE K S, SENTHIL-KUMAR M. Virus-induced gene silencing is a versatile tool for unraveling the functional relevance of multiple abiotic-stress-responsive genes in crop plants. Frontiers of Plant Science, 2014, 5: 323.
[8] BURCH-SMITH T M, ANDERSON J C, MARTIN G B, DINESH-KUMAR S P. Application and advantages of virus-induced gene silencing for gene function studies in plants. The Plant Journal, 2004, 39: 734-746.
[9] KANAZAWA A, INABA J I, SHIMURA H, OTAGAKI S, TSUKAHARA S, MATSUZAWA A, KIM B M, GOTO K, MASUTA C. Virus-mediated efficient induction of epigenetic modifications of endogenous genes with phenotypic changes in plants. The Plant Journal, 2011, 65(1): 156-168.
[10] LU R, MARTIN-HERNANDEZ A M, PEART J R, MALCUIT I, BAULCOMBE D C. Virus-induced gene silencing in plants. Methods, 2003, 30(4): 296-303.
[11] ZHANG C, GHABRIAL S A. Development of bean pod mottle virus-based vectors for stable protein expression and sequence- specific virus-induced gene silencing in soybean. Virology, 2006, 344(2): 401-411.
[12] KACHROO A, GHABRIAL S. Virus-induced gene silencing in soybean. Methods in Molecular Biology, 2012, 894: 287-297.
[13] IGARASHI A, YAMAGATA K, SUGAI T, TAKAHASHI Y, SUGAWARA E, TAMURA A, YAEGASHI H, YAMAGISHI N, TAKAHASHI T, ISOGAI M, TAKAHASHI H YOSHIKAWA N. Apple latent spherical virus vectors for reliable and effective virus-induced gene silencing among a broad range of plants including tobacco, tomato,, cucurbits, and legumes. Virology, 2009, 386(2): 407-416.
[14] GEDLING C R, ALI E M, GUNADI A, FINER J J, XIE K, LIU Y, YOSHIKAWA N, QU F DORRANCE A E. Improved apple latent spherical virus-induced gene silencing in multiple soybean genotypes through direct inoculation of agro-infiltratedextract. Plant Methods, 2018, 14: 19.
[15] LI C, YOSHIKAWA N, TAKAHASHI T, ITO T, YOSHIDA K, KOGANEZAWA H. Nucleotide sequence and genome organization of apple latent spherical virus: a new virus classified into the family comoviridae. Journal of General Virology, 2000, 81(2): 541-547.
[16] YAMAGISHI N, SASAKI S, YAMAGATA K, KOMORI S, NAGASE M, WADA M, YAMAMOTO T, YOSHIKAWA N. Promotion of flowering and reduction of a generation time in apple seedlings by ectopical expression of thegene using the apple latent spherical virus vector. Plant Molecular Biology, 2011, 75(1/2): 193-204.
[17] HUANG Y, HONG H, XU M, YAN J, DAI J, WU J, FENG Z, ZHU M, ZHANG Z, YUAN X, DING X, TAO X. Developmentally regulatedsusceptibility to tomato spotted wilt virus infection. Molecular Plant Pathology, 2020, 21(7): 985-998.
[18] KON T, YOSHIKAWA N. Induction and maintenance of DNA methylation in plant promoter sequences by apple latent spherical virus-induced transcriptional gene silencing. Frontiers in Microbiology, 2014, 5: 595.
[19] LI C, HIRANO H, KASAJIMA I, YAMAGISHI N, YOSHIKAWA N. Virus-induced gene silencing in chili pepper by apple latent spherical virus vector. Journal of Virological Methods, 2019, 273: 113711.
[20] OGATA T, TOYOSHIMA M, YAMAMIZO-ODA C, KOBAYASHI Y, FUJII K, TANAKA K, TANAKA T, MIZUKOSHI H, YASUI Y, NAGATOSHI Y, YOSHIKAWA N, FUJITA Y. Virus-mediated transient expression techniques enable functional genomics studies and modulations of betalain biosynthesis and plant height in Quinoa. Frontiers of Plant Science, 2021, 12: 643499.
[21] YAMAGISHI N, YOSHIKAWA N. Highly efficient virus-induced gene silencing in apple and soybean by apple latent spherical virus vector and biolistic inoculation. Methods in Molecular Biology, 2013, 975: 167-181.
[22] 高鵬飛, 席飛虎, 張澤宇, 胡凱強, 陳凱, 魏文桃, 丁家治, 顧連峰. 植物VIGS技術及其在林業科學中的研究進展. 生物技術通報, 2021, 37(5): 141-153.
GAO P F, XI F H, ZHANG Z Y, HU K Q, CHEN K, WEI W T, DING J Z, GU L F. Research progress of plant VIGS technology and its application in forestry science. Biotechnology Bulletin, 2021, 37(5): 141-153. (in Chinese)
[23] WERNER R C, DUGé DE BERNONVILLE T, GLéVAREC G, LANOUE A, OUDIN A, PICHON O, ST-PIERRE B, COURDAVAULT V, BESSEAU S. ALSV-based virus-induced gene silencing in apple tree (L.). Methods in Molecular Biology, 2020, 2172: 183-197.
[24] 周彥. 果樹病毒載體研究進展. 中國農業科學, 2014, 47(6): 1119-1127.
ZHOU Y. Progresses in study of virus-based vectors of fruit trees. Scientia Agricultura Sinica, 2014, 47(6): 1119-1127. (in Chinese)
[25] YAEGASHI H, YAMATSUTA T, TAKAHASHI T, LI C, ISOGAI M, KOBORI T, OHKI S, YOSHIKAWA N. Characterization of virus-induced gene silencing in tobacco plants infected with apple latent spherical virus. Archives of Virology, 2007, 152(10): 1839-1849.
[26] BURCH-SMITH T M, SCHIFF M, LIU Y, DINESH-KUMAR S P. Efficient virus-induced gene silencing in. Plant Physiology, 2006, 142(1): 21-27.
[27] XU H Y, ZHANG C, LI Z C, WANG Z R, JIANG X X, SHI Y F, TIAN S N, BRAUN E, MEI Y, QIU W L, LI S, WANG B, XU J, NAVARRE D, REN D, CHENG N, NAKATA P A, GRAHAM M A, WHITHAM S A, LIU J Z. The MAPK kinase kinaseregulates cell death and defense responses.Plant Physiology, 2018, 178(2): 907-922.
[28] TIAN S N, LIU D D, ZHONG C L, XU H Y, YANG S, FANG Y, RAN J, LIU J Z. Silencingenhances the susceptibility of soybean to bacterial pathogen through attenuating the activation ofsignaling pathway. Plant Science, 2020, 292: 110386.
[29] LI C, LI K, ZHENG M, LIU X, DING X, GAI J, YANG S., a cytosolic 6-phosphogluconate dehydrogenase, enhanced tolerance to phosphate starvation by improving root system development and modifying the antioxidant system in soybean. Frontiers in Plant Science, 2021, 12: 704983.
[30] KIM K H, LIM S, KANG Y J, YOON M Y, NAM M, JUN T H, SEO M J, BAEK S B, LEE J H, MOON J K, LEE S H, LEE S H, LIM H S, MOON J S, PARK C H. Optimization of a virus-induced gene silencing system with soybean yellow common mosaic virus for gene function studies in soybeans. Plant Pathology Journal, 2016, 32(2): 112-122.
Establishment of ALSV-Induced Gene Silencing in Chinese Soybean Cultivars
DONG YongXin1, WEI QiWei1,2, HONG Hao1, HUANG Ying1, ZHAO YanXiao1, FENG MingFeng1, DOU DaoLong1, XU Yi1, TAO XiaoRong1
1College of Plant Protection, Nanjing Agricultural University/Key Laboratory of Soybean Disease and Pest Control of Ministry of Agriculture and Rural Affairs, Nanjing 210095;2Dazhou Academy of Agricultural Sciences in Sichuan Province, Dazhou 635000, Sichuan
【】 The establishment of apple latent spherical virus (ALSV)-induced gene silencing on domestic soybean cultivars will offer a simple, time-saving and operable system for gene function and genetic breeding of soybean cultivars in China.【】The infectious clones of ALSV which was used to infect plant through agro-infiltration was constructed. A 327 bp cDNA fragment of phytoene desaturase () cDNA was amplified from soybean cultivar Williams 82 and inserted into the pALSV2 vector.The pALSV1 and pALSV2-were used to co-infectplant throughAgroinfiltration, and the ALSV virus particle was purified from systemic infected leaves ofplants at 17 days post infiltration (dpi). The purified virions were mechanically inoculated onto the first-round true leaves of soybean and photobleaching phenotype on the upper non-inoculated leaves was monitored from 10 to 30 dpi. Using the empty ALSV vector as a control, the gene expression levels of ALSV coat protein () gene andin ALSV:GmPDS infected soybean were examined by RT-PCR and quantitative RT-PCR analysis, respectively. 【】At 20 dpi, soybean cultivar Nannong 1138-2 inoculated with ALSV:GmPDS showed photobleaching phenotype in the systemic infected leaves, however Williams 82 did not show gene silencing phenotype of PDS in the systemic infected leaves. The results of qRT-PCR confirmed that the RNA expression level ofin Nannong 1138-2 was significantly reduced but ALSV:was unable to silencein Williams 82. By using the same method, the gene silencing efficiency of other 9 domestic soybean cultivars mediated by ALSV:was examined. Soybean cultivars including Nannong 47, Andou 203, Xiangdou 4, Zhonghuang 13, Shanning 29 and Qihuang 34 all displayed photobleaching phenotype in plant systemic infected leaves after inoculation with ALSV:GmPDS, whilewas not silenced in Hedou 12, Zhonghuang 311 and Shanning 16.【】In this study, an-mediated ALSV viral VIGS vector was constructed. ALSV virions was propagated and purified fromplant. The purified virions were rub inoculated onto the first-round true leaves of soybean. Finally, an efficient gene silencing system was established on a number of domestic soybean cultivars in China.
virus-induced gene silencing (VIGS); soybean; apple latent spherical virus; phytoene desaturase ()

10.3864/j.issn.0578-1752.2022.09.002
2021-11-23;
2022-01-10
國家自然科學基金杰出青年基金(31925032)、海南省重大科技計劃(ZDKJ2021007)
董永鑫,E-mail:2019102035@njau.edu.cn。衛其巍,E-mail:675815483@qq.com。董永鑫與衛其巍為同等貢獻作者。通信作者陶小榮,E-mail:taoxiaorong@njau.edu.cn。通信作者徐毅,E-mail:xuyiqdpd@njau.edu.cn。通信作者竇道龍,E-mail:ddou@njau.edu.cn
(責任編輯 李莉)

