2種常見CRISPR-Cas系統在蘋果中的適用性特征
岳鵬 高海琴 李哲 米志波 鄭博 趙彥敏






摘要:運用生物信息學方法初步分析CRISPR-Cas9和Cpf1在蘋果中的整體適用性特征,以期為蘋果基因組編輯和CRISPR-Cas在蘋果研究中的推廣使用提供一定的參考和便利。結果表明,蘋果染色體中有數量可觀的PAM,平均間隔7 bp堿基有1個5′-NGG、間隔3 bp有1個5′-TTN;也就是說5′-TTN比5′-NGG的出現頻率高。SpCas9、FnCpf1分別有29.0%、26.9%的作用位點幾乎覆蓋了所有染色體基因,個別不能被SpCas9識別的基因能被FnCpf1識別,反之亦然。蘋果的CRISPR靶序列有大量重復,單一靶序列被視為能被Cas蛋白特異識別并有效編輯。在靶序列長度為20 nt時,99.5%的染色體基因可至少被其中1種Cas蛋白編輯,分別具有不同的可編輯度搭配;其中的237個基因只能被1種Cas蛋白編輯,填補了另一種Cas蛋白留下的編輯空白,另有220個染色體基因(0.5%)不能被任一種Cas蛋白編輯,即2種Cas蛋白同時留下編輯空白,沒有互補。
關鍵詞:蘋果;CRISPR-Cas;適用性特征;PAM出現頻率;基因編輯
中圖分類號:S661.101 ??文獻標志碼: A
文章編號:1002-1302(2022)07-0043-08
收稿日期:2021-06-04
基金項目:中國成人教育協會“十四五”成人繼續教育科研規劃重點課題(編號:2021-323ZB);河北省張家口市科技項目(編號:18110300043);河北省張家口市社會科學立項研究課題(編號:2021121)。
作者簡介:岳 鵬(1984—),男,河北張家口人,碩士,講師,從事農林生命科學類和開放教育研究。E-mail:yppolymerase@foxmail.com。
通信作者:趙彥敏,博士,副教授,從事農林生命科學類和開放教育研究。E-mail:zym319@163.com。
蘋果( Malus domestica )是世界上主要果樹作物之一,其產量和質量易受生物和非生物脅迫的影響[1]。因此,了解抗逆性相關基因的功能及調控規律,對培育抗逆性強的品種至關重要[2]。與其他作物相比,果樹具有高度雜合的多倍體基因組且繁育周期長,導致傳統的育種研究進展緩慢[3];而隨著基因組測序工作的完成,對其基因結構、基因通路和基因功能的認識為基因編輯奠定了基礎[4]。高效、易用、省時、低成本的基因編輯技術是賦予果樹重要經濟性狀的捷徑。常間四文重復序列叢集關聯蛋白[CRISPR(clustered regularly interspaced short palindromic repeat)-Cas(CRISPR-associated proteins)]系統具備以上優勢,在研究生物多種類的精確分子機制方面具有極高的應用價值[5]。
當前,基因組編輯技術主要集中在第2類CRISPR-Cas系統[6];它可以使用單個效應蛋白剪切DNA,包括Ⅱ型、Ⅴ型、Ⅵ型[7]。其中,Ⅱ型的Cas9及其失去剪切活性的衍生品dCas9被廣泛應用于多種生物體的基因組操作,包括靶向基因干擾、轉錄激活抑制、表觀遺傳修飾及目標堿基對轉換[8-9]。經典的Cas9蛋白須識別銜接在靶序列(+)3′端形如5′-NGG 的前間隔序列鄰近基序(protospacer-adjacent motif,PAM,+),并在PAM上游第3個堿基處切割雙鏈,形成平頭末端;在人類基因組中約平均間隔8 bp就會有1個5′-NGG[10]。不同于此,Ⅴ型的Cpf1(Cas12a)要識別毗鄰在靶序列(+)5′端形似5′-TTN 的PAM(+),并在PAM下游的同向鏈(+)第18、互補鏈(-)第23堿基處交錯切割,形成有5個突出堿基的黏性末端[11]。作為新型且更小的CRISPR效應蛋白,Cpf1的開發利用有利于突破和克服Cas9在使用中的一些限制,尤其是拓寬了CRISPR-Cas系統的識別范圍,使之能更有效地編輯富含AT堿基的基因組[12]。
CRISPR-Cas9已被大量使用在植物基因功能分析及育種研究中[13-15],其中包括多種果樹[14]。有關蘋果的幾項研究,依次是首次利用CRISPR-Cas9敲除內源 PDS (phytoene desaturase)基因[16],高效傳送CRISPR-Cas9核糖核蛋白到蘋果原生質體操縱DIPM1( DspA/E -interacting proteins of ?M.×domestica )、DIPM2和DIPM4基因[17],優化CRISPR-Cas9的應用條件并敲除PDS和TFL1(terminal flower)基因[3],運用CRISPR-Cas9敲除DIPM4基因的同時又減少外源DNA的殘留[18]。相對地,CRISPR-Cpf1的技術特點更加鮮明,但在2016年首次應用于水稻和煙草后[19],在植物基因組項目中使用的報道較少,目前還未見于果樹研究[2]。
已有的研究結果表明,CRISPR-Cas系統可實際應用于蘋果基因編輯,但只關注了少數幾個基因,且僅限于使用CRISPR-Cas9。本研究對蘋果全基因組序列進行初步分析,嘗試探索2種流行的CRISPR-Cas系統,即CRISPR-Cas9和CRISPR-Cpf1,在蘋果基因組編輯中的整體適用性,首次從PAM的數量和頻率、靶序列的重復和分布及2種Cas蛋白的互補等幾個方面開展討論,并形成PAM位點和靶序列信息庫,以期為蘋果基因組編輯和CRISPR-Cas系統在蘋果研究中的推廣使用提供一定的參考和便利。
1 材料與方法
1.1 數據獲取
蘋果全基因組代表性數據ASM211411v1下載自NCBI網站[20],17條染色體(chromosome,chr)和1條線粒體(mitochondrion,mt)的DNA序列以FASTA格式分別存儲在文本文件中;所有下載的DNA都只有單鏈(+)。相應的基因文件也從NCBI網站下載,其中以列表形式記錄基因在DNA序列上的起止位點等信息。最新發布在NCBI網站上的蘋果基因組數據ASM411538v1質量更高[21],但相應的基因信息不夠完善,只在本研究中作對比和補充。在諸多CRISPR-Cas系統中,效應蛋白SpCas9[化膿性鏈球菌( Streptococcus pyogenes ,Sp)]和FnCpf1[弗郎西斯菌( Francisella novicida ,Fn)]識別的PAM較有代表性,分別為5′-NGG和5′-TTN[10-11];本研究圍繞這2種典型的PAM,嘗試使用個人計算機分析蘋果全基因組。
1.2 PAM計數和間距計算
對蘋果各DNA序列出現的5′-NGG或 5′-TTN 計數,并記錄中間堿基在DNA序列上的位點作為PAM位點;還需要同時累計5′-CCN或 5′-NAA,以實現對互補鏈的搜索。計數期間,單獨計算N所代表的各種堿基占比,并用PAM位點數量與DNA長度的比值表示序列的PAM密度。除線粒體外,合計染色體DNA的各項數據以考量全基因組。
同樣地,分別計算各DNA序列和全基因組的PAM出現頻率。將每2個相鄰PAM位點之差作為間距(用字母 d 表示),其意義為間隔 d 個堿基對存在1個PAM,代表PAM的出現頻率;并累計每個 d的出現次數(用字母n表示),再把全部d升序排列(以t為排列序號),記錄不同d的數量(用字母m 表示)。PAM出現頻率的均值( d? mean)和中值( d? median)計算如下:
d? mean=∑ mi=1d in i∑mi=1n i;若∑ti=1n i≥∑mi=1n i2>∑t-1i=1n i,d? median =d t 。
1.3 計算剪切位點
在DNA序列上從5′-NGG前溯3個堿基或從5′-CCN后推3個堿基,獲得Cas9剪切位點。通過判斷剪切位點是否處于基因的起止位點之間,將對應的PAM位點劃入相應的基因范疇;不屬于任何基因范疇的PAM位點不做標記。
依據Cpf1的剪切特征,在DNA序列上從5′-TTN后推18個堿基或從5′-NAA前溯23個堿基,獲得同向剪切位點,用來判斷相應的PAM位點是否屬于同向鏈基因;從5′-TTN后推23個堿基或從 5′-NAA 前溯18個堿基,獲得逆向剪切位點,用來判斷相應的PAM位點是否屬于互補鏈基因。
1.4 靶序列的截取
根據2種PAM的特點,在DNA序列上分別截取長度為20 nt(不含PAM)的靶序列(target),用PAM位點命名,以FASTA格式儲存;PAM在互補鏈上的,還需要按照堿基配對原則轉換堿基并逆序。對具有同種PAM的靶序列進行重復性搜索,找到單一序列(singleton)和相同序列簇(cluster);簇中的PAM位點有屬于染色體基因的計1分,有屬于染色體基因間隔的計2分,有屬于線粒體基因的計4分,有屬于線粒體基因間隔的計8分,4種分值任意組合可將所有簇歸入15個重復類(用repeat ?N 命名, N 為不大于15的正整數)。重點關注靶序列單一且屬于基因范疇的PAM位點,其數量與基因長度的比值表示基因的可編輯度;按照“基因ID-PAM位點-靶序列”的模式建立簡易信息庫,找到可編輯度最高和最低的基因。
1.5 程序實現
以上操作已被整合到幾個Perl腳本中,并盡量優化算法降低時間復雜度和空間復雜度[22]。其中,為避免耗費大量時間,在判斷PAM位點是否影響基因時,用數組模擬DNA序列,數組索引表示位點,基因區間內的數組元素由基因ID填充,其他為未定義(undef)值;當剪切位點對應的數組元素為基因ID時,將PAM位點劃入此基因范疇。為避免占用大量內存空間導致程序卡頓,在搜索重復靶序列時,把總文件分割成大小合適的幾個子文件,使用散列快速剔除子文件內的相同序列;然后利用每個子文件中序列唯一的特點,使用散列剔除子文件間的相同序列,最終合并成沒有重復序列的總文件。
2 結果與分析
2.1 PAM含量分析
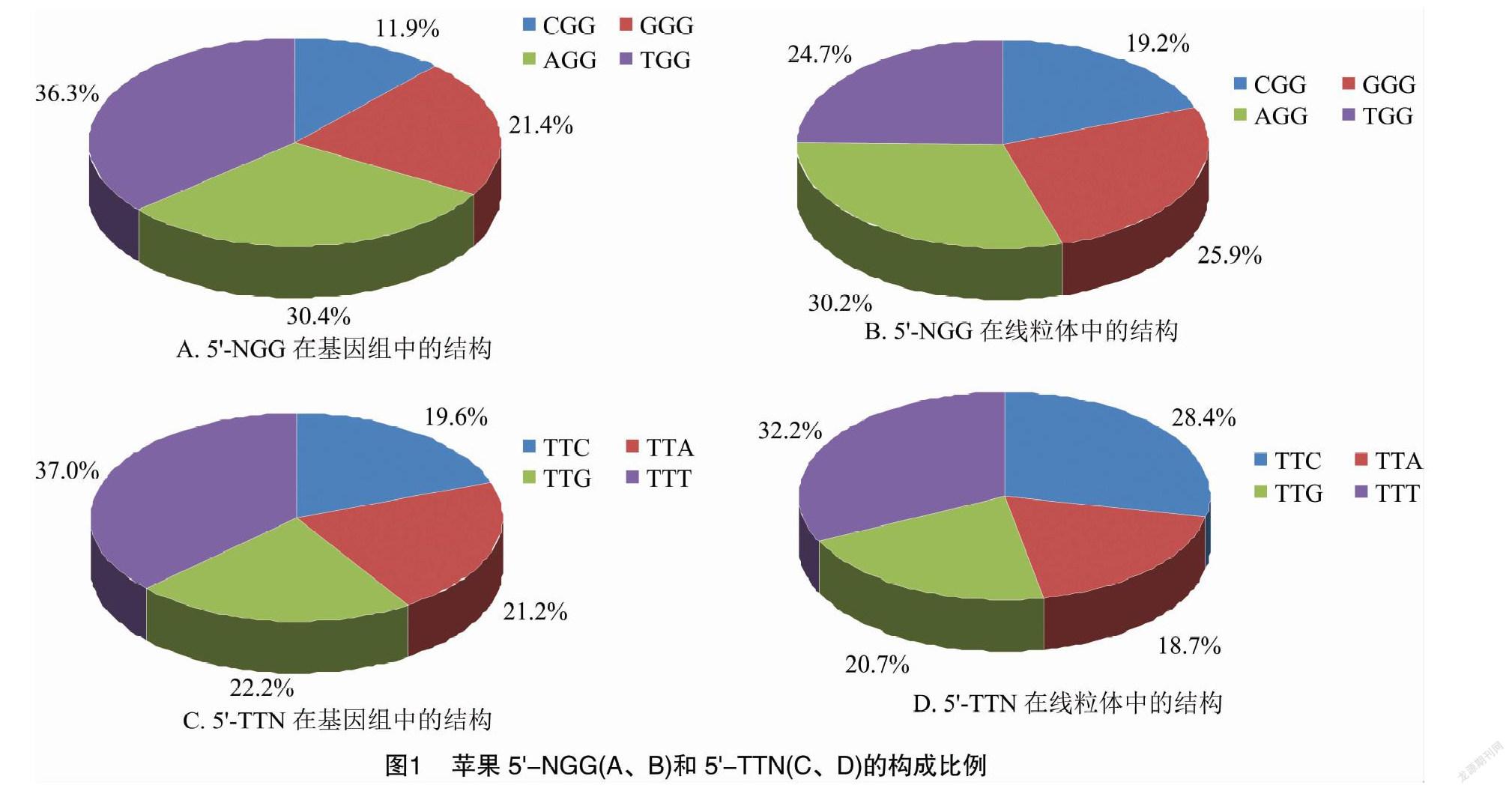
蘋果基因組(ASM211411v1)中5′-NGG總量為48 368 223,chr15的數量最多(4 179 083),chr01的最少(2 291 346),各染色體間差異較大;再結合序列長度估算序列的PAM密度,基因組是0.074,chr10最高(0.078),chr06最低(0.069),相差不大(表1)。在基因組中,CGG、GGG、AGG和TGG在NGG總量中的比例分別為11.9%、21.4%、30.4%、36.3%(圖1-A);在各染色體中的比例與此一致,略有微小波動(表1)。線粒體中的5′-NGG含量為 46 468,但密度達到0.117,高于基因組最高值;CGG、GGG、AGG和TGG的比例分別為19.2%、259%、30.2%、24.7%,與基因組中的同項數據也有明顯差異(圖1-B)。
基因組中5′-TTN的總數為127 635 154,同樣是chr15最多(11 092 974),chr01最少(5 902 134);估算基因組的密度是0.194,依然是chr10最高(0206),chr06最低(0.180),兩者比較接近(表2)。在基因組中,TTC、TTA、TTG和TTT在TTN總量中的比例分別為19.6%、21.2%、22.2%、37.0%(圖1-C);同樣,在各染色體中的比例與此一致(表2)。線粒體中5′-TTN含量為69 296,密度只有0.174 6,低于基因組最低值;TTC、TTA、TTG和TTT的比例分別為28.4%、18.7%、20.7%、32.2%,與基因組中的同項數據也差異明顯(圖1-D)。
作為對比,另一基因組數據(ASM411538v1)的5′-NGG密度為0.084,CGG、GGG、AGG和TGG的比例分別為11.7%、21.5%、30.6%、36.3%;5′-TTN的密度為0.219,TTC、TTA、TTG和TTT的比例分別是19.6%、21.2%、22.4%、36.8%。使用2個不同版本的基因組數據計算出的結果很接近,特別是NGG和 TTN的構成比例幾乎完全一致,可以用同一組餅狀圖表示(圖1-A、圖1-C)。
2.2 PAM出現頻率
在蘋果基因組(ASM211411v1)中,相鄰5′-NGG的間距最小為1 bp,出現次數最多(圖2-A);最大為1 347 bp,出現在chr07的19 065 740~19 067 087位點間(表1)。間距中值是7 bp,表示平均約間隔7 bp堿基就有1個5′-NGG,也說明半數以上的間距不大于7 bp;間距均值為12.0 bp,是受到最大值的影響而產生了偏移(圖2-a)。各染色體情況與此高度一致(表1)。而在線粒體中,間距中值和均值分別為5、8.5 bp,與基因組完全不同(圖2-b)。
基因組中,相鄰5′-TTN的間距最小為1 bp,出現次數最多(圖2-c);最大為1 022 bp,出現在chr07的26 831 153~26 832 175位點間(表2)。間距中值是3 bp,表示平均約間隔3 bp堿基就有1個 5′-TTN,也說明半數以上的間距不大于3 bp;受最大值影響,間距均值是4.5 bp(圖2-c)。同樣,各染色體情況與此一致(表2)。線粒體的間距中值和均值分別為4、5.7 bp,也與基因組完全不同(圖2-d)。使用另一版本的基因組數據(ASM411538v1)計算,可得出類似的結果:5′-NGG的間距中值和均值分別為7、11.9 bp,5′-TTN的間距中值和均值分別為3、4.5 bp;所展示出的變化趨勢也可以繪成同樣的圖像(圖2-a、圖2-c)。
2.3 PAM的基因歸屬
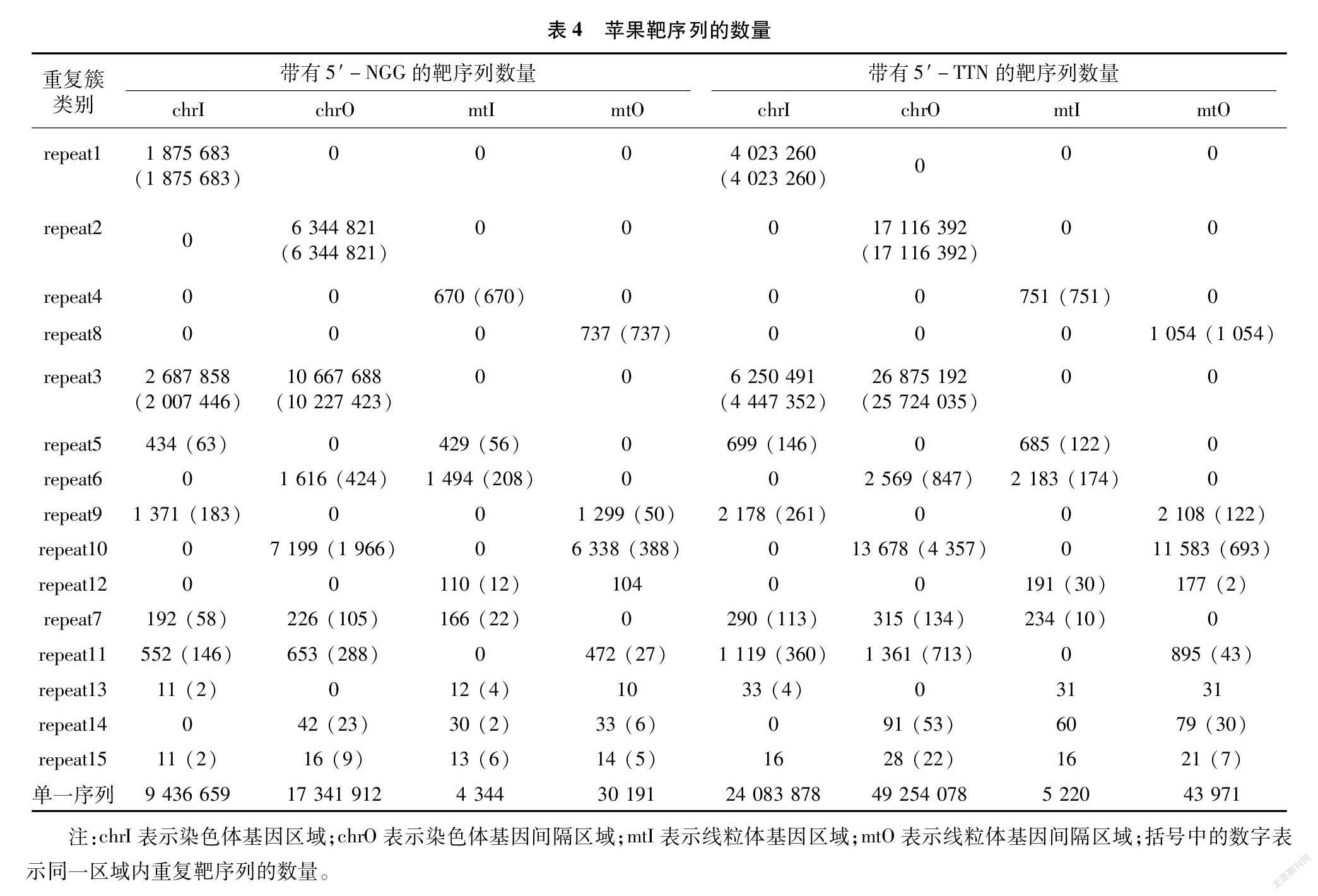
在蘋果基因組中,29.0%的5′-NGG和26.9%的5′-TTN能影響到基因,基本覆蓋了全部43 464個基因(表3)。其中,chr06上ID為114825448的基因含5′-NGG最多,有14 620個;chr16的114822391(表示基因ID,下同)和chr09的114827208基因不含5′-NGG。chr06的114825448基因含5′-TTN最多,有36 069個;chr16的108169786、chr12的108174957、chr11的108174696、chr02的114823832、chr03的114824289和108171505基因都不含5′-TTN。
在線粒體中,15.6%的5′-NGG能作用于全部70個基因,13.5%的5′-TTN覆蓋了98.6%的基因(表3)。其中,ID為13630194的基因含5′-NGG最多,有1 186個;13630239(121 892~121 913位點)和13630229基因含5′-NGG最少,只有5個。13630194基因含5′-TTN最多,有1 337個;13630239基因(121 892~121 913位點)不含5′-TTN。與各染色體相比,線粒體中屬于基因間隔的PAM占比明顯更大(表3)。
2.4 靶序列的重復簇
蘋果的CRISPR靶序列有大量重復,共計15種重復簇(表4)。其中,重復序列只屬于染色體基因的簇劃入repeat1,只屬于染色體基因間隔的為repeat2,只屬于線粒體基因的為repeat4,只屬于線粒體基因間隔的為repeat8,repeat15是重復的靶序列同時出現在上述4個區域中。可以發現,帶有 5′-NGG 的靶序列,染色體基因的648條與線粒體基因中的620條重復;帶有5′-TTN的靶序列,染色體基因中的1 038條與線粒體基因中的966條重復(表4,repeat5、7、13、15)。只屬于染色體基因的,帶有5′-NGG的重復靶序列共有3 883 583條,帶有5′-TTN的重復靶序列共有84 71 496條(表4,repeat1、3、5、9、7、11、13、15括號中的數字);而在線粒體基因中,這2個數值分別為980、1 087(表4,repeat4、5、6、12、7、13、14、15括號中的數字)。
2.5 基因可編輯度
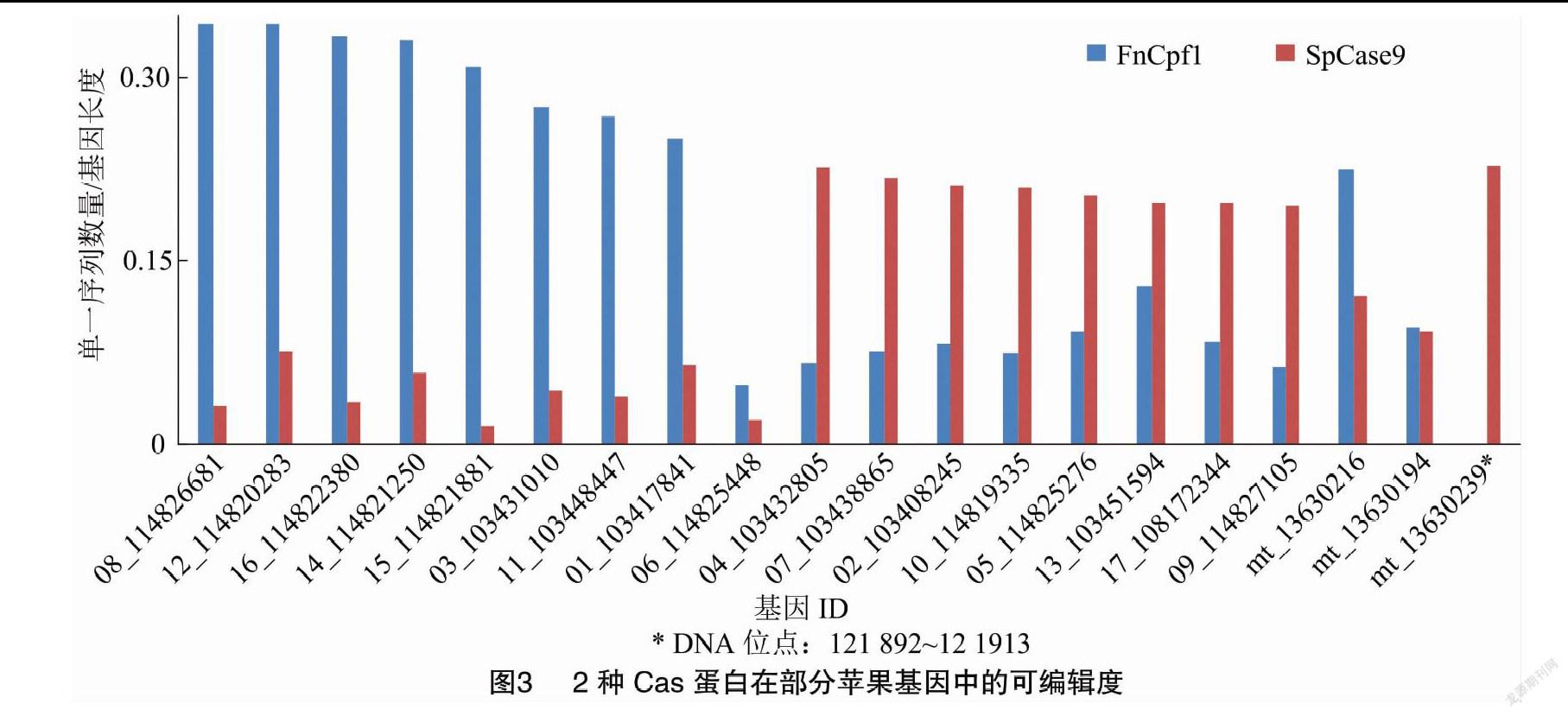
在蘋果各染色體中,帶有5′-NGG的單一靶序列數量為26 778 571,其中屬于基因的有9 436 659,其余都處于基因間隔中;帶有5′-TTN的單一靶序列數量為73 337 956,屬于基因的有240 838 78(表4)。SpCas9對chr04上的103432805基因有最高的可編輯度,為0.226,含有PAM最多的114825448基因的可編輯度僅為0.020(圖3);另有372個基因的可編輯度為0。FnCpf1對chr08上的114826681基因有最高的可編輯度,為0.344,含有PAM最多的114825448基因的可編輯度僅為0.049(圖3);另有305個基因的可編輯度為0。
在線粒體中,帶有5′-NGG的單一靶序列數量為34 535,屬于基因的有4 344;帶有5′-TTN的單一靶序列數量為49 191,屬于基因的有5 220(表4)。SpCas9對13630239基因(121892~121913位點)可能有最高的可編輯度,為0.227,含有PAM最多的13630194基因的可編輯度為0.092(圖3);有4個基因的可編輯度為0。FnCpf1對13630216基因可能有最高的可編輯度,為0.224,含有PAM最多的13630194基因的可編輯度為0.095(圖3);有6個基因的可編輯度為0。
2.6 Cas蛋白的編輯互補
蘋果中的大多數基因可同時被2種Cas蛋白編輯, 分別具有不同的可編輯度搭配(圖3)。 可編輯度為0的基因在全部基因中的占比較小,在各DNA序列上均有分布;其中,chr04上的數量最多,有66個不能被SpCas9編輯、有62個基因不能被FnCpf1編輯(圖4)。經過篩選,共有237個(0.5%)染色體基因、2個(2.9%)線粒體基因能被1種Cas蛋白編輯,填補了另一種Cas蛋白留下的編輯空白(圖4,part Ⅰ、part Ⅲ);共有220個染色體基因(0.5%)、4個(5.7%)線粒體基因不能被任一種Cas蛋白編輯,即2種Cas蛋白同時留下編輯空白,沒有互補(圖4,part Ⅱ)。
3 討論
作為重要的基因編輯工具[23],CRISPR-Cas系統在蘋果基因組中有較好的整體適用性,主要表現在3個方面。一是有數量可觀的PAM分散在蘋果DNA序列的各個角落,出現頻率很高,平均間隔很短。二是Cas蛋白的作用位點幾乎覆蓋了所有基因,個別不能被SpCas9識別的基因卻含有FnCpf1的識別位點,反之亦然。三是擁有單一靶序列的基因占大多數,99.5%的染色體基因和94.3%的線粒體基因都能至少被其中1種Cas蛋白編輯。蘋果DNA序列的測序結果表明,AT堿基的含量高于CG堿基[20-21],導致5′-TTN的數量遠超5′-NGG、5′-TTN 的出現頻率更高、帶有5′-TTN的單一靶序列數量更多,也就是說Cpf1在蘋果基因編輯中有更大的可挖掘潛力。
各染色體的PAM密度、組成和出現頻率幾乎一致,可視作蘋果基因組的整體特征之一。雖然也存在于蘋果細胞中,但線粒體通常被認為是有益的共生生物[24],其DNA不被計入基因組;這一點在本研究中也有突出體現,即與染色體的同項數據相比,線粒體都有明顯差異。目前,對蘋果線粒體基因的編輯還未見報道,其操作過程是否與基因組相同還需要更深入的研究驗證,在本研究中僅是預測性的初步探討;且線粒體DNA體量小、基因少[20],對基因組編輯的影響不大,在特定環境中可不做考慮。葉綠體也有類似情況[25],可在條件成熟時進一步研究討論。
已有的測序結果含有未知堿基,在數億堿基的蘋果DNA序列中比例微小,對多項計算結果的影響可忽略不計[20-21]。但如果2個PAM之間存在未知堿基且結合上下游無法判斷是否存在另一個PAM,就在確定PAM頻率時摒棄這2個PAM的間距,避免出現超長間距的同時,也保證了是在計算相鄰2個PAM的間距。對比人類基因組取間距中值作為PAM的出現頻率,本研究也采用了同樣的取值方法;蘋果基因組平均間隔7 bp堿基就有1個5′-NGG,頻率高于人類基因組的8 bp[10]。
一般,判斷PAM是否屬于基因依據的是其位點是否在基因起止位點間,臨近基因邊緣的PAM就有可能實際作用到了間隔區。不同于此,本研究將判斷依據改進為剪切位點是否在基因起止位點間,既避免了上述問題,也充分挖掘了隱藏的基因PAM。在此基礎上截取的靶序列都具有明確的基因歸屬。靶序列的長度按常規被設定為20 nt,初步分析了因序列重復導致的脫靶情況;根據序列越短重復率越高的共識,可適當增加靶序列的長度提高基因的可編輯度。此外,脫靶的原因還包括相似匹配和種子序列的長度[11],可在未來的研究中做更深入的分析。
基因可編輯度與單一靶序列的數量成正比,與基因自身的長度成反比,表示的是單位長度內含有的備選靶序列密度。可編輯度為0的基因,小部分是因為不含有PAM,大多數是在屏蔽了重復靶序列后,備選數量為0。本研究采用了較嚴格的屏蔽標準,凡是在蘋果DNA序列中出現的重復靶序列均計入重復簇;重復簇的類別劃分較細,15個類別涵蓋了靶序列所在4個區域的所有搭配,方便在試驗設計時有側重地取舍。在蘋果全部基因中,2種Cas蛋白都有占比很小的編輯盲區,FnCpf1要好于SpCas9。盲區重疊的部分所含的224個基因不適宜使用這2種Cas蛋白編輯,可考慮換用識別不同PAM的其他Cas蛋白;其中超過半數的基因(139個)編碼多種RNA,通常在實際研究中較少涉及到。
在Perl腳本的幫助下,各步驟的運算結果都在文本文件中詳細列表構成了信息庫,可直接打開查詢感興趣的信息;也可導入數據庫加以專業化的管理和維護,成為網絡服務平臺的構建基礎,這是開展下一步研究的一個重要方向。
參考文獻:
[1]Arzani A,Ashraf M. Smart engineering of genetic resources for enhanced salinity tolerance in crop plants[J]. Critical Reviews in Plant Sciences,2016,35(3):146-189.
[2]Wang X H,Tu M X,Li Z,et al. Current progress and future prospects for the clustered regularly interspaced short palindromic repeats (CRISPR) genome editing technology in fruit tree breeding[J]. Critical Reviews in Plant Sciences,2018,37(4):233-258.
[3]Charrier A,Vergne E,Dousset N,et al. Efficient targeted mutagenesis in apple and first time edition of pear using the CRISPR-Cas9 system[J]. Frontiers in Plant Science,2019,10:40.
[4]Zhou J H,Li D D,Wang G M,et al. Application and future perspective of CRISPR/Cas9 genome editing in fruit crops[J]. Journal of Integrative Plant Biology,2020,62(3):269-286.
[5]Yan F C,Wang W,Zhang J Q. CRISPR-Cas12 and Cas13:the lesser known siblings of CRISPR-Cas9[J]. Cell Biology and Toxicology,2019,35(6):489-492.
[6]Strecker J,Jones S,Koopal B,et al. Engineering of CRISPR-Cas12b for human genome editing[J]. Nature Communications,2019,10:212.
[7]Makarova K S,Zhang F,Koonin E V. Snapshot:class 2 CRISPR-Cas systems[J]. Cell,2017,168(1/2):328-328e1.
[8]Hu J H,Miller S M,Geurts M H,et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity[J]. Nature,2018,556(7699):57-63.
[9]Moradpour M,Abdulah S N A. CRISPR/dCas9 platforms in plants:strategies and applications beyond genome editing[J]. Plant Biotechnology Journal,2020,18(1):32-44.
[10]Hsu P D,Lander E S,Zhang F. Development and applications of CRISPR-Cas9 for genome engineering[J]. Cell,2014,157(6):1262-1278.
[11]Zetsche B,Gootenberg J S,Abudayyeh O O,et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system[J]. Cell,2015,163(3):759-771.
[12]楊 帆,李 寅. 新一代基因組編輯系統CRISPR/Cpf1[J]. 生物工程學報,2017,33(3):361-371.
[13]Ma X L,Zhu Q L,Chen Y L,et al. CRISPR/Cas9 platforms for genome editing in plants:developments and applications[J]. Molecular Plant,2016,9(7):961-974.
[14]Wang X H,Tu M X,Wang D J,et al. CRISPR/Cas9-mediated efficient targeted mutagenesis in grape in the first generation[J]. Plant Biotechnology Journal,2018,16(4):844-855.
[15]Rodríguez-Leal D,Lemmon Z H,Man J,et al. Engineering quantitative trait variation for crop improvement by genome editing[J]. Cell,2017,171(2):470-480,e8.
[16]Nishitani C,Hirai N,Komori S,et al. Efficient genome editing in apple using a CRISPR/Cas9 system[J]. Scientific Reports,2016,6:31481.
[17]Malnoy M,Viola R,Jung M H,et al. DNA-free genetically edited grapevine and apple protoplast using CRISPR/Cas9 ribonucleoproteins[J]. Frontiers in Plant Science,2016,7:1904.
[18]Pompili V,Costa L D,Piazza S,et al. Reduced fire blight susceptibility in apple cultivars using a high-efficiencyCRISPR/? Cas9-FLP/FRT-based gene editing system[J]. Plant Biotechnology Journal,2020,18(3):845-858.
[19]Endo A,Masafumi M,Kaya H,et al. Efficient targeted mutagenesis of rice and tobacco genomes using Cpf1 from ?Francisella novicida [J]. Scientific Reports,2016,6:38169.
[20]Daccord N,Celton J M,Linsmith G,et al. High-quality de novo assembly of the apple genome and methylome dynamics of early fruit development[J]. Nature Genetics,2017,49(7):1099-1106.
[21]Zhang L Y,Hu J A,Han X L,et al. A high-quality apple genome assembly reveals the association of a retrotransposon and red fruit colour[J]. Nature Communications,2019,10:1494.
[22]Alsuwaiyel M H.Algorithms:design techniques and analysis (revised edition)[M]. Singapore:World Scientific Publishing,2016:20-34.
[23]Koonin E V,Makarova K S,Zhang F. Diversity,classification and evolution of CRISPR-Cas systems[J]. Current Opinion in Microbiology,2017,37:67-78.
[24]Nykky J,Vuento M,Gilbert L.Role of mitochondria in parvovirus pathology[J]. PLoS One,2014,9(1):e86124.
[25]Waters M T,Langdale J A.The making of a chloroplast[J]. The EMBO Journal,2009,28(19):2861-2873.

