毛竹Mariner-like element自主轉座子的鑒定與生物信息學分析*
謝佳敏 周明兵
(浙江農林大學 亞熱帶森林培育國家重點實驗室 杭州 311300)
轉座子廣泛分布在生物基因組內,是生物基因組成的重要部分,在有些植物中轉座子含量可以高達80%以上(Sergeevaetal.,2011),如在小麥(Triticumaestivum)基因中轉座子含量為88%(Chouletetal.,2010),在玉米(Zeamays)基因中轉座子含量為84%(Schnableetal.,2009)。轉座子在基因組內的活動可以對生物基因組產生巨大的影響,主要表現為:轉座子的轉座活動造成基因插入突變,同時,轉座過程中也產生新基因,因此轉座子的活動被認為是生物進化的重要動力之一(Kazazian,2004)。
根據轉座子在生物基因組內轉座模式的不同,將轉座子分為ClassⅠ和ClassⅡ 2類。ClassⅠ類轉座子又叫逆轉錄轉座子(RNA轉座子),在生物體內以RNA為中間體進行轉座,其轉座模式為“復制-黏貼”;ClassⅡ類轉座子又叫DNA轉座子,在生物體內的轉座模式為“剪切-黏貼”。根據序列的相似性,DNA轉座子又可以分為12個不同的超級家族:Tc1/Mariner,hAT,Mutator,Merlin,Transib,P,Piggybac,PIF,CACTA,Crypton,Helitron以及Maverick(Wickeretal.,2007)。
DNA轉座子根據能否發生自主轉座又可分為自主轉座子和非自主轉座子。自主轉座子由于其結構完整,具有編碼完整轉座酶的能力,所編碼的轉座酶可以促使自身發生轉座;而非自主轉座子與自主轉座子相比,其內部的轉座酶編碼序列有所缺失,不能編碼轉座酶,必須依靠相應的自主轉座子編碼的轉座酶作用才能轉座。非自主DNA轉座子既可以是自主DNA轉座子缺失部分序列所產生的衍生物,也可以是僅僅帶有與對應自主DNA轉座子相似的末端倒置重復序列(Terminal Inverted Repeats,TIR)(Liuetal.,2009)。
Mariner-like elements (MLE)家族轉座子是DNA類轉座子Tc1/Mariner超級家族中的重要成員,最早是在研究毛里塔尼亞果蠅(Drosophilamauritiana)白眼基因的一個不穩定突變時發現的(Haymeretal.,1986;Jacobsonetal.,1986)。完整的MLE轉座子的結構比較簡單,主要包括轉座子末端被轉座酶識別并結合的末端顛倒重復序列(TIRs)、中間編碼轉座酶開放閱讀框(Open Reading Frame,ORF)和轉座子插入靶基因組時存在的靶位點重復序列(target site duplications,TSDs)(Robertson,1993)。其中,TIR結構是在基因組中,兩端前后倒置互補的重復DNA序列,TIR是轉座酶所識別及結合的區域。轉座酶的編碼區一般包含2個功能區域,HTH結構域(螺旋-轉角-螺旋空間結構)與DDD(3個天冬氨酸組成的保守空間結構)功能結構域,其中HTH結構域的功能是識別和結合基因組中對應的TIR結構,DDD功能域是催化轉座的催化區域,具有剪切轉座子并整合入基因組的功能。
MLE轉座子結構簡單,在轉座酶的催化作用下,含有其結合序列的TIR結構就有概率發生轉座(Lampeetal.,1996)。利用MLE轉座子的轉座必需元件和轉座酶可以分離的特征,可以構建二元轉座子系統,該轉座子系統具有高載量、高轉座效率、高安全性等突出優點,可將其應用于轉基因、基因功能研究、基因治療等領域。如在鮭魚(Oncorhynchusketa)中發現的Sleeping Beauty,在經過人工改造后,構建成為具有高活性的轉座子(Izsvaketal.,2000;Ivicsetal.,2006),它能在大多數脊椎動物細胞中發生轉座(Liuetal.,2006),還可使所攜帶的基因在動物體內實現穩定整合和長期表達(Horieetal.,2003)。同時利用該轉座子的定向點特異性修復能力與干細胞的多向分化性相結合,就可通過基因修復來治療多種疾病,且無需進行體外細胞培養(Vandendriesscheetal.,2009;Dupuyetal.,2002),是目前利用最廣泛的轉座子基因工具。
毛竹(Phyllostachysedulis)占我國竹類種植面積的近74%,約有627.7萬hm2,是非木材產品中最豐富的自然資源(Jiangetal.,2007)。最新的毛竹基因組測序結果表明,毛竹基因組中轉座子含量高達63.24%(Zhaoetal.,2018)。本課題組從毛竹基因組中克隆得到2個MLE轉座子并成功構建了2個MLE轉座子的體外表達體系(Zhouetal.,2015;2016;2017)。本研究利用最新的毛竹基因組數據系統地鑒定了毛竹基因組中MLE自主轉座子,及對應非自主轉座子,并分析它們的進化模式,研究結果有助于毛竹的基因組進化研究與新的植物轉座子工具開發。
1 材料與方法
1.1 毛竹MLE轉座子完整序列的鑒定 下載最新版的毛竹全基因組序列文件(http:∥bamboo.bamboogdb.org),使用IRF307(Warburton,2004),對毛竹基因組中的TIR結構進行篩選,參數設置為TIR序列長度大于20且小于100,兩側TIR序列的相似度在80%以上。
根據兩端TIR序列坐標,提取坐標間包括TIR的完整DNA序列,利用NCBI數據庫中已發布的植物中完整MLE轉座酶序列,使用Fasta36軟件構建本地MLE蛋白數據庫,使用Fasty36功能,將提取的核酸序列進行翻譯,將e值小于-10、閱讀框連續且具有正常的起始密碼子和終止密碼子的序列視為MLE自主轉座子。使用DNAMan分析獲得的完整MLE自主轉座子的轉座酶ORF,使用在線工具HELIX-TURN-HELIX MOTIF PREDICTION(https:∥npsa-prabi.ibcp.fr)進行HTH結構域的預測,使用在線工具Swiss model對轉座酶的三級結構進行分析,確定其DDD結構域。
1.2 完整MLE轉座子的活性分析 從NCBI數據庫下載水稻(Oryzasativa)、大豆(Glycinemax)和毛竹中已報道的、具有轉座活性的MLE轉座酶(Yangetal.,2014;2006;Jarviketal.,1998;Zhouetal.,2015)。使用Mafft軟件將本次篩選獲得的毛竹MLE自主轉座子的轉座酶與下載的活性MLE轉座酶進行比對(Feschotteetal.,2003),驗證所獲得的毛竹MLE自主轉座子的轉座酶功能區域,預測所得到的MLE轉座子的轉座酶是否具有潛在的轉座活性。
通過NCBI數據庫下載已經發表的部分動植物及微生物中的活性MLE轉座酶完整序列,將本研究得到的毛竹完整MLE轉座酶進行比對構建進化樹,分析其在生物中的進化關系。
1.3 非自主轉座子鑒定 將上述鑒定MLE自主轉座子的TIR序列與IRF307軟件獲得的TIR序列(含有TIR的序列)數據庫進行本地Blast,獲得了高度相似性的TIR序列,提取與MLE自主轉座子TIR一致的TIR序列。將提取的序列與MLE自主轉座子序列通過在線BlastN進行兩兩比對(https:∥blast.ncbi.nlm.nih.gov/Blast.cgi),得到MLE自主轉座子與TIR序列內部序列一致的TIR序列,視為MLE自主轉座子對應的非自主轉座子。
使用Mafft軟件將獲得的MLE非自主轉座子與其對應的MLE自主轉座子進行比對,分析MLE非自主轉座子與自主轉座子各部分缺失的情況。
1.4 插入偏好性規律 根據MLE自主轉座子與非自主轉座子的坐標,在毛竹全基因組中,提取MLE自主轉座子與非自主轉座子側翼25 bp序列,使用TBTools工具的SequenceLogo進行比對和可視化展示(Chenetal.,2020)。
2 結果與分析
2.1 2個潛在 Mariner-like自主轉座子的鑒定 通過IRF307軟件在新版毛竹基因組中共找到了310 845條含有TIR的序列,利用Fasta軟件構建的植物MLE轉座酶本地數據庫,經Fasty36進行篩選,獲得了301個含有TIR結構且擁有完整或部分編碼轉座酶結構的序列(e值小于5)。篩選得到了2個擁有完整MLE轉座酶功能域的MLE轉座子,命名為PhV2MLE1A和PhV2MLE2A。PhV2MLE1A全長3 950 bp,TIR長30 bp(5′TIR:CTCCCTCCGTCCCAGTATATAGGGCGTATA;3′TIR:TATATGCCCTATATACTGGGACGGAGGGAG),閱讀框編碼414個氨基酸(圖1A);PhV2MLE2A全長12 990 bp,TIR長49 bp(5′TIR:CGACTATGAGG TAGTCGTAGCAAGACTTACGACTATGGGATAGTCGT AG;3′TIR:CTACAACTATATTACAGTCGTAAGATC TCCTACGACTATATTACGGTCG),閱讀框編碼372個氨基酸(圖1B)。
通過在線工具HELIX-TURN-HELIX MOTIF PREDICTION和Swiss model,將PhV2MLE1A和PhV2MLE2A編碼的轉座酶進行結構域的在線預測,圖1A和圖1B中分別為PhV2MLE1A和PhV2MLE2A轉座酶閱讀框翻譯序列,下劃線依次為預測得到的HTH結構域和DDD結構域。

圖1 PhV2MLE1A和PhV2MLE2A轉座酶ORF翻譯Fig.1 ORF translation of PhV2MLE1A and PhV2MLE2A transposaseA:PhV2MLE1A轉座酶ORF翻譯圖;B:PhV2MLE2A轉座酶ORF翻譯圖。下劃線依次為預測得到的HTH結構域和DDD結構域。A:ORF translation diagram of PhV2MLE1A transposase;B:ORF translation diagram of PhV2MLE2A transposase.The underline is the predicted HTH domain and DDD domain in sequence.
2.2PhV2MLE1A和PhV2MLE2A具有轉座活性 將PhV2MLE1A和PhV2MLE2A的轉座酶,與已報道的水稻中的活性MLE轉座子Osmar1,Osmar5,Osmar9,Osmar10,Osmar14,Osmar17,Osmar19的轉座酶(Osmar5是目前水稻中發現的唯一擁有天然活性的MLE轉座子),大豆中的活性MLE轉座子Soymar1的轉座酶,毛竹中的完整活性MLE轉座子Phmar1,Phmar2的轉座酶,進行比對(圖2)。PhV2MLE1A和PhV2MLE2A轉座酶的HTH結構域和DDD結構域與已報道的活性轉座酶的對應結構域高度同源(圖2),其中,一些在關鍵位點上的氨基酸完全一致,如在結合區域HTH結構的R、T,DDD結構域的DEKWF、QQDNA、PNSPD,這些關鍵位點的氨基酸決定了轉座酶的功能。PhV2MLE1A和PhV2MLE2A轉座子的TIR結構完全符合已報道的活性MLE轉座子結構特征。
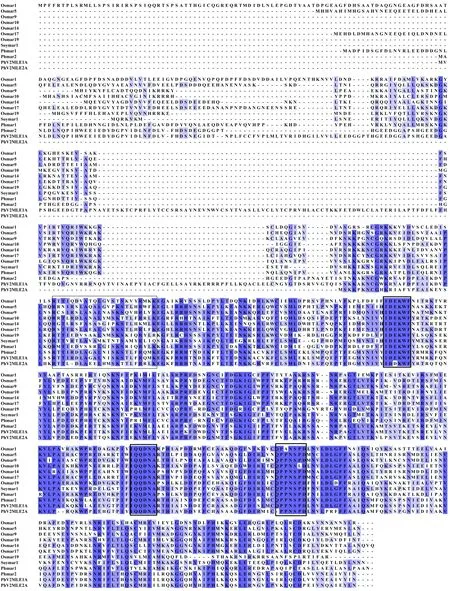
圖2 PhV2MLE1A和PhV2MLE2A轉座酶與水稻、毛竹及大豆中完整MLE轉座酶的比對Fig.2 Comparison of PhV2MLE1A and PhV2MLE2A transposase with complete MLE transposase in rice,Moso bamboo and soybeanOs:水稻;Soy:大豆;Ph:毛竹。細框內為HTH結合域,粗框內為催化域DDD結構。Os:Oryza sativa;Soy:Glycine max;Ph:Phyllostachys edulis.The HTH binding domain is in the thin frame,and the catalytic domain DDD structure is in the thick frame.
同時發現,所篩選得到的PhV2MLE1A轉座子與已發表的Phmar2轉座子相似性極高,認為是同一個MLE轉座子;PhV2MLE2A轉座子為全新發現的一個毛竹MLE轉座子。
圖3中轉座酶的進化關系共分為A和B 2個大類。本次篩選獲得的2個完整MLE轉座子的轉座酶處在A類,與水稻、大豆等植物中的MLE轉座酶十分接近,B類中主要為微生物、昆蟲以及人類中的MLE轉座酶。
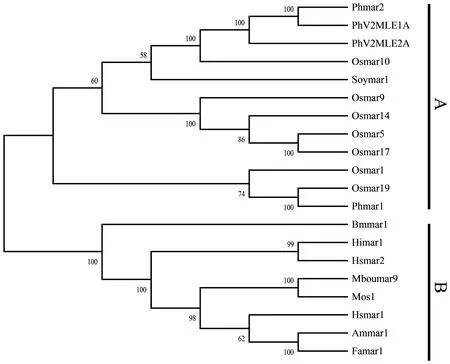
圖3 PhV2MLE1A和PhV2MLE2A的進化關系Fig.3 The evolutionary relationship of PhV2MLE1A and PhV2MLE2APh:毛竹;Os:水稻;Soy:大豆;Bm:野桑蠶;Hi:草蛉;Hs:人;Mbou:螞蟻;Am:蜜蜂;Fa:地蜈蚣;Mos:家蠅。Ph:Phyllostachys edulis;Os:Oryza sativa;Soy:Glycine max;Bm:Bombyx mandarina;Hi:Chrysopinae;Hs:Homo sapiens;Mbou:Messor bouvieri;Am:Apis mellifera;Fa:Forficula auricularia;Mos:Musca domestica.
2.3 非自主MLE轉座子的鑒定 將PhV2MLE1A和PhV2MLE2A的TIR序列與通過IRF307軟件獲得的毛竹TIR序列數據庫,進行本地Blast,獲得了高度相似性的TIR序列,提取對應的非自主轉座子序列,將對應非自主轉座子序列與完整的MLE自主轉座子序列使用BlastN進行兩兩比對之后,PhV2MLE1A共篩選得到4條結構一致的對應非自主轉座子(PhV2MLE1NA1,PhV2MLE1NA2,PhV2MLE1NA3和PhV2MLE1NA4)。PhV2MLE2A共篩選得到4條結構一致的對應非自主轉座子(PhV2MLE2NA1,PhV2MLE2NA2,PhV2MLE2NA3和PhV2MLE2NA4)。圖4為PhV2MLE1A(A)和PhV2MLE2A(B)轉座子與對應的非自主轉座子兩側TIR結構比對情況。
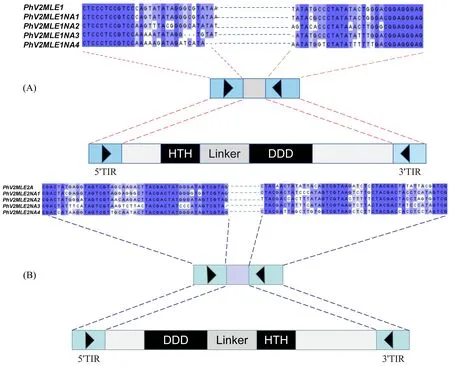
圖4 PhV2MLE1A(A)和PhV2MLE2A(B)及其非自主轉座子的TIR結構Fig.4 TIR structure of PhV2MLE1A(A)and PhV2MLE2A(B)and their non-autonomous transposons
將完整MLE轉座子根據編碼區域及轉座酶的功能分為9個部分:TIR,TIR-M,M-HTH,HTH,HTH-DDD,DDD,DDD-T,T-TIR,TIR(分別為5′TIR結構,5′TIR結構至轉座酶起始密碼子M,起始密碼子M至HTH結構區域,HTH轉座酶結合域,HTH結合域至DDD功能域的連接區域,DDD結構功能域,DDD功能域至終止密碼子T,終止密碼子T至3′TIR結構)。將2個完整MLE轉座子及其非自主轉座子結構比對后,對MLE非自主轉座子與其自主轉座子的各個區域的序列分布的情況進行統計(圖5)。發現除TIR結構依舊完整之外,與完整MLE轉座子相比較,其他非自主轉座子各個部分DNA序列缺失嚴重,且缺失情況并不存在規律。PhV2MLE1NA1和PhV2MLE2NA1保留了大部分HTH和DDD結構域,PhV2MLE1NA2、PhV2MLE1NA3、PhV2MLE1NA4的轉座酶缺失比較嚴重,PhV2MLE2NA2、PhV2MLE2NA3保留了TIR序列及側翼序列,而PhV2MLE2NA4只具備完整的TIR序列。
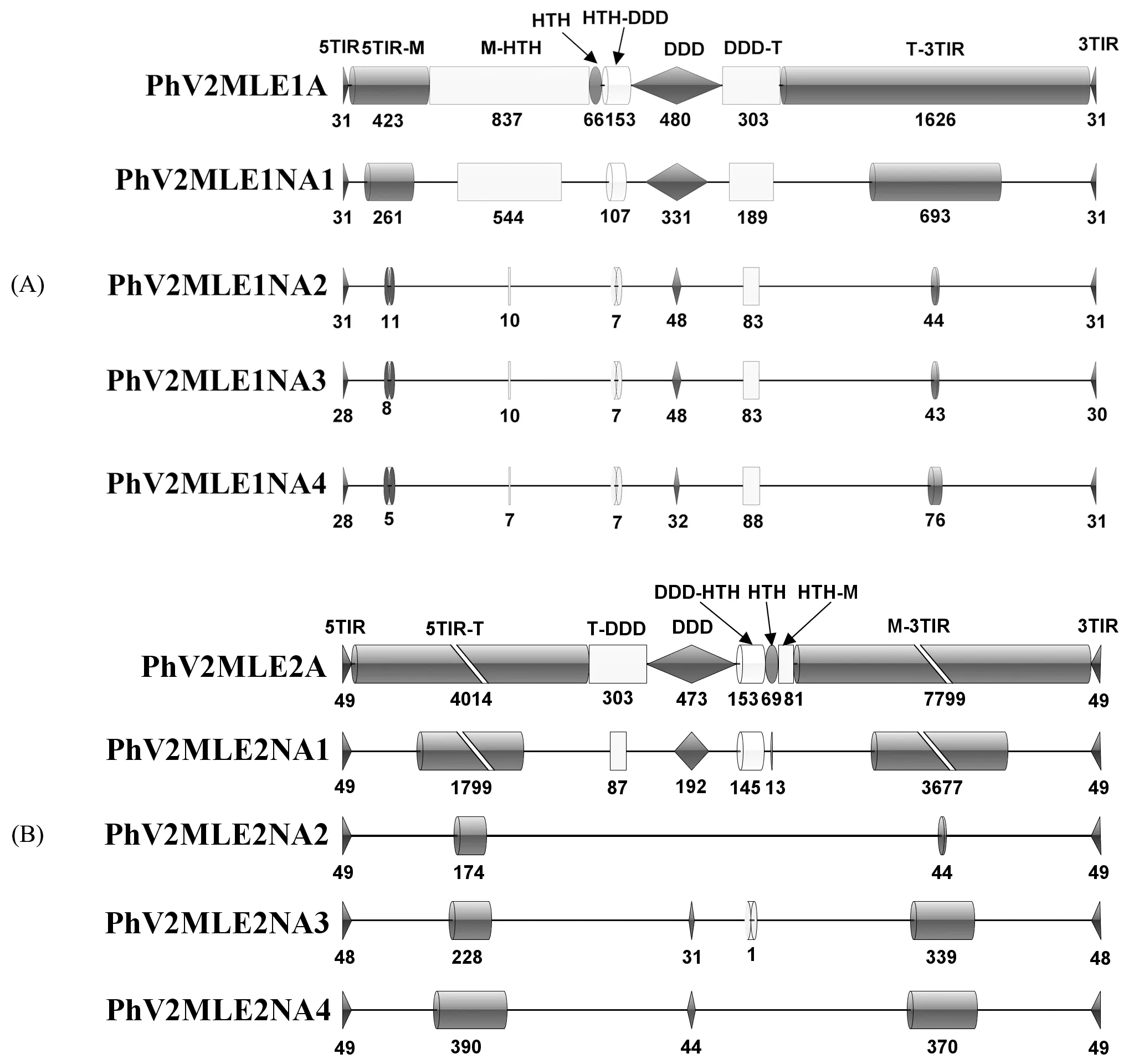
圖5 PhV2MLE1A(A)和PhV2MLE2A(B)及其非自主轉座子結構缺失情況Fig.5 PhV2MLE1A (A)and PhV2MLE2A (B)and their non-autonomous transposons structure deletion
2.4 轉座子插入偏好性規律 對鑒定到的2個MLE自主轉座子及其對應的非自主轉座子的側翼20 bp序列進行比對分析(圖6),PhV2MLE1A和PhV2MLE2A以及它們的非自主轉座子插入位點上下游均為2 bp的TA,MLE轉座子在插入基因組的過程中存在著明顯的插入偏好性。

圖6 PhV2MLE1A及其非自主轉座子(A)和PhV2MLE2A及其非自主轉座子(B)兩側側翼序列富集分析Fig.6 Sequence enrichment analysis on flanking of PhV2MLE1A and its non-antonomous transposon (A)and PhV2MLE2A and its non-antonomous transposon (B)
3 討論
本次研究從MLE轉座子具有特征性的TIR序列入手,首先對新版毛竹全基因組中全部含有TIR結構的序列進行了發掘,再分析具有MLE轉座酶的MLE轉座子序列,全面鑒定了毛竹MLE自主轉座子,也對相應的MLE非自主轉座子進行了鑒定和分析。
MLE轉座子在進化過程中以垂直傳遞為主,也可通過橫向傳遞的方式侵入其他物種基因組中,頻繁轉座,大量擴增自身的拷貝數,從而在宿主基因組長期進化中保留下來(Miskeyetal.,2005;Lampeetal.,2001;Hartletal.,1997)。一個轉座子從侵入宿主基因組到在宿主基因組穩定下來一般要經歷5個階段:1)外源轉座子的侵入;2)高頻轉座以擴增拷貝數;3)通過物種雜交在群體里廣泛擴散;4)大量轉座子積累點突變和插入/缺失突變喪失活性;5)通過轉座子的隨機丟失,宿主基因組和轉座子達到生態平衡。處在第2)階段的MLE轉座子活性最強,目前在基因組鑒定的MLE轉座子大部分處在4)或5)階段,積累了或多或少的突變,部分或全部喪失了轉座能力,成為低活性或非活性的轉座子“化石”。本研究從新版毛竹基因組中共找到了310 845個TIR結構,在這些TIR結構中找到了301個可編碼完整或者部分MLE轉座酶的DNA序列(e值小于-5),但是只有2個可被鑒定為潛在自主轉座子。說明在毛竹的基因組中分布著大量的MLE轉座子,在毛竹進化過程中,絕大多數的MLE轉座子的轉座酶結構發生了缺失,這可能是毛竹進化過程中對于轉座子活動的一種調控,通過對MLE轉座子的結構進行了干預,使其MLE轉座酶結構缺失,從而喪失轉座能力(Mtésetal.,2009)。
本次共找到2條結構完整的MLE轉座子PhV2MLE1A和PhV2MLE2A,將它們的轉座酶與水稻、大豆以及毛竹中已經報道的MLE轉座子的轉座酶比對,發現PhV2MLE1A和PhV2MLE2A轉座酶具有完整的功能結構。通過與動植物中的MLE轉座酶比對構建進化關系,PhV2MLE1A和PhV2MLE2A轉座酶與同屬禾本科(Gramineae)的水稻中的MLE轉座酶相似性非常高,而這些轉座酶已經被確定具有轉座活性。PhV2MLE1A轉座子(Phmar2)已經證明具有轉座活性,因此PhV2MLE2A很可能也具有自主轉座活性,是下一步重點研究對象。Zhou等(2015)報道的另一個毛竹Phmar1轉座子在本次篩選中沒有發現,可能原因是本研究毛竹全基因組測序中所選取的毛竹樣品與Zhou等(2015)克隆轉座子所用毛竹植株存在差異,在不同毛竹個體中MLE轉座子的活動及分布狀態有較大差異。
將結構完整轉座子PhV2MLE1A和PhV2MLE2A序列與其非自主轉座子進行比對后發現,非自主轉座子的缺失情況并不存在規律,轉座子內部的編碼區與非編碼區都存在著序列缺失的情況。這些在進化過程中喪失自主轉座能力的MLE轉座子,當其對應的轉座酶表達時,可以借助轉座酶的催化作用發生轉座,此特性已經在擬南芥(Arabidopsisthaliana)和酵母(Saccharomycescerevisiae)等模式生物中驗證(Zhouetal.,2016)。
通過對2個MLE自主轉座子PhV2MLE1A和PhV2MLE2A,及其對應非自主轉座子插入位置側翼DNA序列比對發現,MLE轉座子插入位點兩側都是2 bp的TA,說明MLE轉座子在轉座過程中存在著明顯的特異性,插入位點選擇有偏好TA的現象(Feschotteetal.,2005)。
4 結論
毛竹基因組資源豐富,是很好的植物基因庫。而轉座子活動是生物進化的重要動力,毛竹基因組中MLE轉座子的分布情況,對研究毛竹基因組的構成與進化有著重要的意義。本研究從毛竹基因組中篩選得到的MLE自主轉座子PhV2MLE1A和PhV2MLE2A,擁有高度保守的TIR結構與完整的編碼轉座酶結構,可以為基因標簽的開發提供新的選擇。另一方面,從自主轉座子與非自主轉座子角度出發,研究了MLE轉座子在基因組中的分布與缺失特點,MLE轉座子在毛竹基因組中的分布與缺失情況并無明顯規律,探究MLE轉座子的活動規律有助于揭示MLE轉座子在毛竹進化過程中的演變規律。

