基于數據庫分析CDCA基因家族在胃癌中的表達及預后意義*
羅 倩,詹雪冰,董芳媛,孫恩濤,王書翰,陳 冰
(1.皖南醫學院病理教研室,安徽 蕪湖 241002; 2.皖南醫學院檢驗學院)
胃癌(Gastric cancer, GC)是全球第五大最常見惡性腫瘤,其致死率在所有腫瘤中排名第三[1]。手術是胃癌患者的主要治療手段,在過去的幾年里診斷和治療有所進步,但由于大部分患者發病初期無明顯癥狀,就診時已進入中晚期,并且超過半數的胃癌根治術術后會有復發或伴有遠處轉移,胃癌患者總體5年生存率低于30%[2]。因此,分子標記物異常表達的檢測可能為胃癌患者早期診斷和預后的判斷提供有效的參考。
細胞分裂周期相關蛋白(cell division cycle-associated,CDCA)家族有8個成員,即CDCA1(NUF2)、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6(CBX2)、CDCA7、CDCA8。CDCAs基因在生命過程中發揮著重要作用,細胞分裂過程中的失調可能導致惡性腫瘤[3]。據報道,CDCA1的缺失抑制了CD103的抗腫瘤活性,降低了黑色素瘤的檢查點反應,促進了黑色素瘤的發生和轉移[4]。CDCA2通過調控PI3K/AKT通路,影響結直腸癌細胞體內外的增殖[5]。CDCA3作為miR-145-5的靶基因,參與調控結直腸癌的增殖、轉移和上皮細胞-間充質轉化[6]。CDCA4是Nrf2信號通路的下游基因,在MCF-7/ADM人乳腺癌細胞系中調控細胞增殖和凋亡[7]。CDCA5通過調節線粒體介導的凋亡、細胞周期調節和PI3k/AKT/mTOR通路激活,在膀胱癌中發揮腫瘤啟動子的作用[8]。CDCA6和CDCA7的異常表達在前列腺癌、乳腺癌、腎透明細胞癌和食管癌中被發現[9-10]。CDCA8的表達可被KIF18B激活,進而影響胰腺導管腺癌的增殖[11]。以上說明CDCAs基因在腫瘤的發生發展過程中扮演著重要的角色。
本研究利用Oncomine數據庫分析CDCAs基因在胃癌中的表達水平,利用GEPIA數據庫驗證CDCAs基因在胃癌中的表達水平,結合Kaplan-Meier Plotter數據庫的臨床信息分析CDCA基因家族成員在胃癌中的預后結果;利用STRING數據庫構建CDCAs基因的蛋白質互作網絡,并得到與CDCA基因家族共表達的基因;利用metascape數據庫將CDCAs基因及其共表達基因的功能信息進行整合,研究其潛在的作用機制。以上生物信息學分析結果有望為尋找胃癌有效的預后標志物提供新思路。
1 對象與方法
1.1Oncomine數據庫分析 本研究利用Oncomine數據庫(https://www.oncomine.org/)分析正常胃組織和胃癌組織。設定篩選條件:(1)gene:CDCA1、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6、CDCA7、CDCA8;(2)fold change >2,P<0.05,gene rank = top 10%。
1.2GEPIA數據庫分析 GEPIA數據庫(http://gepia.cancer-pku.cn/)可利用TCGA和GTEx直接進行CDCAs基因的表達分析。設置限定條件:|Log2FC|Cutoff: 1,P值 Cutoff: 0.05。
1.3Kaplan-Meier Plotter數據庫分析 Kaplan-Meier Plotter(https://kmplot.com/analysis)數據庫采用Kaplan-Meier法對多種腫瘤患者的生存資料進行預后分析。篩選條件如下:(1)cancer:gastric cancer;(2)gene symbol:CDCA1、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6、CDCA7、CDCA8;(3)Split patients by: best cutoff;(4)survival:OS。在進行總生存期(overall survival,OS)分析的同時,計算危險比(hazard radio,HR)、95%置信區間(confidence interval,CI)及LongrankP<0.05檢驗差異,LogrankP<0.05差異有統計學意義。
1.4STRING數據庫分析 STRING數據庫(https://string-db.org/)是一個在線搜索已知的蛋白質互作關系的數據庫,本研究于2020年12月28日訪問STRING數據庫11.0版。選擇Multiple Proteins板塊,條件設置為:(1)List Of Names: CDCA1、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6、CDCA7、CDCA8;(2)Organism: Homo sapiens;(3)minimum required interaction score: medium confidence(0.400)。
1.5metascape數據庫分析 metascape數據庫(http://metascape.org)整合了GO和KEGG等多個數據資源,可完成通路富集和相關基因的蛋白質網絡分析等。條件設置為:(1)Multiple Gene List: CDCA1、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6、CDCA7、CDCA8;(2)Input as species and Analysis as species: H.sapiens。最后進行通路富集分析。
2 結果
2.1Oncomine數據庫中胃癌患者CDCA基因的表達情況 Oncomine數據庫結果顯示:CDCA1、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6、CDCA7、CDCA8在胃癌組織中顯著升高(圖1)。分析三個數據集“Cui Gastric”,“Derrico Gastric”和“Cho Gastric”,得到CDCA基因家族在胃癌組織和正常組織的差異表達分析結果(表1)。
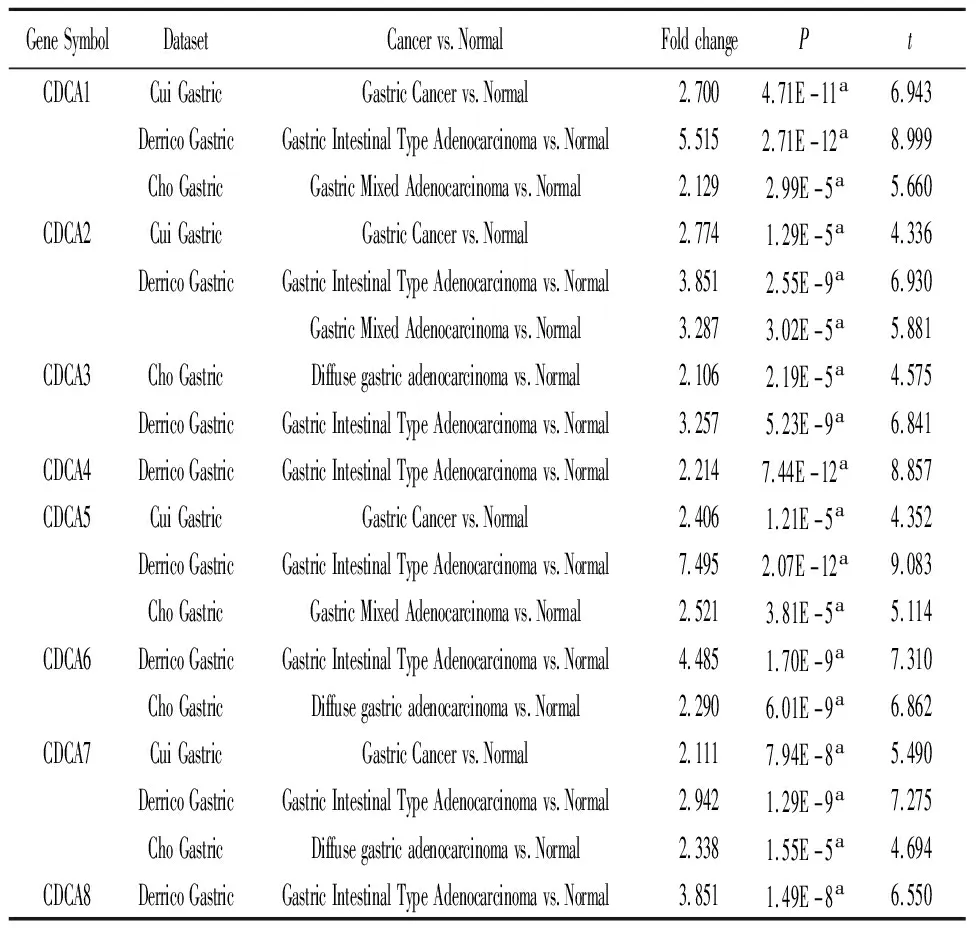
表1 不同類型胃癌與正常肺組織之間CDCA轉錄水平的表達差異顯著
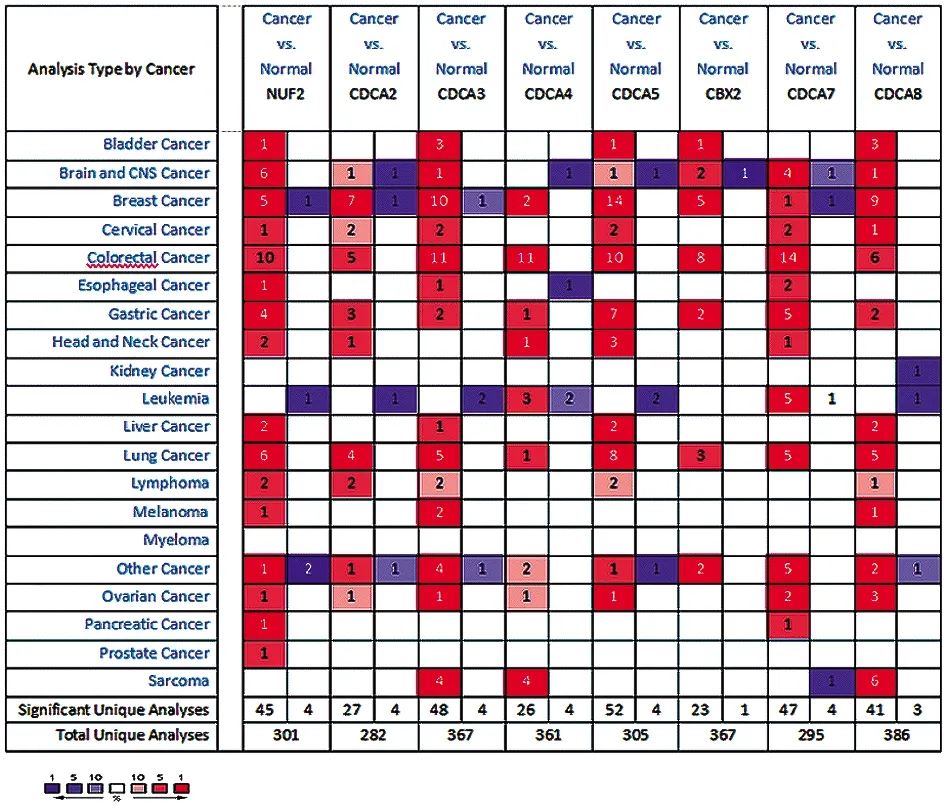
圖1 CDCA在不同腫瘤中的轉錄水平表達
2.2GEPIA數據庫中胃癌患者CDCA基因的表達情況 結果顯示,CDCA1、CDCA2、CDCA3、CDCA4、CDCA5、CDCA6、CDCA7、CDCA8的表達水平在胃癌組織中相對于在正常組織中上調(腫瘤樣本:n=408,正常樣本:n=211,P<0.05)(圖2)。
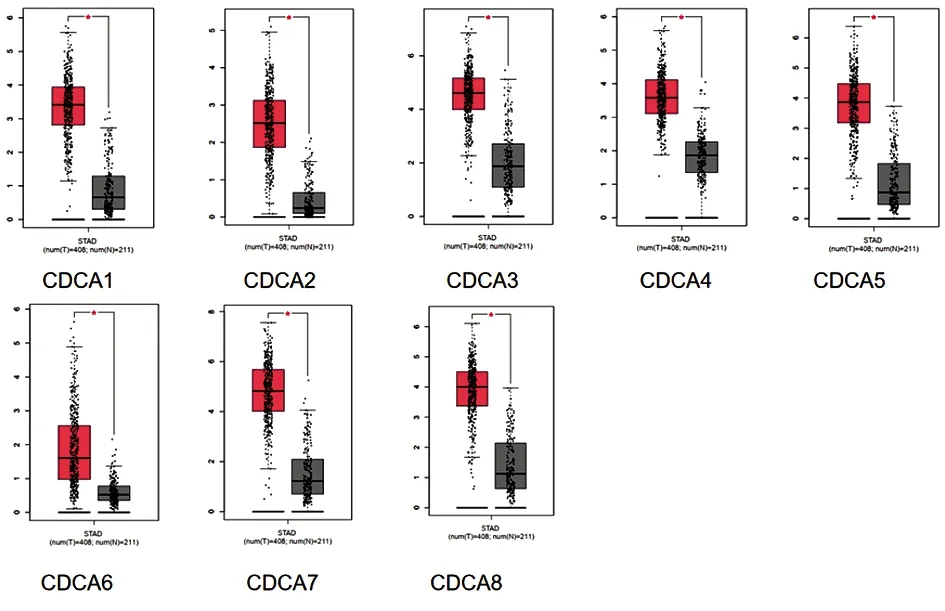
圖2 CDCA基因家族在胃癌組織和正常組織中的表達
2.3Kaplan-Meier Plotter數據庫中CDCA基因家族在胃癌組織中的表達與患者臨床病理資料的生存分析 結果顯示,CDCA1、CDCA2、CDCA3、CDA4、CDCA5、CDCA7、CDCA8的表達高低與預后相關。以HER2(+)/(-)和不同臨床分期為前提條件進行生存分析,CDCA1、CDCA2、CDCA3、CDCA5、CDCA7、CDCA8基因高表達胃癌患者的總生存期較低表達胃癌患者的總生存期顯著延長,而CDCA4基因低表達胃癌患者預后更好(圖3)。見表2、3。
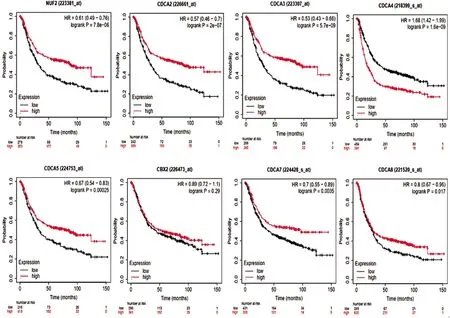
圖3 胃癌患者CDCA基因家族的OS分析
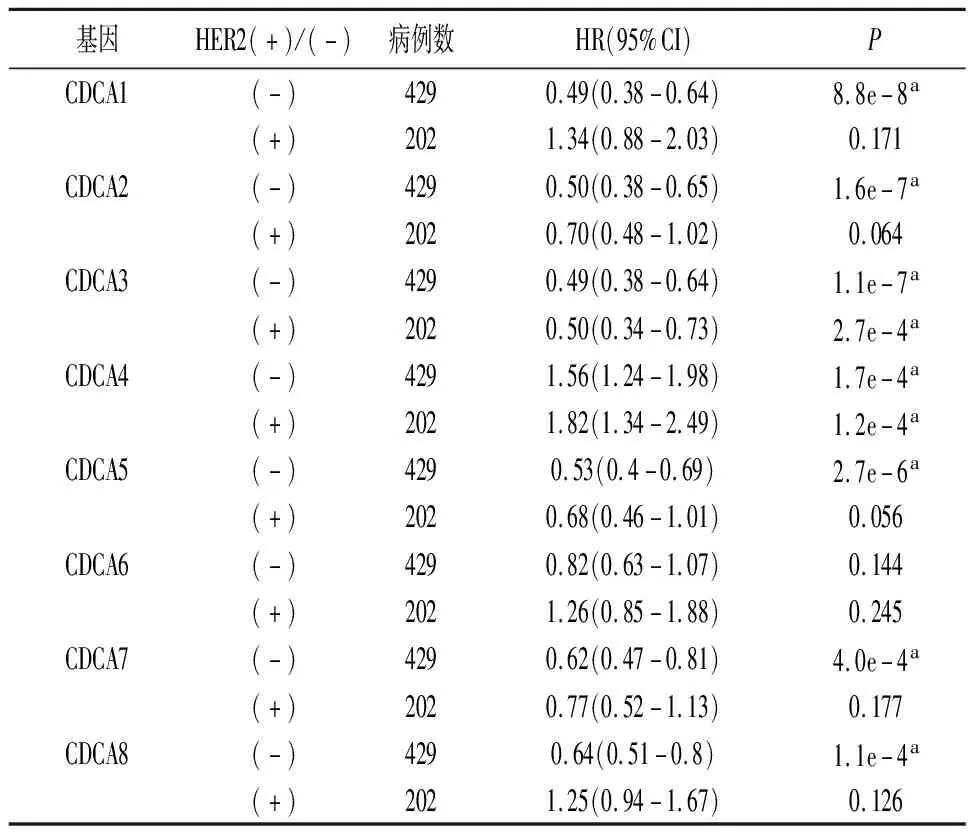
表2 CDCA基因家族表達與胃癌患者HER2(-)/(+)的相關性
2.4STRING數據庫中胃癌患者CDCA基因的通路富集分析 結果顯示,CDCAs基因在胃癌中呈正相關的共表達基因為:CDCA2、CDCA7、CDCA4、CDCA1(NUF2)、CDCA3、CDCA6(CBX2)、CDCA8、CDCA5、NDC80、SPC25、BIRC5、BUB1、AURKB、CDC20、INCENP、SPC24、CCNB1和CENPE,作用評分均大于0.99(圖4a、4b)。
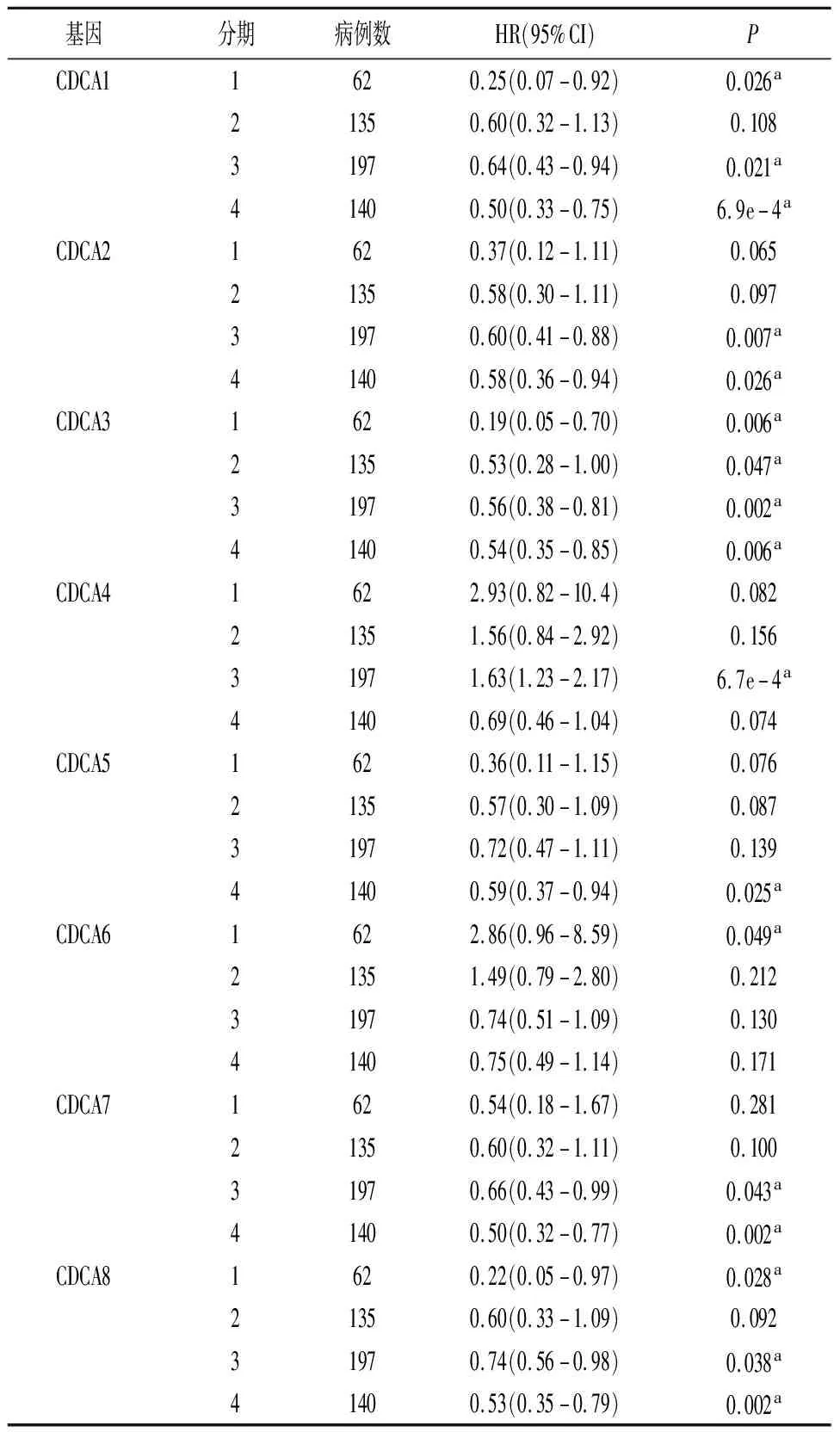
表3 CDCA基因家族表達與胃癌患者分期的相關性
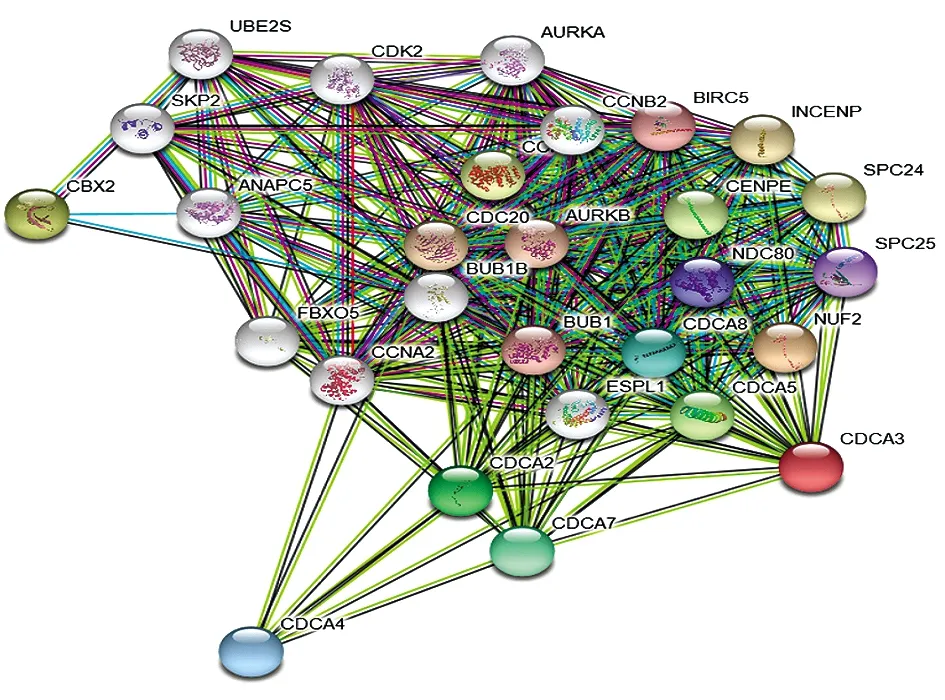
圖4a CDCAs基因基于STRING數據庫的蛋白質互作分析
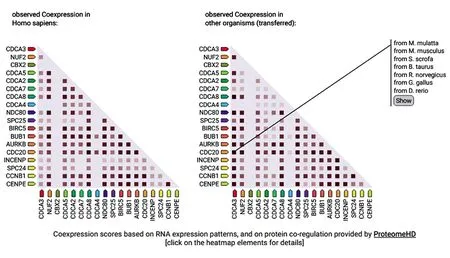
圖4b CDCAs基因基于STRING數據庫的基因共表達分析
2.5metascape數據庫中胃癌患者CDCA基因的功能預測 結果顯示,CDCA基因的改變影響了以下幾個過程:(1)R-HAS-2500257: 分辨姐妹染色單體粘連;(2)M129: PID PLK1通路;(3)M14: PID AURORA B通路;(4)CORUM:127: NDC80著絲點復合體;(5)GO:0030261: 染色體凝縮;(6)GO:0140013: 減數分裂(圖5a、5b和5c)。
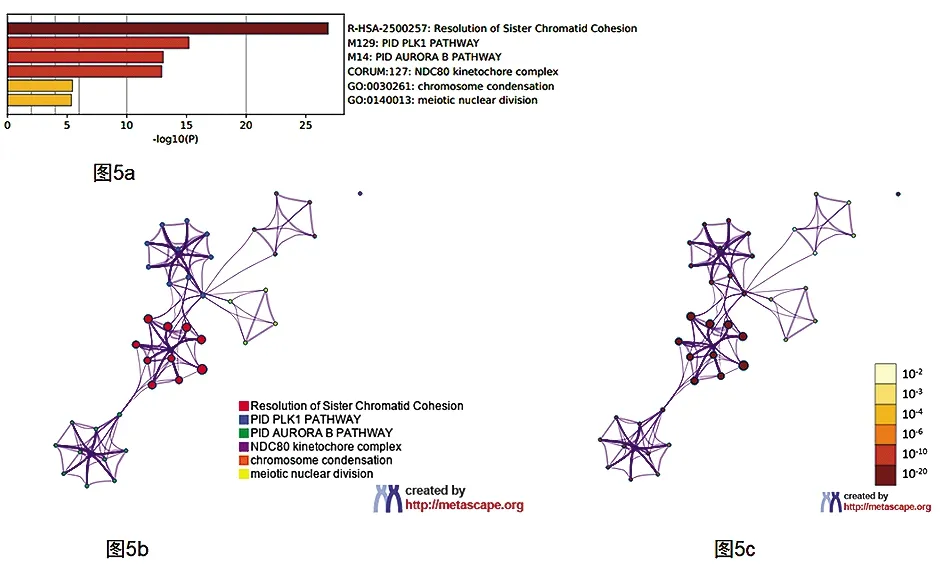
圖5 CDCAs基因的功能以及與CDCAs基因改變顯著相關的功能
3 討論
近年來有研究表明CDCA基因家族可能有助于腫瘤的發展[12],但目前較少有胃癌中CDCA基因家族的表達情況和預后分析的相關報道。細胞周期異常調節所介導的細胞轉化和不受控制的細胞生長為腫瘤的增殖提供了有利條件,因此腫瘤的發生發展離不開細胞分裂[13]。值得注意的是CDCA基因家族部分家族成員在腫瘤中高表達,具有調節腫瘤細胞周期、促進腫瘤細胞增殖、減少腫瘤細胞凋亡的作用,導致預后不良[14]。本研究利用Oncomine數據庫和GEPIA數據庫進行分析,均提示CDCA基因家族8個成員的mRNA在胃癌組織中呈高表達,說明CDCA基因家族成員可能在胃癌的發生發展中起著一定的作用。
有報道CDCA基因家族的一些成員可能在乳腺癌、肺腺癌和胰腺癌中上調,這些基因的高表達水平被認為是這些腫瘤生存的不良預測因素[12]。本研究利用Kaplan-Meier Plotter數據庫進行生存分析,結果表明除CDCA6基因差異無統計學意義,CDCA基因家族另外7個成員中,CDCA1、CDCA2、CDCA3、CDCA5、CDCA7、CDCA8的高表達與胃癌患者的總生存期延長有關,CDCA4在胃癌中的高表達,與胃癌患者的總生存期減少有關。本研究以HER2(+)/(-)和不同臨床分期為前提條件進行分析,CDCA4基因表達下調,CDCA1、CDCA2、CDCA3、CDCA5、CDCA7、CDCA8基因表達部分上調。已有報道胃癌HER2狀態和胃癌的預后有一定的相關性[15]。值得注意的是,本研究中以HER2(+)/(-)和不同臨床分期為前提條件的結果與胃癌患者總生存期結果相比結果有差異,說明CDCA基因家族基因在胃癌組織中的表達情況可能與HER2通路有關,還需進一步研究證實。另外,本研究CDCA6基因的mRNA在胃癌組織中的表達高于正常組織,但其表達水平對胃癌患者生存期無明顯影響,推測可能CDCA6的功能主要作用在于腫瘤的發生,對胃癌患者后期影響較小。
此外,為探討CDCA基因家族成員影響胃癌預后的機制,本研究利用STRING數據庫和metascape數據庫進行了KEGG和GO的分析,找出CDCAs基因表達和突變頻率最高的連鎖基因與胃癌預后的關系,結果是CDCAs基因改變會影響以下途徑:(1)R-HAS-2500257: 分辨姐妹染色單體粘連;(2)M129: PID PLK1通路;(3)M14: PID AURORA B通路;(4)CORUM:127: NDC80著絲點復合體;(5)GO:0030261: 染色體凝縮;(6)GO:0140013: 減數分裂。凝縮后的染色體包含兩個姊妹染色單體,兩單體由著絲粒連接。研究發現,PLK1是有絲分裂激酶信號的主要調節者和協調者,而AURORA B就是有絲分裂激酶成員之一[16]。PLK1通過調節AURORA B的活性確定著絲粒的身份,AURORA B激酶通過磷酸化著絲粒上的NDC80蛋白,調控NDC80寡聚化作用來糾正錯誤的微管-動粒連接,動粒與微管正確連接的前提是姐妹染色單體的正常粘連[16-18]。本研究推測CDCAs基因及其突變頻率最高的連鎖基因可能通過調控PID PLK1和PID AURORA B信號通路影響NDC80著絲點復合體,進而干擾動粒與微管的連接影響姐妹染色單體粘連,最終影響染色體的穩定性,使腫瘤細胞的生長失控。因此,對CDCAs基因表達的調控,有望通過調節上述信號通路影響染色體的穩定性,將胃癌細胞阻滯在有絲分裂期,使胃癌細胞發生凋亡,進而影響胃癌的發生發展。
綜上所述,本研究利用生物信息學,探討CDCAs基因的表達水平與胃癌發生發展的關系,提示CDCAs基因表達水平可能與胃癌的預后相關。篩選CDCAs基因在胃癌中的共表達基因然后進行GO和KEGG富集分析,探討了其可能在胃癌發生發展中的機制。目前,檢測CDCAs基因的異常表達可能為胃癌患者早期診斷和預后提供新思路,并可能為胃癌的預后診療提供新的方法和新的理論依據。同時,深入研究CDCA基因家族成員在腫瘤的發生發展也為研究其他胃腸道腫瘤提供新的思路和方向。

