利用全基因組關聯分析挖掘粳稻直鏈淀粉含量調控基因
鄭洪亮,孫世臣,丁國華,王彤彤,趙宏偉,王敬國,劉化龍,韓 笑,鄒德堂,來永才
(1.黑龍江省農業科學院博士后科研工作站,哈爾濱 150086;2.東北農業大學寒地糧食作物種質創新與生理生態教育部重點實驗室,哈爾濱 150030;3.黑龍江省農業科學院,哈爾濱 150086)
淀粉作為稻米主要成分,占稻米干重90%左右,其組分主要包括直鏈淀粉和支鏈淀粉,其中直鏈淀粉含量是影響水稻蒸煮食味品質重要因素,研究認為稻米直鏈淀粉含量、結構和組成影響膠稠度、糊化溫度[1],且稻米蒸煮食味與直鏈淀粉含量呈顯著負相關,與膠稠度呈顯著正相關[2]。因此,挖掘稻米直鏈淀粉含量QTLs/基因,對于改良稻米品質具有重要意義。
稻米直鏈淀粉含量是由多基因控制的數量性狀,利用正向遺傳學和反向遺傳學方法挖掘到與直鏈淀粉含量相關QTLs/基因。目前,已利用傳統QTL分析方法從32個遺傳群體中定位直鏈淀粉含量相關QTL 141個[3],但由于不同研究人員采用試驗材料遺傳背景差異較大,且選用分子標記不同,導致定位結果差異較大。李修平等進一步采用元分析方法對141個QTL構建一致性圖譜,最終映射得到30個“一致性”QTL[3]。目前,已克隆水稻直鏈淀粉相關基因11個,包括位于第6染色體上的顆粒結合型淀粉合成酶基因Wx,也是公認的水稻直鏈淀粉含量主效基因[4],位于第6和第8染色體上與可溶性淀粉合成酶相關的3個基因SSSI[5]、SSIIa[6]和SSIIIa[7],位于第1、3、5、7、8、9染色體上與ADP葡萄糖焦磷酸化酶相關的6個基因[8]:AGPL1、AGPL2、AGPL3、AGPL4、AGPS1、AGPS2a,以及位于第2染色體上與淀粉分支酶相關的基因SBEIIb[9]。但以上研究結果對于解析稻米直鏈淀粉含量復雜的遺傳基礎仍不充分。
除傳統QTL分析外,全基因組關聯分析是另外一種解析復雜性狀遺傳基礎的重要方法,具有群體構建簡單,表型變異豐富,且可在同一位點上分析多個等位基因,定位精度高等優勢。目前稻米品質相關研究應用廣泛,如Chen等利用收集世界各地527份水稻品種為試驗材料,結合3 916 415個SNP對稻谷籽粒4種貯藏蛋白作全基因組關聯分析,共檢測到34個位點與4種貯藏蛋白顯著關聯,其中有28個位點與已知QTLs/基因處于相同或相近位置[10]。Qiu等以272份秈稻品種構成的自然群體為試驗材料,利用18 824個高質量SNP對粒長、粒寬、長寬比、粒厚、千粒重、堊白度、堊白率、糙米率等10個性狀作全基因組關聯分析,共檢測到38個顯著關聯QTL,其中有5個QTL為已知QTLs/基因[11]。但利用全基因組關聯分析研究粳稻直鏈淀粉含量遺傳基礎及基因挖掘的報道較少。
本研究以收集的295份溫帶粳稻種質組成的自然群體為試驗材料,于2019~2020年測定稻米直鏈淀粉含量,結合高通量測序獲得788 396個多態性SNP作GWAS分析,并針對重要QTL區間挖掘候選基因,以期為解析粳稻直鏈淀粉含量遺傳機制及利用分子育種手段改良稻米品質奠定基礎。
1 材料與方法
1.1 試驗材料
以國內外收集的295份溫帶粳稻種質構成的自然群體為試驗材料,國內材料主要來自于黑龍江、吉林、遼寧和寧夏,國外材料主要來自于日本、韓國、朝鮮和俄羅斯。具體材料明細見表1。
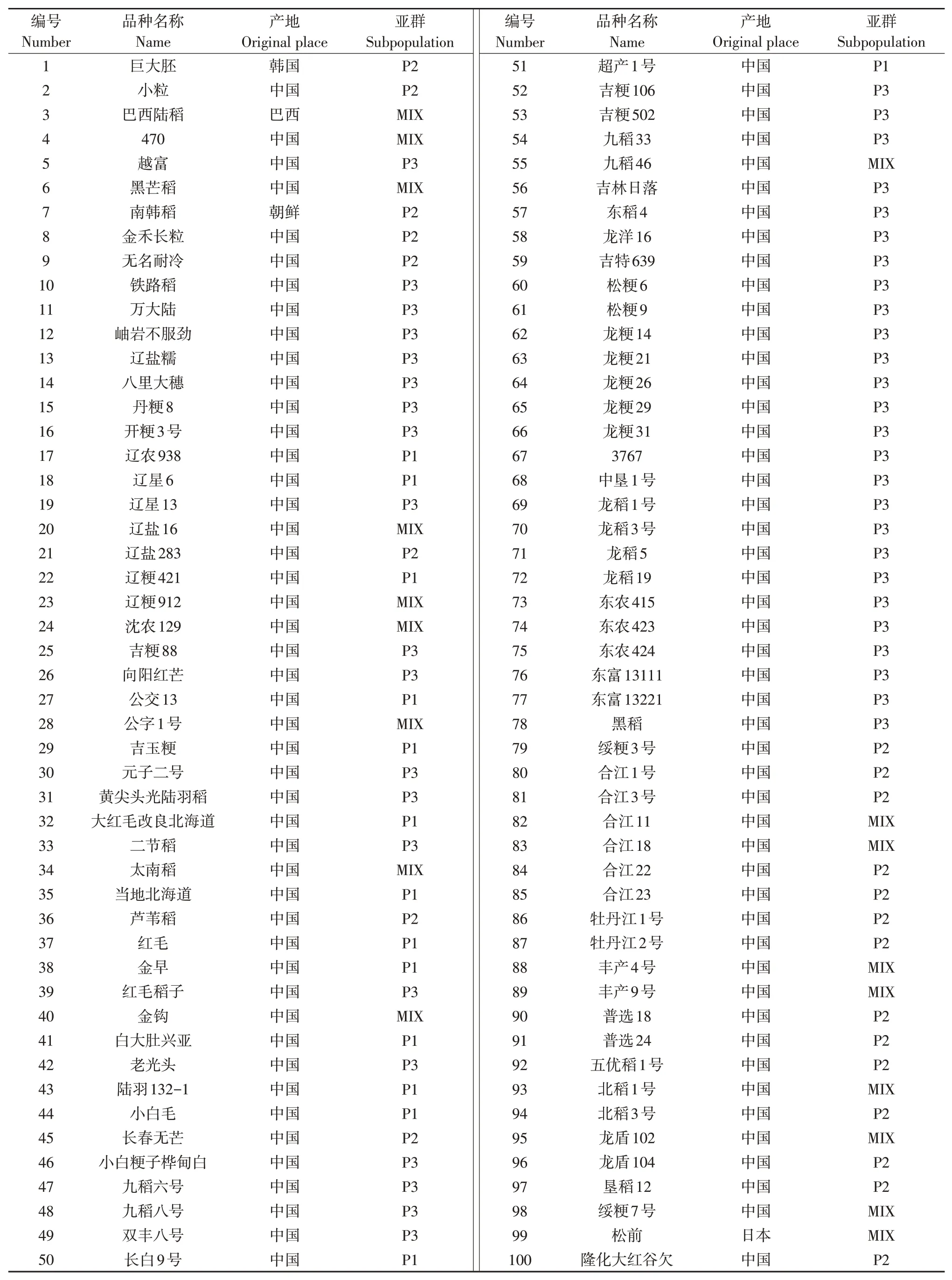
表1 295個粳稻品種具體信息Table 1 Detailed information of 295 japonica rice varieties
1.2 田間種植及稻米直鏈淀粉含量測定
2019~2020年將所有供試材料種植于東北農業大學阿城實驗實習基地水稻試驗基地,4月15日播種,5月20日插秧,插秧密度為30 cm×16.7 cm,4行區,每行40株,田間采用隨機區組設計,3次重復,田間管理同當地大田生產。于水稻成熟后收獲籽粒,置于40℃烘箱中烘干48 h至水分含量為14%,經糙米機、精米機及磨粉機加工后,用于稻米直鏈淀粉含量測定,測定方法參照國家標準(GB/T17891-2017)[12]。
續表
續表
1.3 全基因組重測序、群體結構、親緣關系及連鎖不平衡分析
本研究所用自然群體基因型數據獲取及群體結構、親緣關系、連鎖不平衡分析等工作前期實驗室已完成,具體分析過程及結果如下:①利用Illumina HiSeq XTen平臺作高通量測序,平均測序深度14.62×,以粳稻品種日本晴(IRGSP-1.0)為參考基因組,采用BWA軟件序列比對,采用GATK軟件“Best Practice”作群體SNP檢測,共獲得3 437 749個多態性SNP標記,進一步利用Plink軟件作數據質控,最終篩選出最小等位基因頻率(MAF)>5%,且缺失率(Missing data)<20%的788 396個SNP用于后續分析;②利用ADMIXTURE軟件分析群體結構[13],結果表明,當K=3時,群體CV值最小,因此將群體劃分為3個亞群,并將其對應Q矩陣用于后續關聯分析;③利用Tassel 5.0軟件評估群體材料間親緣關系[14],結果表明,親緣關系系數小于0.1材料占78.8%,>0.5材料僅有0.4%,因此本研究所用群體材料間親緣關系較遠,對后續GWAS分析結果影響較小;④利用PopLDdecay軟件計算得到水稻全基因組r2值[15],依據Huang等方法[16],將r2衰減到最大值一半時對應的物理距離作為LD衰減距離,經計算,位點間最大r2值為0.84,群體LD衰減距離為109.7 kb。
1.4 稻米直鏈淀粉含量全基因組關聯分析
利用Tassel 5.0軟件混合線性模型(Q+K)對稻米直鏈淀粉含量作GWAS分析[14],采用Li等方法通過GEC軟件計算有效獨立SNP數目,最終將P<5.46×10-6作為顯著性關聯閾值[17]。如果在LD區間內有多個顯著SNP存在,則將這些SNP視為同一個QTL,結果中僅列出P值最小的SNP作為峰值SNP,且峰值SNP貢獻率代表QTL貢獻率,以峰值SNP位置上下游分別增加109.7 kb(LD衰減距離)作為QTL區間范圍。GWAS分析結果的曼哈頓圖和Q-Q圖使用R語言中“qqman”軟件包繪制。
1.5 候選基因單倍型分析
將兩年共同檢測且不含已知基因的QTL作為重要QTL,根據區間內基因注釋結果,分析區間內所有基因單倍型。具體操作過程參考文獻[18]:①根據水稻注釋數據庫(http://rapdb.dna.affrc.go.jp/)獲取QTL區間內全部基因;②利用水稻3K RGP的Rice SNP-Seek Database網站提取所有基因非同義突變SNP[19],結合QTL區間內所有SNP確定本研究自然群體最終非同義突變SNP;③對具有非同義突變SNP的所有候選基因作單倍型分析;④對不同單倍型(≥10份材料)直鏈淀粉含量作方差分析,篩選出具有顯著性差異基因,并結合基因功能注釋和前人研究結果確定候選基因。
2 結果與分析
2.1 粳稻品種稻米直鏈淀粉含量表型分析
通過測定2019~2020年295份粳稻材料直鏈淀粉含量。結果表明,直鏈淀粉含量在兩年內均表現出豐富的表型變異且趨勢一致,總體上,2020年各品種直鏈淀粉含量略高于2019年,兩年平均值分別為19.96%和20.37%,變異范圍分別為15.21%~25.34%和15.55%~25.68%,變異系數分別為12.12%和11.59%(見表2)。自然群體偏度值和峰度值絕對值均小于1,表明直鏈淀粉含量兩年表型分布呈近似正態分布,符合典型數量性狀遺傳特征(見圖2)。
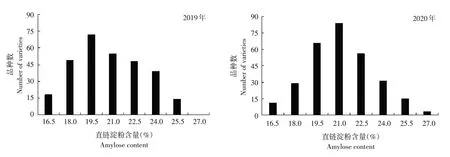
圖2 粳稻群體中直鏈淀粉含量頻率分布Fig.2 Frequency distribution of the amylose content in 295 japonica rice

表2 295份粳稻種質直鏈淀粉含量表型值統計分析Table 2 Phenotypic analysis of amylose content in 295 japonica rice germplasms
2.2 稻米直鏈淀粉含量全基因組關聯分析
利用Tassel 5.0軟件混合線性模型(MLM)對295份粳稻品種直鏈淀粉含量作全基因組關聯分析,曼哈頓圖和QQ散點圖(見圖3)。在顯著性閾值P<5.46×10-6條件下,2019~2020兩年共檢測到與直鏈淀粉含量相關QTL 12個,分布在水稻第3、4、11和12染色體上,貢獻率范圍為8.78%~11.62%。2019年和2020年均檢測到7個QTL,其中qAAC4-2和qAAC12-2在兩年中重復檢測到,qAAC4-2表型貢獻率兩年中分別為11.12%和9.15%,qAAC12-2表型貢獻率兩年中分別為10.62%和10.30%。根據全基因組LD衰減距離,最終將qAAC4-2和qAAC12-2分別定位于水稻第4和12染色體20.27~20.49 Mb和19.14~19.36 Mb(見表3)。
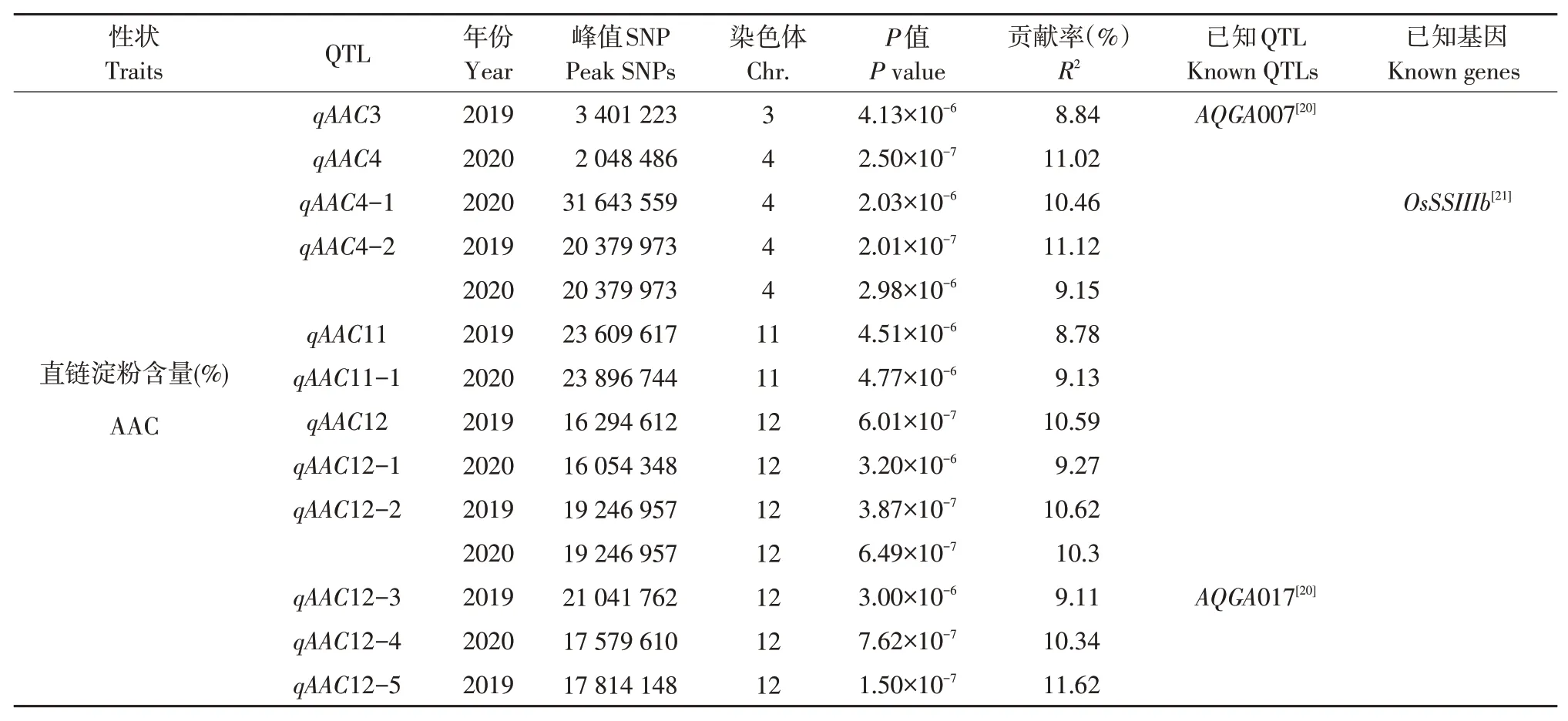
表3 粳稻淀粉相關性狀顯著相關位點Table 3 Significant correlation loci of starch related traits in japonica rice
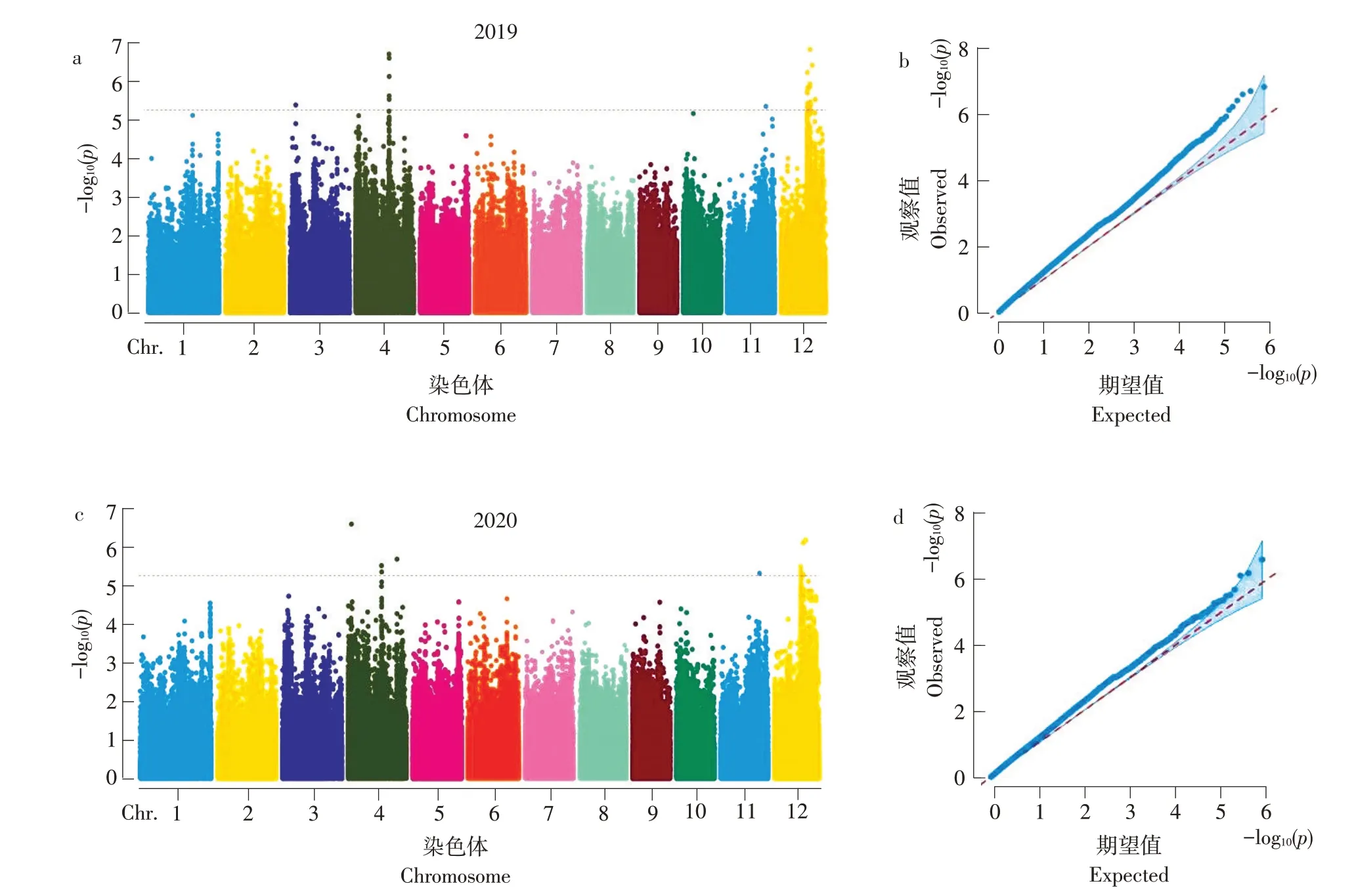
圖3 粳稻直鏈淀粉含量全基因組關聯分析結果曼哈頓圖和QQ散點圖Fig.3 Manhattan plot and quantile-quantile(Q-Q)plots of genome-wide association studies for the amylose content in 295 japonica rice
2.3 稻米直鏈淀粉含量候選基因單倍型分析
針對兩年中同時檢測到的2個QTL(qAAC4-2和qAAC12-2)區間內所有基因分析單倍型。qAAC4-2位于水稻第4染色體20.27~20.49 Mb區間內,該區間包含32個基因,單倍型分析結果表明,共有6個基因不同單倍型直鏈淀粉含量存在顯著差異(見圖4a~f)。
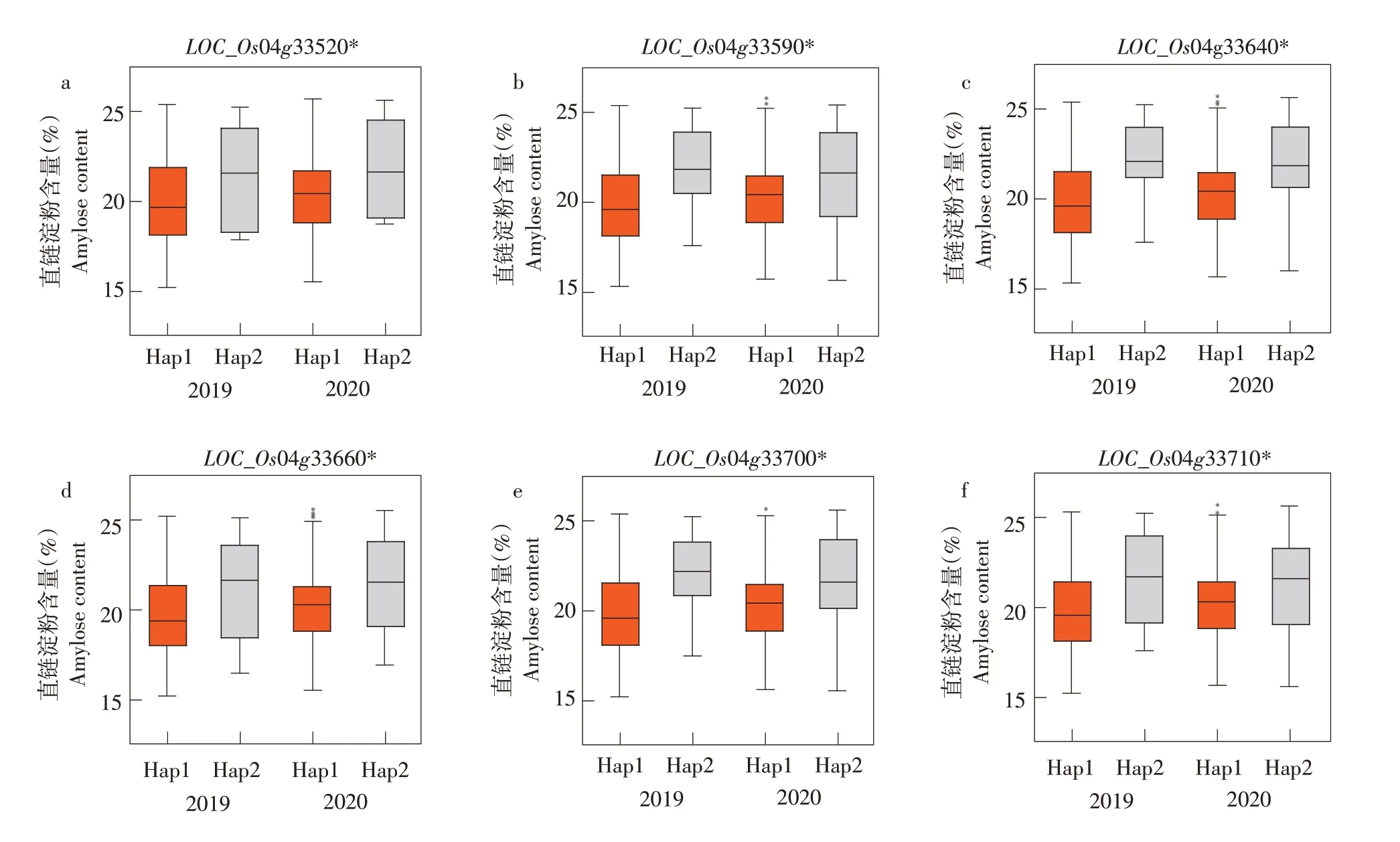
圖4 候選基因不同單倍型之間直鏈淀粉含量箱線圖Fig.4 Boxplots of amylose content between different haplotypes of candidate genes
LOC_Os04g33520非同義突變SNP分為兩種單倍型,Hap2(CA)顯著大于Hap1(TG)(見圖4a);LOC_Os04g33590被非同義突變SNP分為兩種單倍型,Hap2(A)顯 著 大 于Hap1(G)(見 圖4b);LOC_Os04g33640非同義突變SNP分為兩種單倍型,Hap2(T)極顯著大于Hap1(G)(見圖4c);LOC_Os04g33660被非同義突變SNP分為兩種單倍型,Hap2(G)顯著大于Hap1(A);LOC_Os04g33700被非同義突變SNP分為兩種單倍型,Hap2(CCTACC)顯著大于Hap1(TACGTG);LOC_Os04g33710非同義突變SNP共分為兩種單倍型,Hap2(CT)顯著大于Hap1(AG)(見表4)。

表4 候選基因單倍型分組及每種單倍型SNP組成Table 4 Candidate gene haplotype group and the composition of each haplotype SNP
根據基因功能注釋(見表5),LOC_Os04g33640編碼糖苷水解酶,即一種水解糖苷鍵的酶[22],該酶對糖和糖綴合物水解與合成具有調節作用[23],推測其可能影響淀粉鏈長度和分支[24],進而影響直鏈淀粉及支鏈淀粉含量。因此,推測LOC_Os04g33640最可能為qAAC4-2候選基因。另外一個QTL,qAAC12-2位于水稻第12染色體19.14~19.36 Mb區間內,該區間包含27個基因,單倍型分析結果表明,該QTL區間內所有基因不同單倍型直鏈淀粉含量差異不顯著。

表5 候選基因基因注釋Table 5 Candidate gene of gene annotation
3 討論與結論
稻米直鏈淀粉含量除受遺傳因素控制外,溫度、光照、海拔等環境因素及施肥、收獲時期、貯藏時間、碾磨精度等農藝措施均對直鏈淀粉含量有影響[25]。本研究以295份粳稻種質為試驗材料,于2019年和2020年種植于阿城基地,從測定的稻米直鏈淀粉含量看,2019年直鏈淀粉含量平均值為19.96%,變異范圍為15.21%~25.34%,2020年直鏈淀粉含量平均值為20.37%,變異范圍為15.55%~25.68%,其海拔、施肥、收獲時期、貯藏時間、碾磨精度等均不存在差異,而溫度和光照在年際間有較大差異,根據試驗地氣象數據統計,2019年和2020年4~9月份≥10℃活動積溫和日照時數分別為2 764℃、1 272.3 h和2 922℃、1 301.3 h,可知2020年活動積溫和日照時數比2019年分別增加158℃和29 h。說明溫度和光照對稻米直鏈淀粉含量略有影響,且稻米直鏈淀粉含量隨活動積溫和日照時數增加而有所提高。
長期以來針對稻米直鏈淀粉含量遺傳基礎解析開展大量研究,除已克隆主效基因Wx外,近年來通過遺傳群體和自然群體鑒定到眾多QTLs/基因。根據國家水稻數據中心基因數據庫(http://www.ricedata.cn/gene/)和水稻QTL數據庫(https://archive.gramene.org/qtl/)公布信息,將本研究檢測到的12個與直鏈淀粉相關QTL與前人結果作比較,發現部分QTL與前人已定位QTL位于相同、相近區間或包含已克隆淀粉相關基因。如Fan等通過秈稻品種珍汕97和H94雜交得到的F1構建雙倍單倍體(DH)群體為試驗材料[20],利用水稻218個SSR標記位點,檢測到兩個與直鏈淀粉含量相關QTL(AQGA007,AQGA017),分別與本研究2019年檢測到的qAAC3和2020年檢測到的qAAC12-3位于相同區間。另外,本研究2020年檢測到與直鏈淀粉含量相關QTL(qAAC4-1)區間內包含可溶性淀粉合酶Ⅲ基因OsSSIIIb,與Zhao等研究結果一致[21]。表明本研究全基因組關聯分析的檢測結果具有較高準確性,同時也縮小已定位QTL區間。
由于傳統基因圖位克隆需耗費大量人力和時間,因此克隆影響復雜性狀QTL一直是植物遺傳學家和分子生物學家面臨的重大挑戰。采用GWAS方法作QTL分析,并針對QTL區間內所有基因作單倍型分析,篩選出不同單倍型表型值之間存在顯著性差異候選基因,再結合基因注釋和前人研究結果挖掘候選基因,可提高候選基因篩選效率及準確性[26]。本研究針對兩年共同檢測到且不含已知基因的兩個重要QTL(qAAC4-2、qAAC12-2)分析候選基因單倍型,結果發現qAAC12-2區間內所有基因不同單倍型直鏈淀粉含量差異不顯著,而qAAC4-2區間內共有6個基因不同單倍型直鏈淀粉含量存在顯著差異,其中編碼糖苷水解酶的LOC_Os04g33640,對糖和糖綴合物水解與合成具有調節作用。研究表明,α-淀粉酶和β-葡萄糖苷酶均含有這種糖基水解酶結構域,可水解淀粉產生糊精、低聚糖和單糖,從而降低淀粉積累[23-24]。因此,推測LOC_Os04g33640可能具有類似α-淀粉酶功能。下一步將重點針對LOC_Os04g33640開展轉基因功能驗證和分子育種利用,為粳稻品質改良提供理論依據。

