基于微型DNA條形碼的黃粉蝶亞科的物種鑒定
孫永嶺,孫穎慧,趙 宇,李 嬌,冀國澳,王 瑩
(德州學院生命科學學院,山東德州253023)
黃粉蝶亞科(Coliadinae)隸屬于鱗翅目(Lepidoptera),粉蝶科(Pieridae),是蝴蝶中較常見的類群。體中形,色彩較為素雅,多為黃色、橙色或白色,全世界已知11 屬240 種,中國記載有6 屬38 種。黃粉蝶亞科幼蟲卵化后喜食植物葉片與幼嫩莖稈,對農(nóng)林業(yè)造成嚴重危害。迄今為止,我國黃粉蝶亞科幼蟲及卵在形態(tài)學上無法進行精準鑒別,即對有害蝶類昆蟲的鑒定主要依據(jù)其成蟲的外部形態(tài),但因?qū)⒂紫x飼養(yǎng)到成蟲再鑒別這一階段費時費力,所以探討準確、快速、有效的黃粉蝶亞科鑒定方法很有必要。
近20年來,采用分子生物學技術對物種鑒定和分類的影響巨大,可以解決傳統(tǒng)分類法無法高效鑒定蝶類昆蟲的問題。經(jīng)典分類學方法具有局限性,很難快速、有效地對所得生物樣本進行鑒定。因此,2002年Tautz 等人[1]首先提出了DNA 分類的概念。隨后,Herbert[2-3]所提出的線粒體COI 基因指定片段(648bp)為基礎的DNA條形碼(Minimalist-barcode)在新型物種基因鑒定分析方面的優(yōu)勢已經(jīng)在學術界受到了廣泛關注。但是DNA條形碼技術也存在不足,DNA 容易降解這一特性制約了條形碼的適用范圍。當樣本質(zhì)量不高,存在DNA 降解情況時,常常難以獲得完整的DNA 條形碼信息,給不同類物種的有效鑒定工作帶來極大困擾[4]。
微型DNA 條形碼(Minimalist-barcode,簡寫為mini-barcode)技術主要是通過對中國自然科學博物館各種鳥類標本的DNA 條形碼鑒定設計得出的[5]。對于存在于博物館里面的,且年代比較久遠的生物標本或者化石中的古老物種,由于時間的流逝,DNA 片段會丟失或者受到干擾,DNA 條形碼技術使用會受到很大限制。因此,Hebert 首次明確地選取了一個物種的200bp 長度的條形碼并用來進行物種鑒定的微型識別基因序列,由于在200bp 指定長度的一個指定物種序列比較容易無需進行擴增鑒定即可直接獲得,且通過數(shù)據(jù)采集分析和綜合研究所得到的結(jié)果表明,該物種序列指定長度的一個指定物種序列長度在野外原生物種微型識別鑒定中也同樣適用,所以Hebert 在論文中明確提出了微型DNA 條形碼這一概念。微型DNA 條形碼技術是DNA 條形碼技術的應用拓展與補充延伸,通過此種技術可以更加準確、便捷的對蝶類昆蟲進行鑒定,為物種鑒定等相關研究工作提供有力支撐。對于有害物種的鑒定,傳統(tǒng)的方法存在一定的弊端。同時,在樣品標本的批量采集、運輸和保存等各個方面,也會導致標本DNA 發(fā)生降解,難以得到完整的DNA 條形碼信息。據(jù)此,本研究以黃粉蝶亞科為研究重點對象,主要探究并討論微型DNA 條形碼對黃粉蝶亞科物種在鑒定應用方面的實施性與可行性。
1 材料與方法
1.1 材料
虛擬實驗中的黃粉蝶亞科36 個樣本COI 片段序列均來自于BOLD 數(shù)據(jù)庫(Barcoding of life data),包括黃粉蝶亞科6 屬38 種36 個樣本,詳細序列編號見附錄1 的3—38;實體實驗中黃粉蝶亞科的2 個樣本材料來自山東德州岔河,分別為斑緣豆粉蝶Calias erate 和豆粉蝶Coilas hyale。
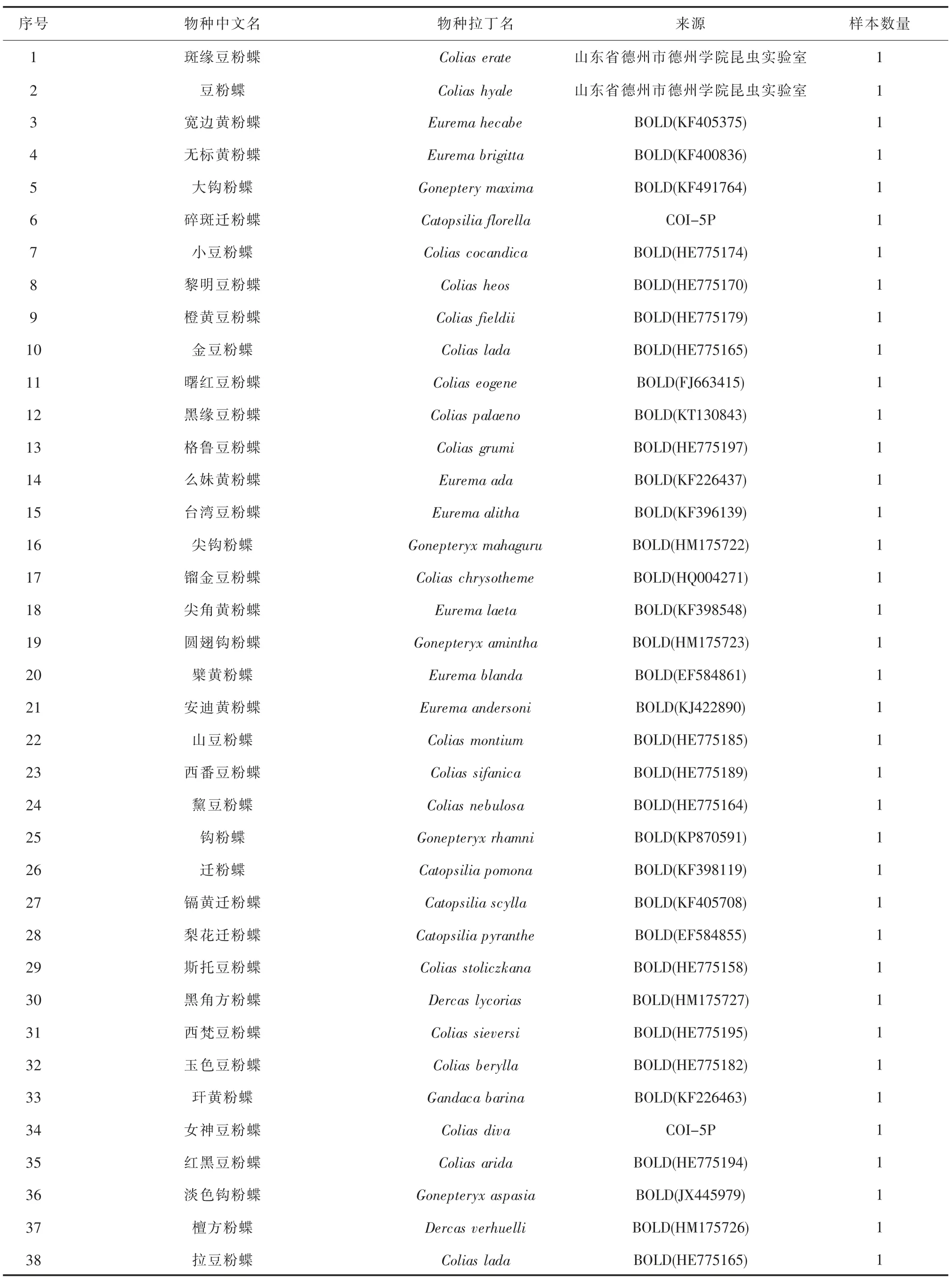
附錄1 物種樣本數(shù)量及來源Appendix 1 Origin and amounts of the samples used in this study
1.2 方法
1.2.1 虛擬實驗
將BOLD 數(shù)據(jù)庫中已經(jīng)下載的黃粉蝶亞科36 個樣本COI 基因局部序列用Clustal X[6]軟件進行比對,并將比對之后形成的數(shù)據(jù)集按照圖1表示的方式依次進行截取,將其截成長度為648bp、200bp、50bp 幾個合適的片段數(shù)據(jù)集,分別標記為full-barcodeA,full-barcodeB,full-barcodeC,然后用MAGE7.0 軟件對這3 個片段數(shù)據(jù)集依次構(gòu)建系統(tǒng)發(fā)育樹(Neighbor-Joining,Saitou&Nei,1987),參數(shù)為Kimura-2-parameter,再根據(jù)計算結(jié)果最終得出變異位點、簡約位點、種內(nèi)差異、種間差異這4 個數(shù)據(jù)。
1.2.2 實體實驗
總基因組的提取:從標本蝴蝶同側(cè)取下1—2 只足,提取總DNA。
①將取下的足剪碎后放入0.2 mL 離心管中依次加入15 μL 提取緩沖液GA(5 mmol/L )、10 μL(20 mg/mL)的蛋白酶K 溶液,渦旋混勻10 s 后放在56 ℃的溫度下孵育3 h,使樣本充分降解消化,等完成后再次進行簡短離心。
②加入25 μL 緩沖液GA 混勻,再加入50 μL 沖液GB 和1 μL Carner RNA 儲存液,渦旋混勻10 s 后敲擊至沉淀消失,然后進行簡短離心以去除管蓋內(nèi)壁的液滴。
③加入50 μL 預冷的無水乙醇,輕輕顛倒混勻后放置5 min 再簡短離心以去除管蓋內(nèi)壁的液滴。
④取上清液移至一個吸附柱CR2 中,以12 000 rpm 離心30 s,棄廢液。
⑤向吸附柱CR2 內(nèi)加入500 μL 的緩沖液GD,再以12 000 rpm 離心30 s,棄廢液。
⑥繼續(xù)向吸附柱CR2 內(nèi)加入600 μL 的漂洗液PW,以12 000 rpm 離心30 s 棄廢液,并重復一次。
⑦再將材料以12 000 rpm 離心2 min,倒掉廢液,將吸附柱CR2 放置3 min 左右。
⑧將吸附柱CR2 放入1 個干凈的離心管中,加入20~50 μL 的洗脫緩沖液TB,放置2~5 min 后以12 000 rpm 離心2 min。將溶液收集到離心管中。此時離心管中的溶液即為提取到的總DNA。
1.2.3 PCR 擴增
COI 基因擴增引物為 (LEPF1:5’-ATTCAACCAATCATAAAGATATTGG-3’; LEPR1:5’-TAAACTTCTGGATGTCCAAAAAATCA-3’),PCR 反應體系總共加入25 μL 溶液,其中各項比例為雙蒸水9.5 μL,mix 12.5 μL,引物LEPF11 μL,引物LEPR1 溶液1 μL,提取到的DNA1 μL,混合完成后放入離心機簡短離心至混勻。將PCR 擴增儀設定為:第1 步預變性94 ℃5 min;第2 步變性94℃1 min;第3 步退火55℃1 min;第4 步延伸
72℃2 min。重復以上的2 到4 步30 次,最后在72 ℃下延伸10 min。設定完成后將混合溶液放入PCR 擴增儀,大約擴增2 個小時22 分鐘。
1.2.4 制膠電泳
按照1:100 的比例進行配膠,經(jīng)0.1%低熔點的瓊脂糖凝膠電泳切膠分離后,找出所需長度的DNA 片段,用DNA 凝膠回收試劑盒(Tiangen 公司),作為測序的模板送至北京擎科新業(yè)生物技術有限公司測序。
1.2.5 測序結(jié)果
測序結(jié)果與虛擬實驗中36 個樣本序列結(jié)合,以DNA-barcode 指定序列5’端200 bp 為分析基礎,構(gòu)建系統(tǒng)發(fā)育樹,參數(shù)為Kimura-2-parameter。
2 結(jié)果與分析
2.1 虛擬實驗
虛擬實驗中3 個片段數(shù)據(jù)集full-barcodeA,full-barcodeB,full-barcodeC 的序列解析結(jié)果(見表1),即已算出變異位點、簡約位點、種內(nèi)差異、種間差異,且以這3 個片段數(shù)據(jù)集構(gòu)建的NJ 樹分別見圖1-A(648 bp 系統(tǒng)發(fā)育樹)、圖1-B(200 bp 系統(tǒng)發(fā)育樹)、圖1-C(50 bp 系統(tǒng)發(fā)育樹)。


圖1 基于36 個物種條形碼構(gòu)建系統(tǒng)發(fā)育樹Figure 1 The Phylogenetic tree based of 36 species
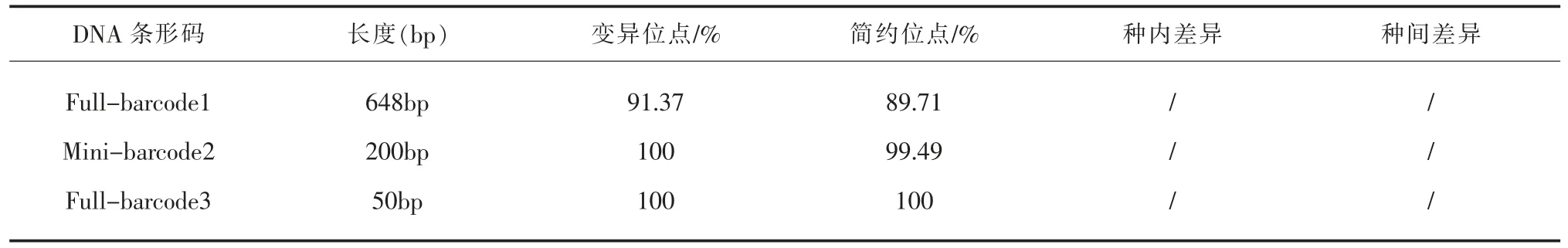
表1 全長DNA 條形碼與微型DNA 條形碼的對比Table 1 The compare of DNA barcode and Minimalist-barcode
2.2 實體實驗
將PCR 擴增及測序得到6 條648 bp 的序列,與虛擬實驗中得到的36 個樣本序列合并后得到38 條序列的數(shù)據(jù)集,由該數(shù)據(jù)集5’端200 bp 序列構(gòu)建的NJ 樹(見圖2)。
3 討論
在本實驗中,我們以寬帶紙弄蝶Hasora hurama、沖繩絨毛弄蝶Hasora chromus 為外群,構(gòu)建NJ 樹,并且從NJ 樹上分析。虛擬實驗full-barcodeA 的NJ 樹中(見圖1-A),臺灣豆粉蝶Eurema alitha、尖鉤粉蝶Gonepteryx mahaguru、黑角方粉蝶Dercas lycorias、檀方粉蝶Dercas verhuelli 位于一個分支,親緣關系較近;full-barcode B的NJ 樹中(見圖1-B),無標黃粉蝶Eurema brigitta、尖鉤粉蝶Gonepteryx mahaguru、黑角方粉蝶Dercas lycorias、檀方粉蝶Dercas verhuelli 位于一個分支,親緣關系較近;full-barcodeC 的NJ 樹中(見圖1-C),尖角黃粉蝶Eurema laeta、黑緣豆粉蝶Colias palaeno、女神豆粉蝶Colias diva、鎦金豆粉蝶Colias chrysotheme、玕黃粉蝶Gandaca barina 位于一個分支,親緣關系較近。實體實驗中,測序得到的兩種黃粉蝶亞科物種斑緣豆粉蝶Colias erate、豆粉蝶Colias hyale 序列5’端200bp 在NJ(見圖2)樹上得到完全區(qū)分。
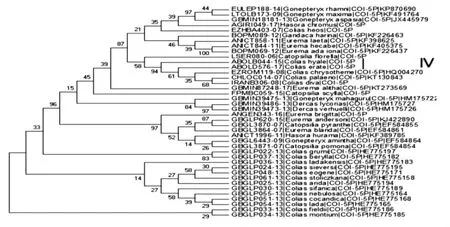
圖2 基于38 個物種條形碼構(gòu)建系統(tǒng)發(fā)育樹Figure 2 The Phylogenetic tree based of 38 species
在實驗中,當被區(qū)分的序列的長度依次是648bp、200bp、50bp 的時候,其變異位點在0.913 7 與1 之間,簡約位點的變動區(qū)間在0.897 1 到1 之間,種內(nèi)差異為0.010-0.017,種間差別為0.152-0.191。以上這些數(shù)據(jù)基本上都可以用來表示微型DNA barcode 與完整的barcode 在遺傳距離關系上起著相似的作用。所以微型DNA 條碼類似于完整的DNA 條碼,且根據(jù)分子進化生物學統(tǒng)計數(shù)據(jù)顯示物種的種內(nèi)差異在1%~2%之間,種間差異在10%~20%之間[7],虛擬實驗結(jié)果表明full-barcodeB 符合這個差異范圍,即微型DNA 條碼可以比較方便的去分辨物種。
但是由于目前微型DNA 條形碼使用較少,可靠性還需要大量數(shù)據(jù)進行檢驗,在動物分類學中有一定的可靠性,但是在其他物種中會存在很多問題,這些問題需要我們?nèi)ブ饌€發(fā)現(xiàn)并一一解決,從而使微型DNA 條形碼能夠作為一個可靠的標記基因進行物種鑒定。由于微型DNA 條碼序列過短易受其他因素的影響,會使最后的結(jié)果產(chǎn)生偏離,且在使用微型DNA 條形碼進行物種鑒定時還可能存在種間差異過大的問題,所以想要解決目前存在的問題,還需要科研人員進一步去分析解決。

