基于有限突變模型和大規模數據的19個常染色體STR的實際突變率研究
劉志勇,任賀,陳沖,張京晶,張曉夢,石妍,石林玉,陳瀅,程鳳,賈莉,陳曼,范慶煒,張家榕,李萬婷,王萌春,任子林,劉雅誠,倪銘,孫宏鈺,嚴江偉
研究報告
基于有限突變模型和大規模數據的19個常染色體STR的實際突變率研究
劉志勇2,任賀3,陳沖4,張京晶5,張曉夢1,石妍4,石林玉1,陳瀅4,程鳳1,賈莉4,陳曼6,范慶煒7,張家榕1,李萬婷1,王萌春1,任子林8,劉雅誠4,倪銘8,孫宏鈺2,嚴江偉1
1.山西醫科大學法醫學院,太原 030001 2.中山大學中山醫學院法醫學系,廣州 510080 3.北京警察學院,北京 102202 4.北京通達首誠司法鑒定所,北京 100192 5.北京華彥科技有限公司司法鑒定所,北京 100192 6.南方醫科大學法醫學院,廣州 510515 7.川北醫學院法醫學系,南充 637000 8.北京輻射醫學研究所,北京 100850
短串聯重復序列(short tandem repeat, STR)已廣泛用于法醫學親子鑒定和個體識別中,但STR的突變可能會影響其結果的解釋。在大多數類似研究中,由于忽略“隱性”突變現象,STR的突變率被低估。鑒于此,為獲得更加準確的STR實際突變率,本研究使用Slooten與Ricciardi提出的有限突變模型和大規模數據,對28,313例(78,739個體)中國北京漢族已確認親生關系的親子鑒定案的20個常染色體STR基因座(和;由于有限突變模型中未包含的矯正參數,因此本文實際計算其余19個STR基因座的突變率)進行了調查。結果發現,所有基因座均存在突變現象,總計發生1665個突變事件,包括1614個一步突變,34個兩步突變,8個三步突變和9個非整步突變。基因座特異性的平均實際突變率在三聯體中為0.00007700 ()~0.00459050 (),在二聯體中為0.00000000 ()~0.00344850()。此外,本研究還分析了表面和實際突變率、三聯體和二聯體突變率、父源和母源的突變率之間的關系。研究表明,實際突變率多大于表面突變率,而且1*/2* (表面突變率)的比值通常也大于1/2 (實際突變率) (1*,1;2*,2分別是一步和兩步的突變率),即更多的“隱性”突變被釋放出來。而且父源和母源的三聯體和二聯體的突變率也有存在差異。隨后,將這些突變率數據與已發表的中國其他漢族人口的相關研究進行比較,展現出了STR突變率的時間與區域差異。由于樣本量大,本研究中還報告了一些少見的突變事件,例如同卵雙胞胎突變和“假四步突變”等。綜上所述,本研究通過大量數據獲得了接近真實的STR突變率的估計值,不僅可為中國法醫DNA數據庫和群體遺傳學數據庫提供重要的基礎數據,也對開展法醫學個體識別、親權鑒定和遺傳學研究具有重要的意義。
常染色體STR;有限突變模型;親子鑒定;中國漢族人群;突變分析
STR (short tandem repeat)是人類基因組中非常豐富的一類遺傳標記[1],由于其高度的多態性[2,3],被廣泛應用于個體識別、親子鑒定和群體遺傳學等領域。然而,在大多數STR基因座中都可以觀察到突變事件,一般認為復制滑脫是導致STR突變的主要機制[4]。這些突變現象往往會對法醫學實踐中證據權重評價產生影響,比如在計算累積父權指數(cumula-tive paternity index, CPI)時需要特別考慮突變現象,過高過低的突變率數據都不利于得出客觀結論,因此獲得STR基因座的準確突變率是非常重要的。
STR基因座突變率的實際評估計算中,往往會受抽樣群體大小的影響[5]。盡管已有研究者發表大量關于STR基因座突變率的論文,但基于較少的樣本量,其STR突變率的計算結果可能存在偏差。除了以上因素外,突變率的不同計算方法也會對STR實際突變率計算產生影響。在已報道的多數研究中STR突變率一般是通過直接計數法來計算的5~8],并且將三聯體和二聯體的數據進行了合并。然而,該方法沒有考慮“隱性突變(hidden mutation) ”現象,因為人們普遍認為當發生突變時,突變步數越少越真實,但實際并非總是如此,即某些突變會被隱藏。鑒于通過三聯體和二聯體的計算得到的STR基因座突變率有一定差異,因此合并三聯體和二聯體的突變數據可能是不合理的。即,直接計數法對于STR的實際突變率的估計有一定偏差。
針對以上抽樣群體尺度與計算方法的問題,本研究采用了大量親子鑒定數據和Slooten與Ricciardi[9]的更為嚴謹的有限突變模型計算方法。基于28,313例已確認親生關系的中國漢族人群親子鑒定案例,分別計算了三聯體和二聯體兩種情形的19個常染色體STR基因座實際突變率。本文詳細比較了表面突變率(直接計數法)和實際突變率(有限突變模型),并與其他多個群體的STR突變率進行了對比分析。最后,本文還討論了影響STR突變率的幾個因素,比如突變年齡與來源、等位基因長度、基因座雜合度等。
1 材料與方法
1.1 數據來源
本研究涉及的基因座突變數據來自2009~2019年間中國北京漢族人群的常規親子鑒定案例的統計結果,該數據已獲得北京通達首誠司法鑒定所許可使用。所有數據均采用匿名化處理,不涉及個體相關除突變信息以外的其他STR基因座具體分型,也不涉及家系情況。本次統計分析的親子鑒定案例的CPI值均大于10,000,達到親子鑒定的確認標準(未能確認的案例被除外),且突變的STR基因座已經過多個試劑盒驗證。總計28,313例(78,739個體)親子鑒定數據被納入,包括22,113例三聯體(trio’s)和6200例二聯體(duo’s) (父親–孩子:2342例;母親–孩子:3858例)。該數據涉及的STR基因座包括CODIS 系統()和。由于有限突變模型[9]中未包含基因座的計算矯正參數,因此本研究中共包括除之外其余19個STR基因座的突變率計算。
1.2 突變率計算
在已確認親生關系的親子鑒定案例中,當發現STR等位基因違反孟德爾遺傳定律時,認為該STR基因座發生突變事件[10]。在計算過程中,突變步數與來源的判定根據Brinkmann等[10]和Weber等[11]的描述,本研究中所有可疑的STR突變事件均再次通過同一試劑盒和其他試劑盒進行檢測,確保分型正確。所有STR基因座的三聯體和二聯體父源母源突變事件的統計結果見附表1。在突變率計算過程中,一方面使用直接計數法公式*=n/N計算表面突變率,其中n是觀察到的突變事件的數目,N為減數分裂的發生次數;另一方面在得到表面突變率后,通過概率矯正因子矩陣A矯正得到實際突變率(見方程(I))。其中A_{k, l}代表觀測到k步突變,實際是l步突變的概率。對于任一A_{k, l},Slooten和Ricciardi[9]基于NFI的參考數據集(2085個體)的等位基因型頻率,隨機組合一對夫婦,模擬生成子代的基因型,其中從母親遺傳一個沒有突變的等位基因,但從父親遺傳得到一個實際有l步突變的等位基因,即已知實際突變步數,然后通過觀察得到表面突變步數。上述過程通過計算機模擬重復100,000次,計算不同突變步數的頻率,進而估計最終突變轉移概率A_{k, l}。該模型假設:(1)當發生不確定來源的突變時,認為父源與母源發生的概率相同,均為0.5;(2)當觀察到的突變事件可以同時解釋為相同的突變增加或者突變減少時,該模型方法不予區分。本文采用模型中下列矩陣方程(I)計算實際突變率。

在“方程(I) ”中,k*為表面為k步的突變率,0*為表面不發生突變的概率;k代表實際為k步的突變率,0為實際不發生突變的概率;Ak,l是表面突變步數為k,而實際步數為l的概率;*為基因座特異性表面突變率;為基因座特異性實際突變率。為了便于理解該模型,方程(Ⅱ)列出了方程(I)的展開式。以TH01為例,統計計算得到了表面突變率k*后,再根據Slooten和Ricciardi[9]提供的矯正矩陣A_{k, l},可以結合方程(Ⅱ)反解出實際突變率k,其具體的計算過程見附表2。本文采用在線計算器(https://www.numberempire.com/matrixbinarycalculator. php)進行矩陣求逆。
方程(Ⅱ)
2 結果與分析
2.1 突變分析
本項研究中,在28,313例確認的親子鑒定案例(22,113例三聯體和6200例二聯體)的20個常染色體STR基因座中共發現了1665個突變事件,所有基因座均觀察到突變現象。基于有限突變模型計算得到的三聯體和二聯體的基因座特異性實際突變率和總突變率數據顯示(表1,不包含基因座):三聯體基因座特異性平均實際突變率范圍為0.00007700 ()~0.00459050 (),二聯體為0.00000000 ()~0.00344850 ();基因座具有較高的父源突變率(0.00767100)和總突變率(0.00459050),具有較高的母源突變率(0.00237900),與Jin等[12]基于直接計數法的研究結果一致。
同卵雙胞胎(monozygotic, MZ)是由單個精子和單個卵子結合形成受精卵后,分裂成兩個胚胎形成的,其發生率約為1/250[13]。從理論上講,同卵雙胞胎擁有相同的基因組,然而研究者在STR[14]、拷貝數變異(CNV)[15]和單核苷酸多態性(SNP)[16]中觀察到了一些差異。本次研究中,在10對MZ中發現了相同突變,包括7例男性和3例女性(附表3),突變基因座為(2例)、(2例)、(2例)、、、和,且均為一步突變。在已公開發表的論文中,尚未報道同時在MZ中發現不一致的STR突變,但Wang等[17]報道了一對MZ中其中一個個體的基因座出現三等位基因模式的案例。在親子鑒定實踐中,一般較少會在同一個體觀察到多個突變。如果觀察到,其可能會干擾鑒定人得出親子關系的正確結論。在本次研究的28,313例親子鑒定案件中,共發現27例孩子的基因型中可觀察到2個突變的事件,發生率為0.00095363 (27/28,313,20個常染色體STR基因座) (附表4A);同時也發現3例孩子的基因型中可觀察到3個突變的事件(附表4B)。在這30例的多基因座突變案例中,本研究使用了總計超過39個基因座的多個STR試劑盒對親子關系進行了確認。
2.2 表面與實際突變率的比較
基于直接計數法和有限突變模型,計算得到每個STR基因座的表面突變率(*)與實際突變率(),三聯體的相關突變率數據見附表5,二聯體相關數據見附表6。在使用有限突變模型的矩陣計算(方程(I))中,本研究發現有三個負值的實際突變率,即三聯體組的和基因座的M2;二聯體組的基因座的F1(M,F分別代表母源和父源突變)。為了查找原因,本研究分析了和基因座在三聯體中的突變特征,發現母源突變中可觀察到一步和三步突變,但未觀察到兩步突變;同理,二聯體組的基因座僅在父源中觀察到兩步突變,但未觀察到一步突變。即表明,實際突變率負值可能是在某STR基因座可觀察到高步數突變,但未觀察到某個低步數突變的情況下發生的(即非連續的突變步數現象)。換言之,即這幾個負值可能在提示潛在低步數突變應該會被觀察到,但實際中并沒有觀察到,這已經超過了有限突變模型的可解釋范疇。因此在數據處理過程中,把三個負值實際突變率設為零。如圖1所示,在父源和母源突變之間有一個明顯的界限,特別是在三聯體的一側(圖1A),這可能提示三聯體與二聯體的計算得到的STR突變率是有差異的,盡管這種差異可能來自于三聯體與二聯體本身,或者來自于樣本量的差異,但可間接說明以往直接合并三聯體與二聯體數據進行計算存在不合理之處。此外,從該圖也可發現在三聯體和二聯體中,和基因座的表面和實際突變率都較低。與之相反的是,和基因座在三聯體中的表面和實際突變率均較高,而和基因座在二聯體中的表面和實際突變率較高。為了進一步分析有限突變模型的校正效果,繪制了實際突變率與表面突變率比值圖(/*) (圖2)。該圖表明,除父源的和(/*≈1)和母源的基因座(/*≈1)外,其余基因座的突變率均得到了較大程度的矯正(/*>1)。就矯正程度而言,二聯體組的基因座的效應表現得更為明顯(與Slooten和Ricciardi[9]的研究結果相似),尤其是和基因座。也就是說有限突變模型通過參數矯正的方式,將更多的隱性的突變挖掘出來。然而,在親子鑒定過程中,需要計算有突變率參與的父權指數時,目前一般采用直接計數法計算得到平均STR突變率(比如男性取0.002;女性取0.001~0.0005)來進行。若為了更嚴謹的計算,可采用三聯體和二聯體的父源、母源或者STR基因座特異性突變率,當然也可以使用本次研究基于有限突變模型計算獲得的STR基因座特異性實際突變率。從理論上講,計算單個基因座父權指數時,采用上述兩種算法得到的突變率,結果是會存在差異,但對總體的鑒定結論的影響有限。

表1 三聯體與二聯體STR基因座特異性實際突變率
“”表示實際突變率;“F”表示父源;“M”表示母源。

圖1 三聯體和二聯體表面和實際突變率熱圖
A:三聯體情形;B:二聯體情形;“*”表示表面突變率;“”表示實際突變率。
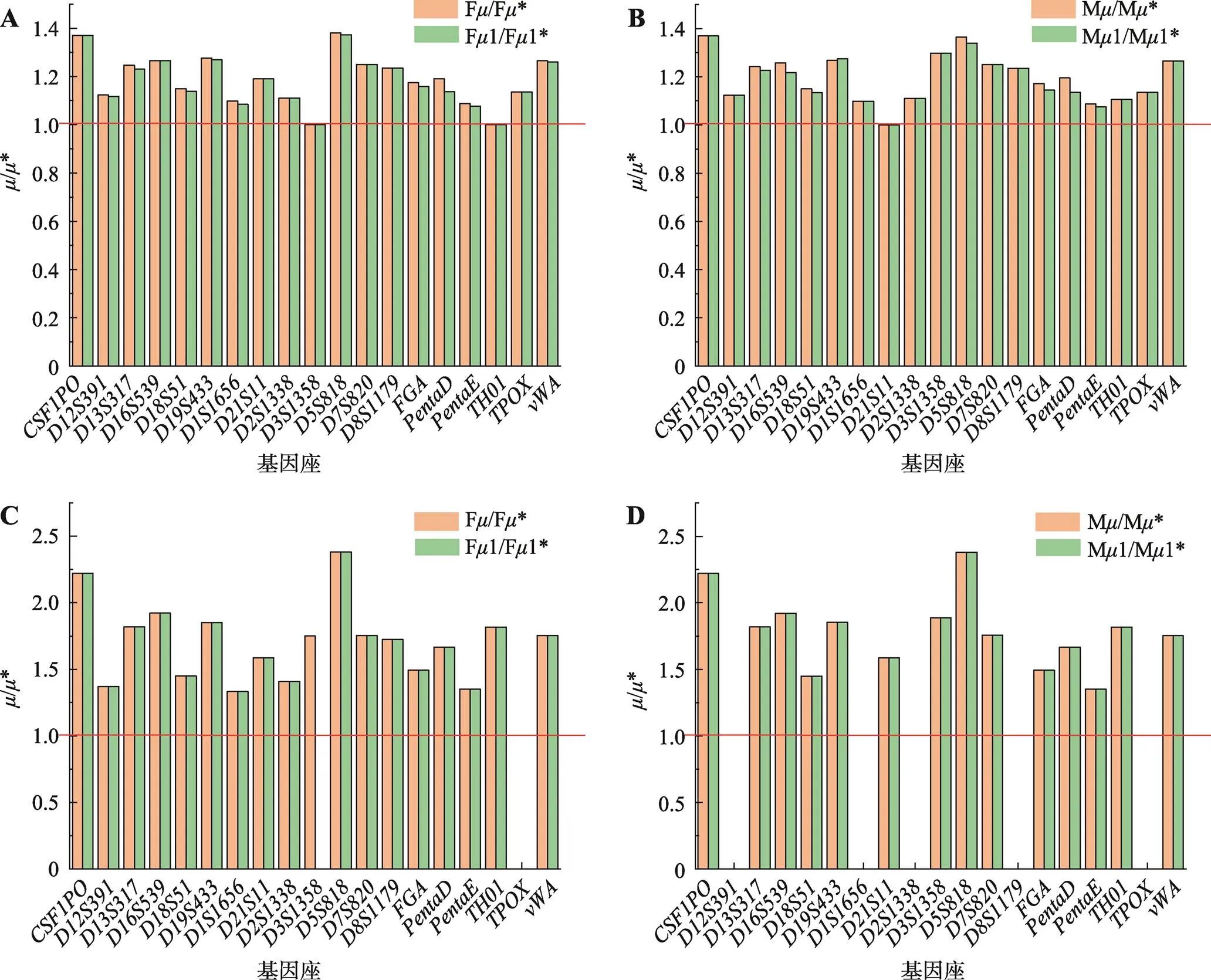
圖2 表面突變率與實際突變率比較
“A”和“B”代表三聯體組父源(F)和母源(M)的/*比值;“C”和“D”分別代表二聯體組父源(F)和母源(M)的/*比值;空白處表示比式的分母為零的情況。
2.3 三聯體和二聯體組突變率的比較
在以往基于直接計數法研究中,一般將三聯體和二聯體組的數據進行合并計算突變率。然而,三聯體和二聯體得到的突變率水平是有一定差異,其在2.1和2.2中已進行部分闡述。在大多數STR基因座中,三聯體父源的實際突變率值大于二聯體的父源相關值(圖3,綠色>黑色);三聯體母源的實際突變率值大于二聯體的母源相關值(圖3,粉色>藍色);它們的比值表現出相似的規律(圖3),特別是在基因座。與此同時,本研究也發現存在相反趨勢的基因座,如三聯體組的、和二聯體組的等,因此將三聯體和二聯體的數據合并是不合理的,它們之間的差異是存在且不宜忽略的。
2.4 父源和母源突變率的比較
在1665個突變事件中,共觀察到1178例父源突變和293例母源突變事件,同時有194例突變來源不明(父源或母源),其中父源突變占多數,為70.75% (圖4A)。在三聯體組中,總F為0.06405550,M為0.01937639 (表1);二聯體中,總F為0.05146300,M為0.01220500 (表1)。從以上數據可以得知,父源突變率一般較母源突變率高,在三聯體和二聯體組中分別約為3.3倍和4.2倍,其與以往的研究結果一致[11,12]。Jónsson等[18]對冰島1548個三聯體進行的新生突變(de novo mutations, DNMs)研究也表明父源的突變數量約是母源的4倍,這也側面反映了本次研究的結果。本研究還比較了不同突變來源的基因座特異性突變率(圖5)。如圖5A顯示,三聯體中絕大多數基因座不同突變來源計算得到的突變率具有一定差異性,除和基因座外,父源突變率是母源突變率2倍以上。而二聯體組也顯示了相似的趨勢(圖5B)。這可能是男性與女性形成配子時經歷的細胞分裂次數不同,而男性需要產生更多的配子的緣故[10]。可以預知,使用突變來源特異性基因座突變率將有助于親子關系等法醫學鑒定得出的更加客觀而準確結論。另外,為了說明不同突變來源的年齡范圍,本研究制作了父源(圖4B)和母源(圖4C)突變年齡的扇形圖,從中可以看到父源突變的年齡范圍比母源突變的年齡范圍更廣,這可能是由于男性在較長時間內可以保持一定的生殖能力,而女性不可以的原因。

圖3 三聯體和二聯體中Fμ、Mμ及其比值的比較
“trio’s”表示三聯體組;“duo’s”表示二聯體組;空白處表示比式的分母為零的情況。

圖4 本次研究中突變事件來源、年齡和步數的比較
A:突變來源分布;B:父源突變年齡分布;C:母源突變年齡分布;D:突變步數分布。
2.5 突變步長與突變方向分析
在所有的突變事件中,共觀察到1614個一步突變、34個兩步突變、8個三步突變和9個非整步突變。一步突變占總突變事件的96.94%,是主要的突變類型,逐步突變模型(stepwise mutation model, SMM)可以解釋一步突變為主的現象[19,20]。在其他非一步突變中,兩步突變占2.04%,三步突變占0.48%,而非整步突變占0.54% (圖4D)。發生非整步突變的基因座分別為:(3例)、(2例)、、、和;突變來源為父親的6例,母親的1例,未能確定突變來源的2例;非整步的突變方向,其中7例突變減少,1例突變增加,另有1例不能確定突變方向;關于非整步突變的步數,0.1、0.2、0.3步均有觀察到,其中有5例含有0.2步突變,2例有0.3步突變,1例有0.1步突變,另有1例為0.3或者0.1步突變。就本研究觀察到的9例非整步突變而言,突變減少和包含0.2步突變是占多數的,詳細信息展現在附表7中。為了進一步分析有限突變模型對于STR基因座等位基因不同突變步數的突變率影響,本研究計算了三聯體中發生一步突變與兩步突變的比值(圖6)。在父源(圖6A)中,1*/2*的值普遍大于1/2,特別是在基因座(本次研究:1*/2*≈104;1/2≈73),這表明由于有限突變計算模型的使用,1/2之間的差異正在減小。這些規律與Slooten與Ricciardi[9]的研究相似(1*/2*≈75;1/2≈58),也就是說更多的高步數隱性突變被挖掘出來。在母源(圖6B)基本上也顯示了這一規律,但發現了具有相反趨勢的基因座,如、和,根據該模型作者的解釋[9],這可能是由于模型本身不完美的原因造成的。
盡管有限突變計算模型中不考慮突變方向(突變減少/增加),但本研究仍然統計了等位基因減少和增加的分布情況。在1665次突變事件中(附表8),總計有706次突變減少(mutation loss),703次突變增加(mutation gain),256次為不確定突變方向或者分組;突變減少與增加(loss/gain)之比約為1∶1,與Lan等[21]的研究相似。Brinkmann等[10]報道認為突變的發生與等位基因的長度存在一定關聯,長的等位基因比短的等位基因更易于發生突變;Shao等[5]認為較長的等位基因傾向于發生突變減少事件,而短的等位基因則更傾向于發生突變增加事件。為了探究等位基因長度與突變方向之間的關系,參照Ge等[22]和Xu等[23]的方法,將每個STR基因座的等位基因根據長度分為三組,即較短(short group)、中等(medium group)、較長(long group)等位基因組,詳細的統計數據見附表8。從表中可計算得知在以上三個分組中,總的突變次數分別為105、1042、397,其中中等等位基因長度組發生的突變事件最多(約為67.49%),較長等位基因長度組次之(約為25.71%),而較短等位基因長度組最少(約為6.80%),也即中等及較長等位基因更易于發生突變,較短的等位基因發生突變較少,這與Xu等[23]的研究結論一致;較短等位基因組突變減少與增加(loss/gain)的比值約為0.49 (<1),中等組為0.86,較長組為1.72 (>1),本研究結果也表現出等位基因“短變長,長變短”的規律,這也得到Shao等[5]、Ge等[22]、Xu等[23]、Dupuy等[24]的支持,其可能與STR的序列結構有關。
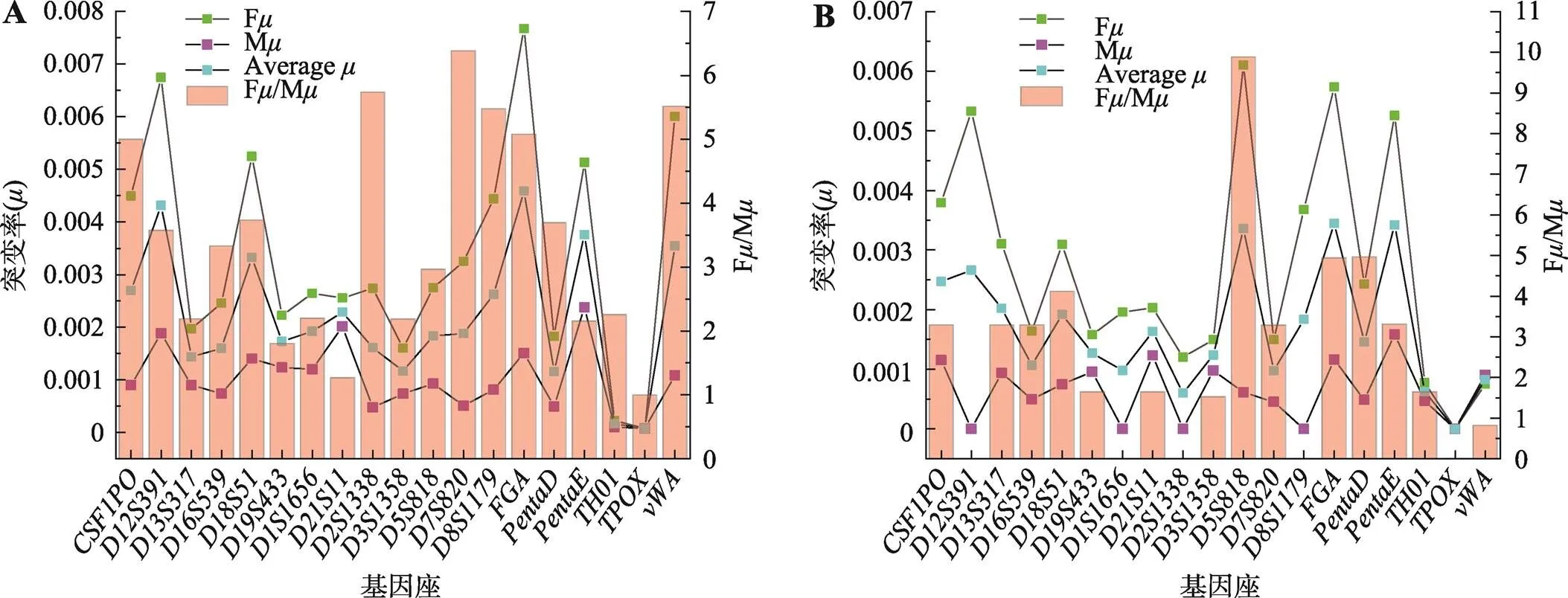
圖5 不同突變來源的基因座特異性突變率比較
A:三聯體情形;B:二聯體情形;空白處表示比式的分母為零的情況。

圖6 三聯體父源與母源的突變步長分析
A:父源情形;B:母源情形;空白處表示比式的分母為零的情況。
2.6 雜合度與實際突變率的關系
一般認為,雜合度高的基因座其突變率也較高,也即突變率高可能是基因座雜合度高的一個原因。據此根據已有文獻報道的雜合度數據[25],分析了每個STR基因座的雜合度和實際突變率的關系,使用Origin軟件繪制線性擬合圖(圖7),Pearson?s= 0.68454。結果也相似地表明,雜合度較低的STR基因座突變率普遍較低,如、基因座,這與前人的研究相一致[12]。
3 討論
不同的突變率計算方法結果可能存在潛在的偏差,比如傳統的直接計數法在處理突變數據時未考慮到“隱性突變”現象[26,27]。此處以兩個確認的三聯體親子鑒定為例進行說明(下文“a”為正整數)。案例一:某STR基因座母親的分型是M=(a, a+1),孩子是C=(a, a+1),父親是F=(a–1, a+1),假設真實的遺傳情況為孩子C的“a+1”來自母親M的“a+1”,“a”來自父親F的“a–1”或者“a+1”一步突變后的“a”,即實際發生一步突變。從表面上看,可能會認為孩子C的“a+1”遺傳自父親F的“a+1”,而“a”遺傳自母親M的“a”,此時表面上會認為沒有突變發生,即“突變發生有無認識偏差”型隱性突變(Type Ⅰ型);案例二:某STR基因座母親的分型是M=(a+1, a+1),孩子是C=(a, a–1),父親是F=(a, a–1),假設真實遺傳情況為孩子C的“a–1”來自母親M的“a+1”兩步突變后的“a–1”,“a”來自父親F的“a”,即實際發生兩步突變。從表面上看,可能會認為孩子C的“a–1”遺傳自父親F的“a–1”,“a”遺傳自母親M的“a+1”一步突變后的“a”,此時表面上會認為發生一步突變,因為當不知道突變真相時,一般判斷為較短的突變步數,即“真實突變步數認識偏差”型隱性突變(Type Ⅱ型)。類似地,隱性突變也可以出現在二聯體中。在上述案例中,表面觀察到的突變被定義為表面突變(apparent mutation, ApM),而不可見的真實突變被定義為實際突變(actual mutation, AcM)。因此,如果不考慮隱性突變(比如直接計數法),突變率可能就會被低估。盡管研究者無法直接從表面的STR分型突變現象中洞察真實的突變,但Slooten和Ricciardi[9]通過突變模擬的方式研究了該隱性突變現象,并提供了可接近真實突變率的計算模型和矯正參數。該模型根據突變的等位基因是否超過STR等位基因分型標準品(ladder)的范圍L,可分為有限突變模型(restricted mutation model)和無限突變模型(unrestricted mutation model),而真實的生物學過程可能介于這兩個模型之間。當不知道突變的等位基因是否超過Ladder時,無限突變模型可能是合適的。然而,基于大量數據研究調查發現[9],突變的等位基因不包括在Ladder中是一種非常少見的現象,本次研究的數據也證明這一點。綜合上述,鑒于使用簡單的有限突變模型是具有一定合理性的,本研究擬采用該模型計算實際STR突變率。
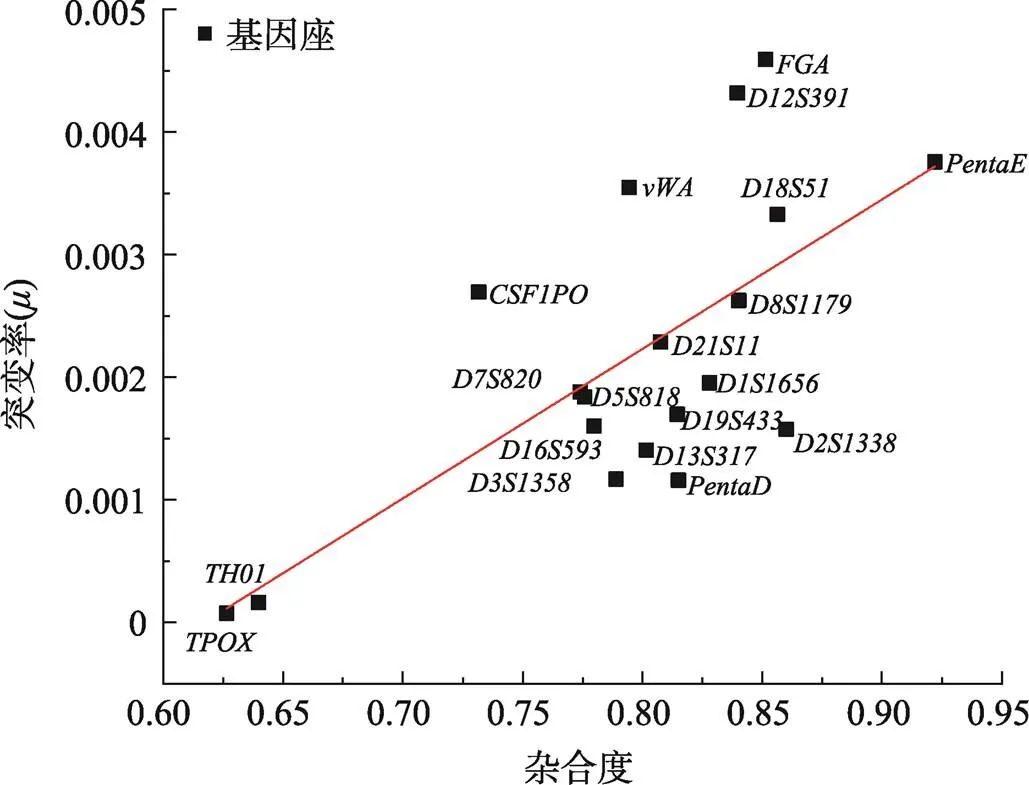
圖7 19個常染色體STR基因座雜合度與實際突變率的關系
基于以上有限突變模型計算獲得的STR基因座實際突變率,本研究一方面為了觀察其是否有時間差異,收集了Yan等[28]于2006年報道的與本研究具有相同來源的北京漢族突變率數據,這組數據調查了6532例親子鑒定案例中的19,754個樣本的17個常用STR基因座特異性突變率, 這些基因座均包含在本次研究中;另一方面為了觀察本研究的北京漢族與中國其他漢族人群突變率的地域差異,收集了中國其他地區或省(華北、華東、華南地區和河南、上海、貴州、河北、云南、福建、廣東等省)漢族群體的突變率數據(附表9)。如各基因座突變率比較的散點圖所示(圖8),在時間變化方面:共享的17個STR基因座中,除和外,其余13個基因座的突變率在14年間中增幅較大(紅色方框與黑色圓點),尤其是和的突變率增加將近一倍;在地域變化方面,不同漢族群體間的STR基因座特異性突變率具有一定差異,特別是在和基因座,這些差異可能來自于多個地域來源樣本的遺傳結構不同[29,30],或是使用群體樣本量不同,或使用的計算模型方法不同。
親子鑒定實踐中,有時可能遇到假突變現象(fake mutation),其應引起足夠的重視。例如,本次研究中在一例確認的三聯體親子鑒定中基因座發現其基因型不符合遺傳規律的現象,稱之為“假四步突變(fake four-steps mutation) ”:假設父AF=8,母親M=12和孩子C=8 (圖9)。當使用四個STR擴增試劑盒(PowerPlex?21 (Promega,美國)、MicroreaderTM21 (閱微基因,北京)、MicroreaderTM23sp-B (閱微基因,北京)、AGCU 17+1 system (中德美聯,無錫))確認基因型后,得到相同的結果。起初,懷疑母親和孩子之間可能發生了四步突變,即母親的等位基因“12”突變為“8”。為了進一步證實這個猜想,首先使用ForenSeq DNA Signature Prep Kit (Verogen, 美國)在MiSeq FGx儀器(Illumina,美國)中進行測序,并使用ForenSeq通用分析軟件(Verogen,美國)分析原始數據。測序結果顯示(圖9),母親的等位基因“8”發生了丟失,其序列與孩子的等位基因“8”是一致的。為了繼續尋找母親等位基因丟失的原因,使用primer5軟件設計了基因座的引物(F:TGGAAGCTGCATAGAC;R:ACTCCATACAAATGCAAGAGA),并進行了克隆測序(圖9)。如紅色方框所示(圖9),母親的等位基因“8”的核心區上游第17 bp處發生單堿基突變,由正常的“C”突變為“T”。以上研究表明,基因座引物結合區的突變可能是多個試劑盒未能成功擴增出母親等位基因“8”的原因。實際中,真正的四步突變是非常少見的,當遇到可疑的四步突變時,測序有時是很必要的。

圖8 中國10個不同地區或省漢族人群突變率比較
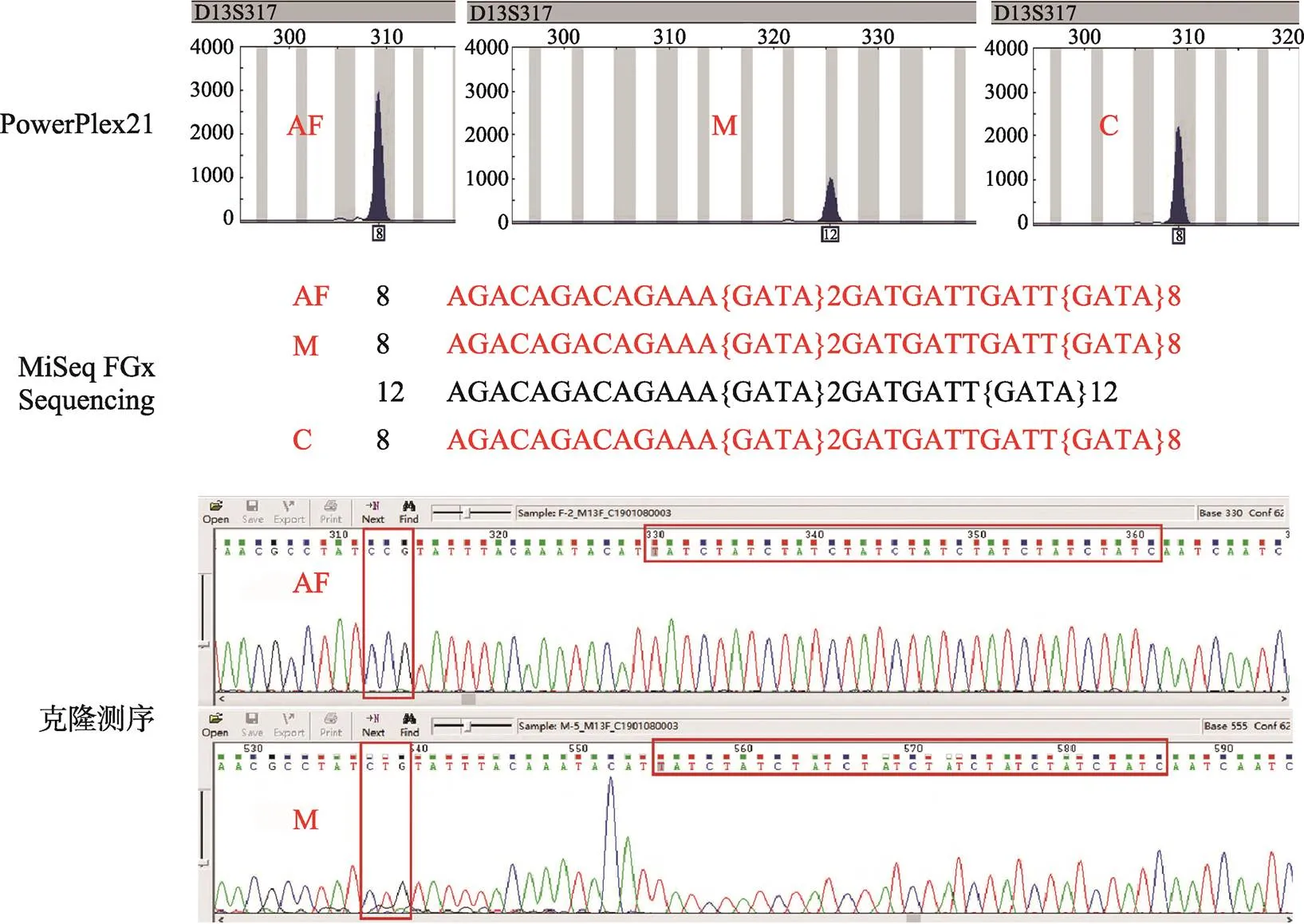
圖9 基于三種方法對D13S317基因座的測序結果
本研究在28,313例確認的親子鑒定案例中,共發現1665個突變事件。基于以上突變數據和有限突變模型,分別計算了三聯體和二聯體案例中的父源與母源的19個常染色體STR基因座的特異性實際突變率。與直接計數法計算突變率相比,本次的計算結果揭示了更多的隱性突變現象,因此獲得的突變率數值理論上更加接近真實情況。本研究不僅可為中國法醫DNA數據庫和群體遺傳學數據庫提供重要的基礎數據,也對開展法醫學個體識別、親權鑒定和遺傳學研究具有重要的意義。
附加材料詳見文章電子版www.chinagene.cn。
[1] Subramanian S, Mishra RK, Singh L. Genome-wide analysis of microsatellite repeats in humans: their abundance and density in specific genomic regions., 2003, 4(2): R13.
[2] B?r W, Brinkmann B, Budowle B, Carracedo A, Gill P, Lincoln P, Mayr W, Olaisen B. DNA recommendations. Further report of the DNA Commission of the ISFH regarding the use of short tandem repeat systems. International Society for Forensic Haemogenetics,, 1997, 110(4): 175–176.
[3] Butler JM. Short tandem repeat typing technologies used in human identity testing., 2007, 43(4): 2–5.
[4] Kornberg A, Bertsch LL, Jackson JF, Khorana HG. Enzymatic synthesis of deoxyribonucleic acid, XVI. oligonucleotides as templates and the mechanism of their replication., 1964, 51(2): 315–323.
[5] Shao CC, Lin MX, Zhou ZH, Zhou YQ, Shen YW, Xue AM, Zhou HG, Tang QQ, Xie JH. Mutation analysis of 19 autosomal short tandem repeats in Chinese Han population from Shanghai., 2016, 130(6): 1439–1444.
[6] Li HX, Peng D, Wang Y, Wu RG, Zhang YM, Li R, Sun HY. Evaluation of genetic parameters of 23 autosomal STR loci in a southern Chinese Han population., 2018, 45(4): 359–364.
[7] Wang HD, Kand B, Su N, He M, Zhang B, Guo YX, Zhu BF, Liao SX, Zeng ZS. Evaluation of the genetic parameters and mutation analysis of 22 STR loci in the central Chinese Han population,, 2017, 131(1): 103–105.
[8] Qu N, Zhang XC, Liang H, Ou XL. Analysis of genetic polymorphisms and mutations at 23 autosomal STR loci in Guangdong Han population., 2019, 38: e16–e17.
[9] Slooten K, Ricciardi F. Estimation of mutation probabilities for autosomal STR markers., 2013, 7(3): 337–344.
[10] Brinkmann B, Klintschar M, Neuhuber F, Hühne J, Rolf B. Mutation rate in human microsatellites: influence of the structure and length of the tandem repeat., 1998, 62(6): 1408–1415.
[11] Weber JL, Wong C. Mutation of human short tandem repeats., 1993, 2(8): 1123–1128.
[12] Jin B, Su Q, Luo HB, Li YB, Wu J, Yan J, Hou YP, Liang WB, Zhang L. Mutational analysis of 33 autosomal short tandem repeat (STR) loci in southwest Chinese Han population based on trio parentage testing., 2016, 23: 86–90.
[13] Taylor MJ, Fisk NM. Prenatal diagnosis in multiple pregnancy., 2000, 14(4): 663–675.
[14] Wurmb-Schwark NV, Schwark T, Christiansen L, Lorenz D, Oehmichen M. The use of different multiplex PCRs for twin zygosity determination and its application in forensic trace analysis., 2004, 6(2): 125–130.
[15] Xu YJ, Li TT, Pu T, Cao RX, Long F, Chen S, Sun K, Xu R. Copy number variants and exome sequencing analysis in six pairs of Chinese monozygotic twins discordant for congenital heart disease., 2017, 20(6): 521–532.
[16] Weber Lehmann J, Schilling E, Gradl G, Richter DC, Wiehler J, Rolf B. Finding the needle in the haystack: differentiating “identical” twins in paternity testing and forensics by ultra-deep next generation sequencing., 2014, 9: 42–46.
[17] Wang LF, Yang Y, Zhang XN, Quan XL, Wu YM. Tri-allelic pattern of short tandem repeats identifies the murderer among identical twins and suggests an embryonic mutational origin., 2015, 16: 239–245.
[18] Jónsson H, Sulem P, Kehr B, Kristmundsdottir S, Zink F, Hjartarson E, Hardarson MT, Hjorleifsson KE, Eggertsson HP, Gudjonsson SA, Ward LD, Arnadottir GA, Helgason EA, Helgason H, Gylfason A, Jonasdottir A, Jonasdottir A, Rafnar T, Frigge M, Stacey SN, Magnusson OT, Thorsteinsdottir U, Masson G, Kong A, Halldorsson BV, Helgason A, Gudbjartsson DF, Stefansson K. Parental influence on human germline de novo mutations in 1,548 trios from Iceland., 2017, 549(7673): 519–522.
[19] Valdes AM, Slatkin M, Freimer NB. Allele frequencies at microsatellite loci: the stepwise mutation model revisited., 1993, 133(3): 737–749.
[20] Klintschar M, Dauber EM, Ricci U, Cerri N, Immel UD, Kleiber M, Mayr WR. Haplotype studies support slippage as the mechanism of germline mutations in short tandem repeats., 2010, 25(20): 3344–3348.
[21] Lan Q, Wang HD, Shen CM, Guo YX, Yin CY, Xie T, Fang YT, Zhou YS, Zhu BF. Mutability analysis towards 21 STR loci included in the AGCU 21?+?1 kit in Chinese Han population., 2018, 132(5): 1287–1291.
[22] Ge JY, Budowle B, Aranda XG, Planz JV, Eisenberg AJ, Chakraborty R. Mutation rates at Y chromosome short tandem repeats in Texas populations,, 2009,3 (3): 179–184.
[23] Xu W, Wang YQ, Zhang DD, Wang DX, Zhou L, Ye XL, Zhu CG, Shi YZ. Mutation analysis of 21 autosomal short tandem repeats in Han population from Hunan, China.. 2019, 46(3): 254–260.
[24] Dupuy BM, Stenersen M, Egeland T, Olaisen B. Y-chromosomal microsatellite mutation rates: differences in mutation rate between and within loci,, 2004, 23(2): 117–124.
[25] Xie BB, Chen L, Yang YR, Lv YX, Chen J, Shi Y, Chen C, Zhao HY, Yu ZL, Liu YC, Fang XD, Yan JW. Genetic distribution of 39 STR loci in 1027 unrelated Han individuals from northern China., 2015, 19: 205–206.
[26] Chakraborty R, Stivers DN, Zhong YX. Estimation of mutation rates from parentage exclusion data: applications to STR and VNTR loci., 1996, 354(1):41–48.
[27] Vicard P, Dawid AP. A statistical treatment of biases affecting the estimation of mutation rates., 2004, 547(1–2): 19–33.
[28] Yan JW, Liu YC, Tang H, Zhang QX, Huo ZY, Hu SN, Yu J. Mutations at 17 STR loci in Chinese population., 2006, 162(1–3): 53–54.
[29] Lu DJ, Liu QL, Wu WW, Zhao H. Mutation analysis of 24 short tandem repeats in Chinese Han population., 2012, 126(2): 331–335.
[30] Qian XQ, Yin CY, Ji Q, Li K, Fan HT, Yu YF, Bu FL, Hu LL, Wang JW, Mu HF, Haigh S, Chen F. Mutation rate analysis at 19 autosomal microsatellites., 2015, 36(14): 1633–1639.
Actual mutational research of 19 autosomal STRs based on restricted mutation model and big data
Zhiyong Liu2, He Ren3, Chong Chen4, Jingjing Zhang5, Xiaomeng Zhang1, Yan Shi4, Linyu Shi1, Ying Chen4, Feng Cheng1, Li Jia4, Man Chen6, Qingwei Fan7, Jiarong Zhang1, Wanting Li1, Mengchun Wang1, Zilin Ren8, Yacheng Liu4, Ming Ni8, Hongyu Sun2, Jiangwei Yan1
Short tandem repeat (STR) markers have been widely used in forensic paternity testing and individual identification, but the STR mutation might impact on the forensic result interpretation. Importantly, the STR mutation rate was underestimated due to ignoring the “hidden” mutation phenomenon in most similar studies. Considering this, we useSlooten and Ricciardi’s restricted mutation model based on big data to obtain more accurate mutation rates for each marker. In this paper, the mutations of 20 autosomal STRs loci (and; The restricted model does not include the correction factor of, this paper calculates remaining 19 STR loci mutation rates) were investigated in 28,313 (Total: 78,739 individuals) confirmed parentage-testing cases in Chinese Han population. As a result, total 1665 mutations were found in all loci, including 1614 one-steps, 34 two-steps, 8 three-steps, and 9 nonintegral mutations. The loci-specific average mutation rates ranged from 0.00007700 () to0.00459050 () in trio’s and 0.00000000 () to 0.00344850 () in duo’s. We analyzed the relationship between mutation rates of the apparent and actual, the trio’s and duo’s, the paternal and maternal,respectively. The results demonstrated that the actual mutation rates are more than the apparent mostly, and the values of1*/2*(apparent) are also greater than1/2 (actual) commonly (1*,1;2*,2 are the mutation rates of one-step and two-step). Therefore, the “hidden” mutations are identified. In addition, the mutations rates of trio’s and duo’s, the paternal and maternal, exhibit significant difference. Next, those mutation data are used to do a comparison with the studies of other Han populations in China,which present the temporal and regional disparities. Due to the large sample size, some rare mutation events, such as monozygotic (MZ) mutation and “fake four-step mutation”, are also reported in this study. In conclusion, the estimation values of actual mutations are obtained based on big data, they can not only provide basic data for the Chinese forensic DNA and population genetics databases, but also have important significance for the development of forensic individual identification, paternity testing and genetics research.
autosomal STR; restricted mutation model; parentage testing; Chinese Han population; mutational analysis
2021-06-02;
2021-08-18
國家自然基金重點項目(編號:82030058)資助[Supported by the National Natural Science Foundation of China (No. 82030058)]
劉志勇,博士研究生,研究方向:法醫遺傳學。E-mail: liuzhy255@mail2.sysu.edu.cn
任賀,碩士,副教授,研究方向:法醫遺傳學。E-mail: snoopy_fr@sohu.com
劉志勇和任賀并列第一作者。
嚴江偉,博士,教授,研究方向:法醫基因組學。E-mail: yanjw@sxmu.edu.cn
10.16288/j.yczz.21-197
2021/8/31 17:30:44
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210831.1400.001.html
(責任編委: 朱波峰)

