細胞內大片段DNA數據存儲的多RS碼交織編碼
陳為剛,葛奇,王盼盼,韓明哲,郭健
(1天津大學微電子學院,天津300072;2教育部合成生物學前沿科學中心,天津大學,天津300072;3天津大學化工學院,天津300072)

人工合成脫氧核糖核酸(DNA)作為一種有潛力的數據存儲介質,存儲密度高,可用時間久,保存能耗低,有望成為未來海量離線數據存儲的重要選擇之一[1-7]。美國半導體工業協會(SIA)與半導體研究公司(SRC)在2021年1月發布《半導體十年計劃》,將DNA數據存儲列為與硬盤、固態硬盤、磁帶并列的大量數據的主要存儲方式之一,成為未來全球存儲產業競爭的重要方向[8]。DNA數據存儲的模式主要包括:短片段寡核苷酸池(Oligo pool)存儲[9-18]、細胞內DNA存儲[19-26]等。短片段的寡核苷酸池存儲,借助DNA的高通量芯片合成與測序技術[18],發展迅速,但是在大規模均衡擴增、復制成本方面仍存在很大挑戰[12]。細胞內DNA數據存儲,尤其是細胞內大片段DNA存儲,借助體內組裝方法實現短DNA片段組裝成長片段,借助體內復制實現高效擴增,復制成本低,在大規模數據分發等場景或有潛在應用價值。近年來,合成生物學發展迅速,尤其酵母基因組的人工合成與基于酵母的同源組裝取得了很大進展[27-37]。在此基礎上,前期我們設計組裝了一條約254 kb的酵母人工染色體,存儲了37.8 KB圖片與視頻數據,除了能可靠復制,未見其他明顯生物功能,綜合考慮信息編碼、合成組裝、復制穩定性、采用三代納米孔測序儀讀出等問題,實現了細胞內的外源數字信息寫入,并基于三代納米孔測序器件實現了快速便攜讀出[19]。目前長基因組的合成與組裝難度大、成本高,借助細胞增殖的復制成本低,納米孔測序可實現便攜式快速讀出。綜合以上幾個特點,大片段DNA存儲非常類似只讀光盤(CD)的早期發展階段,該種存儲模式稱為“酵母光盤”或“DNA光盤”模式。本文針對DNA數據存儲的“光盤”模式設計編碼與數據恢復方法,并結合實測數據開展仿真研究。
在數據存儲領域,糾錯編碼是保證數據可靠性的重要手段。根據香農信息論的信道容量與信道編碼的基本理論,糾錯碼需要與寫入/讀出的錯誤特點匹配,才能實現可靠與高效的數據存儲[38-39]。目前,數字通信領域的幾種重要糾錯碼已經在體外DNA數據存儲中進行了嘗試。例如,數字噴泉碼用于糾正寡核苷酸分子丟失造成的刪除錯誤[10],里德-所羅門(RS)碼糾正堿基刪除與隨機錯誤[12-13],低密度奇偶校驗(LDPC)碼與RS碼構成的乘積碼糾正刪除與隨機錯誤[15]等。而體內大片段DNA存儲的編碼方法,采用LDPC碼與偽隨機序列構成的水印碼,針對三代納米孔測序的高錯誤率,重點考慮難以處理的堿基插入/刪節錯誤[19]。該方法的編碼效率較低,為1.19 bit/bp,距離4堿基{A,T,G,C}表示信息的理論極限密度2 bit/bp仍有較大差距。細胞內的數據存儲框架,與針對細菌等微生物的基因組從頭(de novo)進行測序組裝非常類似,需要測序讀段從頭組裝的過程,需要考慮組裝后重疊群(contig)的特點,進一步得到完整的數據。因此,為適配二代高通量測序的高精度、組裝的重疊群可能存在缺失片段的特點,同時提高堿基承載有效數據的效率,研究便于擴展的信息編碼方法,對降低寫入成本、開展大片段DNA數據存儲具有重要價值。
基于上述考慮,針對細胞內大片段DNA數據存儲,為實現信息編碼方法適配于測序、讀段組裝的錯誤特點,設計了基于多個高碼率里德-所羅門(RS)碼的符號交織編碼方法;提出數據DNA與自主復制序列(autonomously replicating sequence,ARS)交替鑲嵌,構建大片段DNA數據存儲一般結構的方法。讀取端匹配于二代高通量測序特點,設計了基于不同參數(k-mer)組裝多個重疊群、根據ARS定位重疊群位置實現數據段合并、使用高碼率RS碼的糾刪糾錯算法糾正殘留錯誤的處理流程。研究方法上,為了給從頭合成與測序“濕”實驗提供靈活的實驗前驗證與評估,建立了基于計算機的仿真流程,搭建了擴增與測序模型,利用前期的254 kb存儲專用人工染色體的真實測序數據[19]進行校準,對編碼方案、恢復方法進行了仿真驗證。仿真實驗證實,在保證端到端可靠寫入與讀出的前提下,本方法實現的大片段DNA的數據部分邏輯密度為1.973 bit/bp,即使考慮生物功能單元開銷,堿基總體邏輯密度仍高達1.947 bit/bp,高于目前基于寡核苷酸池的存儲方法(目前報道的最高密度為1.57 bit/nt[10]),非常接近2 bit/bp,充分說明了大片段DNA存儲的優勢。
1 大片段DNA數據存儲的編碼方法
大片段DNA數據存儲的邏輯結構設計,不同于基于寡核苷酸池(oligo pool)的數據存儲,索引與引物(或類似單元,例如酵母人工染色體中的骨架)所占的比例相對較低,在堿基利用率上具有一定優勢[11,19]。數據讀取階段,需要先對測序讀段進行從頭(de novo)組裝,類似新物種的基因組從頭(de novo)測序。然后,利用糾錯碼對殘留的錯誤進行糾正,得到完全無錯的數據DNA序列,該過程與傳統基因組測序不同。因此,設計大片段DNA數據存儲的糾錯編碼方案,需要與測序讀段組裝后的錯誤特點相匹配。同時,與生物研究中的基因組組裝要求不同,根據數據存儲與讀取的特點,面向數據存儲的讀段組裝以及后續處理,需要算法有較低復雜度,能在接近實時的情況下實現數據可靠讀出,而基因組的從頭組裝一般對處理時間的要求并不苛刻。
考慮上述特點,提出基于多個RS碼交織編碼得到數據DNA單元,進一步與ARS序列交替鑲嵌,構建體內數據存儲人工染色體,形成高效率的大片段DNA數據存儲基本結構。針對大片段DNA的二代高通量測序數據,結合現有的讀段組裝軟件實現重疊群快速組裝,利用ARS序列定位重疊群、RS碼糾錯糾刪譯碼,實現數據的快速恢復,其工作流程如圖1(a)所示。本文的大片段DNA設計方法包括以下幾個要素:高碼率的RS碼,交替嵌入的ARS序列以及尺度可變的組合方法。實際流程中,將數據寫入大片段DNA,也即DNA的合成組裝過程,需要借助酵母實現;數據的復制也是借助酵母自身繁殖的過程;核酸提取與建庫等是酵母研究中基本操作。進一步,將酵母人工染色體引入大腸桿菌進行富集或直接對酵母進行操作,提取核酸、建庫,得到測序數據。前期工作中,我們使用長度為254 886 bp的人工染色體初步證明該方法的可行性,但在更大的長度,實現人工染色體的分離具有難度,也非常具有研究價值,本文不對該問題進行研究。從大片段DNA的合成組裝到二代測序輸出,依據信息論的研究范式,一般稱其為“信道”,本文采用仿真的方法描述該“信道”[圖1(a)]。該仿真的“信道”是經過前期254 kb存儲專用人工DNA序列的測序數據訓練校準的,更接近真實實驗,這是本文研究的特色之一。
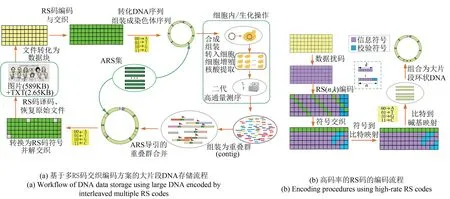
圖1 面向大片段DNA數據存儲的高碼率RS碼編碼方法Fig.1 Encoding scheme using a very high code rate RScodes for data storage with large DNA
1.1 多個極高碼率的RS碼符號交織的編碼方法
提出的設計方案中糾錯碼采用RS碼。設計方案與大片段DNA數據存儲流程中的錯誤類型能實現較好匹配。RS碼是一種高效、可同時糾正刪除(erasure,或稱為“擦除”)與隨機錯誤的多進制循環碼,可獲得理論上最大的最小距離(minimum distance),稱為最小距離最大可分(MDS)碼;同時,高碼率的RS碼,冗余符號所占比例較低,編譯碼復雜度較低,可支撐數據存儲的快速譯碼讀出[39-40]。正是由于這些特點,經過優化處理的RS碼在硬盤、光盤、固態硬盤以及分布式存儲等領域都獲得了廣泛應用[41-47]。借鑒RS碼的成功經驗,本文針對二代高通量測序錯誤率低、能高效組裝成重疊群的特點,設計了極高碼率(R=0.987)的多個RS碼的交織編碼方法,并基于此方法構建DNA數據存儲單元。
數量不等的DNA數據單元與不同的ARS和載體結合,構造了可變長度的大片段DNA數據存儲結構。不同的ARS組成了可選的ARS序列集合[48],根據目前的相關研究結果,ARS的數量較多,能滿足本文的設計方案。ARS序列集合在本文的設計中有兩個作用:一是與流程中的“濕”操作有關,面向寫入側的實際體內組裝與擴增,支持大片段DNA在酵母體內的可靠組裝、復制[19,35-37];二是流程中的信息處理,在數據讀取時,作為組裝的重疊群的標志(類似“路標”),確定組裝的重疊群在整個人工染色體大片段DNA中的位置,便于實現數據恢復。
編碼流程如圖1(b)所示,具體包括以下步驟:
步驟1:數據擾碼。也即將數據與已知的偽隨機序列疊加[7]。由于數據可能存在長的連續的“0”或者“1”,采用擾碼能減少連續比特的數量,從而減少后續長連續堿基的數量,降低測序與合成的難度,減少難以處理的堿基的插入與刪節(insertion/deletion)錯誤[2,10,12]。
步驟2:RS碼編碼。選擇的RS碼為定義在有限域GF(212)上的RS碼(4095,4040,t=27),其碼長為4095個符號,信息位部分的長度為4040個符號,可以糾正55個符號刪除或27個符號錯誤,碼率為0.987。每個符號包含12 bit,一個RS碼的碼字包含的比特數量為49 140 bit。該RS碼所定義的有限域為GF(212),階數較高,但是僅包含55個冗余符號,碼率非常高,考慮到RS碼的編譯碼復雜度與冗余符號的數量直接相關,采用該參數的RS碼具有可行的編譯碼實現復雜度,復雜度遠低于文獻中采用的冗余符號數量高達65 536×15%、定義在GF(216)的RS碼[12,39]。
步驟3:多RS碼符號交織。根據選擇的RS碼字的數量P,將其按照列的方式進行排序,然后將其分解為若干組P×P的單元,對每一個單元分別按照圖1(b)所示的對角循環的方式進行交織,得到P個數據分組。每個數據分組包含P個RS碼符號,從而實現了符號交織,每個分組的大小為4095個符號。在圖1(b)中,僅用5個碼字的交織為例展示原理[17,39]。在本文的仿真案例中P=100。
步驟4:將RS碼碼字轉化為比特分組。每個數據段對應的4095個符號,轉化為49 140 bit,為一個基本分組。
步驟5:比特分組轉碼為DNA數據序列。按照相鄰兩個比特轉化為1個堿基,來自一個GF(212)上的一個符號轉化得到的12 bit,映射為相鄰的6個堿基。采用該種轉化,有利于RS碼發揮其糾正突發刪除能力強的優勢。一個RS碼碼字轉化為24 570 bp的DNA數據序列。
步驟6:DNA數據序列與ARS、載體等組合,構成完整的大的環狀DNA。選擇長度較短的P-1個ARS序列,然后按照交替組合的方式,進一步添加載體骨架序列,得到一個環形染色體序列,作為大片段數據存儲的基本單元。
1.2 細胞內數據存儲的大片段DNA的通用編碼設計
在我們以前的工作中,初步驗證了酵母人工染色體用于數據存儲的可行性與穩定性。在本文提出長片段DNA數據存儲的一般框架:選擇P個DNA數據段與自主復制序列(ARS)交替鑲嵌組合,進一步添加載體,構成一種酵母內數據存儲通用大片段DNA結構。該方法靈活選擇編碼DNA數據單元與ARS的數量,也可在一定范圍內改變數據單元大小、數據單元承載有效數據量的大小(也即改變RS碼的碼率),構成一個規模與效率都可變的長DNA數據存儲統一框架。設計中,編碼DNA數據單元可能出現ARS序列相似度非常高的情況,但是出現概率較低。選用的100組ARS序列的最小長度為57 bp,則理論上數據DNA部分出現該序列的概率非常低,約為1/457,因此在數據處理中無需對該問題進行處理。其余ARS序列的長度均高于57 bp,出現的概率會更低。
第一個可變參數為人工環形染色體包含的數據單元數量P,在確定每個數據單元的大小后,可以根據數據量以及大片段DNA的合成組裝策略靈活選擇單元數量。第二個可變參數為RS碼的信息符號數量,為進一步提高恢復的可靠性,可以減少每個RS碼包含的信息符號的數量,提高RS碼的糾錯能力。還可以將組裝使用的測序數據覆蓋度為約束,確定錯誤率,以此來調整RS碼的參數。進一步,本設計結構的各個要素,例如編碼方法、ARS單元等均具有可擴展性。例如,也可以采用其他的糾錯編碼方法構建數據單元,從而匹配采用不同寫入或讀取模式的需要。利用糾正插入與缺失錯誤的編碼方案,設計了與本文方法類似的結構,用于三代納米孔測序場景[19];可根據宿主菌的情況,靈活設計復制起始位點(origin of replication,酵母中為ARS)集合、載體序列,從而構建適合不同宿主菌的編碼方案。
作為一個例子,本文中我們采用了定義在有限域GF(212)上的RS碼(4095,4040,t=27)的構建的編碼方法,可以滿足設計長度為幾十萬到幾百萬堿基的人工染色體(圖2)。具體展示了三個設計實例:第一是2 489 847 bp的長序列的方案,存儲了一張照片和一份中文文本;第二是兩條1.25 Mb的長序列的設計,分別存儲了一張照片以及用于填充的文本文件;第三是10條大約250 kb的長序列的設計,該長度與我們之前的實驗驗證研究相似,相關結論可以借用。根據數據單元的組裝結構,可得到該編碼方法的編碼效率與邏輯密度。本方法采用的RS碼碼率為R=4040/4095。數據部分邏輯密度為2 bit/bp×4040/4095=1.973 bit/bp。在第一種方案中,考慮鑲嵌的ARS序列以及載體序列,總體邏輯密度為1.947 bit/bp。在其他兩種方案中,由于載體所占比例增加,總體邏輯密度略有下降,見表1。上述邏輯密度均高于目前文獻中四堿基編碼的邏輯密度。本文提供的編碼方法與數據恢復方法,可在該邏輯密度下實現可靠數據讀取,非常接近4個堿基存儲數據的理論邏輯密度,也即2 bit/bp。
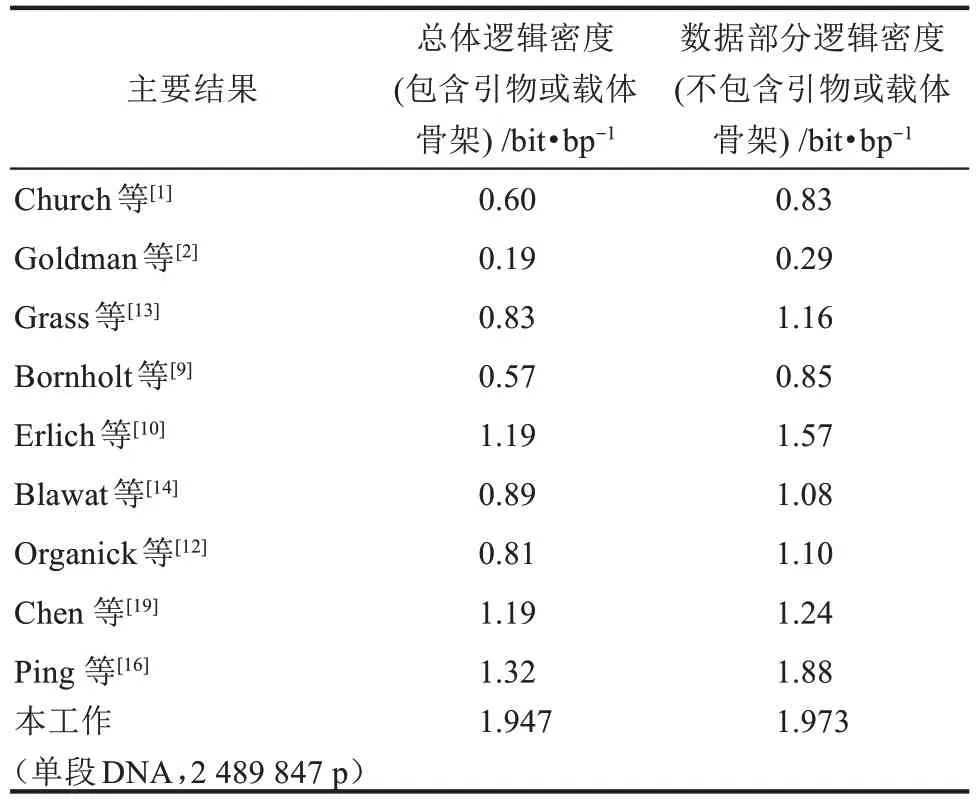
表1 不同編碼方法的堿基邏輯密度比較Tab.1 Base logical density using different encoding schemes
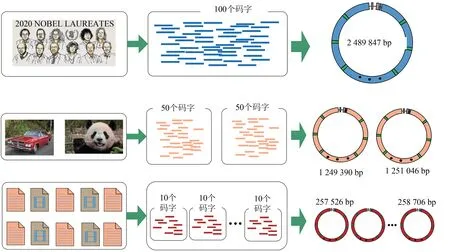
圖2 不同數量數據段組合構建不同長度的大片段DNAFig.2 Building of variant-length large DNA integrating different number of data blocks
2 數據恢復策略:ARS導引的重疊群合并與RS碼糾錯糾刪方法
針對提出的大片段DNA數據存儲結構,設計了面向二代高通量測序的數據恢復方法。大片段DNA數據存儲的讀取,與新物種的基因組測序、從頭組裝非常類似,目標均是得到“完美”的、沒有任何堿基錯誤的基因組。新物種的基因組從頭測序,對實時性要求低,可對參數反復調整以得到最優結果[49-55]。大片段DNA數據存儲的讀出,對算法實時性要求高,傳統的生物信息學處理流程并不適用。針對這一特點,數據恢復時,無需在讀段組裝步驟獲得“完美”序列,利用糾錯碼糾正組裝后的殘留錯誤,降低數據讀取的整體復雜度,但是需要實現糾錯碼及其譯碼方法與組裝方法的適配。
本文提出的方法面向數據DNA長度在Kb到Mb級。該長度的DNA適用于常用的二代測序雙端讀取(paired-end)讀段的高效組裝,例如基于de Bruijn圖的組裝方法[53],典型的組裝軟件有Velvet[54]或ABySS[55]等。組裝得到的序列依據內嵌的RS碼可實現糾錯,得到“完美”人工染色體序列。該方法與傳統基因組測序組裝的主要差別是:可以在較低的測序覆蓋度下得到“完美”的基因組序列,并且在設計大片段DNA時,在數據段避免了重復序列、長連續堿基等,讀段組裝與恢復方法更為有效。
基于上述思路,提出的數據恢復方法如圖3所示,具體步驟為:
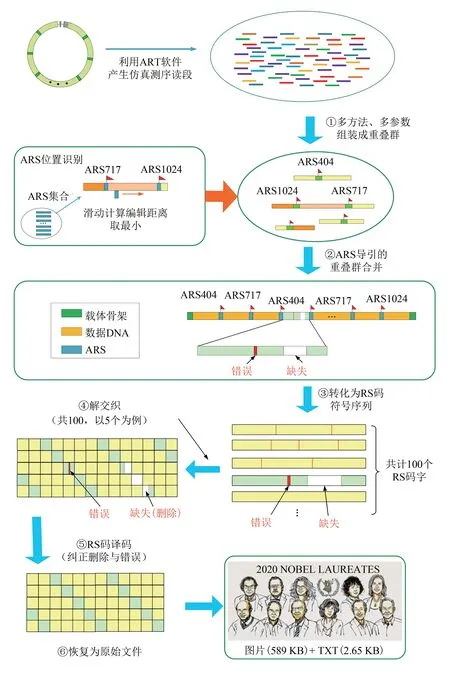
圖3 基于短讀段從頭組裝、ARS引導的多重疊群合并、RS碼糾錯糾刪的數據恢復流程Fig.3 Data readout processes
步驟1:利用Velvet或ABySS等二代序列組裝軟件在多個不同長度的k-mer值下實現二代測序讀段的組裝,得到一組重疊群;該過程同時實現了基于de Bruijn圖的數據預糾錯,能糾正二代測序中存在的單堿基替換、插入與缺失錯誤。
步驟2:識別出每個重疊群中的ARS序列,根據ARS序列確定數據讀段的位置。ARS位置的識別依據包括插入與刪節錯誤的編輯距離,在本文中我們采用了一種魯棒的識別策略,只要識別序列與ARS序列的編輯距離小于該ARS序列長度的20%,判斷為該ARS存在。識別出ARS位置后,將ARS兩側對應的測序讀段,放入該數據段對應的緩存區,直到所有包含ARS序列(或部分ARS序列)的讀段被全部標記與分配完畢。
步驟3:對每一個數據讀段所對應的部分測序讀段,進行大數合并,得到每條數據讀段的合并序列。如果某部分讀段不存在測序數據,則標記該部分片段為符號刪除,如果在某些位置,無法進行大數判決,也標注為刪除;進一步將其轉化為RS碼符號序列。
步驟4:根據分組交織順序對P個數據段進行解交織,得到P個存在錯誤與刪除的RS碼碼字。
步驟5:解交織得到RS碼的P個碼字,分別進行糾錯、糾刪除譯碼,得到數據段。
步驟6:根據RS碼的譯碼得到的數據段恢復原始文件,實現比特到文件的恢復。
提出的數據讀取方法有以下幾個顯著特點。首先,使用基于de Bruijn圖的不同軟件和參數的組裝方法得到的重疊群具有一定獨立性,對大片段DNA的不同部分有不同的覆蓋度。本文中,為降低讀取復雜度與讀取成本,我們采用低覆蓋度的測序數據,例如20×到30×。在低的覆蓋度下,不同的k-mer值產生的de Bruijn圖的結構有很大的差別,進一步考慮到后續處理方法不同,會得到差別很大的一組重疊群。傳統的基因組的組裝目標是得到大的重疊群,本文的組裝目標是得到盡可能多的重疊群去覆蓋數據部分。因此,借用通信中的“分集合并”(diversity and combination)的思想,充分利用重疊群的多樣性(diversity),可提高數據段的覆蓋度。二代測序讀段的錯誤率本身并不高,因此組裝的重疊群的錯誤率往往較低,RS碼需解決的主要問題是缺失部分數據的恢復。
然后,利用鑲嵌在數據段之間的ARS序列實現每個重疊群的位置判斷,ARS序列充當了一種分布式路標,實現了與大片段DNA數據存儲的特點較好匹配。從生物功能方面,該設計也使得大片段DNA在酵母內的組裝與傳代更為穩定[19,35]。最后,多個重疊群利用ARS定位并合并后,由于ARS缺失或者所有重疊群不能覆蓋某區域,會造成數據的某些部分的缺失,將大數判決后存在缺失數據部分標注為刪除(erasure),可以充分發揮RS碼糾刪能力,提高糾錯效率。進一步采用交織與解交織,可將組裝后數據段中的大段序列缺失轉化為隨機符號的刪除錯誤,防止單個數據塊譯碼失敗[39,47]。這是本文設計的交織的RS碼方案的特色所在,較好地匹配了組裝后重疊群的特點,實現了整體優化。前已提及,本文未對DNA組裝以及隨著酵母增殖進行復制的過程進行建模。事實上,DNA組裝過程一般采用能保證完整性與正確性的方法。在酵母增殖過程中,在254 kb長度的人工染色體中堿基出錯的概率非常低,測試了100代的12個樣本中未在數據DNA部分觀察到任何堿基錯誤;但是,長度達到2.5 Mb的人工染色體,超過目前酵母承載外源DNA實驗結果的上界,存在不確定性,可能會存在大片段的缺失。針對大片段的丟失,目前的設計方案最大可容忍33 000 bp的大片段缺失。存在大片段缺失時,整個序列會發生整體移位(shift),考慮到ARS序列是分布式嵌入的,基于ARS的重疊群的定位仍可以工作,這也是提出的分布式嵌入ARS序列的優點。
本方法的另一特點是測序與譯碼的復雜度較低。提出的恢復方法可以在較低的測序覆蓋度下完成數據恢復,因此需要緩存處理的總測序數據量較少,從而使得組裝處理、重疊群合并等步驟的處理復雜度較低。進一步,采用的RS碼碼率很高,校驗符號的數量僅為55,根據RS碼的特點,其譯碼復雜度較低。設計中通過糾刪除與交織技術,充分挖掘RS碼的糾錯能力,仍能實現在20×測序覆蓋度下實現可靠恢復,整體復雜度較低,能在較高的效率實現數據可靠恢復。
仿真實驗中,假定人工染色體的測序數據與宿主基因組數據是分離的。該條件可以通過生化操作或測序數據預處理實現。生化處理可根據人工染色體的特性將其分離,在前期針對254 kb的實驗中,將人工染色體轉入大腸桿菌進行富集。但是,在更大規模的人工染色體,例如Mb長度級別的人工染色體,轉入大腸桿菌的方法存在困難。將Mb長度級別的人工染色體分離的操作仍然需要根據人工染色體與宿主染色體之間的關聯,并進行進一步設計,這也是目前我們正在開展的工作。在測序數據預處理方面,可開展宿主與人工染色體的混合測序,然后先將測序數據與宿主菌的已知基因組進行比對,再將基因組數據剔除。優點是該方法處理準確度較高。缺點是增加測序數據處理的總量,例如酵母的基因組的堿基數量大約為12 Mb,與設計的2.5 Mb的序列相比,數據量大約是人工染色體序列的4.8倍。
3 實驗結果與分析
本文設計了一個長度為2.5 Mb的用于數據存儲的酵母人工染色體作為仿真測試實例。高通量測序過程利用二代測序數據的仿真軟件ART[56],得到了雙端讀取的PE150測序仿真數據。然后,開展從測序讀段的數據恢復實驗,驗證提出的大片段DNA編碼方法在二代高通量測序下的優越性,也即實現了測序數據特點、從頭組裝方法以及糾錯編碼的匹配,從而能憑借非常小的編碼冗余實現了非常高的邏輯密度。本部分主要介紹基于仿真測序數據的測試驗證方法。
3.1 仿真測序數據校準與分析
本文建立了基于計算機的長DNA片段數據存儲仿真平臺,如圖4所示。目前長片段DNA存儲框架中,基因組合成與組裝過程產生的錯誤遠少于測序產生的錯誤。因此,在數據恢復中需要應對的錯誤主要來自高通量測序。仿真實驗中,選擇產生測序讀段的ART軟件模擬測序過程。本研究雖未開展直接的合成與測序實驗,我們利用前期的“濕”實驗數據[13]對本文的仿真方法進行了校準與驗證,使得仿真結果具有較好可信度,一定程度上實現了“濕”實驗與仿真設計的融合,使得仿真過程更為合理。進一步,我們分析了仿真的測序數據與端到端的存儲恢復性能。
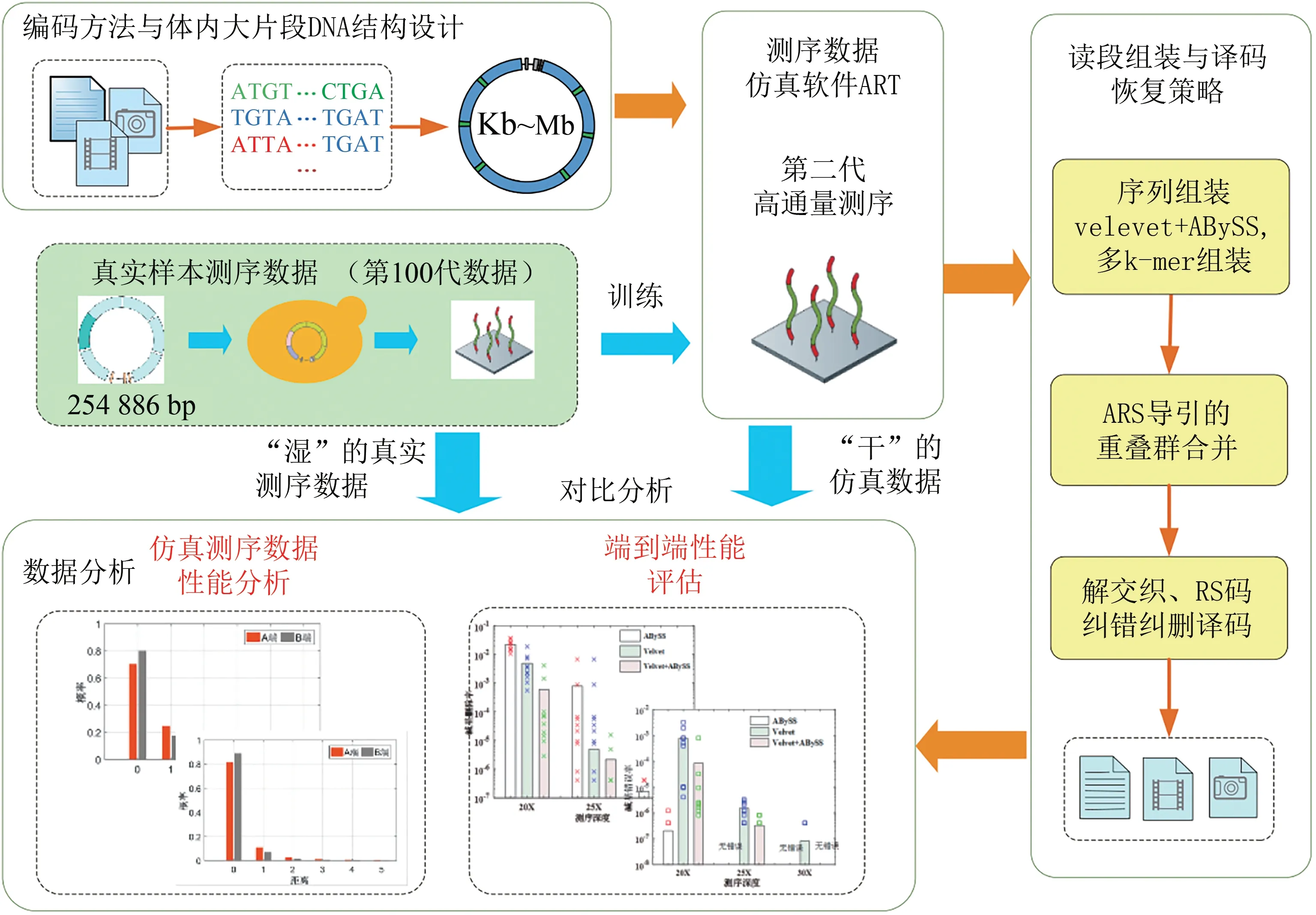
圖4 基于計算機仿真的編碼大片段DNA體內存儲驗證流程Fig.4 Verification procedures using computer simulation for proposed encoding method and construction scheme of large DNA chunks in living cells
數據存儲的大片段DNA與物種的基因組存在一定差別。為利用ART軟件產生更符合實際情況的測序數據,我們先采用以前構建的數據存儲人工染色體的二代測序數據[19]對ART軟件進行參數訓練。具體參數訓練與校準中,針對254 886的人工染色體,我們采用的實際測序數據的覆蓋度超過200×。然后,利用訓練的參數生成針對本文設計的大片段DNA的二代測序數據。我們獨立產生了10組30×的數據并開展10次獨立實驗,總的測序數據量也達到了總覆蓋度超過300×。圖5和圖6給出了生成的仿真測序數據與我們前期實驗得到的254 kb人工染色體測序數據的對比。圖5比較了讀段中測序錯誤的數量。從該圖可以看出,發生錯誤的讀段的數量在20%左右。仿真產生的讀段的質量略差于實測數據,這可以更好地說明本文提出方法的糾錯能力。圖6比較了讀段中測序與處在讀段中位置的關系。可以看出,二代測序讀段中包含插入、缺失與替代錯誤,錯誤率在10-4~10-3左右。插入與缺失的錯誤率明顯低于替代錯誤概率。從圖中可看出生成的測序數據特征與實際二代測序數據特征是非常一致的,這說明利用仿真的方法生成測序數據是具有較好可行性的。考慮到目前大片段DNA的組裝仍然是非常有挑戰性的任務,本文的仿真方法可以在實驗前提供更為全面的評估,提高實驗效率。
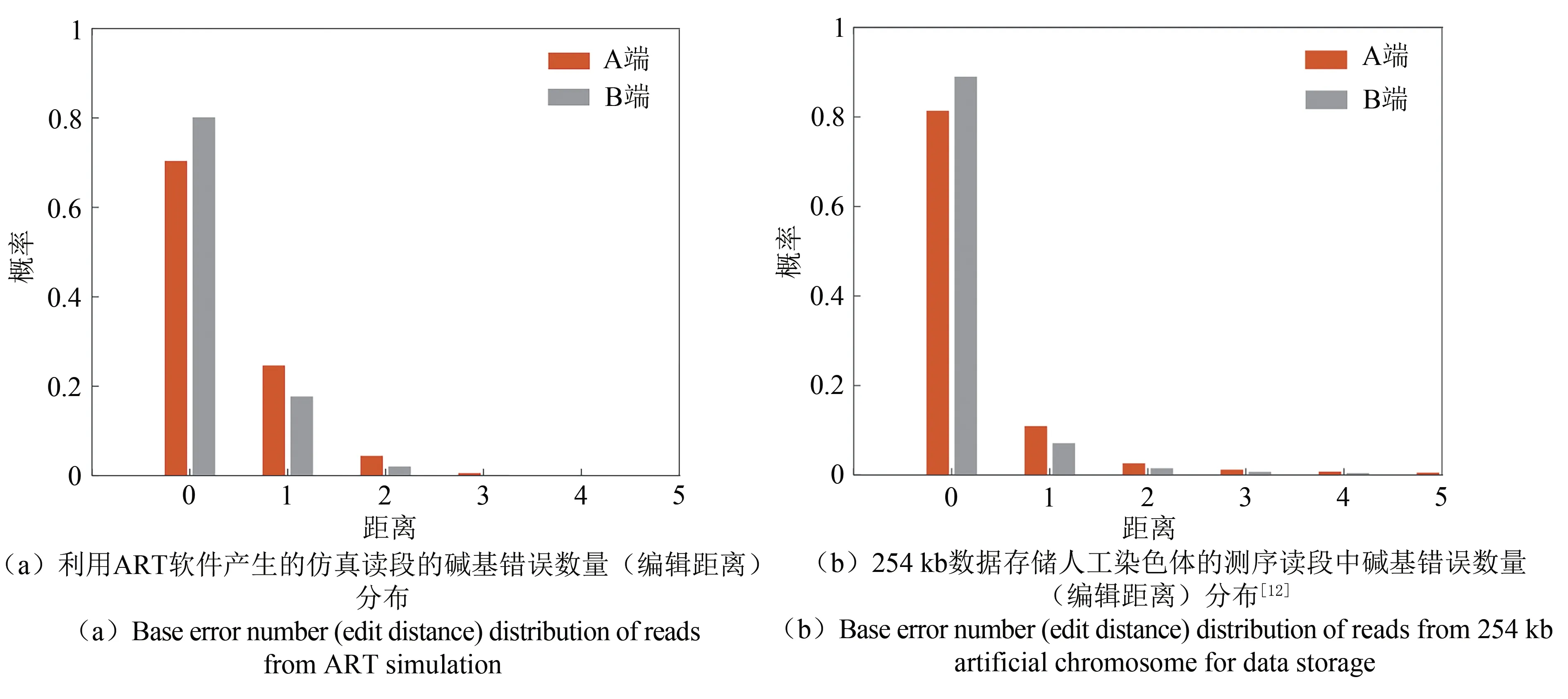
圖5 仿真讀段與實際測序讀段的堿基錯誤數量(編輯距離)分布Fig.5 Base error number(edit distance)distribution in simulation and real sequencing reads.
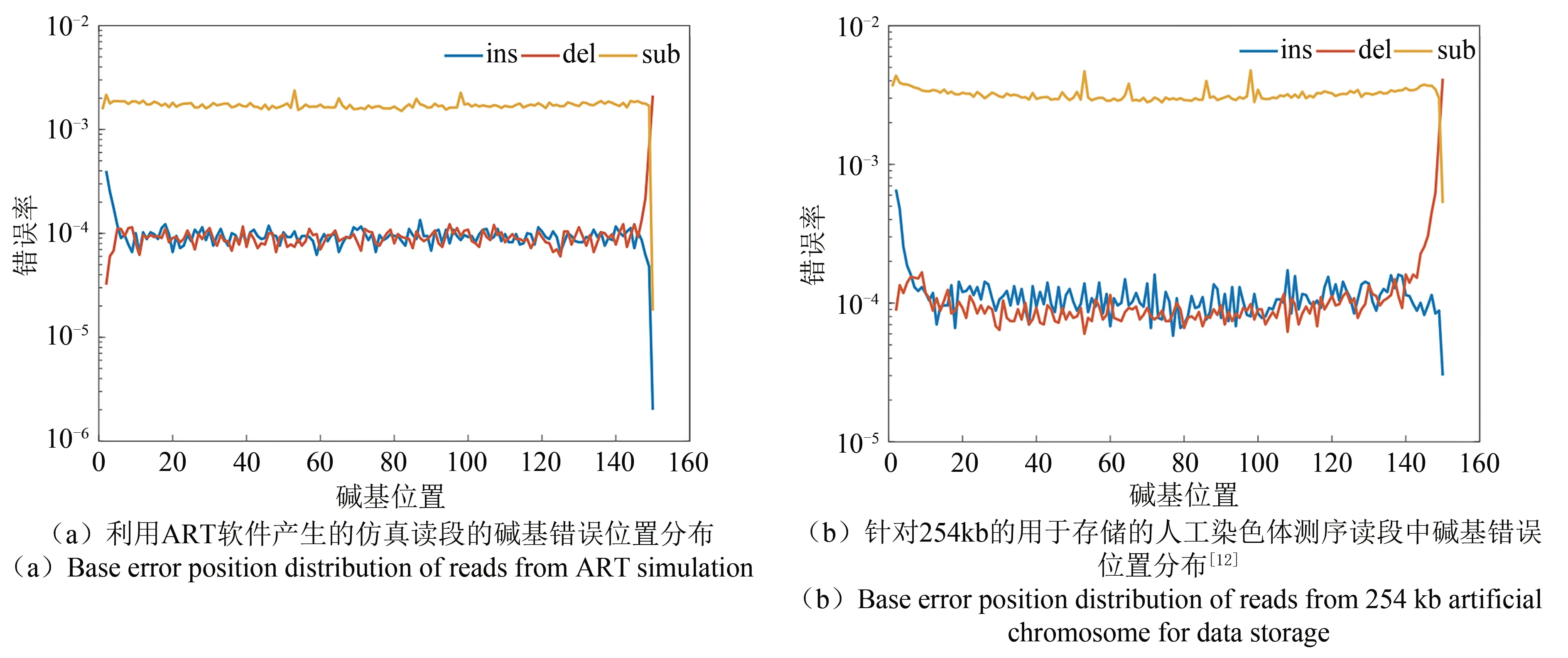
圖6 仿真與實測讀段的堿基錯誤隨著位置變化情況Fig.6 Base error position distribution in simulation and real sequencing.
3.2 譯碼恢復性能與分析
以100個RS碼字的2.5 Mb的基因組為例開展仿真研究。在該模型中,影響數據恢復性能的主要參數是測序數據覆蓋度,我們在不同的覆蓋度下對編碼方案、數據恢復方法進行了仿真測試與分析。測序覆蓋度(coverage),體現了對用于存儲的DNA的處理復雜度,與成本、信息處理硬件設備復雜度等密切相關。一般而言,二代高通量測序是基于合成的測序方法,測序覆蓋度越高,讀取成本會越高,測序時間會越長;高覆蓋度的測序讀段越多,需要的數據緩存的硬件復雜度與計算量都迅速增長。因此,本文參照傳統的信息存儲設備的特點,致力于在相對較低的測序覆蓋度下,實現沒有任何堿基錯誤的快速、“完美”的數據讀出。
測試中我們選用覆蓋度為20×、25×、30×,每個覆蓋度用ART生成10組獨立測試數據,進行10次獨立的平行組裝與譯碼實驗。選用的組裝軟件為Velvet與ABySS,每種組裝方法選擇若干不同k-mer值,表2給出了具體仿真結果。圖7給出了不同測序覆蓋度下的組裝錯誤特性說明,證實提出的多RS碼交織并執行糾刪糾錯方案的合理性。從圖7可以看出,當測序覆蓋度增加時,多軟件多參數的組裝方法的性能不斷改善,殘留的錯誤與刪除數量快速下降,從而可以實現可靠恢復。在測序覆蓋度為20×時,單獨的Velvet與ABySS組裝的錯誤率均在該方案的糾錯能力的邊緣(糾正1.34%的符號刪除,或者糾正0.66%的符號錯誤,圖7),存在數據恢復失敗的情況,詳見表2。考慮到組裝與重疊群合并策略的波動較大,圖7給出了每次實驗的具體結果(如圖中的“×”和“□”)。
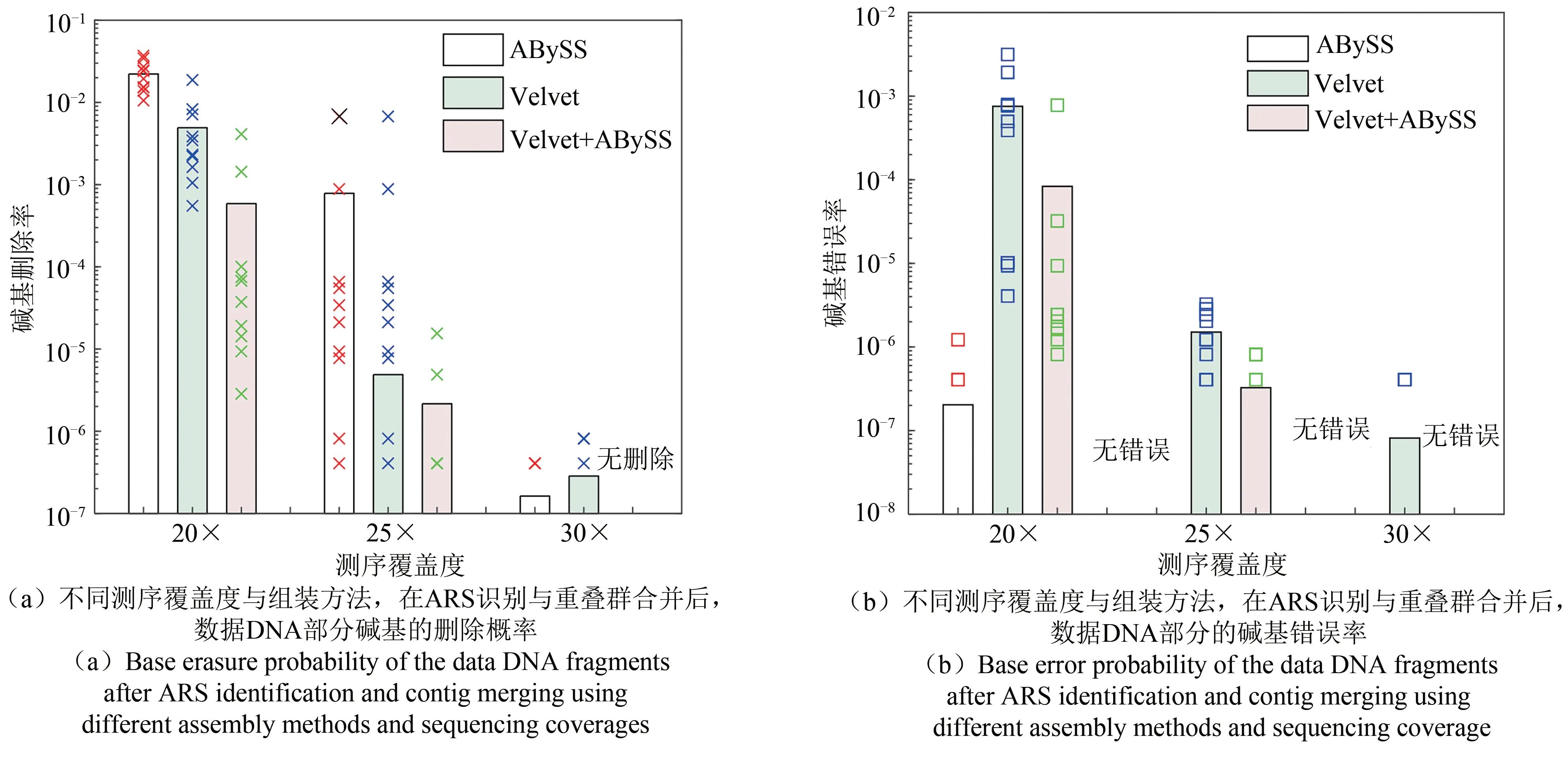
圖7 不同測序覆蓋度與組裝方法,在ARS識別與重疊群合并后,數據DNA部分錯誤分布Fig.7 Base error distribution of the data DNA after ARSidentification and contig merging using different assembly methods and sequencing coverage
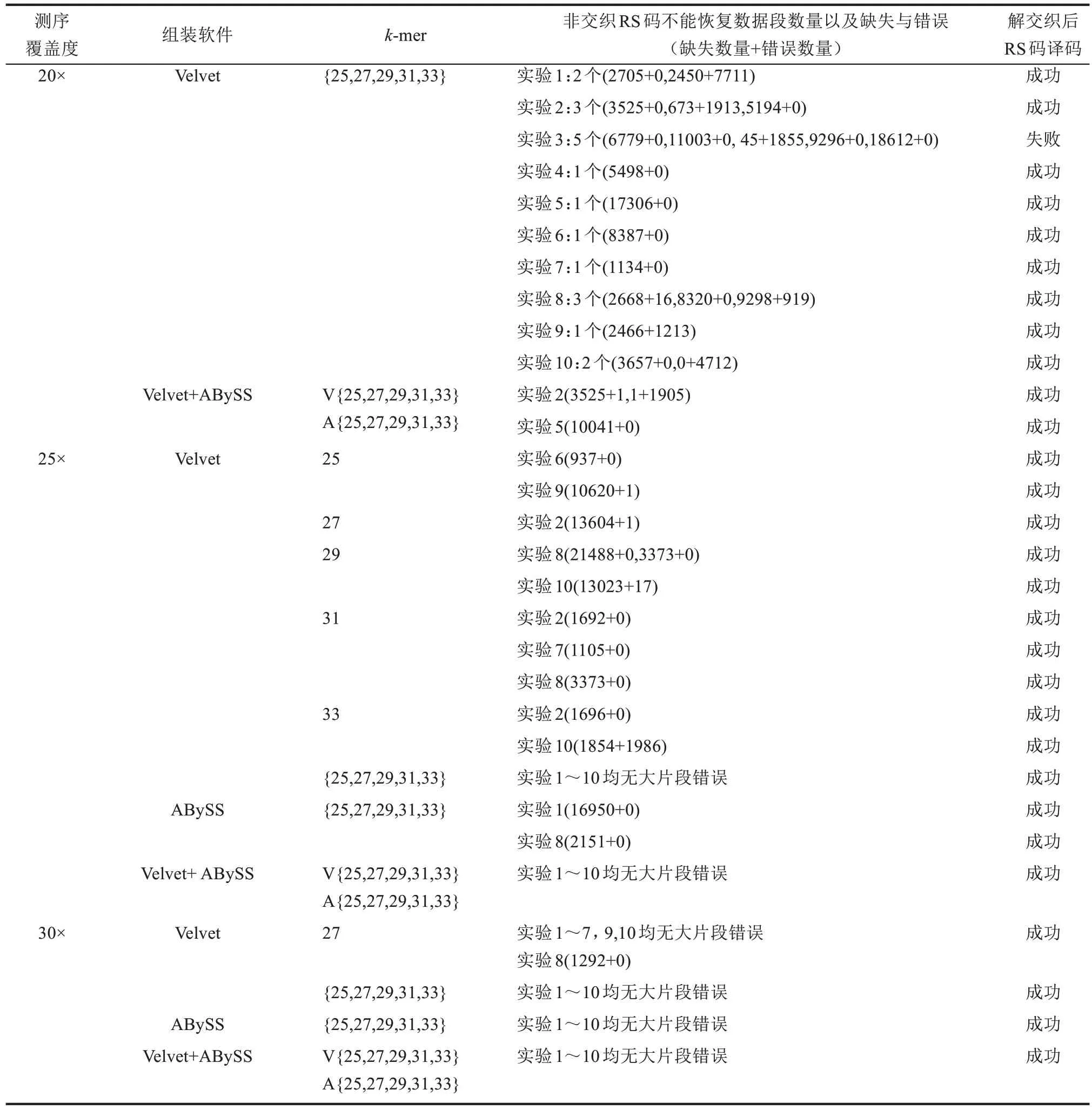
表2 采用交織多個RS碼的數據恢復分析Tab.2 Data recovery analysis using interleaved multiple RScodes
從表2的仿真結果可以看出,當覆蓋度為25×和30×時,所有方案的10次平行實驗均譯碼成功,驗證了該編碼方法與數據恢復方法的魯棒性。同時也可看出,采用多方法、多k-mer與采用單k-mer的結果相比較,讀段組裝的性能有明顯改善,片段缺失與錯誤均明顯減少。表2中僅列出了大片段的錯誤情況,這些組裝、ARS識別后的錯誤經過交織,可充分利用多個高碼率RS碼的糾錯能力,獲得非常高的成功率,實驗測試中在25×與30×下數據恢復都是成功的。表2中還列出了若不采用交織方案,僅采用相同參數的RS碼獨立編碼每個數據段不能成功譯碼的所有情況。若不采用多RS碼交織,每個數據塊采用單獨的RS碼編碼,由于高碼率的RS碼的糾錯糾刪能力有限(糾正Nerasure=55個刪除,或者Nerror=27個錯誤,或者2×Nerror+Nerasure≤55),會存在某些片段不能正確譯碼的情況,不能完全恢復數據。
在20×測序數據下,采用單個k-mer組裝的重疊群錯誤率很高,不能正確譯碼,本部分數據量較大,未在表2中列出,詳細的信息見本文在期刊官網html文件的補充材料表。但是,多個k-mer值組合的情況仍獲得較好的性能。首先,采用Velvet軟件多k-mer組裝,10次獨立實驗,僅有第三次會發生解交織后的譯碼失敗,其他情況均正確譯碼。進一步,在Velvet與ABySS混合組裝中,所有10組獨立實驗,均獲得了增益,解交織后全部譯碼成功。
進一步,根據表2中的數據(第4列)也可看出,本文提出的框架,二代高通量測序讀段組裝后的錯誤主要是大片段的數據缺失錯誤,標記為刪除。這是本文主要設計出發點:實現糾錯編碼、組裝方法與測序方法的匹配。第一,考慮到RS碼有理論上最優的糾正刪除錯誤能力,本文提出采用RS碼來糾正這些突發刪除,可取得非常好的效果,能憑借較小的編碼冗余度獲得可靠數據恢復,可以實現高邏輯密度存儲的可靠存儲。第二,多RS碼交織避免了某些數據段缺失過多無法恢復的情況。
4 結語與展望
為利用細胞內數據處理與存儲的優勢,本文提出一種針對大片段DNA數據存儲的融合碼率為0.987的RS碼與符號交織的高效編碼方法。提出的編碼方法實現了將數據文件編碼到多個DNA數據單元,DNA數據單元進一步與ARS交替鑲嵌組合,構建了靈活的細胞內DNA數據存儲結構,實現數據在大片段DNA中的存儲。進一步,基于二代高通量測序讀段、采用提出的數據恢復方法,可實現可靠的高效率DNA數據讀出。該方法實現了交織的多RS碼的編碼方法、大片段DNA邏輯結構、二代高通量測序、從頭組裝方法的多要素匹配,從而能夠實現非常高的堿基邏輯密度,總體邏輯密度達到1.947 bit/bp,高于目前的主要設計方法,非常接近理論的2 bit/bp。
基于前期的實際生物實驗與香農信息論的經典研究方法,提出的大片段DNA數據存儲的設計方法,實現了編碼方法與細胞內大片段存儲信道的匹配,將信息論的研究方法擴展到了合成生物學領域。后續研究將把更多的系統影響因素納入考慮,例如三代測序、堿基識別方法、測序條形碼[57-58]等,為研究者提供更全面、更準確、更系統化的大片段DNA數據存儲仿真平臺,為研究更接近傳統存儲系統形態的DNA存儲提供依據。
該存儲模式中,酵母內大片段DNA的從頭合成與組裝的“濕”實驗是目前技術難度最大、成本最高的部分[27-37]。設計的Mb級別的DNA是否適合在酵母內合成與組裝,組裝難度如何,都是值得進一步深入分析的問題。之前的實驗研究中,僅完成了254 kb的大片段DNA數據存儲的全流程實驗驗證,在一定程度上證明本設計思路具有可行性[19]。到目前,針對已存在的基因序列,可以構造2 Mb以上的酵母人工染色體(YAC)[59-60]。但是,對于來自數字世界轉化來的Mb級別以上的DNA數據序列,尚無嚴格的實驗證實。因此,如何進一步突破單個細胞內的存儲長度,挑戰數據存儲的容量上限,并研究其與宿主細胞的相互影響,尤其是Mb級別的完全外源的人工DNA的組裝、復制穩定性以及與生物本身基因組的相互作用等問題,都需進一步實驗研究。在合成生物學“設計-構建-測試-學習”的閉環策略中,針對數據存儲專用的人工染色體,在254 kb正在初步完成該閉環策略[19]。進一步通過“學習”能否構建Mb級別的細胞內存儲機制,本文只是完成了“設計”步驟,后續還需要更為深入研究,包括構建、穩定性測試分析等工作。同時,在外部數據體內存儲的場景,大片段DNA在細胞內的處理(例如組裝、分離等),是合成生物學的重要基礎問題[25,32-37],期望在未來取得更大的進展,不僅推動DNA數據存儲的發展,也促進合成生物學本身的發展。
針對細胞內長片段DNA存儲(“DNA光盤”)的應用場景,考慮到目前長片段DNA的組裝成本高,類似早期只讀光盤的發展,可通過大量用戶共享一次數據寫入的成本(“母盤”的成本)才能獲得應用優勢。考慮到用于存儲數據的染色體,借助酵母增殖的復制成本低,能效高,是一種極為高效的生物計算模式。前期研究已初步說明了該類染色體可以有效承載數據,能隨著生命過程快速復制,并且便于讀出,證明該模式適合數據大規模分發。數據分發(例如通過介質克隆或網絡傳輸),都需要一定成本,利用生命過程的數據大規模復制與分發,即使與基于寡核苷酸池的DNA存儲相比,在成本方面仍具有優勢,其量化評估需要綜合考慮的因素較多,可以將其作為未來研究的方向。

